2. 四川鹿鸣丘高原生态农业科技有限公司, 若尔盖 624500
2. Sichuan Lumingqiu Plateau Eco-agriculture Technology Limited Company, Ruoergai 624500, China
线粒体(mitochondria)是细胞中进行生物能量代谢的重要细胞器,也是重要的遗传信息载体。线粒体DNA(mitochondrial DNA,mtDNA)作为细胞核外遗传物质,相较于核DNA具有分子结构简单、母系遗传、变异率高、进化速度快等特点。mtDNA的特性对研究动物种群的遗传多样性、种群结构和进化具有重要意义[1]。mtDNA包括一些编码基因和非编码区内的复制控制区(control region或displacement loop,D-Loop)。mtDNA D-Loop中还存在两个高变区(high variable,HV)[2]:HV1和HV2,HV1位于D-Loop序列的5′端,HV2位于近3′端,而且D-Loop区的变异有严重的碱基转换偏好[3-4]。mtDNA D-Loop控制区受进化的压力较小,进化速率较快,其碱基替换率是mtDNA其它区段的2.8~5.0倍[5],遗传变异程度较高,而且常出现串联重复序列,这些特点使得mtDNA的D-Loop控制区分子标记已成为研究物种分子系统发生、物种亲缘关系和家畜驯化进程的有利工具[6-10]。随着时间的推移,母本谱系随着个体的进化而进化,线粒体基因组也会随着环境变化而产生相应的适应性突变[2],随着突变的逐渐固定,不同的线粒体谱系从一个或多个共同的起源中产生,形成汇聚相应线粒体基因组特征的mtDNA单倍型[11]。mtDNA单倍型能够区分出动物的品种和品系,并赋予动物优势或劣势[2],这对于物种资源的保护与利用非常重要。
近些年来,随着分子生物学技术的飞速发展,人类在动物线粒体基因组测序的研究上有了质的飞跃。比如,已经在GenBank数据库中公布的藏猪、内江猪、乌金猪、雅南猪、金华猪、梅山猪等中国地方家猪,重庆野猪、云南野猪、福建野猪等中国野猪以及杜洛克猪、长白猪和大白猪等引入猪种在内的几十个猪种的线粒体基因组的全序列[12-18]和上千条部分序列[19-21]。这些工作为基于mtDNA基因组序列研究猪种间和种内群体间亲缘关系远近以及系统发育关系奠定了基础。
藏猪主要分布于青藏高原地区,包括云南迪庆、四川阿坝及甘孜、甘肃合作以及西藏自治区山南、林芝、日喀则、昌都等地方[22-23]。由于近年来动物繁育方法不断改进,太过于追求生产性能的提高,加之生态环境的改变,致使许多拥有丰富遗传资源的地方猪种数量锐减,纯种藏猪的分布范围和数量也在不断减少。因此,最大限度地保存藏猪的遗传多样性是藏猪保护与利用面临的突出问题。阿坝藏族羌族自治州若尔盖地区由于独特的地理位置和恶劣的交通环境,导致该区域藏猪的开发与利用程度低,保障了若尔盖地区藏猪血缘纯正。本研究对若尔盖地区藏猪mtDNA D-Loop高变区部分序列进行了测定,并结合在GenBank上已报道的其他地区藏猪的mtDNA D-Loop序列,从线粒体DNA的角度对若尔盖地区藏猪的遗传结构和系统进化地位进行研究,并采用了生物信息学的数据处理方法,探讨若尔盖地区藏猪的遗传多样性以及与其它报道过的猪品种的亲缘关系,为合理保护和利用藏猪的遗传资源,培育新品种提供科学依据,对本来就匮乏的遗传资源认识是一个有力的补充,对保护此类稀有猪种具有极其重要的参考意义。
1 材料与方法 1.1 试验材料本次试验动物为四川省阿坝藏族羌族自治州若尔盖地区的求吉乡、包座乡、巴西乡、降扎乡、占哇乡、崇尔乡、热尔乡、冻列乡和甘肃省甘南藏族自治州迭部县益哇乡等9个乡镇的80头藏猪(表 1)。用耳号钳剪耳缘组织3~5 g,置于含1 mL 75%乙醇的1.5 mL离心管中带回实验室,于-20 ℃保存,备用。
|
|
表 1 若尔盖地区藏猪采样统计表 Table 1 The samples of Tibetan pig in Ruoergai area |
利用天根DNA(DP304,北京)提取试剂盒提取藏猪耳组织的DNA,具体试验操作步骤按照其使用说明书进行。
1.2.2 PCR引物的设计根据公布在GenBank数据库中甘南藏族自治州的藏猪mtDNA全序列(登录号:KC493609),利用引物设计软件Primer 5.0对藏猪的mtDNA D-Loop高变区设计扩增引物。上游引物5′-CAGTCAACATGCGTATCAC-3′;下游引物5′-TGTGGTAGATTGGCGTAA-3′。引物由北京擎科生物科技有限公司成都分公司合成。
1.2.3 PCR反应和序列测定PCR反应体系为:2×Taq PCR Master Mix(KT201) 10 μL,ddH2O 6 μL,DNA模板2 μL,上下游引物各1 μL。PCR反应扩增条件为:94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,30个循环;最终72 ℃延伸10 min;16 ℃保存。扩增的PCR产物用1%的琼脂糖凝胶电泳检测其片段大小,检测到目的片段的扩增产物由北京擎科生物科技有限公司成都分公司进行纯化、回收与测序,并且公司直接反馈正反向测序拼接的序列结果。测定的样本序列已提交GenBank数据库,登录号为MK947106~MK947122。
1.3 数据处理与分析应用DNAMAN8.0软件[24]对测定的80头藏猪的mtDNA D-Loop部分序列进行序列同源性比对分析。应用DNASTAR[25]软件编辑序列,利用Clustal X 1.81软件[26]对编辑好的序列进行对齐。应用DNAsp5.0软件[27]计算对齐序列的单倍型数(number of haplotypes,h)、单倍型多样度(haplotype diversity,Hd)、核苷酸多样度(nucleotide diversity,Pi)、平均核苷酸差异数(average number of nucleotide difference,k)。应用Network5.10软件[28]对单倍型序列进行网络关系分析。应用MEGA6.0软件[29]统计对齐序列的平均碱基组成、多态位点数、简约信息位点数、总体转换/颠换比率(the overall transition/transversion bias,R)等;以GenBank数据库中9种家猪和23种野猪(表 2),以及非洲疣猪(Phacochoerus africanus,登录号DQ409327)的mtDNA D-Loop区序列作为外类群[13],采用邻接法(NJ)构建系统进化树进行分析。
|
|
表 2 GenBank数据库中使用的线粒体基因的家猪和野猪信息 Table 2 Information on mitochondrial genes of domestic and wild boars used in GenBank |
若尔盖地区藏猪mtDNA D-Loop高变区经扩增、测序、比对编辑后,获得有效长度为435 bp的核苷酸序列(不包括串联重复序列),以及由10个碱基组成的20~30个(平均约25个)不等的串联重复序列TACACGTGCG。该段序列与甘南藏族自治州的藏猪mtDNA全序列进行比对,其位于387~821 bp之间的碱基位置,占线粒体D-Loop区全长的41.77%(不包括串联重复序列)。
用MAGE6.0软件对80条核苷酸序列进行碱基分析发现,T、C、A和G 4种核苷酸的平均比例分别为26.52%、26.35%、29.94%和17.19%,A+T含量(56.46%)明显高于G+C含量(43.54%),可见藏猪mtDNA D-Loop高变区富含A和T,也体现出哺乳动物线粒体DNA碱基组成的基本特点。利用DNAsp5.10软件对若尔盖地区80头藏猪的mtDNA D-Loop区的部分序列进行单核苷酸多态性(single nucleotide polymorphism,SNP)分析,共检测到14个多态位点,占所测核苷酸的3.22%,说明该序列突变率较小,有较好的稳定性,其中有9个单一变异位点(singleton)和5个简约信息位点(parsimony informative sites),且核苷酸的替代全部是转换,可见序列突变还远远未达到饱和,详见表 3。
|
|
表 3 若尔盖地区藏猪的单倍型分布及频率 Table 3 Haplotype distribution and frequency of Tibetan pig in Ruoergai area |
由表 3和图 1可知,若尔盖地区9个乡镇藏猪群体的80条mtDNA D-Loop高变区核苷酸序列存在14个变异位点,鉴定了17个单倍型(Hap_1~Hap_17)。其中,共享单倍型8个,特有单倍型9个,Hap_1和Hap_15单倍型是4个乡镇(降扎乡、益哇乡、热尔乡和冻列乡)群体的共享单倍型,表明这4个乡镇藏猪群体存在两个共同的母系祖先单倍型。若尔盖地区藏猪群体间的特有单倍型数差异较大,除了包座乡、巴西乡和崇尔乡藏猪没有特有单倍型之外,其它6个乡镇群体都存在特有单倍型,其中,降扎乡藏猪的特有单倍型数量最多,占单倍型总数的17.65%(3/17),而热尔乡、求吉乡、占哇乡、迭部县益哇乡和冻列乡分别为2、1、1、1和1个。并且,单倍型分布的频率差异较大,Hap_1单倍型频率最大,为26.25%(21/80),Hap_15、Hap_7、Hap_9、Hap_13和Hap_11频率依次降低,分别为17.50%、8.75%、7.5%、7.5%和6.25%,其余11个单倍型频率都低于6.25%。
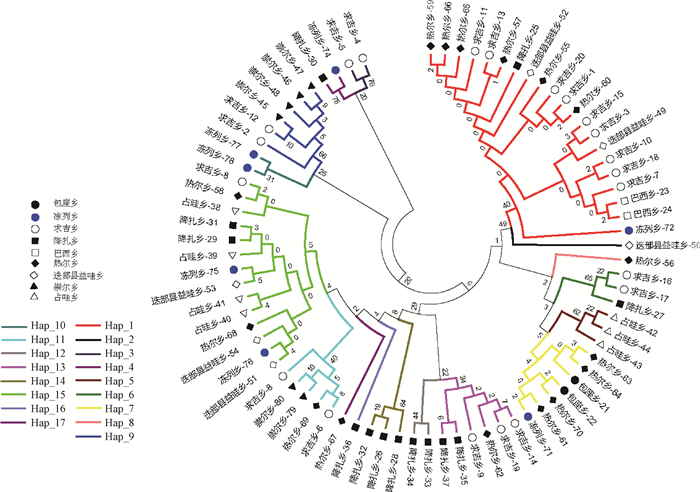
|
图 1 若尔盖地区藏猪的mtDNA控制区单倍型分布 Fig. 1 Distribution of mtDNA control region haplotype of Tibetan pig in Ruoergai area |
由表 4可知,若尔盖地区藏猪单倍型多样度(Hd)、核苷酸多样度(Pi)和平均核苷酸差异数(k)分别为0.881、0.004 66和2.028。无论从单倍型数、单倍型多样度还是核苷酸多样度来看,降扎乡藏猪遗传多样性最高,而占哇乡藏猪的Pi和k最低。占哇乡由于特殊的地理位置,处于山谷之中,相对较为封闭,该群体长期自然条件产生的突变类型在该群体中得到积累,可能由于与外界交流少,群体内近亲高度繁殖,使其遗传多样性比较有限。
|
|
表 4 若尔盖地区藏猪群体的遗传多样性 Table 4 The genetic diversity of Tibetan pig populations in Ruoergai area |
同时对若尔盖地区9个乡镇藏猪的80条核苷酸序列进行Tajima检验,其Tajima’s D值为-0.797,差异不显著(P>0.100),符合中性突变。
2.4 若尔盖地区藏猪群体之间基于mtDNA D-Loop区部分序列的平均遗传距离利用MEGA6.0软件以Nucleotide:p-distance模型和缺失数据成对删除的方式,对获得的80条核苷酸序列计算若尔盖地区藏猪之间的平均遗传距离。由表 5可知,若尔盖地区藏猪群体间遗传距离较小,平均遗传距离为0.004 66,其中降扎乡和冻列乡藏猪间的遗传距离最大为0.006 90,包座乡和益哇乡藏猪间的遗传距离最小为0.002 16。
|
|
表 5 基于mtDNA D-Loop区部分序列的藏猪群体间的平均遗传距离 Table 5 The mean genetic distance between Tibetan pig populations based on mtDNA D-Loop region partial sequence |
本研究对获得的17条单倍型序列在Clustal X 1.81软件中进行比对后,用MEGA6.0以Kimura 2-parameter模型建立NJ树,并对拓扑图进行了自展检验(Bootstrap),重复抽样次数为1 000次。所构建的系统进化树见图 2,若尔盖地区9个乡镇的藏猪很明显的分成了2个支。用Network 5.10软件进行网络关系分析也证实了这一点(图 3)。
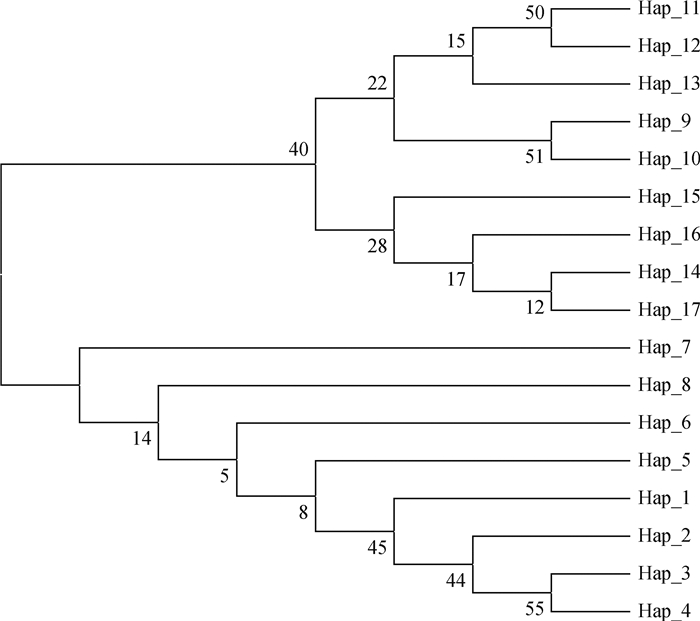
|
节点旁数字为1 000次Bootstrap检验后置信度值(%) Numbers at nodes indicate Bootstrap values (%) with 1 000 replicates 图 2 若尔盖地区藏猪基于mtDNA D-Loop区单倍型序列构建的系统发育树 Fig. 2 Phylogenetic tree constructed based on haplotype sequences of mtDNA D-Loop region of Tibetan pig in Ruoergai area |
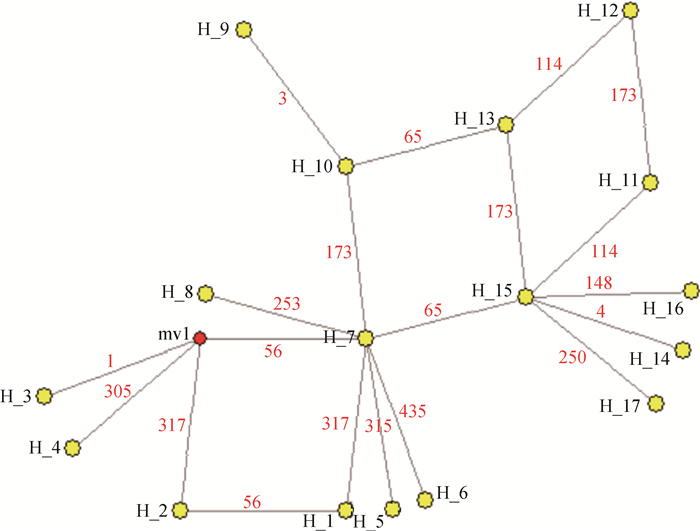
|
图 3 若尔盖地区藏猪mtDNA D-Loop区单倍型的网络关系 Fig. 3 Network relationship of haplotypes of mtDNA D-Loop region of Tibetan pig in Ruoergai area |
为了进一步研究若尔盖地区藏猪的起源,从GenBank数据库中获取已提交的中国地方猪、中国和国外野猪、以及欧美引入猪种的mtDNA D-Loop高变区的序列(表 2),共计32条序列。应用DNAsp5.0软件进行单倍型分析可知,若尔盖地区藏猪的特有单倍型除了Hap_2之外,其余的8种特有单倍型在32个序列中均未发现。可见,若尔盖地区藏猪的特有单倍型具有很强的品种特异性与地域性。同时,Hap_1、Hap_2、Hap_7、Hap_11和Hap_15也是其他地区猪的共享单倍型,详见表 6。
|
|
表 6 若尔盖地区藏猪与其它猪种共享单倍型 Table 6 Shared haplotypes by Tibetan pig in Ruoergai area and other pig breeds |
本研究以GenBank数据库中的序列DQ409327的非洲肯尼亚疣猪的mtDNA D-Loop部分序列作为外类群,利用MEGA5.10软件应用邻接法(NJ)构建了若尔盖地区藏猪和中国地方猪、野猪和国外野猪以及欧美引入猪种mtDNA D-Loop部分序列的系统发育树,详见图 4。

|
节点旁数字为1 000次Bootstrap检验后置信度值(%) Numbers at nodes indicate Bootstrap values (%) with 1 000 replicates 图 4 基于不同猪种部分mtDNA D-Loop序列构建的系统进化树 Fig. 4 Neighbor joining tree based on the mtDNA partial D-Loop sequences of different pig breeds |
从图 4可以看出,所构建的NJ系统树明显地将外类群的非洲疣猪与其它猪种分开,野猪间单倍型都聚为一类,而马来西亚野猪与其它野猪遗传距离较远;野猪与家猪的部分单倍型不能分开,不过所有的欧美引种猪聚为一类;若尔盖地区藏猪的单倍型Hap_1和Hap_2与福建野猪聚为一类,单倍型Hap_9与云南迪庆的藏猪聚为一类,单倍型Hap_8与雅南猪聚为一类,单倍型Hap_10与贵州可乐猪聚为一类。降扎乡、热尔乡和冻列乡藏猪的单倍型在整个系统进化树中分布较广,三者的母源血统遗传复杂,彼此之间以及与其它乡镇猪种间基因交流较多。总的来说,若尔盖地区藏猪在进化树中比较分散,说明该地区藏猪母源血统遗传复杂,彼此之间基因交流多,且若尔盖地区藏猪与其它猪种含有多种共享单倍型,母缘的亲缘关系较近。
3 讨论 3.1 若尔盖地区藏猪mtDNA D-Loop区的序列特征Ursing和Arnason首次报道猪线粒体基因组序列全长为16 679 bp[30],但是随后的研究发现,该序列长度不是绝对的[12-13],因为在线粒体的控制区内存在不同数量的串联重复序列GTACACGTGC,其变化范围为14~29个[31],而在本研究中,若尔盖地区藏猪全部是以TACACGTGCG为串联重复单位,串联重复序列数量为20~30个。刘靖闻等[32]对甘南州135头蕨麻猪mtDNA D-Loop全序列分析发现其表现出3种长度变异,其中4个序列长度为1 199 bp,3个序列长度是1 319 bp,127个序列长度为1 219 bp。在广西和贵州3个猪种中也存在这种情况[33-34]。造成这种长度差异除了少数碱基缺失或插入外,其主要是由于D-Loop区内的串联重复序列的拷贝数不同所致。除此之外,其他地区的藏猪、家猪以及杜洛克猪、长白猪、大白猪和约克夏猪等引进猪种均是以TACACGTGCG为串联重复单位(表 2中不同猪种的mtDNA信息)。不过,山东8个猪种线粒体的控制区串联重复序列为ACGTGCGTAC[35]。D-Loop区内串联重复序列长度均是10 bp,但是串联重复序列的碱基组成不同,具体原因有待进一步研究。
本研究中分析的若尔盖地区藏猪mtDNA D-Loop部分序列长度为435 bp,该段序列位于甘南藏族自治州藏猪mtDNA全序列的387~821 bp,占线粒体D-Loop区全长的41.77%(不包括重复序列),处于高变区HV1和HV2之中。同时,在不考虑插入、缺失的条件下,共检测到14个变异位点,占所测核苷酸的3.22%,说明该核苷酸序列突变率小,有较好的稳定性。其中有9个单一变异位点和5个简约信息位点,且核苷酸的替代全部是转换,可见序列突变还远远未达到饱和。山东猪种核苷酸替换以转换为主,转换明显多于颠换,转换/颠换总体比率为8.839[35]。本试验中,核苷酸序列的突变没有出现碱基的颠换,可能是由于获得435 bp高变区序列不完整,其位于HV1和HV2之间,没有涵盖到颠换的碱基片段。
mtDNA分子是由很不均一的片段构成的,G+C含量为21%~50%,其中无脊椎动物为21%~43%,脊椎动物为37%~50%[36]。本试验中,G+C含量为43.54%,处于变化范围的中间值。另外,A+T含量为56.46%,显著高于G+C含量。其中,A+T的含量可以反映出序列的变异性,具有高比例的A+T可能是D-Loop序列变异较快的原因之一,同时也说明mtDNA D-Loop高变区富含A、T,表现出很强的碱基偏倚性,这一结果与已报道的猪[8]、绵羊[9]、家禽[6]等动物的研究结果一致。
3.2 基于mtDNA D-Loop区部分序列的若尔盖地区藏猪遗传多样性分布规律单倍型多样度(Hd)[37]和核苷酸多样度(Pi)[38]是衡量群体mtDNA遗传变异的两个重要指标,Hd值和Pi值越大,说明群体的多样性程度越高,遗传多样性越丰富,反之,多样性程度越低,遗传多样性越贫乏。郭永博和蔡原[23]对西藏4个藏猪群体(林芝、山南、昌都和日喀则)遗传多样性的研究发现,Hd和Pi值分别为0.538±0.036和0.001 98、0.816±0.029和0.003 51、0.883±0.014和0.004 31、0.420±0.110和0.001 27。合作、迪庆、甘孜州、阿坝州的4个藏猪群体 Hd值分别为0.963、0.930、0.303和0.830,Pi值分别为0.008 20、0.005 50、0.000 700和0.003 60[22]。本研究中,若尔盖地区藏猪Hd和Pi分别为0.881和0.004 66,比西藏林芝、山南、日喀则、甘孜州、阿坝州藏猪的Hd和Pi都偏大,说明若尔盖地区藏猪遗传多样性程度高,受人工选择的影响程度小。同时,对17个单倍型进行的区域分布研究发现,无论从单倍型数、单倍型多样度还是核苷酸多样度来看,降扎乡藏猪都是最高的,说明了降扎乡藏猪的遗传多样性最丰富。联合国粮农组织历年关于家养动物报告中都涉及到有关遗传多样性调查的内容,要求不断改进和完善保存理论及保持机制,不断加大各区域保种项目的投资力度。因此,若尔盖地区藏猪遗传资源的保护过程中应该遵循遗传多样性原则进行优先保护。
3.3 若尔盖地区藏猪系统发生关系线粒体基因组的突变是通过母系世代进化而来的,并固定下来形成mtDNA单倍型[11]。若尔盖地区藏猪共享单倍型共有8个,特有单倍型9个,其中Hap_1和Hap_15单倍型是降扎乡、益哇乡、热尔乡和冻列乡群体的共享单倍型,表明这4个乡镇藏猪群体存在两个共同的母系祖先单倍型。有研究表明,包括藏猪在内的家猪来源于亚洲野猪和欧洲野猪[39-40],而且是多次驯化事件的结果[13],就不同藏猪群体而言存在着共同的母系起源,但在几千年的驯化中形成了不同的进化分支,由于生活环境差异,各群体也存在一定数量的特有单倍型,且频率差异较大[41],其中藏猪的进化分支有一定的地理特征,即群体特异性,欧洲起源家猪与亚洲起源家猪及藏猪明显的分为两个不同的进化支,在藏猪群体中西藏藏猪与甘孜藏猪处于同一个进化支,而合作藏猪形成了一个相对独立的进化分支,阿坝藏猪在各个进化支中都有出现[22],若尔盖地区藏猪的基因型也广泛的与中国地方家猪、野猪聚在一起。而若尔盖地区藏猪中除了包座乡、巴西乡和崇尔乡藏猪没有特有单倍型,其它6个乡镇群体都存在特有单倍型,其中,降扎乡藏猪的特有单倍型最多,占单倍型总数的17.65%。可能是由于崇尔乡藏猪没有经过长期的自然选择和人工选择,受到的人工选育程度低,致使该群体没有形成自己独特的遗传结构,其余6个群体虽然在遗传结构上有独特性,但它们都属于一个地区,群体间有密切的关系。本研究中,Hap_1单倍型出现的频率最大,为26.25%,已报道的阿坝州藏猪的mtDNA D-Loop序列也属于该单倍型。mtDNA单倍型能够区分出动物的品种和品系,并赋予动物优势和劣势[2]。有研究表明,线粒体DNA的基因型与猪的肉质[42]有一定的关系。此外,它还与猪的繁殖能力和产仔数有很大的关系[43-44]。可见,本研究获得的17种线粒体DNA的单倍型,为后续若尔盖地区藏猪的保护与利用提供了重要的基础资料。不过,由于若尔盖地区收集到的藏猪的数量有限,要明确分析该地区藏猪的起源及遗传背景,还需扩大样本量作进一步的研究。
4 结论本研究收集了若尔盖地区9个乡镇80头藏猪的耳组织,获得80条藏猪mtDNA D-Loop高变区(435 bp)序列,鉴定了17个单倍型,Hap_1和Hap_15单倍型是降扎乡、益哇乡、热尔乡和冻列乡藏猪群体的共享单倍型,表明这4个乡镇藏猪群体存在两个共同的母系祖先单倍型。构建的NJ分子系统进化树将若尔盖地区藏猪分为2支,而加入中国地方家猪、野猪和引种猪后,若尔盖地区藏猪在进化树中比较分散,说明该地区藏猪母源血统遗传复杂,彼此之间基因交流多。本研究进一步证实了若尔盖地区藏猪比西藏林芝、山南、日喀则、甘孜州、阿坝州藏猪的遗传多样性程度高,受到人工选择强度低,应强化对若尔盖地区藏猪遗传资源的保护与利用。
| [1] | GVOZDANOVIĆ K, MARGETA V, MARGETA P, et al. Genetic diversity of autochthonous pig breeds analyzed by microsatellite markers and mitochondrial DNA D-loop sequence polymorphism[J]. Anim Biotechnol, 2019, 30(3): 242–251. |
| [2] | RUIZ-PESINI E, MISHMAR D, BRANDON M, et al. Effects of purifying and adaptive selection on regional variation in human mtDNA[J]. Science, 2004, 303(5655): 223–226. |
| [3] |
姚一博, 贾媛, 夏小婷, 等. 湘西黄牛mtDNA D-loop区遗传多样性研究[J]. 中国牛业科学, 2017, 43(5): 11–13, 27.
YAO Y B, JIA Y, XIA X T, et al. Genetic diversity of mitochondrial DNA D-loop region in Xiangxi cattle[J]. China Cattle Science, 2017, 43(5): 11–13, 27. (in Chinese) |
| [4] |
薛艳洁, 潘娜, 李明云. 基于线粒体COⅡ基因和D-loop区序列对江西安南光唇鱼(Acrossocheilus)物种鉴定及系统发育位置分析[J]. 生物学杂志, 2019, 36(1): 25–28.
XUE Y J, PAN N, LI M Y. Species identification and phylogenetic position of Acrossocheilus fasciatus from Annan in Jiangxi Province based on mitochondrial COⅡ and D-loop region sequences[J]. Journal of Biology, 2019, 36(1): 25–28. (in Chinese) |
| [5] | ROSEL P E, HAYGOOD M G, PERRIN W F. Phylogenetic relationships among the True Porpoises (Cetacea:Phocoenidae)[J]. Mol Phylogenet Evol, 1995, 4(4): 463–474. |
| [6] |
谢艾轩, 吴启超, 李思德, 等. 四川麻鸭mtDNA全序列的克隆和生物信息学分析[J]. 畜牧兽医学报, 2017, 48(3): 436–445.
XIE A X, WU Q C, LI S D, et al. Complete sequence cloning and bioinformatics analysis of Sichuan Sheldrake duck mitochondrial genome[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(3): 436–445. (in Chinese) |
| [7] | CSIZMÁR N, MIHK S, JÁVOR A, et al. Genetic analysis of the Hungarian draft horse population using partial mitochondrial DNA D-loop sequencing[J]. PeerJ, 2018, 6: e4198. |
| [8] |
王志敏, 徐亚欧, 孙艳, 等. 乌金猪mtDNA D-Loop区的遗传多样性[J]. 吉首大学学报:自然科学版, 2018, 39(2): 65–69.
WANG Z M, XU Y O, SUN Y, et al. Genetic diversity of mtDNA D-Loop sequences in Wujin pig[J]. Journal of Jishou University:Natural Science Edition, 2018, 39(2): 65–69. (in Chinese) |
| [9] |
梁瑞圆, 陈晓勇, 孙洪新, 等. 五个地方绵羊种群mtDNA D-loop区系统进化及遗传多样性分析[J]. 华北农学报, 2018, 33(1): 135–142.
LIANG R Y, CHEN X Y, SUN H X, et al. Phylogenety and genetic diversity of five local sheep breeds on mtDNA D-loop[J]. Acta Agriculturae Boreali-Sinica, 2018, 33(1): 135–142. (in Chinese) |
| [10] | STONEKING M, SOODYALL H. Human evolution and the mitochondrial genome[J]. Curr Opin Genet Dev, 1996, 6(6): 731–736. |
| [11] | WALLACE D C, RUIZ-PESINI E, MISHMAR D. mtDNA variation, climatic adaptation, degenerative diseases, and longevity[J]. Cold Spring Harb Symp Quant Biol, 2003, 68: 479–486. |
| [12] | LI M Z, JIN L, MA J D, et al. Detecting mitochondrial signatures of selection in wild Tibetan pigs and domesticated pigs[J]. Mitochondrial DNA Part A, 2016, 27(1): 747–752. |
| [13] | WU G S, YAO Y G, QU K X, et al. Population phylogenomic analysis of mitochondrial DNA in wild boars and domestic pigs revealed multiple domestication events in East Asia[J]. Genome Biol, 2007, 8(11): R245. |
| [14] | WANG R M, WU X H, CHENG X Z, et al. The complete mitochondrial genome of Dabieshan black pig (Sus scrofa) and a phylogenetic study[J]. Mitochondrial DNA Part B, 2018, 4(1): 876–877. |
| [15] | NGUYEN H D, BUI T A, NGUYEN P T, et al. The complete mitochondrial genome sequence of the indigenous I pig (Sus scrofa) in Vietnam[J]. Asian-Australas J Anim Sci, 2017, 30(7): 930–937. |
| [16] | XU Y Y, TIAN X X, CHEN L L, et al. The complete mitochondrial genome of Juema pig Sus scrofa (Suina:Suidae) from southern Gansu[J]. Mitochondrial DNA Part A, 2016, 27(5): 3697–3698. |
| [17] | VO T T B, NGUYEN H D, BUI T A, et al. Phylogenomic analysis and gene organization of mitogenome from Mong Cai pig in Vietnam[J]. Curr Sci, 2019, 116(9): 1566–1571. |
| [18] | YANG J, WANG J, KIJAS J, et al. Genetic diversity present within the near-complete mtDNA genome of 17 breeds of indigenous Chinese pigs[J]. J Hered, 2003, 94(5): 381–385. |
| [19] | JIN L, ZHANG M W, MA J D, et al. Mitochondrial DNA evidence indicates the local origin of domestic pigs in the upstream region of the Yangtze River[J]. PLoS One, 2012, 7(12): e51649. |
| [20] | ZHANG J X, JIAO T, ZHAO S G. Genetic diversity in the mitochondrial DNA D-loop region of global swine (Sus scrofa) populations[J]. Biochem Biophys Res Commun, 2016, 473(4): 814–820. |
| [21] | TOUMA S, SHIMABUKURO H, ARAKAWA A, et al. Maternal lineage of Okinawa indigenous Agu pig inferred from mitochondrial DNA control region[J]. Asian-Australas J Anim Sci, 2019, 32(4): 501–507. |
| [22] | JIAO T, ZHAO S G, WANG C, et al. Mitochondrial DNA D-loop diversity of Tibetan pig populations[J]. Philipp Agric Sci, 2019, 92(4): 362–369. |
| [23] |
郭永博, 蔡原. 西藏藏猪遗传多样性研究[J]. 生物技术通报, 2015, 31(5): 224–230.
GUO Y B, CAI Y. The study of genetic diversity in Tibetan pig of Tibet[J]. Biotechnology Bulletin, 2015, 31(5): 224–230. (in Chinese) |
| [24] | SU Y F, LIU Y C, CHEN Y M, et al. Detection and phylogenetic analysis of porcine epidemic diarrhea virus in central China based on the ORF3 gene and the S1 gene[J]. Virol J, 2016, 13(1): 192. |
| [25] | ZHANG J X, YANG B C, WEN X C, et al. Genetic variation and relationships in the mitochondrial DNA D-loop region of Qinghai indigenous and commercial pig breeds[J]. Cell Mol Biol Let, 2018, 23(1): 31. |
| [26] | THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The Clustal_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 4876–4882. |
| [27] | LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451–1452. |
| [28] |
赵拴平, 贾玉堂, 徐磊, 等. 安徽东流水牛线粒体D-Loop区遗传多样性与系统进化分析[J]. 西北农林科技大学学报:自然科学版, 2016, 44(12): 21–27.
ZHAO S P, JIA Y T, XU L, et al. Genetic variation and phylogeny of Dongliu Buffalo based on mitochondrial DNA D-Loop[J]. Journal of Northwest A&F University: Natural Science Edition, 2016, 44(12): 21–27. (in Chinese) |
| [29] | TAMURA K, STECHER G, PETERSON D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725–2729. |
| [30] | URSING B M, ARNASON U. The complete mitochondrial DNA sequence of the pig (Sus scrofa)[J]. J Mol Evol, 1998, 47(3): 302–306. |
| [31] | GHIVIZZANI S C, MACKAY S L D, MADSEN C S, et al. Transcribed heteroplasmic repeated sequences in the porcine mitochondrial DNA D-loop region[J]. J Mol Evol, 1993, 37(1): 36–47. |
| [32] |
刘靖闻, 张勇, 赵兴绪. 甘南州蕨麻猪mtDNA D-环序列的起源及遗传多样性分析[J]. 中国兽医学报, 2013, 33(6): 933–938.
LIU J W, ZHANG Y, ZHAO X X. Genetic diversity and origin of Gannan Juema pig by mitochondrial DNA D-loop sequence[J]. Chinese Journal of Veterinary Science, 2013, 33(6): 933–938. (in Chinese) |
| [33] |
陈祥, 丁玫, 孙鹃, 等. 贵州3个地方猪种的mtDNA D-loop序列遗传多样性分析[J]. 畜牧与兽医, 2015, 47(5): 55–59.
CHEN X, DING M, SUN J, et al. Genetic diversity analysis of mtDNA D-loop sequences in three local pig breeds in Guizhou[J]. Animal Husbandry & Veterinary Medicine, 2015, 47(5): 55–59. (in Chinese) |
| [34] |
綦文晶, 何剑雄, 郭晓萍, 等. 广西3个猪种线粒体DNA D-loop序列遗传多样性及系统进化研究[J]. 基因组学与应用生物学, 2016, 35(9): 2367–2374.
QI W J, HE J X, GUO X P, et al. The genetic diversity and phylogenetic relationships among three Guangxi pig breeds based on sequences of mtDNA D-loop[J]. Genomics and Applied Biology, 2016, 35(9): 2367–2374. (in Chinese) |
| [35] |
王继英, 郭建凤, 孙守礼, 等. 山东猪种mtDNA D-loop区部分序列遗传多样性及系统进化研究[J]. 畜牧兽医学报, 2009, 40(6): 792–799.
WANG J Y, GUO J F, SUN S L, et al. The genetic diversity and phylogenetic relationships among Pig breeds of Shandong province based on partial sequence of mtDNA D-loop region[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(6): 792–799. (in Chinese) |
| [36] | JIA S G, CHEN H, ZHANG G X, et al. Genetic variation of mitochondrial D-loop region and evolution analysis in some Chinese cattle breeds[J]. J Genet Genomic, 2007, 34(6): 510–518. |
| [37] | NEI M, TAJIMA F. DNA polymorphism detectable by restriction endonucleases[J]. Genetics, 1981, 97(1): 145–163. |
| [38] | LI W H, SADLER L A. Low nucleotide diversity in man[J]. Genetics, 1991, 129(2): 513–523. |
| [39] | GIUFFRA E, KIJAS J M, AMARGER V, et al. The origin of the domestic pig:independent domestication and subsequent introgression[J]. Genetics, 2000, 154(4): 1785–1791. |
| [40] | WATANABE T, HAYASHI Y, KIMURA J, et al. Pig mitochondrial DNA:polymorphism, restriction map orientation, and sequence data[J]. Biochem Genet, 1986, 24(5-6): 385–396. |
| [41] | LARSON G, DOBNEY K, ALBARELLA U, et al. Worldwide phylogeography of wild boar reveals multiple centers of pig domestication[J]. Science, 2005, 307(5715): 1618–1621. |
| [42] | FERNÁNDEZ A I, ALVES E, FERNÁNDEZ A, et al. Mitochondrial genome polymorphisms associated with longissimus muscle composition in Iberian pigs[J]. J Anim Sci, 2008, 86(6): 1283–1290. |
| [43] | TSAI T S, RAJASEKAR S, JOHN J C S. The relationship between mitochondrial DNA haplotype and the reproductive capacity of domestic pigs (Sus scrofa domesticus)[J]. BMC Genet, 2016, 17(1): 67. |
| [44] | JOHN J C S, TSAI T S. The association of mitochondrial DNA haplotypes and phenotypic traits in pigs[J]. BMC Genet, 2018, 19(1): 41. |



