2. 江苏省农业科学院动物免疫工程研究所, 南京 210014
2. Institute of Animal Immune Engineering, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China
产仔数是猪育种中母猪生产性能测定的一个重要指标,也是一个十分重要的繁殖性状和经济性状[1-2]。由于猪的产仔数性状遗传力(0.10~0.15)非常低和性别限制,所以用传统的选择手段进行猪产仔数的育种比较困难[3-4]。产仔数受许多因素的影响,其中胚胎附植是一个重要影响因素,因为大部分胚胎会在猪胚胎附植期(妊娠13~24 d)死亡,所以这一时期成为影响猪产仔数的重要时期,如何减少这一时期的胚胎死亡成为亟待解决的问题[5]。
猪胚胎附植开始于妊娠第12~14天[5-6],在第18天进入附植高峰[7],在第24天左右结束[8]。在胚胎附植过程中,卵巢分泌一些物质,使子宫发育到胚胎附着的接受状态,胚泡再根据子宫内膜附植点的信号着床[9]。在妊娠30 d时,用6头高育种值母猪和6头低育种值母猪进行了卵巢基因表达分析,鉴定了27个差异表达基因与猪产仔数QTL紧密关联[10]。另外,在胚胎附植过程中给母猪注射雌激素,鉴定出一些由于破坏妊娠而导致的异常基因表达[11]。尽管胚胎附植对产仔数有影响,但目前人们对猪的胚胎附植准确调控机制了解甚少。梅山猪是优秀的高繁中国本土品种[12],具有产仔数多、泌乳能力强、肉质鲜美等优点, 妊娠第30天的梅山猪胚胎死亡率(21%)比大白猪(45%)低很多[13],这就说明,高繁母猪的胚胎附植率更高[14-15]。近年来,高通量测序技术的迅速发展,为人们进一步挖掘猪胚胎附植关键基因提供了新的途径[16]。
本研究以梅山猪为试验动物,利用转录组测序(RNA sequencing, RNA-seq)分析猪胚胎附植卵巢中期和后期差异表达基因,获得了一些有意义的结果。
1 材料与方法 1.1 试验动物试验动物来自于江苏农林职业技术学院梅山猪保种场,以胚胎附植期的梅山猪母猪(第5~7胎次)为研究对象,按猪场常规进行饲喂和管理,同时在自由采食及饮水条件下,注意进行疾病的防治和行为体况的观察。在同场、相同管理条件下进行单圈饲养,进行同期发情处理,用相同种公猪配种两次, 以最后一次配种为0 d计,在妊娠第18(附植中期)和24天(附植后期)进行屠宰,各屠宰2头,采集卵巢组织样,立即放置于液氮保存。
1.2 RNA提取与检测采用TRIzol /氯仿方法[17]从猪卵巢组织中提取总RNA,之后用超微量紫外分光光度计(Thermo NANODROP 2000 Spectrophotometer, USA)检测RNA浓度,再用1.5%的琼脂糖电泳检测RNA完整性。RNA质量评估标准:OD260 nm/OD280 nm值为1.8~2.2,OD260 nm /OD230 nm为1.7~2.2,RNA电泳出现18S和28S两条带,条带清晰,无降解(图略)。
1.3 转录组测序(RNA-seq)流程采用illumina Hiseq2000测序平台的双端100 bp测序模式对样本进行高通量测序。原始数据经过引物与adaptor序列去除, 最终保留高质量的碱基片段。保留的序列(remained reads)片段用于后续RNA-Seq进行模块分析。转录组测序流程:1)根据碱基质量控制与过滤及Read长度范围控制对原始数据进行质量控制筛查;2)与参考基因组(NCBI, Sus scrofa Scrofa11.1)进行外显子、内含子、UTR非编码区,上、下游调控区进行基因组比对,进而进行基因表达量分析和基因差异表达分析。差异基因表达分析以后,又分成两个流程:1)依次进行转录本预测识别、基因结构模型的识别优化、转录本表达量计算分析、转录本差异表达分析、转录本功能预测和蛋白结构域分析;2)生物通路(代谢通路和信号转导通路)分析预测、GO分子功能注释和GO功能富集度分析,采用KEGG(kyoto encyclopedia of genes and genomes,www.genome.jp/kegg/)生物通路数据库进行富集度分析(enrichment analysis)。
1.4 测序数据的基因组比对将样本预处理后,序列与参考基因组序列进行全基因组序列比配,计算序列的全基因组覆盖情况和RNA的表达量。序列比配需要考虑到RNA结构特点,采用Tophat软件完成基因组比配。比配参数和比配的条件:1)允许2个错配碱基模式;2)允许每个read测序序列最多20个最优比配记录;3)考虑可变剪切情况,分割片段区域(segment)的分段长度设定等于read长度的1/2;4)允许分段内错配碱基数目最多2 bp;5)最大插入和删除长度设定为3 bp;6)可变剪接位置比配要求0错配,即保证完全比配;7)最小异构体比例系数(min isoform-fraction)设定为0.15。该设置含义:如果有跨越2个外显子的跳跃(junction)由S个测序片段所覆盖,假设让外显子A的平均测序深度为D,比B的平均测序深度高。如果S/D < 设定的最小异构体比例系数(设置为0.15), 则不认为当前存在外显子跳跃(junction)存在。8)最小内含子长度(minimum intron length)设置为50 bp;9)最大内含子长度(maximum intron length)设置为50 kb;10)对跳跃(junction)探测的比配条件允许最多40个最优比配记录,允许2 bp错配。
1.5 已知基因表达量和差异性计算分析采用将序列拼装比配在基因组序列上,并通过统计学方法估算编码RNA表达量及用于后续样本差异表达量比较分析。通过Tophat软件对cDNA、mRNA转录组测序数据(RNA-Seq)reads的基因组比配,获得可针对剪切位点比配的基因组比配数据(spliced alignments)。对于两端测序片段(paired-end RNA-Seq);通过参考基因区域内比配read标准化算法(Cufflinks软件,版本号:2.2.1)对每个片段做独立比配处理,该算法在拼接过程中将叠加的比配片段进行联通,最后估计拼接转录本的丰度,即表达量。RNA表达量计算采用FPKM计算度量指标(FPKM- fragments per kilobase of transcript extro per million fragments mapped)。FPKM含义:以每百万比配序列每1 kb长度作RNA表达量指标,其中转录本长度和总比配read数目用于归一化表达量数值。在差异表达分析部分所计算的表达量,笔者采用的参数设置是考虑基于RNA注释坐标兼容并符合比配要求的read序列,即compatible-hits-norm模式(兼容比配表达量计算模式)。
笔者将试验样本(条件)之间的lg ratio与其中某个条件表达量的lg值作比较。假设转录本a和b表达量的比值为Y (Y=FPKMa/FPKMb), 两条件下表达量的比值Y的对数值(lgY)可以作为检验统计量,采用负伯努利分布计算检验统计量T(T=E (lgY)/Var (lgY), E为期望值,Var为变异方差), 得出差异表达显著值。此计算转录本的差异表达方法和计算表达量的方法具有高度的灵敏性。
2 结果 2.1 转录组测序数据质量控制转录组测序数据质量系列评估结果表明,胚胎附植中期卵巢(卵_18 d)和后期卵巢(卵_24 d)的组织样测定出的序列中,常见基因序列按照占比大小,卵_18 d样品:外显子编码序列(CDS_exons,42.81%)、内含子(introns,16.82%)、3′-外显子非翻译区(3′UTR_exons,8.03%)和5′-外显子非翻译区(5′UTR_exons 5.82%),卵_24 d样品:CDS_exons(47.60%)、3′UTR_exons (7.63%)、Introns (7.18%)和5′UTR_exons (5.93%)。
基因组各分段测序数量分布结果表明,两个样品(卵_18 d和卵_24 d)的曲线图基本一致,都是先升高后降低,峰值出现的地方一致,测序数(read number)从基因全长(5′→3′)5%左右陡增,在40%的时候有一个急速降低,随之到70%的时候达到峰值,然后快速下降。即转录组测序覆盖了基因组95%(从5%~100%)的序列,且在覆盖的基因组中,每一段的测序数量大都在200 000~350 000(图 1 A, B)。A、T、G、C 4碱基频率结果表明,两个样品(卵_18 d和卵_24 d)所测序列前10个碱基的核苷酸频率波动比较大,之后趋于平稳,其中卵_18 d的C和G两碱基的核苷酸频率始终高于A和T两碱基,平均高于0.2(图 1 C);卵_24 d的A、T、G和C碱基的核苷酸频率则始终缠在一起,维持在0.24~0.26(图 1 D)。
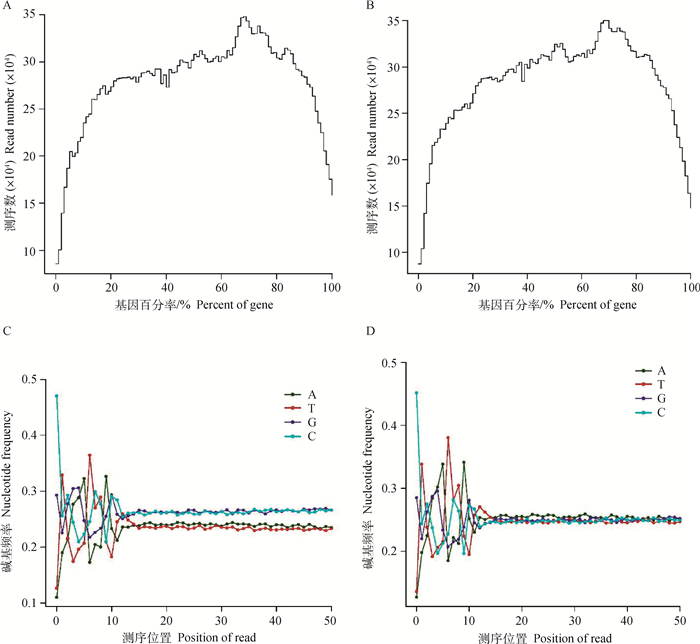
|
A、B.卵_18 d和卵_24 d基因组各分段测序数量分布;C、D.卵_18 d和卵_24 d基因测序序列A、T、G和C 4碱基频率 A, B.Quantity distribution of reads for genome of ovary_18 d and ovary_24 d; C, D.Frequency of A, T, G, C for reads of ovary_18 d and ovary_24 d 图 1 质量控制分析 Figure 1 Results of quality control analysis |
RNA-seq可以获得基因组序列的类别:正义外显子、反义外显子、正义内含子、反义内含子、正义3′-非编码区、反义3′-非编码区、正义5′-非编码区、反义5′-非编码区、正义编码序列、反义编码序列、正义下游1 kb、反义下游1 kb、正义上游1 kb和反义上游1 kb,14类别在饼形图中按顺时针依次排列(图 2 A, B)。表明,胚胎附植中期卵巢(卵_18 d)和后期卵巢(卵_24 d)两个样品的测序结果中,各类别所占比例有相同,也有不同。其中,相同点:正义外显子和反义外显子类别所占比例最大,且份额基本相等,二者之和(全部外显子)约占总类别1/3,其次为反义编码序列和正义编码序列;不同点:卵_18 d的正义内含子和反义内含子所占比例(10%,9%)比卵_24 d的(7%,7%)大,同时卵_18 d的正义上游1 kb和反义上游1 kb所占比例(4%,3%)比卵_24 d的大(3%,3%)。
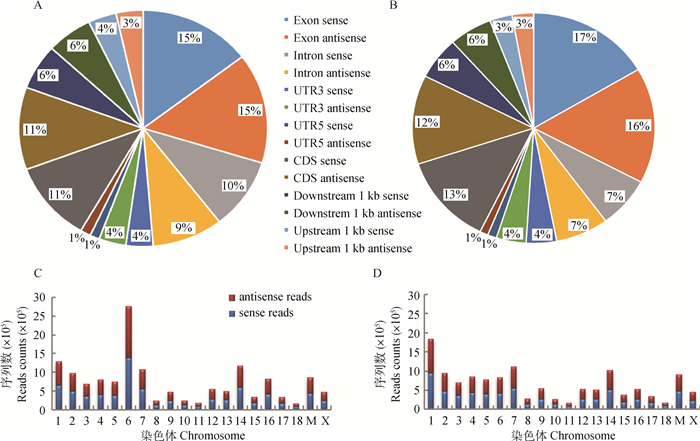
|
A、C.卵_18 d;B、D.卵_24 d A, C. Ovary_18 d; B, D.ovary_24 d 图 2 转录组测序获得的基因组序列分类(A、B)和在基因组各染色体测到的序列数量(C、D) Figure 2 Classification (A, B)of genomic sequence and genome alignment counts distribution(C, D) in each chromosome by RNA-seq |
RNA-seq分别获得胚胎附植中期卵巢(卵_18 d)和后期卵巢(卵_24 d)组织样14 579 752和13 119 531个测序序列(reads)。其中卵_18 d在6号染色体中得到的reads最多,其次是14、1、7、16、M、2号染色体;卵_24 d在1号染色体中得到的reads最多,其次是7、14、M、2、4、6号染色体(图 2 C, D),这说明卵巢在猪胚胎附植两个时期reads所在位置有较大的差异,即两个时期的表达基因存在较大的差异。正义序列(sense reads)指的是基因组正义链中的比配序列数量,反义序列(antisense reads)指的是基因组反义链中的比配序列数。结果表明,胚胎附植期卵_18 d和卵_24 d两个组织样的表达基因检测到的外显子序列最多,即大部分检测的基因都在猪胚胎附植中期和后期发挥着作用,且附植中期的主效调控基因可能位于6号染色体,附植后期的主效调控基因可能位于1号染色体。
2.3 猪胚胎附植中期和后期卵巢基因表达量检测猪胚胎附植中期卵巢(卵_18 d)和后期卵巢(卵_24 d)两个组织样分别检测出16 159个和16 373个有表达量的基因,卵_18 d和卵_24 d的基因表达量最高值分别为2.96×106和2.97×106,基因表达量最低值分别为3.44×10-231和1.05×10-260,平均值分别为955.31和1 075.62。这些结果表明,猪胚胎附植后期卵巢(卵_24 d)相较于中期卵巢(卵_18 d),有更多的基因表达,且基因的表达量更高(无论是平均数还是峰值);卵_18 d有1 198个基因的表达量高于100,卵_24 d的为1 357个;卵_18 d有316个基因的表达量高于500,卵_24 d的为394个;卵_18 d有159个基因的表达量高于1 000,卵_24 d的为209个;卵_18 d有46个基因的表达量高于1 000,卵_24 d的为53个。
两个组织样中最高表达量的基因是同一个基因,为EN19669,位于线粒体中(序列位点:6 443~6 509),该基因在卵_18 d和卵_24 d中的表达量:2.96×106和2.97×106。两个组织样中表达量最高的已知基因也相同:MT-ATP8,位于线粒体染色体中(序列位点:8 958~9 162),在卵_18 d和卵_24 d中的表达量:129 142和104 391。卵_18 d组织样中表达量排前13位的基因均位于线粒体中,表达量第14位(染色体中第1位)的位于14号染色体中,为EN11278(序列位点:79 769 815~79 769 890),表达量前50位的基因中,有35个基因位于线粒体中。卵_24 d组织样中表达量排前12位的基因均位于线粒体中,表达量第13位(染色体中第1位)的位于14号染色体中,为EN11278基因(与卵_18 d相同),表达量前50位的基因中,有34个基因位于线粒体中。两个时期卵巢组织样中表达量前50位的基因中,线粒体基因占了≥60%,即线粒体基因很可能在猪胚胎附植调控中发挥着重要作用。
2.4 猪胚胎附植中期和后期卵巢差异表达基因分析猪胚胎附植中期卵巢(卵_18 d)和后期卵巢(卵_24 d)两个组织样之间有2 682个显著差异表达基因(P < 0.05),包含1 998个上调基因和684个下调基因(图 3A);显著差异表达基因中又包含684个极显著差异表达基因(P < 0.01, q < 0.05), 分别为581个上调基因和103个下调基因(卵_24 d相较于卵_18 d) (图 3A, B, D)。图 3C中卵_18 d和卵_24 d样品检测出差异表达基因的表达量均呈中间大、两头小的分布,且前者的平均表达量低于后者。火山图表明,卵_18 d和卵_24 d之间有比较多的显著差异表达基因(红点)(图 3 D),热图表明猪胚胎附植卵巢中期和后期差异表达基因的mRNA表达量大多为10~100(101~102)(图 3 E)。
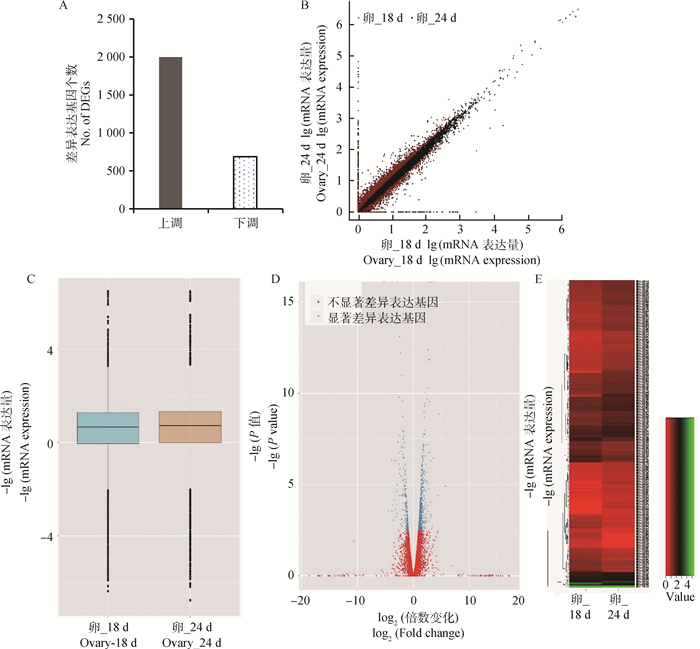
|
A~E.上调和下调基因数、差异基因mRNA表达量散点图、盒形图、火山图和热图 A-E.Number of up-regulated DEGs and down-regulated DEGs, scatter plots of DEGs mRNA expression intensity, box plots, volcano, heat maps 图 3 猪胚胎附植中期(卵_18 d)和后期(卵_24 d)卵巢组织差异表达基因分布 Figure 3 Distribution of differentially expressed genes (DEGs) between ovary_18 d and ovary_24 d |
卵_18 d和卵_24 d之间差异最大的基因为EN19684,位于线粒体中(序列位点:12 805~12 864),差异最大的染色体基因为EN11278,位于14号染色体中(序列位点:79 769 815~79 769 890),差异最大的已知基因为TIMP1,位于X染色体中(序列位点:42 058 788~42 110 182)。
2.5 猪胚胎附植中期和后期卵巢差异表达基因GO功能富集分析对胚胎中期和后期的卵巢组织差异表达基因进行GO功能富集,生物学过程结果表明(图 4A):卵巢两个时期之间的差异表达基因有2 954个富集的生物学过程(BP),其中前38个为显著富集(P < 0.05),前12位(P=0)显著富集的生物学过程分别为内皮细胞活化、蛋白丝氨酸/苏氨酸磷酸酶活性的调节、硫酸乙酰肝素蛋白多糖分解代谢过程、建立平面极性的蛋白质定位、膜组件、维持蛋白质在质膜中的位置、长链脂肪酸代谢过程、过氧化物酶体的长链脂肪酸输入、双链断裂修复的正调控、蛋白多糖的分解代谢过程、硫化合物分解代谢过程和维持蛋白质在膜中的位置。
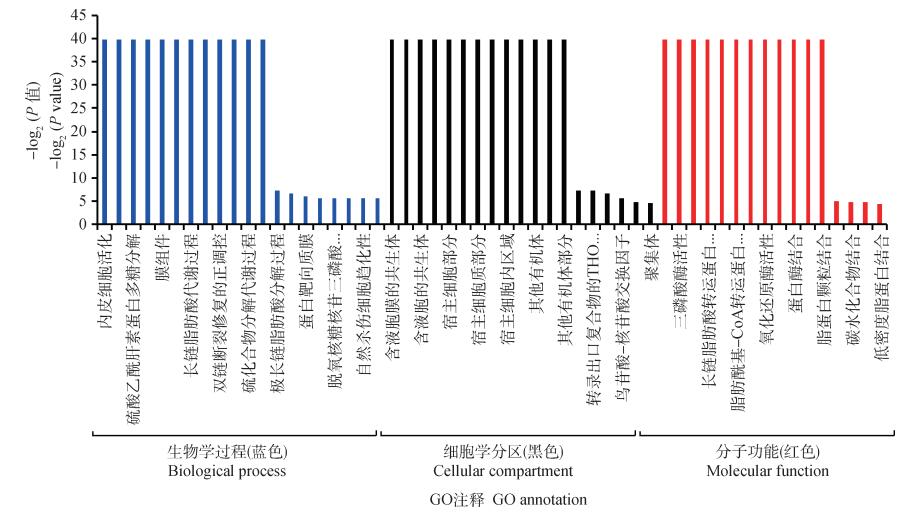
|
图 4 猪胚胎附植中期(Day 18)和后期(Day 24)卵巢组织差异表达基因的GO注释分布 Figure 4 GO annotation distribution of differential expression genes between ovary_18 d and ovary_24 d detected by RNA-seq |
细胞学分区结果表明(图 4),卵巢两个时期(胚胎附植中期和后期)之间的差异表达基因有381个富集的细胞学分区(CC),其中前19个为显著富集(P < 0.05),前13位(P=0)显著富集的细胞学分区:含液泡膜的共生体、含液膜的共生体、宿主细胞质、宿主细胞部分、宿主细胞内部分、宿主细胞质部分、体外空间、宿主细胞内区域、宿主细胞、其他有机体、其他有机体细胞和其他有机体部分。分子功能结果表明,卵巢两个时期之间的差异表达基因有630个富集的分子功能(MF),其中前16个为显著富集(P < 0.05),前6位(P=0)显著富集的分子功能分别为FAD-AMP裂解酶(环化)活性、三磷酸酶活性、过氧化物酶体脂肪酰基-CoA转运蛋白活性、长链脂肪酸转运蛋白活性、脂肪酰基转运蛋白活性和脂肪酰基-CoA转运蛋白活性。
2.6 猪胚胎附植中期和后期卵巢差异表达基因Pathway分析Pathway (生物通路)分析采用KEGG(kyoto encyclopedia of genes and genomes,www.genome.jp/kegg/)生物通路数据库进行富集度分析(enrichment analysis)。对卵巢组织胚胎中期和后期的差异表达基因进行生物通路(pathway)分析,结果表明(图 5),卵巢两个时期(胚胎附植中期和后期)之间的差异表达基因在185条生物通路上存在差异,其中有7条生物通路上存在极显著差异(P < 0.01),它们依次为PI3K-Akt信号通路、Hippo信号通路、昼夜周期、乙型肝炎通路、2-氧代羧酸代谢、HIF-1信号通路和化学致癌作用系统,接下来(0.3 < P < 0.05)依次为肾素-血管紧张素系统、不饱和脂肪酸的生物合成、ECM受体相互作用、缬氨酸 & 亮氨酸和异亮氨酸的生物合成、苯丙氨酸,酪氨酸和色氨酸生物合成等。
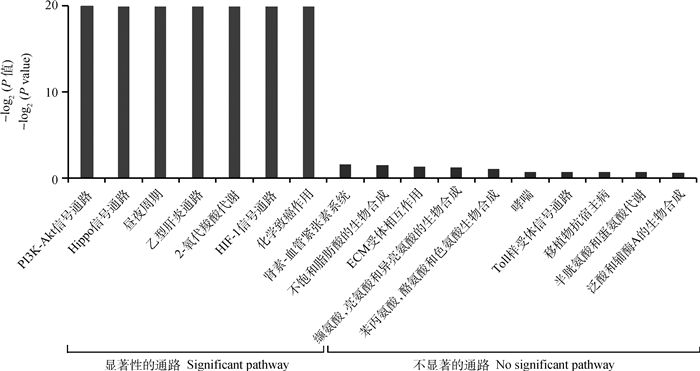
|
图 5 猪胚胎附植中期(Day 18)和后期(Day 24)卵巢组织差异表达基因的生物通路注释 Figure 5 Pathway annotation distribution of differential expression genes between tissues of ovary_18 d and ovary_24 d detected by RNA-seq |
提高胚胎附植时期胚胎存活率是提高猪产仔数的一个重要措施[14-15],另外猪的胚胎附植阶段比人长很多,且没有侵入子宫上皮细胞,这使猪成为一个很好的模式动物,用于研究人的胚胎附植期相关疾病[18]。研究胚胎附植需要找到这一繁殖活动的关键调控基因,RNA-seq(转录组测序)技术为这个研究提供了新的机会[19]。猪的胚胎附植开始于妊娠第13~14天,妊娠第18~24天附植完成[5, 8]。本研究中,成功测定了梅山猪妊娠第18天(胚胎附植中期)和第24天(胚胎附植后期)卵巢组织样中的差异表达基因,这些结果可以帮助人们了解关键基因调控猪胚胎着床,发现更多的影响胎仔数的候选基因,这些基因可以用于动物育种中的标记辅助选择(MAS)[20]。
外显子和内含子测序比对结果表明,胚胎附植期卵_18 d和卵_24 d两个组织样的表达基因检测到的外显子序列最多,即大部分检测的基因都在猪胚胎附植中期和后期发挥着作用,且附植中期的主效调控基因有可能在6号染色体,附植后期的主效调控基因有可能在1号染色体。研究表明,在猪6号染色体上可能存在影响窝产仔数的QTL[21]和一个可终止胎儿发育的基因或基因群[22];影响猪产仔数的雌激素受体基因(ESR)和胰岛素样生长因子受体(IGF1R)均位于1号染色体上[23],且ESR与IGF1R基因的表达量与排卵数呈显著相关[24]。
基因表达量结果表明,猪胚胎附植后期卵巢(卵_24 d)相较于中期卵巢(卵_18 d),有更多的基因表达,且基因的表达量更高;两个时期卵巢组织中表达量前50位的基因中,线粒体基因占了60%以上,即线粒体基因在猪胚胎附植调控中发挥着重要作用。两个组织样中最高表达量的基因是同一个基因,为EN19669,表达量最高的已知基因也相同,为MT-ATP8,两个基因均位于线粒体中。MT-ATP8的功能为参与ATP的合成从而为细胞发育提供能量[25]。线粒体是哺乳动物卵子发生和早期胚胎发育中的发育调节的一个连接点[26],动态调节线粒体在胚胎植入前和胚胎干细胞中发挥作用[27]。在小鼠中,高水平的线粒体代谢对头部发育至关重要[28]。差异表达基因分析结果表明,猪胚胎附植中期卵巢(卵_18 d)和后期卵巢(卵_24 d)两个组织样之间有2 682个显著差异表达基因(P < 0.05),包含1 998个上调基因和684个下调基因;显著差异表达基因中又包含684个极显著差异表达基因(P < 0.05, Q < 0.05), 包含581个上调基因和103个下调基因。这说明附植中期和后期卵巢组织同一基因的表达量出现了很大的变化,85%的基因表达上调,这些上调的基因很可能与母猪妊娠建立有关[16]。
前3位显著富集的生物学过程:内皮细胞活化、蛋白丝氨酸/苏氨酸磷酸酶活性的调节、硫酸乙酰肝素蛋白多糖分解代谢过程,研究表明,这几种生物学过程对胚胎植入过程中的胚胎生长和母体组织重构是必不可少的[29-30];细胞学区分为宿主细胞质、宿主细胞部分、宿主细胞内部分,这与猪卵巢内卵泡成熟和排卵的生理活动相一致;分子功能:FAD-AMP裂解酶(环化)活性、三磷酸酶活性、过氧化物酶体脂肪酰基-CoA转运蛋白活性。胚胎附植是一种磷脂酸结合过程,由各种激素调节胎儿发育和改变母体的生理机能,以支持怀孕[31]。卵巢两个时期(胚胎附植中期和后期)之间的差异表达基因在185条生物通路上存在差异,其中前3位极显著差异的生物通路依次为PI3K-Akt信号通路、Hippo信号通路、昼夜周期。
4 结论本研究结果表明,猪胚胎附植中期的主效调控基因有19%位于6号染色体,附植后期的主效调控基因有14%位于1号染色体;相对于附植中期,卵巢更侧重于胚胎附植后期的调控;线粒体基因在猪胚胎附植调控中发挥着重要作用;卵巢附植中期和后期差异表达基因功能以内皮细胞活化为主,生物通路以PI3K-Akt信号通路为主。
| [1] | LINVILLE R C, POMP D, JOHNSON R K, et al. Candidate gene analysis for loci affecting litter size and ovulation rate in swine[J]. J Anim Sci, 2001, 79(1): 60–67. DOI: 10.2527/2001.79160x |
| [2] | LIN H C, LIU G F, WANG A G, et al. Effect of polymorphism in the leukemia inhibitory factor gene on litter size in Large White pigs[J]. Mol Biol Rep, 2009, 36(7): 1833–1838. DOI: 10.1007/s11033-008-9387-0 |
| [3] | JOHNSON R K, NIELSEN M K, CASEY D S. Responses in ovulation rate, embryonal survival, and litter traits in swine to 14 generations of selection to increase litter size[J]. J Anim Sci, 1999, 77(3): 541–557. DOI: 10.2527/1999.773541x |
| [4] |
付言峰, 王爱国, 李兰, 等. 猪胚胎附植期Eph A4的组织表达及其多态性对产仔数的影响[J]. 中国农业大学学报, 2013, 18(3): 128–137.
FU Y F, WANG A G, LI L, et al. Expression of Eph A4 in tissues during embryo implantation and its polymorphism influence on litter size in pigs[J]. Journal of China Agricultural University, 2013, 18(3): 128–137. (in Chinese) |
| [5] | DANTZER V. Electron microscopy of the initial stages of placentation in the pig[J]. Anat Embryol, 1985, 172(3): 281–293. DOI: 10.1007/BF00318976 |
| [6] | REN Q, GUAN S, FU J L, et al. Expression of tissue inhibitor of metalloproteinases-3 messenger RNA and protein in porcine endometrium during implantation[J]. Mol Biol Rep, 2011, 38(6): 3829–3837. DOI: 10.1007/s11033-010-0498-z |
| [7] | FU Y F, LI L, LI B X, et al. Long form leptin receptor and SNP effect on reproductive traits during embryo attachment in Suzhong sows[J]. Anim Reprod Sci, 2016, 168: 57–65. DOI: 10.1016/j.anireprosci.2016.02.026 |
| [8] | KYRIAZAKIS I, WHITTEMORE C T. Whittemore's science and practice of pig production[M]. 3rd ed.Oxford, UK: Blackwell Publishing, 2006. |
| [9] | PSYCHOYOS A. Uterine receptivity for nidationa[J]. Ann New York Acad Sci, 1986, 476(1): 36–42. |
| [10] | FERNANDEZ-RODRIGUEZ A, MUNOZ M, FERNANDEZ A, et al. Differential gene expression in ovaries of pregnant pigs with high and low prolificacy levels and identification of candidate genes for litter size[J]. Biol Reprod, 2010, 84(2): 299–307. |
| [11] | ROSS J W, ASHWORTH M D, WHITE F J, et al. Premature estrogen exposure alters endometrial gene expression to disrupt pregnancy in the pig[J]. Endocrinology, 2007, 148(10): 4761–4773. DOI: 10.1210/en.2007-0599 |
| [12] |
周艳红, 付言峰, 赵为民, 等. 胚胎附植期梅山猪EphrinA1全编码区克隆及生物信息学分析[J]. 江苏农业学报, 2015, 31(2): 350–356.
ZHOU Y H, FU Y F, ZHAO W M, et al. Coding sequence cloning and bioinformatics analysis of EphrinA1 during embryo implantation in Meishan swine[J]. Jiangsu Journal of Agricultural Sciences, 2015, 31(2): 350–356. DOI: 10.3969/j.issn.1000-4440.2015.02.020 (in Chinese) |
| [13] | BAZER F W, THATCHER W W, MARTINAT-BOTTE F, et al. Conceptus development in large white and prolific Chinese Meishan pigs[J]. J Reprod Fertil, 1988, 84(1): 37–42. DOI: 10.1530/jrf.0.0840037 |
| [14] | WU Y P, WANG A G, LI N, et al. Association with TGF-β1 gene polymorphisms and reproductive performance of large white pig[J]. Reprod Domest Anim, 2010, 45(6): 1028–1032. DOI: 10.1111/j.1439-0531.2009.01480.x |
| [15] | WANG G Y, KONG L J, HU P, et al. Effect of polymorphism in the peroxisome proliferator-activated receptor gamma gene on litter size of pigs[J]. Mol Biol Rep, 2011, 38(3): 1807–1812. DOI: 10.1007/s11033-010-0296-7 |
| [16] | COSTA V, ANGELINI C, DE FEIS I, et al. Uncovering the complexity of transcriptomes with RNA-seq[J]. J Biomed Biotechnol, 2010, 2010: 853916. |
| [17] | SAMBROOK J, RUSSELL D W. Molecular cloning:A laboratory manual[M]. 3rd ed.New York: Cold Spring Harbor Laboratory Press, 2001. |
| [18] | LEE K Y, DEMAYO F J. Animal models of implantation[J]. Reproduction, 2004, 128(6): 679–695. DOI: 10.1530/rep.1.00340 |
| [19] | WANG W C, QIN Z Y, FENG Z X, et al. Identifying differentially spliced genes from two groups of RNA-seq samples[J]. Gene, 2013, 518(1): 164–170. DOI: 10.1016/j.gene.2012.11.045 |
| [20] | HUMPOLICEK P, TVRDON Z, URBAN T. Interaction of ESR1 gene with the FSHB and MYOG genes:Effect on the reproduction and growth in pigs[J]. Anim Sci Pap Rep, 2009, 27(2): 105–113. |
| [21] | WILKIE P J, PASZEK A A, BEATTIE C W, et al. A genomic scan of porcine reproductive traits reveals possible quantitative trait loci (QTLs) for number of corpora lutea[J]. Mamm Genome, 1999, 10(6): 573–578. DOI: 10.1007/s003359901047 |
| [22] | WADA Y, AKITA T, AWATA T, et al. Quantitative trait loci (QTL) analysis in a Meishan×Göttingen cross population[J]. Anim Genet, 2000, 31(6): 376–384. DOI: 10.1046/j.1365-2052.2000.00696.x |
| [23] | KMIEC M, TERMAN A, ZIEMAK J. Associations between polymorphism of the estrogen receptor gene (ESR/AvaI, ESR/PvuⅡ) and reproduction traits of Polish Landrace boars[J]. Folia Universitatis Agriculturae Stetinensis Zootechnica, 2005. |
| [24] |
颉孝贤, 王起山, 何侃, 等. 影响梅山猪繁殖性能的生理结构及遗传因素研究进展[J]. 畜牧兽医学报, 2012, 43(3): 329–335.
XIE X X, WANG Q S, HE K, et al. The research progress in the physiological structure and hereditary factors influencing the reproduction performance of meishan pig[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(3): 329–335. (in Chinese) |
| [25] | FLAQUER A, BAUMBACH C, LADWIG K H, et al. Mitochondrial genetic variants identified to be associated with posttraumatic stress disorder[J]. Transl Psychiatry, 2015, 5(3): e524. DOI: 10.1038/tp.2015.18 |
| [26] | VAN BLERKOM J. Mitochondrial function in the human oocyte and embryo and their role in developmental competence[J]. Mitochondrion, 2011, 11(5): 797–813. DOI: 10.1016/j.mito.2010.09.012 |
| [27] | HARVEY A, GIBSON T, LONERGAN T, et al. Dynamic regulation of mitochondrial function in preimplantation embryos and embryonic stem cells[J]. Mitochondrion, 2011, 11(5): 829–838. DOI: 10.1016/j.mito.2010.12.013 |
| [28] | ZHOU X, ANDERSON K V. Development of head organizer of the mouse embryo depends on a high level of mitochondrial metabolism[J]. Dev Biol, 2010, 344(1): 185–195. DOI: 10.1016/j.ydbio.2010.04.031 |
| [29] | REYNOLDS L P, BOROWICZ P P, VONNAHME K A, et al. Animal models of placental angiogenesis[J]. Placenta, 2005, 26(10): 689–708. DOI: 10.1016/j.placenta.2004.11.010 |
| [30] | CENCIČ A, GUILLOMOT M, KOREN S, et al. Trophoblastic interferons:Do they modulate uterine cellular markers at the time of conceptus attachment in the pig?[J]. Placenta, 2003, 24(8-9): 862–869. DOI: 10.1016/S0143-4004(03)00135-8 |
| [31] | ZHOU Q Y, FANG M D, HUANG T H, et al. Detection of differentially expressed genes between Erhualian and Large White placentas on day 75 and 90 of gestation[J]. BMC Genomics, 2009, 10: 337. DOI: 10.1186/1471-2164-10-337 |



