2. 江苏省重点动物传染病和人畜共患病预防控制联合创新中心, 扬州 225009
2. Key Joint Center of Infectious Diseases and Zoonosis Prevention and Control of Jiangsu Province, Yangzhou 225009, China
1994年H9N2亚型AIV在我国广东地区首次分离到,随后在我国大部分地区家禽群体中广泛流行[1]。H9N2 AIV单独感染鸡引起轻微的呼吸道症状,可以排毒,很少引起死亡,当与其他病毒(如新城疫病毒、传染性支气管炎病毒等)发生混合感染,会导致呼吸道症状加重,死亡率上升[2]。水禽通常是自然携带,不发病,可以自然携带多种亚型禽流感病毒,成为AIV的储存宿主。在中国,活禽市场是家禽零售的重要场所,不同来源的家禽聚集在一起,再以批发或者零售的方式销售到城市的各个角落。因此,活禽市场成为不同来源家禽共同存在的场所,有研究表明病毒可以在活禽市场持续存在,不断感染进入市场的家禽,同时极有可能发生基因重组,形成新的流行株[3-4],特别是H9N2、H6N6之类的低致病性AIV。在中国流行的H9N2 AIV主要分为G1-like和Y280-like,其八个基因片段的组成也不断发生变化,目前流行的优势基因型是S基因型或G57基因型[5]。近年来,有文献报道S基因型H9N2亚型AIV可以为H7N9[6-8]、H5N2[9-11]、H10N8[12]等亚型AIV提供内部基因,并可以跨种间传播感染人类,给公共卫生带来巨大威胁。因此,对活禽市场H9N2 AIV的流行病学监测,尤其是对鹅、鸭等水禽来源的H9N2 AIV的监测工作不容忽视[13]。
1 材料与方法 1.1 试验材料样本处理液为含有抗生素的pH7.0~7.4的甘油-磷酸盐缓冲液(phosphate buffer saline, PBS);H1、H3、H4、H5、H6、H7、H9、H10、H11亚型血清及新城疫病毒阳性血清、1%鸡红细胞均由扬州大学农业部畜禽传染病重点开放实验室制备并提供。无特定病原体(specific pathogen free, SPF)鸡胚购自北京梅里亚维通实验动物技术有限公司。RNA提取试剂盒购自北京全式金生物技术有限公司。PCR引物及测序由金斯瑞生物科技有限公司合成和完成。
1.2 试验方法 1.2.1 样品采集与病毒分离2016年10月至2017年9月期间,定期每月于华东地区某活禽市场采集活禽的喉头棉拭子和泄殖腔棉拭子样品(合成1管,记作1份),将采集的样品尿囊腔接种10日龄SPF鸡胚,扩增病毒。对于鸡胚尿囊液无效价的样品,要用鸡胚盲传三次。通过血凝-血凝抑制试验(HA-HI)分离、筛选出H9N2 AIV阳性样品。
1.2.2 病毒RNA提取及RT-PCR根据北京全式金生物技术有限公司生产的RNA提取试剂盒及提供的说明书提取分离病毒RNA,并进行反转录获得cDNA。根据各个片段参考序列设计引物(详见表 1),并根据引物Tm值设计PCR程序。经1%琼脂凝胶电泳检测,切取阳性条带胶块,进行胶回收,将胶回收产物送南京金斯瑞生物科技有限公司测序。
|
|
表 1 RT-PCR扩增H9N2禽流感病毒各个基因片段的引物序列 Table 1 RT-PCR amplification primers of each gene of H9N2 avian influenza virus |
应用DNASTAR、DNAMAN等软件编辑、分析序列,从NCBI和GISAID数据库下载H9N2 AIV标准参考株[14](序号1~7)和近年来本实验室分离株(序号8~14)(详见表 2),运用MEG6.0构建各个基因片段的系统发生树。
|
|
表 2 H9N2 AIV参考毒株背景资料 Table 2 Background of H9N2 AIV strains used for analysis |
2016年10月至2017年9月,共计采样12次,采集127个禽群、1 178份样品,分离毒株由多至少依次为H9、H5、H7、H3、NDV、H6、H4、H11、H1、H10,H9亚型AIV分离率最高,多达100个分离株(详见图 1);H9N2亚型AIV在不同宿主中的分离率差别较大,鸡源H9分离株93株,鹅源H9分离株5株,鸭源H9分离株2株;本研究对7株分离自水禽的H9N2亚型AIV展开研究,经RT-PCR检测,确定7个分离株均为H9N2病毒(病毒背景信息详见表 3)。

|
图 1 不同月份采样量及H9亚型AIV分离情况 Figure 1 Sampling amount at different month and isolation of H9 subtype AIV |
|
|
表 3 活禽交易市场7株水禽源H9N2 AIV分离株的背景资料 Table 3 Background of seven H9N2 AIV isolated from waterfowl in LPM |
对7个H9N2病毒分离株各基因核苷酸序列进行比对,并利用邻接法(Neighbor-Joining method)绘制各个基因的系统发生树,结果显示不同基因片段遗传发生存在不同的进化趋势。
2.2.1 血凝素(hemagglutinin,HA)测序表明7株H9N2 AIV的HA基因编码区均为1 683 bp,为完整的开放阅读框(open reading frame,ORF),无核苷酸的插入。HA基因裂解位点(即由基因推导出的氨基酸序列第335—338位)均为RSSR,是典型的低致病性禽流感病毒特征性序列[14]。HA基因核苷酸相似性为98.2%~100.0%,推导出的氨基酸相似性为98.0%~100.0%。系统发生树(图 2)显示,分离株与参考株Y280/97处于同一个大分支,均属于Y280-Like,核苷酸相似性为95.0%~96.0%,氨基酸相似性为91.0%~91.7%。比较近几年的分离株的HA基因,发现本研究的7个分离株进化趋势具有较高的一致性,均属于Group A,与SQ1602(2016)遗传距离较近,核苷酸酸相似性为97.8%~98.6%,氨基酸相似性为97.3%~98.0%。

|
▲.H9N2 AIV分离株;●.参考株 ▲. H9N2 AIV isolate; ●. Reference virus 图 2 7株分离株的HA、NA基因与参考序列构建的遗传进化树 Figure 2 Phylogenetic relationship of HA and NA gene of seven isolates with reference sequence |
测序表明7株H9N2 AIV的NA基因编码区均为1 401 bp,为完整的ORF,无核苷酸插入。NA基因相似性为96.2%~100.0%,推导出的氨基酸相似性为94.7%~100.0%。由系统发生树(图 2)可见,分离株与参考株BJ/94虽然不在同一个分支,但是遗传距离最近,故可以划归为BJ94-Like,与BJ/94的核苷酸相似性为93.4%~94.6%、氨基酸相似性为90.3%~91.0%,分离株核苷酸和氨基酸均发生了较大程度的变异。比较近几年的分离株NA基因,该基因有分化趋势,而本研究的7个分离株进化趋势具有一定的一致性。7个分离株与往年的分离株分属于BJ94-Like的两个Group,7个分离株均属于Group A,除JS21之外的其他6个分离株与SQ1602(2016)遗传距离较近,核苷酸相似性为97.1% ~98.3%,氨基酸相似性为96.9%~97.6%;JS21与AH329(2016)遗传距离较近,核苷酸相似性为99.8%,氨基酸相似性为99.6%。
2.2.3 碱性聚合酶2 (polymerase basic protein 2, PB2)测序表明7株H9N2 AIV的PB2基因编码区均为2 280 bp,为完整的ORF。PB2基因核苷酸相似性为99.1%~100.0%,推导出的氨基酸相似性为99.1%~99.9%,进化趋势具有较高的一致性。由系统发生树(图 3A)可见, 分离株与参考株G1/97处于同一个大分支,均属于G1-Like,核苷酸相似性为95.6%~96.3%,氨基酸相似性为95.4%~ 96.1%。比较近几年分离株的PB2基因,本研究的7个分离株进化趋势具有较高的一致性。分离株PB2基因与属于G1-Like其他参考株属于同一分支,但是明显不属于同一个Group,大致上可分为2个Group。分离株遗传距离与GD1601(2016)较近,均属于Group A,核苷酸及氨基酸相似性为98.4%~98.8%、98.1%~98.8%。
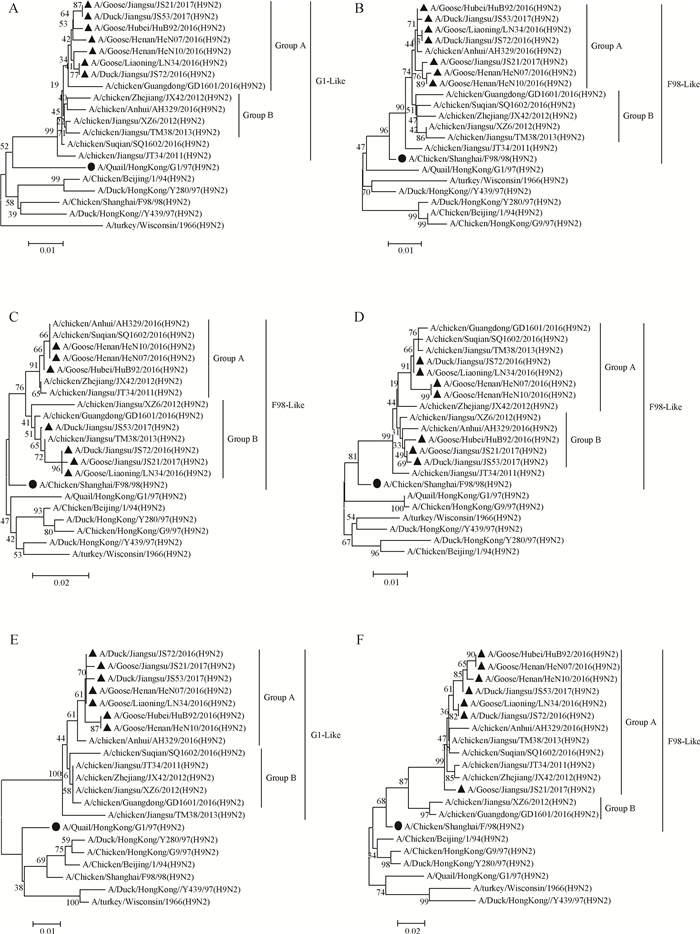
|
A. PB2基因;B. PB1基因; C. PA基因;D. NP基因; E. M基因;F. NS基因;▲.H9N2 AIV分离株;●.参考株 A.PB2 gene; B.PB1 gene; C.PA gene; D.NP gene; E.M gene; F.NS gene; ▲.H9N2 AIV isolate; ●.Reference virus 图 3 7株分离株的PB2、PB1、PA、NP、M、NS基因与参考序列构建的遗传进化树 Figure 3 Phylogenetic relationship of PB2, PB1, PA, NP, M, NS gene of seven isolates with reference sequence |
测序表明7株H9N2 AIV的PB1基因编码区均为2 274 bp,为完整的ORF。PB1基因核苷酸相似性为98.0%~100.0%,推导出的氨基酸相似性为97.7~100.0%。由系统发生树(图 3B)可见,分离株与参考株F98/98处于同一个大分支,均属于F98-Like,核苷酸相似性为97.3%~98.0%,氨基酸相似性为97.6%~98.4%。比较近几年分离株的PB1基因,可大致上分为两个Group,本研究的7个分离株进化趋势具有较高的一致性,与AH329(2016)均属于Group A,核苷酸及氨基酸相似性为98.4%~99.3%、97.7%~99.2%。
2.2.5 酸性聚合酶蛋白(polymerase acidic protein, PA)测序表明7株H9N2 AIV的PA基因编码区均为2 151 bp,为完整的ORF。PA基因核苷酸相似性为99.3%~100.0%,推导出的氨基酸相似性为99.4%~100.0%,进化趋势具有较高的一致性。由系统发生树(图 3C)可见,分离株与参考株F98/98处于同一个分支,均属于F98-Like,核苷酸相似性为98.6%~99.0%,氨基酸相似性为98.6%~99.0%。比较近几年分离株的PA基因,可大致上分为两个Group,有两个分化趋势,分离株HuB92、HeN07和HeN10位于Group A,与AH329(2016)遗传距离较近,核苷酸及氨基酸相似性为99.3%~99.9%、99.0%~99.4%;分离株LN34、JS72、JS21和JS53位于Group B,与TM38(2013)遗传距离较近,核苷酸及氨基酸相似性为98.4%~98.7%、99.0%~99.2%。
2.2.6 核蛋白(NP)测序表明7株H9N2 AIV的NP基因编码区均为1 497 bp,无核苷酸插入,为完整的ORF。NP基因核苷酸相似性为97.8%~100.0%,氨基酸相似性为98.2%~100.0%,进化趋势具有较高的一致性。由系统发生树(图 3D)可见,分离株与参考株F98/98遗传距离最近,归为F98-Like,核苷酸相似性为96.9%~98.2%,氨基酸相似性为98.0%~98.6%。比较近几年分离株的NP基因,可大致上分为两个Group,有两个分化趋势,分离株LN34、JS72、HeN07和HeN10位于Group A,与SQ1602(2016)遗传距离较近,核苷酸及氨基酸相似性为98.0%~100.0%、98.6%~99.8%;分离株HuB92、JS21和JS53位于Group B,与AH329(2016)遗传距离较近,核苷酸及氨基酸相似性为98.0%~99.8%、98.8%~99.0%。
2.2.7 基质蛋白(matrix protein, M)测序表明7株H9N2 AIV的M基因编码区均为982 bp,可分为两个ORF。M基因核苷酸相似性为99.1%~ 100.0%,氨基酸相似性为99.1%~100.0%,进化趋势具有较高的一致性。由系统发生树(图 3E)可见,分离株属于同一分支,与参考株G1/97遗传距离最近,均归为G1-Like,核苷酸相似性为94.6%~95.2%,氨基酸相似性为92.9%~93.6%,同源性较低。比较近几年分离株的M基因,可大致上分为两个Group,本研究的7个分离株与AH329(2016)均属于Group A,遗传距离较近,核苷酸及氨基酸相似性为98.8%~99.4%、98.4%~99.1%。
2.2.8 非结构蛋白(Nonstructural protein, NS)测序表明7株H9N2 AIV的NS基因编码区均为838 bp,均为完整的ORF,可编码2种蛋白。NS基因核苷酸相似性为96.7%~100.0%,氨基酸相似性为96.3%~100.0%,进化趋势具有较高的一致性。由系统发生树(图 3F)可见,分离株NS基因与参考株F98/98同属于一个大的分支,归为F98-Like,大致上可分为2个Group,核苷酸相似性为94.0%~94.4%,氨基酸相似性为91.2%~93.9%,同源性较低。比较近几年分离株的NS基因,可大致上分为两个Group,本研究的7个分离株均属于Group A。属于Group A分离株JS21并未与其他6个分离株聚类在一起。分离株与TM38(2013)同源性较高,核苷酸及氨基酸相似性为97.8%~99.3%、97.4%~99.3%。
3 讨论H9N2亚型禽流感作为一种低致病性禽流感,在我国流行范围较广,四季均可发生,无明显的季节性,尤以秋冬季节病毒分离率较高(详见图 1),这可能与AIV喜寒恶热的生物学特性有关。由H9N2 AIV分离数据分析,一方面,该亚型的分离率是各亚型中最高的;另一方面,该亚型在水禽(主要是鸭、鹅)中的分离率要明显低于陆生禽(主要是鸡)。有报道称,鸡是H9N2 AIV的易感宿主,鸭、鹅等水禽是该病毒的天然储存宿主[1],所以对水禽来源的H9N2 AIV进行持续的流行病学调查,对于防控正在流行的H9N2 AIV优势株或即将流行的H9N2 AIV潜在优势株是有一定意义的。
对2016年10月至2017年9月分离到的5个鹅源H9N2 AIV分离株、2个鸭源H9N2 AIV分离株各个基因片段序列进行编辑处理后,结合F98等H9亚型经典参考株及本实验室近年来上传于GISAID数据库中的H9亚型毒株序列(详见表 2)绘制出各基因片段系统发生树,结果表明7个分离株均属于当今流行的H9N2 AIV基因型——S基因型[5]。总体上,对系统发生树进行分析,分离株各个基因片段遗传进化均存在一定的差异,其中4个基因(HA、PB2、PB1和M)遗传进化较为一致。分离株的HA基因遗传进化趋势一致,与SQ1602(2016)同源性较高,分离株中除了JS21的NA基因与AH329(2016)同源性较高外,均与SQ1602(2016)同源性较高,分离株的HA基因和NA基因可能有着类似的基因来源和遗传进化趋势;PB1基因和M基因遗传进化趋势一致,均与AH329(2016)同源性较高,PB1基因和M基因可能有着类似的基因来源和遗传进化趋势;PB2基因遗传进化趋势一致,与GD1601(2016)同源性较高;NS基因同处于Group A,与TM38(2013)同源性较高,但分离株JS21与其他六株病毒遗传距离较远;PA基因和NP基因均处于不同的Group,可能有着不同的基因来源和遗传进化趋势。综合分析结果,分离株LN34和JS72均在2016年12月份分离,宿主来自不同地区,在活禽市场也是分布在不同的禽群,但是两个毒株的基因片段同源性极高,进化趋势几乎一致,病毒极有可能是持续存在于活禽市场的病毒,造成进入市场交易的家禽感染;分离株HeN07和HeN10都是在2016年11月的同一个鹅群分离到,但是其NS基因在发生树上(图 3F)并不聚类在一起,说明病毒还可能存在重组的现象;分离株JS21的NA基因和NS基因均不与其他六个分离株聚类在一起,提示可能存在变异现象。
2013年,世界卫生组织称:禽流感的监测重点在活禽市场,活禽市场这一AIV污染地的存在,加速了AIV发生基因重组和变异的进程,为新型AIV的出现提供了条件。但是,取缔活禽市场交易,短期内不可能完成,因此应该更加重视对活禽市场的流行病学监测,及时反馈当前AIV的流行情况,为AIV的防控提供参考支持是可行的。我们的研究结果也反映出由于没有采取生物安全的措施,病毒易在活禽市场持续存在且易发生重组,交易的家禽极易发生交叉感染。因此,活禽市场的家禽、特别是水禽在AIV的演化、维持和扩散过程中起重要作用,对活禽市场进行长期不间断的流行病学监测是非常重要的。
4 结论华东地区活禽市场水禽体内携带的7株H9N2 AIV与当前家禽优势流行基因型一致,定期监测活禽市场家禽中循环存在的AIV,对了解潜在的流行株的演化和出现有重要作用。
| [1] | WEBSTER R G, BEAN W J, GORMAN O T, et al. Evolution and ecology of influenza A viruses[J]. Microbiol Rev, 1992, 56(1): 152–179. |
| [2] |
谷长勤, 罗玲, 胡薛英, 等. H9N2亚型禽流感病毒诱导蛋鸡免疫器官细胞凋亡的动态变化[J]. 中国兽医学报, 2008, 28(1): 12–14.
GU C Q, LUO L, HU X Y, et al. Apoptosis of the immune organ in layers inoculated experimentally with H9N2 subtype avian influenza virus[J]. Chinese Journal of Veterinary Science, 2008, 28(1): 12–14. (in Chinese) |
| [3] |
张烨, 李晓丹, 邹淑梅, 等. 2009~2013年我国活禽市场环境样本中禽流感病毒的检测[J]. 病毒学报, 2015, 31(6): 615–619.
ZHANG Y, LI X D, ZOU S M, et al. Detection of avian influenza virus in environmental samples collected from live poultry markets in China during 2009-2013[J]. Chinese Journal of Virology, 2015, 31(6): 615–619. (in Chinese) |
| [4] |
段晓冬, 沈丹, 张海明, 等. 2013年广州市活禽农贸市场禽流感流行病学调查[J]. 中国动物检疫, 2014, 31(11): 33–36, 43.
DUAN X D, SHEN D, ZHANG H M, et al. Epidemiological investigation of avian influenza in retail live poultry markets in Guangzhou in 2013[J]. China Animal Health Inspection, 2014, 31(11): 33–36, 43. DOI: 10.3969/j.issn.1005-944X.2014.11.010 (in Chinese) |
| [5] |
顾敏, 彭大新, 刘秀梵. 我国H9N2亚型禽流感病毒的流行和进化特点[J]. 生命科学, 2015, 27(5): 531–538.
GU M, PENG D X, LIU X F. Features of the evolution and epidemiology of H9N2 subtype avian influenza virus in China[J]. Chinese Bulletin of Life Sciences, 2015, 27(5): 531–538. (in Chinese) |
| [6] | KAGEYAMA T, FUJISAKI S, TAKASHITA E, et al. Genetic analysis of novel avian A(H7N9) influenza viruses isolated from patients in China, February to April 2013[J]. Euro Surveill, 2013, 18(15): 20453. |
| [7] | LAM TT Y, WANG J, SHEN Y Y, et al. The genesis and source of the H7N9 influenza viruses causing human infections in China[J]. Nature, 2013, 502(7470): 241–244. DOI: 10.1038/nature12515 |
| [8] | LEE R T C, GUNALAN V, VAN T D, et al. A new piece in the puzzle of the novel avian-origin influenza A (H7N9) virus[J]. Biol Direct, 2013, 8: 26. DOI: 10.1186/1745-6150-8-26 |
| [9] | ZHAO G, GU X B, LU X L, et al. Novel reassortant highly pathogenic H5N2 avian influenza viruses in poultry in China[J]. PLoS One, 2012, 7(9): e46183. DOI: 10.1371/journal.pone.0046183 |
| [10] | GU M, HUANG J Q, CHEN Y X, et al. Genome sequence of a natural reassortant H5N2 avian influenza virus from domestic mallard ducks in eastern China[J]. J Virol, 2012, 86(22): 12463–12464. DOI: 10.1128/JVI.02315-12 |
| [11] | MI Z Q, LIU W, FAN H, et al. Complete genome sequence of avian influenza virus A/chicken/Jiangsu/1001/2013(H5N2), demonstrating continuous reassortance of H5N2 in China[J]. Genome Announc, 2013, 1(4): e00469–13. |
| [12] | CHEN H Y, YUAN H, GAO R B, et al. Clinical and epidemiological characteristics of a fatal case of avian influenza A H10N8 virus infection:A descriptive study[J]. Lancet, 2014, 383(9918): 714–721. DOI: 10.1016/S0140-6736(14)60111-2 |
| [13] | JIN Y, YU D, REN H G, et al. Phylogeography of Avian influenza A H9N2 in China[J]. BMC Genomics, 2014, 15: 1110. DOI: 10.1186/1471-2164-15-1110 |
| [14] |
古小兵. 华东地区2011-2012禽流感流行病学调查及近三年H9N2禽流感病毒遗传进化分析[D]. 扬州: 扬州大学, 2013.
GU X B. The epidemiology survey of avian influenza virus in eastern China during 2011 to 2012 and the genetic evolution analysis of H9N2 subtype avian influenza virus in last three years[D]. Yangzhou: Yangzhou University, 2013. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=y2418955 |



