2. 中国农业大学动物科技学院, 北京 100193
2. College of Animal Science and Technology, China Agricultural University, Beijing 100193, China
一直以来疾病给养殖业造成严重的经济损失,特别是一些大规模传染性疾病,如猪蓝耳病、奶牛乳房炎、羔羊大肠杆菌腹泻、马流感等。目前,控制各类病原菌的主要方法是抗生素治疗,但长期使用导致病原菌耐药性问题日趋严重。另外,病原的高度变异会造成疫苗免疫失败,使得家畜免疫力下降。目前许多家畜传染病的分子机制仍未被揭示,国内外学者希望通过遗传改良降低重大疫病发生的可能性,但是抗病性状属于复杂性状,遗传力较低,如奶牛体细胞评分(Somatic cell score,SCS)在不同胎次中的遗传力不同,第1、2、3胎的遗传力(h2)分别为0.092、0.151、0.181[1]。猪对呼吸道疾病易感性的h2为0.14,对伪狂犬病毒疫苗免疫应答h2为0.05[2],对干扰素α的h2为0.08[3],均属低遗传力性状[4],抗病育种进展缓慢。利用基因表达芯片(Microarray)和二代测序技术(Next generation sequencing,NGS)研究人和家畜某些特异疾病的发病机制和宿主的细胞反应发现,病原刺激会导致宿主某些基因表达量发生变化[5-7]。但是基因的表达受控于复杂的基因网络和环境因素,包括病原致病力、饲养条件、应激等,这给分子标记辅助选择带来极大的干扰和难度。因此,仅从遗传方面研究复杂的抗病机制受到一定限制。从近年来的研究数据发现,通过研究“病原-机体-环境”的互作关系,有望深入解析家畜抗病/易感相关基因的表达调控机制。
表观遗传为病原微生物与机体细胞间关系的研究架起了重要桥梁,它是指DNA序列未发生变化,而基因表达发生可遗传的变化,进而导致表型的变异[8]。DNA甲基化是最早发现且最为稳定的表观遗传修饰之一,通常基因启动子区的DNA甲基化修饰会抑制基因的表达[8]。白细胞分化抗原4(CD4) 分子是一种相对分子质量为55 000的单链跨膜糖蛋白,主要在胸腺和成熟的T细胞表面表达,在宿主抵抗病毒、细菌等病原体感染中发挥重要作用[9-11]。目前报道,在三大家畜(猪、牛、羊)上CD4基因多态性均与疾病的易感相关[10, 12-14],而CD4基因DNA甲基化的调控机制研究较少。在家禽肿瘤等研究报道中,J. Luo等[15]研究揭示,鸡脾被马立克氏病毒(MDV)感染后,CD4基因表达量上调,并发现CD4启动子的DNA甲基化水平高低与鸡马利克氏疾病的易感性密切相关。本文主要就CD4基因DNA甲基化修饰对免疫反应的调控机制及其在家畜抗病育种中的应用进行综述和展望。
1 CD4+T细胞在免疫系统中的作用及CD4的生物学特性动物血液与机体的免疫功能密切相关,血液中的一些常规生化指标是反映机体健康状况和抗病能力的重要参数。细胞免疫是动物免疫系统的重要组成部分,在清除病原菌入侵中发挥重大作用,而成熟的T细胞在细胞免疫中居中心地位。血液中成熟的T淋巴细胞根据其表面标志和功能可分为CD4+辅助T细胞和CD8+细胞毒性T细胞。CD4+辅助T细胞的主要生物功能是通过分泌的细胞因子,扩大和增强机体免疫应答过程,激活巨噬细胞,进而促进体液免疫。CD4+T细胞的功能主要是“辅助”CD8+T细胞发挥免疫作用。但是CD4+T细胞不是功能单一的一类细胞,而是功能复杂的细胞群,其杀伤和清除病原的作用不应该被忽视[16]。CD4前体细胞(Th0) 在抗原递呈细胞作用下分化为Th1和Th2细胞,在正常生理情况下,Th1和Th2细胞保持动态平衡。一旦这种平衡被打破,由于各类细胞分泌的特征性细胞因子的作用不同,机体则表现出异常的免疫反应。如Th1细胞占优势则主要发挥促炎作用,产生TNFα和IL2等细胞因子;Th2细胞占优势则主要发挥抗炎作用,诱导产生IL4、IL5和IL13等特征性细胞因子[17]。特别是在炎症反应中,当机体受到异己抗原攻击时,Th1和Th2细胞中某一亚群功能升高,另一亚群功能降低,该现象为Th1/Th2漂移,即表现出促炎和抗炎反应的不平衡。
CD4与MHC(主要组织相容性复合体)Ⅱ类分子多肽区结合,主要表达于CD4+辅助T细胞表面。试验证实,CD4基因敲除的动物,无法产生成熟的CD4+T细胞。而成熟的CD4+T细胞同时表达TCR和CD3分子,能够识别抗原、介导免疫应答并参与免疫调节。因此,CD4能够指导CD4+辅助T细胞的正常分化与成熟,维持CD4+T细胞正常的免疫功能。哺乳动物CD4蛋白由单一基因组的CD4基因编码。目前已克隆鉴定了人、猴、鼠、兔、牛、猫、狗等动物的CD4基因序列。牛CD4基因定位于5号染色体,该基因全长16 kb,包含7个外显子,6个内含子,编码395个氨基酸[18]。2012年,有研究人员利用生物信息学手段预测并成功克隆猪CD4基因的启动子与增强子[19]。通过将猪CD4基因的启动子和增强子与人和鼠CD4基因相比较,证明猪CD4的启动子和增强子在人源细胞中调控基因表达的效率明显高于猪和鼠。山羊CD4基因于2012年被首次克隆,所克隆的基因全长cDNA序列为1 555 bp,开放阅读框为1 368 bp,编码455个氨基酸[20]。氨基酸序列比对分析表明,山羊与绵羊CD4的氨基酸序列相似性为98%,与小鼠、人和猪CD4的氨基酸序列相似性分别为66%、74%和81%。另外,CD4在山羊淋巴中的表达量最高,在睾丸中表达量较低,表明山羊CD4蛋白是一种免疫分子[20]。
2 CD4+T细胞与家畜抗病性抗病性(Disease resistance)是指宿主忍耐病原感染,缩短病原在体内的生活周期,抵御疾病影响的能力。这种抗病力受环境的影响,但可以稳定地遗传给后代。在不同的属、品种、个体之间存在这种抗病力的遗传变异,从而构成了对疾病抗性选择的遗传基础[21]。与之对应的便是易感性(Susceptibility),是由遗传因素与环境因素综合作用形成的患病风险程度。而由遗传因素决定的易于患某种或某类疾病的倾向性称为遗传易感性。研究发现,许多疾病都存在遗传易感性,由细菌和病毒引起的某些传染病也存在遗传易感性[22]。
近些年研究证明,CD4+T细胞在宿主抗病性中发挥关键作用[18]。由葡萄球菌引起的奶牛乳房炎主要表现为CD4+T细胞数量增加,而CD4+/CD8+T细胞的比值在感染金黄色葡萄球菌的牛群中远低于未感染的牛群[23]。感染链球菌乳房炎的奶牛CD4+T和CD8+T细胞数同步增长。当繁殖与呼吸综合征病毒感染猪外周血后,CD4+T细胞数量在不同的感染时期存在动态变化[24]。猪CD4+T细胞数量会随着年龄的增长而增长,20和35日龄健康大白猪体内CD4+/CD8+T细胞比值均低于1[10]。刘国民[25]对无角多赛特羊与本土苏尼特羊、乌珠穆沁羊之间适应性和抗病性的差异做了比较分析,采用流式细胞术测定了3个品种羊的外周血液T淋巴细胞CD4+、CD8+表达率,无角多赛特羊CD4+、CD8+及CD4+/CD8+低于苏尼特羊和乌珠穆沁羊,表明引入品种和地方品种的抗病力有所差异。在马的复发性葡萄膜炎中,CD4细胞膜蛋白组丰度的显著降低与马自发免疫性疾病显著相关[26]。当流感病毒感染人、猪、马、禽、鼠等动物,可引发T淋巴细胞介导的适应性免疫反应,包括CD4+辅助T淋巴细胞和CD8+毒性T淋巴细胞,从而发挥抗病作用[27]。近几年研究发现,受表观遗传调控的CD4基因转录表达水平与T细胞的发育直接相关[28]。“遗传-表观遗传假说”认为,环境因素(如病原菌、毒素、激素等)对人和动物的致病主要是通过DNA甲基化和组蛋白修饰等表观基因组起作用[29]。因此,CD4基因的表观遗传修饰将受到遗传学家们的更多关注。
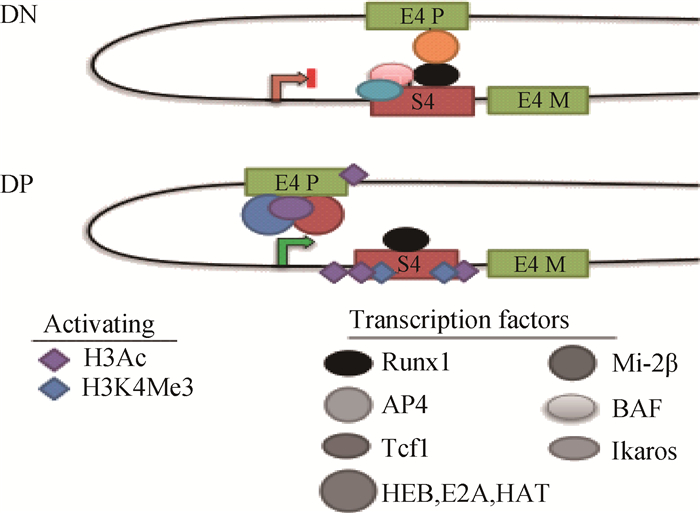
3 CD4基因表观遗传修饰对免疫过程中T细胞分化的作用胸腺细胞在胸腺中经历了阴性和阳性选择,最终成为免疫功能成熟的T细胞,进入血流后称为外周T淋巴细胞,随血液循环进入脾或淋巴结,行使免疫功能。CD4基因第一外显子的甲基化座位发生特异性DNA去甲基化而促进CD4表达,这一位点的持续去甲基化使双阴性(Double-negative,DN)细胞发育为双阳性(Double-postive,DP)细胞,继而发育为CD4单阳性(CD4 single-positive,CD4SP)T细胞[30]。在成熟T细胞(如αβT细胞)的发育中,CD4的动态调控是由多个顺式作用元件(表 1)结合转录因子(表 1)完成的,可精确调节基因的转录起始与转录效率。
两个正向元件E4 P、E4 M和一个沉默元件S4可共同调控成熟T细胞表达。T细胞系的E4 P元件为339 bp DNA片段,具有指导报告基因CD4活化的作用[32-33]。在双阴性(DN)细胞向双阳性(DP)细胞转化过程中,E4 P缺失时,可维持CD4表达,H3Ac和H3K4Me3并未改变,证明E4 P在缺失后,成熟的增强子E4 M发挥作用,从而维持CD4座位的表观遗传状态。在成熟的CD4+T细胞分化中,E4 P缺失后序列分析验证Tcf1、E2A、HEB等转录因子可与E4 P结合[34]。ChIP分析证明, 这些转录因子与E4 P结合可直接促进双阳性细胞CD4的表达[35-36]。CD4 S4是一个434 bp的中心调控元件。在双阴性细胞中,CD4的表达受到S4的抑制,S4又可与E4 P增强子互作,潜在阻止CD4启动子活化,互作可由与S4结合的Runx1介导。核小体重塑复合物BAF可与Runx1转录因子结合调控对S4的可接近性[37],促进转录沉默(图 1,DN)。另外,转录因子Ikaros和BAF及其他染色质重塑复合物相关,对双阴性细胞的CD4抑制必不可少。缺失S4可使HAT p300招募于E4 P座位上,增加双阴性细胞CD4基因启动子H3K9Ac程度。核小体重塑和去乙酰化复合物的ATPase亚单位(mi-2β)是一个CD4表达的正向调控子,可能通过将组蛋白乙酰化转移酶(HATs)招募到E4 P或S4元件发挥作用(图 1,DP)。尽管S4位点发生H3Ac和H3K4me3,但是CD4 DNA甲基化的水平仍然很高[31]。
|
图 1 T细胞发育中CD4基因表达的调控 Figure 1 CD4 regulation during T cell development |
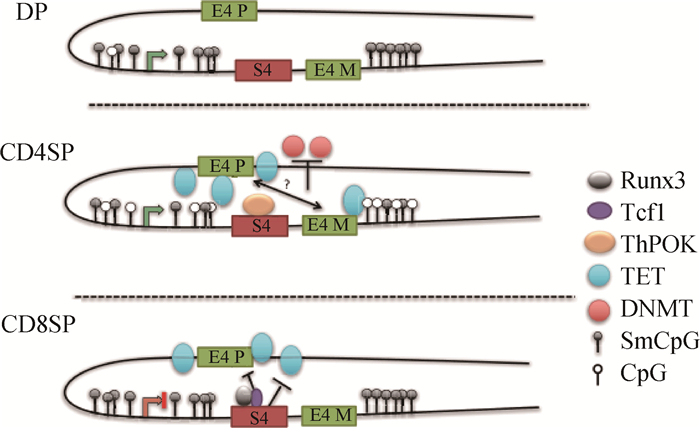
在CD4+T细胞分化中,DNA去甲基化对维持CD4表达非常重要[38]。因为T细胞从双阴性转化为双阳性时,细胞分化降低,CD4单阳性T细胞在复制过程中甲基化程度降低,揭示了细胞的主动去甲基化机制而不是缺失甲基化维持机制。在CD4+CD8+双阳性细胞分化为CD4单阳性T细胞时,利用氧化重亚硫酸盐扩增测序分析了CD4座位5hmC的甲基化,此座位5hmC甲基化水平与基因活化相关,主要富集于基因组区域[39]。双阳性细胞的CD4座位通常为高甲基化状态,当细胞经过MHC-Ⅱ限制性选择转变为CD4单阳性细胞后,E4 P招募TET蛋白经历主动去甲基化,ThPOK(T helper inducing POZ/Krueppel-like factor)转录因子结合于S4元件上发生阳性选择(图 2,DP→CD4SP)。TET(Ten eleven translocation)蛋白是DNA去甲基化过程中的一种重要的酶,可以催化5-甲基胞嘧啶(5-mC)转化为5-羟甲基胞嘧啶(5-hmC),TET对于维持干细胞的多能性有重要作用。在CD4单阳性细胞中,缺失E4 P后,CD4座位仍维持高甲基化状态,更相似于CD8单阳性细胞CD4甲基化模式(图 2,CD8SP)。因此,在成熟的CD4+T细胞中,CD4座位E4 P的低甲基化状态对细胞发育分化是必不可少的。在CD8单阳性细胞中,S4可维持CD4座位高甲基化水平。此外,MHC Ⅱ限制性选择的细胞可重新指导CD8+T细胞分化,由于缺少ThPOK,CD4座位发生高甲基化(图 2,CD8SP),揭示ThPOK对DNA去甲基化是必要的。这些结果证明,顺式作用元件的DNA甲基化机制对CD4表达至关重要。为阐明转录因子如何结合这些顺式作用元件,可以设计Dnmts和TET酶试验来证明CD4和CD8单阳性细胞的阳性选择。
|
图 2 细胞分化过程中CD4座位的DNA甲基化模式[31] Figure 2 Model of methylation dynamics at the CD4 locus during cell differentiation[31] |
DNA甲基化现象不仅能独立遗传,而且在基因表达调控、基因组印记、X染色体失活、转座子抑制、机体功能障碍以及复杂疾病的发生等方面有着重要的调控作用[40-41]。利用全基因组DNA甲基化谱筛选的差异甲基化区域(DMR)是指在不同组织类型或疾病状态下的细胞水平上发生的差异甲基化区域。S.Ecker等通过对CD4+T淋巴细胞和CD8+T淋巴细胞等34个样本近190万个CG位点进行分析,得到了人类第6、20和22号染色体的DNA甲基化图谱[42]。通过分析试验组与对照组的DMRs,结合表观基因组注释信息以寻找新的抗性基因和重要的DNA甲基化标记(DNA methylation markers),有助于了解正常和疾病状态下不同基因相互作用的网络调控关系[43]。这方面研究不仅可为病原感染、肿瘤、糖尿病等复杂疾病的早期诊断和治疗提供新方法,而且还可为家畜抗病育种提供理论基础。
CD4基因DMRs的高甲基化水平主要被鉴定于第一外显子区域,分别延伸至转录起始位点上游和下游-0.7~+3.2 kb区域,这一区域的DNA甲基化程度与CD4表达紧密相关。尽管之前有研究证明DNA甲基化不参与CD4的沉默,但这些结果是利用DNA甲基化转移酶抑制剂5-azacytidine所得到,更可能是由于5-azacytidine对细胞产生毒性而掩盖了DNA甲基化对CD4表达抑制的机理,而降低抑制剂的剂量使CD4表达抑制[37, 44]。将DNMT缺失的初始CD8+T细胞转化到淋巴细胞减少的小鼠体内,导致CD4被强烈激活而使细胞稳定增殖。在DNMT缺失的小鼠初始T细胞中CD4能够正常表达。虽然经典的重亚硫酸盐测序方法不能分辨5mC和5hmC甲基化,但是CD4座位的甲基化CpGs仍存在5hmCs,可促进CD4表达[45]。
4.3 DNA甲基化标记与家畜抗病育种近年来,随着测序和芯片技术的发展,病原感染后宿主表观基因组尤其是全基因组DNA甲基化谱的研究取得了较大进展[46]。L. Jin等[47]构建了青年猪和中年猪骨骼肌的全基因组DNA甲基化图谱,发现一些肿瘤相关基因DNA甲基化的表达模式与年龄有关,随着年龄的增长疾病的风险也随之增加。M. Y. Song等[48]构建了金黄色葡萄球菌感染泌乳奶牛外周血细胞后的DNA甲基化免疫共沉淀(Methylated DNA immunoprecipitation, MeDIP)图谱,揭示了奶牛免疫相关基因的表达受DNA甲基化不同模式的调控。流感病毒感染或dsRNA转染宿主细胞后,白介素IL6和IL32基因表达上调而DNMT活性降低,揭示DNA甲基化标记参与免疫基因表达[39, 49]。非洲猪瘟病毒可通过调控组蛋白H3甲基化标记调节异染色质化,抑制宿主基因表达,有利于猪瘟病毒的复制[50]。王海飞[51]构建了猪外周血单核细胞抗病毒反应的DNA甲基化图谱,分析PolyI:C刺激PBMCs 24 h后,DNA甲基化可通过抑制启动子转录活性而抑制PACSIN1基因的表达,基因低表达可促进促炎细胞因子的生成,增强免疫应答反应。可见,病毒等病原体感染的宿主免疫反应受DNA甲基化标记的调控。
5 CD4基因DNA甲基化标记与家畜抗病力关系的研究进展 5.1 猪CD4基因DNA甲基化标记与抗病力的关系猪是人类最早驯化的家畜之一,为人类提供了重要的动物蛋白质来源。因此,猪的健康状况与食品安全和人类健康息息相关。近些年,一些大规模传染性疾病,如猪瘟、猪繁殖与呼吸综合征、猪流感等对养猪业造成了不可估量的损失。有研究证实,中国地方猪种的抗病性能明显强于国外培育猪种[52-54],但这种强抗病性能经历了几千年的持续表型选择,遗传进展依然十分缓慢。
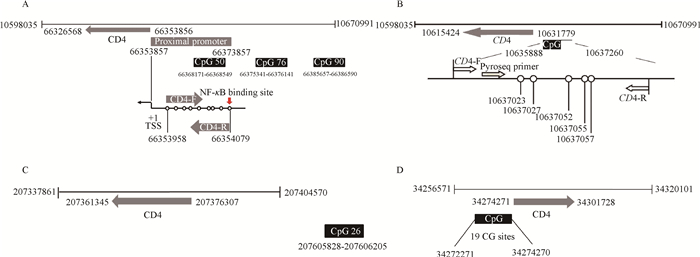
由于CD4基因与动物对疾病的感染密切相关,近年来,国内外针对猪CD4基因多态性与表达的相关性展开了一些研究。刘会杰等[9]揭示,不同品种猪CD4基因表达量存在显著差异;进一步分析发现,尽管CD4为同一基因型,但品种间同一CpG位点具有10%的DNA甲基化差异,初步证明CD4启动子DNA甲基化修饰与品种特异性相关。许金根[10]证明,CD4基因单倍型与T淋巴细胞亚群存在显著关联。但是针对分子抗病育种而言,分子标记辅助选择对于抗特异性疾病育种的准确度较高,对于一般抗病力的育种研究和应用存在较多困难。X. S. Wang等[55]利用MeDIP-chip和Microarray技术,建立dsRNA转染猪肾细胞系PK15的体外模型,从细胞水平筛选猪CD4基因启动子甲基化标记,分析CD4基因DMR主要位于启动子66 353 857~66 373 857 bp区域,通过NFκB结合位点(图 3A,向下箭头所示,转录起始位点上游-223~-102 bp)调控其表达,初步揭示了宿主CD4基因启动子通过NFκppa结合位点的DNA甲基化阻止转录因子结合,从而抑制CD4基因表达,揭示CD4可能参与宿主细胞抗病毒感染[56-57]。“CpG 50”和“CpG 76”距离转录起始位点(Transcriptional Start Site,TSS)上游15、23 kb位置(图 3A),可能位于proximal enhancer的E4 P和distal enhancer的E4 D内。这些座位的甲基化调控抗性机制值得我们继续深入探索,将为挖掘猪重要抗病性状功能基因、提高猪的分子抗病育种效率奠定分子基础,对于实现猪肉健康生产、保障食品安全具有十分重要的意义。
|
黑色框代表启动子区CpG岛 Black boxes represent CpG islands in promoter region 图 3 猪(A)、牛(B)、羊(C)、马(D)的CD4基因启动子区CpG岛结构图 Figure 3 CpG island in the promoter region of CD4 gene in swine (A), cattle (B), sheep (C), horse (D) |
奶牛乳房炎是造成奶牛业经济损失最严重的常见疾病之一[14, 58],引起奶牛乳房炎的主要病原菌包括金黄色葡萄球菌、链球菌和大肠杆菌。目前,主要通过乳汁中的体细胞数(SCC)来判定奶牛是否患有乳房炎,由于SCC遗传力较低,通过传统的降低SCC的遗传改良方法提高乳房炎的抗性效果不稳定[8],因此国内外学者们正努力探索通过表观遗传途径去降低奶牛乳房炎尤其是隐性乳房炎发生的可行性。
在急性炎症中,T淋巴细胞表达CD4分子而被活化。奶牛临床乳房炎CD4基因启动子CpG岛(图 3B)的DNA甲基化状态显著影响血液CD4基因的表达[18]。结合临床兽医诊断,在乳汁SCC较高的情况下,泌乳期感染牛的CD4基因启动子区呈现高甲基化状态,基因表达受到抑制,感染奶牛CD4+T淋巴细胞的比例显著降低。另外,不同胎次、不同场区对DNA甲基化水平无显著效应,但患病状态与DNA甲基化水平显著相关,这可能是乳房炎导致CD4基因DNA甲基化水平降低的重要原因之一[18]。金黄色葡萄球菌是引起奶牛乳房炎重要的病原菌之一,泌乳期奶牛乳腺被感染后,会引起乳汁SCC的急剧升高,经研究发现,这种致病菌会导致奶牛外周血白细胞基因组的组蛋白H3K27三甲基化修饰发生显著改变[59]。在乳房炎牛和健康牛的CD4基因上,H3K27三甲基化水平存在显著差异并影响其表达。这些研究表明,奶牛的乳房炎抗性受CD4基因启动子甲基化和H3K27甲基化调控。因此,奶牛CD4基因DNA甲基化标记(图 3B,CpG: 10 636 888~10 637 260) 可望作为重要的候选表观遗传标记研究其抗乳房炎的功能。
5.3 羊CD4基因DNA甲基化标记与抗病力的关系羊的毛用和肉用品质是养羊业一直以来关注的重要生产性能,面对世界养羊业的变革,养羊开始从毛用羊向肉用及毛肉兼用羊转变。在肉羊养殖生产中,引进羊对当地环境是否适应,常常是导致疾病发生和生产性能降低的主要原因[25]。因此,对抗逆性和抗病性进行探讨,有利于指导养羊实践生产。检测羊血液的CD4+T、CD8+T表达率可以监测细胞免疫水平,间接评价羊的抗病力。研究表明,日粮不同的营养水平对羊CD4+T和CD8+T细胞浓度有显著的影响,营养水平高低对免疫力的提高有显著作用[60]。营养、季节等外界环境因素均是影响羊抗病性状的主要因素。最新的研究揭示,羊外周血PBMCs CD4+T细胞亚群被活化,分泌IL17A细胞因子,证明了反刍动物T细胞发育中细胞因子的动态变化[61]。另外,蓝舌病毒感染羊的血液白细胞后,CD4+、CD8+细胞亚群大量降低,并且可维持3~10 d,反映病毒感染机体后的免疫抑制机制[62]。通过比较犬新孢子感染羊胎盘前、中、后3个时期的免疫反应,同时分析胎盘T淋巴细胞CD3、CD4、CD8抗原的分布和频率,得到CD4+细胞数显著降低,证实T淋巴细胞在羊疾病感染中的免疫作用[63]。
目前,尚无羊疾病抗性方面的CD4+T细胞和免疫相关基因CD4的DNA甲基化修饰的研究。羊CD4基因位于5号染色体,登录号NC_022297.1,转录起始位点(TSS)上游最近的“CpG岛26”位于TSS上游200 kb处(图 3C),这一远端甲基化岛对羊CD4调控可能并不发挥作用,但在TSS上游2 000 bp处存在30个CpG位点,这类密度相对较低的CpG shore可能是潜在发挥作用的重要CpG位点。这为研究表观遗传修饰包括DNA甲基化、组蛋白修饰对羊等小反刍动物疾病发生发展的调控作用奠定试验依据。今后有望通过寻找与疾病抗性相关的表观遗传标记及靶基因,选择抗病个体,结合育种手段培育出山羊和绵羊抗病新品系。
5.4 马CD4基因DNA甲基化标记与抗病力的关系中国马业正处在从传统马业向现代马业转型的过程中,急需培育出适应我国自然环境的马匹新品种(系)。赵一萍[64]对优良地方品种蒙古马免疫相关基因进行研究,揭示了蒙古马抗病力的遗传特性,研究对比了蒙古马和纯血马的血液生化、免疫、抗氧化等指标,判断在正常生理情况下蒙古马和纯血马的血液指标差异,从总体上掌握2个品种马抗病力的情况。J. A. Gim等[65]利用MeDIP-seq技术筛选马在运动前后的全基因组水平DNA甲基化差异区域,主要挖掘出马运动后富集的细胞分化与黏附通路,而反转座元件也呈现出高甲基化状态,揭示了马在运动后表观遗传性状的重塑。最近的研究发现,西尼罗病毒疫苗重组蛋白CD40L可以刺激马外周血产生CD8+T细胞而不产生CD4+T细胞,诱导健康马匹的体液免疫反应和细胞免疫反应[66]。马复发性葡萄膜炎(Equine recurrent uveitis,ERU)是一种眼睛的自发性免疫系统疾病,导致眼睛炎症反应,据报道,ERU马匹CD4+T细胞表面膜蛋白丰度显著高于健康马匹,为人类研究眼睛自发性免疫系统疾病提供模型[26]。S. A. Durward-Akhurst等[67]从患马肌纤维炎症(Immune-mediated myositis,IMM)的马匹肌肉中分离CD4+T细胞,结果证实95% IMM马匹肌肉中CD4+T细胞占优势,仅有28% IMM马匹肌肉中存在CD8+T细胞。对马血液中浆细胞样树突状细胞分析,鉴定出较少的CD4分子表达,但是能够高表达Runx2,其作用机制值得深入分析[68]。
至今尚无马CD4基因DNA甲基化修饰的研究,利用NCBI和UCSC数据库比对马CD4基因序列,CD4基因位于马6号染色体,基因登录号NC_009149.2,3条可变剪切mRNA序列(XM_001497051.4、XM_005610883.2、XM_014740654.1),每条可变剪切转录起始位点前2 000 bp处无CpG岛,但有少量CG位点,如XM_001497051.4序列TSS上游2 000 bp处存在19个CG位点(图 3D),这为马CD4分子的DNA甲基化调控提供关键信息。因此,今后期望从表观遗传学角度解析我国不同地区特色马种质资源的保护、利用和创新,将为指导地方品种马的杂交改良、品种、品系的培育提供理论基础。
6 总结与展望家畜疾病抗性表现的优劣程度,是决定家畜产品产量及质量的关键因素。CD4分子可维持正常的CD4+T细胞分化成熟,使其发挥重要的免疫功能。对家畜基因组上甲基化修饰的CD4表达量变化及调控的系统深入研究,有助于全面了解家畜复杂疾病抗性形成的分子基础。由于CD4基因的DNA甲基化修饰会直接影响CD4+T细胞功能或数量的改变,这就能一定程度地改变机体的免疫应答能力或抗病性能。利用CD4甲基化标记能够间接选择对某种特异病原具有抗性作用的家畜,这将为分子标记辅助选育奠定扎实的理论基础。CD4基因的甲基化修饰研究已报道与猪和奶牛的免疫应答以及多种疾病相关,但羊和马这方面的研究鲜见报道。因此,亟待研究者们加强CD4基因甲基化在牛和猪抗病育种方面的应用推广研究,以及增加其在羊和马方面的应用基础研究。
| [1] | MIGLIOR F, GONG W, WANG Y, et al. Short communication: genetic parameters of production traits in Chinese Holsteins using a random regression test-day model[J]. Dairy Sci, 2009, 92(9): 4697–4706. DOI: 10.3168/jds.2009-2212 |
| [2] | ROTHSCHILD M F, HILL H T, CHRISTIAN L L, et al. Genetic differences in serum-neutralization titers of pigs after vaccination with pseudorabies modified live-virus vaccine[J]. Am J Vet Res, 1984, 45(6): 1216–1218. |
| [3] |
王超, 赵书红, 朱猛进. 猪抗病育种的相关问题及研究进展[J]. 中国畜牧杂志, 2014, 50(22): 67–72.
WANG C, ZHAO S H, ZHU M J. Related problems and research progress in anti-disease breeding of pig[J]. Chinese Journal of Animal Science, 2014, 50(22): 67–72. DOI: 10.3969/j.issn.0258-7033.2014.22.014 (in Chinese) |
| [4] |
朱猛进, 吴珍芳, 赵书红. 猪抗病育种研究进展及对几个认识问题的讨论[J]. 中国畜牧兽医, 2007, 34(4): 63–67.
ZHU M J, WU Z F, ZHAO S H. The study progresses of porcine anti-disease breeding and discussions of several problems[J]. China Animal Husbandry & Veterinary Medicine, 2007, 34(4): 63–67. (in Chinese) |
| [5] | ROTGER M, DANG K K, FELLAY J, et al. Genome-wide mRNA expression correlates of viral control in CD4+ T-cells from HIV-1-infected individuals[J]. PLoS Pathogens, 2010, 6(2): e1000781. DOI: 10.1371/journal.ppat.1000781 |
| [6] | GLADUE D P, ZHU J, HOLINKA L G, et al. Patterns of gene expression in swine macrophages infected with classical swine fever virus detected by microarray[J]. Virus Res, 2010, 151(1): 10–18. DOI: 10.1016/j.virusres.2010.03.007 |
| [7] | LI L, XU Z F, ZHOU Y, et al. Global effects of catecholamines on Actinobacillus pleuropneumoniae gene expression[J]. PLoS One, 2012, 7(2): e31121. DOI: 10.1371/journal.pone.0031121 |
| [8] |
王晓铄, 俞英. 表观遗传对炎症的调控机制及其在奶牛乳房炎抗病育种中的应用前景[J]. 遗传, 2010, 32(7): 663–669.
WANG X S, YU Y. Regulation mechanisms of epigenetics on inflammation and its perspective on breeding for mastitis resistance in dairy cattle[J]. Hereditas (Beijing), 2010, 32(7): 663–669. (in Chinese) |
| [9] |
刘会杰, 王晓铄, 王志军, 等. 大白、长白猪CD4基因启动子多态性及表达差异研究[J]. 中国畜牧杂志, 2014, 50(1): 10–14.
LIU H J, WANG X S, WANG Z J, et al. Study on genetic polymorphism in the promoter of CD4 gene and its expression in Yarkshire and Landrace[J]. Chinese Journal of Animal Science, 2014, 50(1): 10–14. (in Chinese) |
| [10] |
许金根. 猪T淋巴细胞亚群的候选基因分析及表达谱研究[D]. 北京: 中国农业大学, 2013.
XU J G. Analysis of candidate genes and study of expression profile for porcine T lymphocyte subpopulations[D]. Beijing: China Agricultural University, 2013. (in Chinese) |
| [11] |
王晓铄. dsRNA转染猪肾细胞后DNA甲基化抑制剂对全基因组DNA甲基化及表达的调控[D]. 北京: 中国农业大学, 2013.
WANG X S. Regulation of DNA methyltransferases inhibitor on genome-wide methylation and expression in porcine kidney epithelial cells after transfected by synthetic viral-like dsRNA[D]. Beijing: China Agricultural University, 2013. (in Chinese) |
| [12] | BOSCARIOL R, PLEASANCE J, PIEDRAFITA D M, et al. Identification of two allelic forms of ovine CD4 exhibiting a Ser183/Pro183 polymorphism in the coding sequence of domain 3[J]. Vet Immunol Immunopathol, 2006, 113(3-4): 305–312. DOI: 10.1016/j.vetimm.2006.05.015 |
| [13] |
范景胜. 山羊CD4基因外显子6多态性与精子内化外源DNA能力的相关性研究[D]. 重庆: 西南大学, 2009.
FAN J S. Association of goat (Capra hircus) CD4 gene exon 6 polymorphism with ability of sperm internalizing exogenous DNA[D]. Chongqing: Southwest University, 2009.(in Chinese) http://d.wanfangdata.com.cn/Thesis/Y1459499 |
| [14] | HE Y, CHU Q, MA P, et al. Association of bovine CD4 and STAT5b single nucleotide polymorphisms with somatic cell scores and milk production traits in Chinese Holsteins[J]. J Dairy Res, 2011, 78(2): 242–249. DOI: 10.1017/S0022029911000148 |
| [15] | LUO J, YU Y, ZHANG H M, et al. Down-regulation of promoter methylation level of CD4 gene after MDV infection in MD-susceptible chicken line[J]. BMC Proc, 2011, 5(S4): S7. |
| [16] |
孙君重, 肖文华, 于力. CD4+ T淋巴细胞功能研究新进展[J]. 中国实验血液学杂志, 2010, 18(2): 544–548.
SUN J Z, XIAO W H, YU L. Recent research advances on function of CD4+ T lymphocytes-review[J]. Journal of Experimental Hematology, 2010, 18(2): 544–548. (in Chinese) |
| [17] | BÄCKDAHI L, BUSHELL A, BECK S. Inflammatory signalling as mediator of epigenetic modulation in tissue-specific chronic inflammation[J]. Int J Biochem Cell Biol, 2009, 41(1): 176–184. DOI: 10.1016/j.biocel.2008.08.023 |
| [18] | WANG X S, ZHANG Y, HE Y H, et al. Aberrant promoter methylation of the CD4 gene in peripheral blood cells of mastitic dairy cows[J]. Genet Mol Res, 2013, 12(4): 6228–6239. DOI: 10.4238/2013.December.4.10 |
| [19] |
林晓晨. 猪CD4基因启动子与增强子调控基因表达的特性研究[D]. 长春: 吉林大学, 2012.
LIN X C. Characterization of gene expression regulated by CD4 gene promoter and enhancer of porcine[D]. Changchun: Jilin University, 2012. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10183-1012370645.htm |
| [20] |
郑双艳, 赵永聚, 许宝华, 等. 山羊CD4基因的克隆及组织表达分析[J]. 西南大学学报:自然科学版, 2012, 34(2): 1–8.
ZHENG S Y, ZHAO Y J, XU B H, et al. Cloning of goat CD4 gene and its tissue expression[J]. Journal of Southwest University: Natural Science Edition, 2012, 34(2): 1–8. (in Chinese) |
| [21] |
李立明.
流行病学[M]. 北京: 人民卫生出版社, 2008.
LI L M. Epidemiology[M]. Beijing: People's Medical Publishing House, 2008. (in Chinese) |
| [22] |
冷伟建. 脂多糖受体复合物基因多态性与感染易感性相关研究[D]. 南京: 第一军医大学, 2006.
LENG W J. Investigation of the association between genetic polymorphisms of lipopolysaccaride receptor complex and susceptibility of infection[D]. Nanjing: First Military Medical University, 2006. (in Chinese) http://industry.wanfangdata.com.cn/dl/Detail/Periodical?id=Periodical_zgjjyx200509016 |
| [23] | RIVAS A L, SCHWAGER S J, GONZÁLEZ R N, et al. Multifactorial relationships between intramammary invasion by Staphylococcus aureus and bovine leukocyte markers[J]. Can J Vet Res, 2007, 71(2): 135–144. |
| [24] |
孙庆申, 霍庆莉, 蔡雪晖, 等. 猪繁殖与呼吸综合征病毒感染猪外周血CD4、CD8分子动态变化研究[J]. 黑龙江畜牧兽医, 2008(4): 14–15.
SUN Q S, HUO Q L, CAI X H, et al. Dynamic analysis of CD4 and CD8 molecules in peripheral blood of pigs inoculated with PRRSV[J]. Heilongjiang Animal Science and Veterinary Medicine, 2008(4): 14–15. (in Chinese) |
| [25] |
刘国民. 无角多赛特、苏尼特、乌珠穆沁羊种间抗病性比较[D]. 呼和浩特: 内蒙古农业大学, 2011.
LIU G M. Comparative of disease resistivity between Poll Dorset sounite and Ujmuqin sheep[D]. Hohhot: Inner Mongolia Agricultural University, 2011. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10129-1011178440.htm |
| [26] | DEGROOTE R L, UHL P B, AMANN B, et al. Formin like 1 expression is increased on CD4+T lymphocytes in spontaneous autoimmune uveitis[J]. J Proteomics, 2017, 154: 102–108. DOI: 10.1016/j.jprot.2016.12.015 |
| [27] | BAHADORAN A, LEE S H, WANG S M, et al. Immune responses to influenza virus and its correlation to age and inherited factors[J]. Front Microbiol, 2016, 7: 1841. |
| [28] | FEIL R, FRAGA M F. Epigenetics and the environment: emerging patterns and implications[J]. Nat Rev Genet, 2012, 13(2): 97–109. |
| [29] |
党永辉, 李生斌, 孙中生. 重性抑郁障碍发病的表观遗传调控假说[J]. 遗传, 2008, 30(6): 665–670.
DANG Y H, LI S B, SUN Z S. The role of epigenetic regulation in etiology of major depressive disorder[J]. Hereditas (Beijing), 2008, 30(6): 665–670. (in Chinese) |
| [30] | LANDOLFI M M T, SCOLLAY R, PARNES J R. Specific demethylation of the CD4 gene during CD4 T lymphocyte differentiation[J]. Mol Immunol, 1997, 34(1): 53–61. DOI: 10.1016/S0161-5890(96)00104-6 |
| [31] | ISSUREE P D, NG C P, LITTMAN D R. Heritable gene regulation in the CD4:CD8 T cell lineage choice[J]. Front Immunol, 2017, 8: 291. |
| [32] | SAWADA S, LITTMAN D R. Identification and characterization of a T-cell-specific enhancer adjacent to the murine CD4 gene[J]. Mol Cell Biol, 1991, 11(11): 5506–5515. DOI: 10.1128/MCB.11.11.5506 |
| [33] | SAWADA S, SCARBOROUGH J D, KILLEEN N, et al. A lineage-specific transcriptional silencer regulates CD4 gene expression during T lymphocyte development[J]. Cell, 1994, 77(6): 917–929. DOI: 10.1016/0092-8674(94)90140-6 |
| [34] | SAWADA S, LITTMAN D R. A heterodimer of HEB and an E12-related protein interacts with the CD4 enhancer and regulates its activity in T-cell lines[J]. Mol Cell Biol, 1993, 13(9): 5620–5628. DOI: 10.1128/MCB.13.9.5620 |
| [35] | HUANG Z F, XIE H M, IOANNIDIS V, et al. Transcriptional regulation of CD4 gene expression by T cell factor-1/β-catenin pathway[J]. J Immunol, 2006, 176(8): 4880–4887. DOI: 10.4049/jimmunol.176.8.4880 |
| [36] | CHI T H, WAN M M, ZHAO K J, et al. Reciprocal regulation of CD4/CD8 expression by SWI/SNF-like BAF complexes[J]. Nature, 2002, 418(6894): 195–199. DOI: 10.1038/nature00876 |
| [37] | HENSON D M, CHOU C, SAKURAI N, et al. A silencer-proximal intronic region is required for sustained CD4 expression in postselection thymocytes[J]. J Immunol, 2014, 192(10): 4620–4627. DOI: 10.4049/jimmunol.1302374 |
| [38] | SELLARS M, HUH J R, DAY K, et al. Regulation of DNA methylation dictates Cd4 expression during the development of helper and cytotoxic T cell lineages[J]. Nat Immunol, 2015, 16(7): 746–754. DOI: 10.1038/ni.3198 |
| [39] | TSAGARATOU A, ÄIJO T, LIO C W, et al. Dissecting the dynamic changes of 5-hydroxymethylcytosine in T-cell development and differentiation[J]. Proc Natl Acad Sci U S A, 2014, 111(32): E3306–E3315. DOI: 10.1073/pnas.1412327111 |
| [40] | LI W, SUN W, LIU L, et al. IL-32: A host proinflammatory factor against influenza viral replication is upregulated by aberrant epigenetic modifications during influenza A virus infection[J]. J Immunol, 2010, 185(9): 5056–5065. DOI: 10.4049/jimmunol.0902667 |
| [41] | LI M Z, WU H L, LUO Z G, et al. An atlas of DNA methylomes in porcine adipose and muscle tissues[J]. Nat Commun, 2012, 3: 850. DOI: 10.1038/ncomms1854 |
| [42] | ECKER S, CHEN L, PANCALDI V, et al. Genome-wide analysis of differential transcriptional and epigenetic variability across human immune cell types[J]. Genome Biol, 2017, 18: 18. DOI: 10.1186/s13059-017-1156-8 |
| [43] | NOVIK K L, NIMMRICH I, GENC B, et al. Epigenomics: genome-wide study of methylation phenomena[J]. Curr Issues Mol Biol, 2002, 4(4): 111–128. |
| [44] | ZOU Y R, SUNSHINE M J, TANIUCHI I, et al. Epigenetic silencing of CD4 in T cells committed to the cytotoxic lineage[J]. Nat Genet, 2001, 29(3): 332–336. DOI: 10.1038/ng750 |
| [45] | HUANG Y, PASTOR W A, SHEN Y H, et al. The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing[J]. PLoS One, 2010, 5: e8888. DOI: 10.1371/journal.pone.0008888 |
| [46] | FLANAGAN J M. Host epigenetic modifications by oncogenic viruses[J]. Br J Cancer, 2007, 96(2): 183–188. DOI: 10.1038/sj.bjc.6603516 |
| [47] | JIN L, JIANG Z, XIA Y D, et al. Genome-wide DNA methylation changes in skeletal muscle between young and middle-aged pigs[J]. BMC Genomics, 2014, 15: 653. DOI: 10.1186/1471-2164-15-653 |
| [48] | SONG M Y, HE Y H, ZHOU H K, et al. Combined analysis of DNA methylome and transcriptome reveal novel candidate genes with susceptibility to bovine Staphylococcus aureus subclinical mastitis[J]. Sci Rep, 2016, 6: 29390. DOI: 10.1038/srep29390 |
| [49] | TANG B K, ZHAO R H, SUN Y, et al. Interleukin-6 expression was regulated by epigenetic mechanisms in response to influenza virus infection or dsRNA treatment[J]. Mol Immunol, 2011, 48(8): 1001–1008. DOI: 10.1016/j.molimm.2011.01.003 |
| [50] | SIMES M, RINO J, PINHEIRO I, et al. Alterations of nuclear architecture and epigenetic signatures during African swine fever virus infection[J]. Viruses, 2015, 7(9): 4978–4996. DOI: 10.3390/v7092858 |
| [51] |
王海飞. 猪外周血单核细胞抗病毒反应的转录组及全基因组DNA甲基化调控研究[D]. 北京: 中国农业大学, 2016.
WANG H F. Genome-wide analysis of transcriptome and DNA methylation regulation in antiviral response of porcine peripheral blood mononuclear cells[D]. Beijing: China Agricultural University, 2016. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10019-1016085422.htm |
| [52] | LIANG W, LI Z H, WANG P, et al. Differences of immune responses between Tongcheng (Chinese local breed) and Large White pigs after artificial infection with highly pathogenic porcine reproductive and respiratory syndrome virus[J]. Virus Res, 2016, 215: 84–93. DOI: 10.1016/j.virusres.2016.02.004 |
| [53] | CLAPPERTON M, BISHOP S C, GLASS E J. Innate immune traits differ between Meishan and Large White pigs[J]. Vet Immunol Immunopathol, 2005, 104(3-4): 131–144. DOI: 10.1016/j.vetimm.2004.10.009 |
| [54] | HALBUR P G, ROTHSCHILD M F, T HACKER B J. Differences in susceptibility of Duroc, Hampshire, and Meishan pigs to infection with a high virulence strain (VR2385) of porcine reproductive and respiratory syndrome virus (PRRSV)[J]. J Anim Breed Genet, 1998, 115(1-6): 181–189. DOI: 10.1111/jbg.1998.115.issue-1-6 |
| [55] | WANG X S, AO H, ZHAI L W, et al. Transcriptional regulation of CD4 gene expression in porcine kidney epithelial cells by virus-like double-stranded RNA and DNA methyltransferase inhibitor[J]. Genet Mol Res, 2014, 13(2): 3346–3355. DOI: 10.4238/2014.April.29.13 |
| [56] |
王晓铄, 俞英, 刘会杰, 等. 基于表达谱芯片挖掘抗猪瘟病毒相关基因及其在不同猪瘟抗体水平下的表达差异[J]. 畜牧兽医学报, 2014, 45(1): 14–22.
WANG X S, YU Y, LIU H J, et al. Mining anti classic swine fever virus-related genes based on Microarray and analyzing their transcriptional expression under different CSF-antibody levels[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(1): 14–22. (in Chinese) |
| [57] | WANG X S, AO H, ZHAI L W, et al. Genome-wide effects of DNA methyltransferase inhibitor on gene expression in double-stranded RNA transfected porcine PK15 cells[J]. Genomics, 2014, 103(5-6): 371–379. DOI: 10.1016/j.ygeno.2013.10.005 |
| [58] |
丁伯良, 冯建忠, 张国伟.
奶牛乳房炎[M]. 北京: 中国农业出版社, 2011.
DING B L, FENG J Z, ZHANG G W. Dairy mastitis[M]. Beijing: China Agriculture Press, 2011. (in Chinese) |
| [59] | HE Y H, YU Y, ZHANG Y, et al. Genome-wide bovine H3K27me3 modifications and the regulatory effects on genes expressions in peripheral blood lymphocytes[J]. PLoS One, 2012, 7(6): e39094. DOI: 10.1371/journal.pone.0039094 |
| [60] |
刘小刚, 侯先志, 李大彪, 等. 营养限制及补偿对羔羊体重及外周血液CD4+和CD8+ T淋巴细胞的影响[J]. 动物营养学报, 2010, 22(4): 934–942.
LIU X G, HOU X Z, LI D B, et al. Effects of feed restriction and compensation on body weight, CD4+ and CD8+ T-lymphocytes in peripheral blood of lambs[J]. Chinese Journal of Animal Nutrition, 2010, 22(4): 934–942. (in Chinese) |
| [61] | WATTEGEDERA S R, CORRIPIO-MIYAR Y, PANG Y, et al. Enhancing the toolbox to study IL-17A in cattle and sheep[J]. Vet Res, 2017, 48: 20. DOI: 10.1186/s13567-017-0426-5 |
| [62] | CHATZINASIOU E, CHAINTOUTIS S C, DOVAS C I, et al. Immunosuppression in sheep induced by cyclophosphamide, bluetongue virus and their combination: Effect on clinical reaction and viremia[J]. Microb Pathog, 2017, 104: 318–327. DOI: 10.1016/j.micpath.2017.01.048 |
| [63] | ARRANZ-SOLÍS D, BENAVIDES J, REGIDOR-CERRILLO J, et al. Systemic and local immune responses in sheep after Neospora caninum experimental infection at early, mid and late gestation[J]. Vet Res, 2016, 47: 2. DOI: 10.1186/s13567-015-0290-0 |
| [64] |
赵一萍. 蒙古马免疫相关基因表达研究及脾脏表达谱分析[D]. 呼和浩特: 内蒙古农业大学, 2013.
ZHAO Y P. The expression study of immune related genes and the analysis of spleen expression profiles of Mongolian horse[D]. Hohhot: Inner Mongolia Agricultural University, 2013. (in Chinese) https://wuxizazhi.cnki.net/lunwen-1013249784.html |
| [65] | GIM J A, HONG C P, KIM D S, et al. Genome-wide analysis of DNA methylation before-and after exercise in the thoroughbred horse with MeDIP-seq[J]. Mol Cells, 2015, 38(3): 210–220. DOI: 10.14348/molcells.2015.2138 |
| [66] | LIU S A, HAQUE M, STANFIELD B, et al. A recombinant fusion protein consisting of West Nile virus envelope domain Ⅲ fused in-frame with equine CD40 ligand induces antiviral immune responses in horses[J]. Vet Microbiol, 2017, 198: 51–58. DOI: 10.1016/j.vetmic.2016.12.008 |
| [67] | DURWARD-AKHURST S A, FINNO C J, BARNES N, et al. Major Histocompatibility complex Ⅰ and Ⅱ expression and lymphocytic subtypes in muscle of horses with immune-mediated myositis[J]. J Vet Intern Med, 2016, 30(4): 1313–1321. DOI: 10.1111/jvim.14371 |
| [68] | ZIEGLER A, MARTI E, SUMMERFIELD A, et al. Identification and characterization of equine blood plasmacytoid dendritic cells[J]. Dev Comp Immunol, 2016, 65: 352–357. DOI: 10.1016/j.dci.2016.08.005 |



