2. 安徽农业大学动物科技学院, 合肥 230036
2. College of Animal Science and Technology, Anhui Agricultural University, Hefei 230036, China
真核生物体内分布着大量的小RNA,这些不同的小RNA参与基因表达调控。秀丽隐杆线虫中lin-4(一种小RNA)时序性负调控lin-14蛋白表达,这是首次发现的小RNA所参与的基因沉默作用[1]。之后,科研工作者发现存在2类非编码小RNA:小干扰RNA(Small interfering RNA,siRNA)和微小RNA(microRNA,miRNA)。2006年,多个实验室先后发现了另一种新型的非编码小RNA,这种小RNA能够特异性结合Piwi蛋白,因此将其命名为piRNA(Piwi-interacting RNA)[2-3]。随后piRNA成为非编码小RNA研究领域的一个热点,先后在果蝇、线虫、斑马鱼、小鼠、大鼠等动物研究中发现piRNA在生殖细胞及干细胞分化[4]、种系DNA完整性[5]、性别决定[6]、胚胎发育[7]、免疫防御[8]及癌症预测[9-10]等方面有重要作用。本文主要围绕piRNA的生物发生、Piwi与piRNA复合体发挥生物学作用的方式、piRNA在雌性动物中的生物学作用这3个方面对近期的研究进展进行综述,为深入探究piRNA调控雌性动物繁殖机制提供参考。
1 piRNA概述 1.1 piRNA的产生途径2006年A. Aravin等[2]以3月龄C57BL/6J雄性小鼠为研究对象,利用吸附柱离心法从输精管中分离得到一种小RNA,并通过蛋白印记法检测到这种高表达量的小RNA与MILI(一种Piwi亚蛋白)相互作用,他们将其命名为piRNA。之后,对piRNA的生物学来源研究发现,基因组上基因间区大量的piRNA簇经双向或单向转录形成piRNA前体,再由内切酶剪切产生piRNA[11];此外,在mRNA的3′非翻译区(3′ UTR)及基因组中某些长的非编码区域也可产生piRNA[12]。这些新生的piRNA通常被转运出细胞核,在胞质中被加工形成成熟的piRNA发挥生物学功能。piRNA的产生机制分为初级piRNA途径和次级piRNA途径,主要围绕Slicer活性剪切的乒乓扩增循环(Ping-pong amplification cycle)[13]过程完成piRNA的成熟加工。E.M.Weick等[14]对piRNA产生的“乒乓”循环模型进行了总结:生殖细胞的细胞核中piRNA簇转录产生的长链pre-piRNA被转运到细胞质中,它被Ago3识别结合形成Ago3-piRNA复合体,然后piRNA 5′端被线粒体外膜的核酸内切酶Zuc剪切形成5′端磷酸化的piRNA,随后与Piwi或Aub蛋白结合且5′端变为尿嘧啶,3′端也被剪切;在之后的“乒乓”扩增循环过程中,结合了piRNA的Aub可以识别与piRNA互补的RNA序列(例如来源于piRNA簇的另一链的RNA)并对其进行剪切,剪切形成的piRNA又与Ago3结合形成复合体,反过来诱导剪切与之互补的RNA,这样就生成了与循环中最初piRNA一致的序列,如此不断循环下去(图 1)。该模型对理解piRNA发生机制、调控通路、生物学功能有重要意义。之后,科研工作者对piRNA的“乒乓”途径研究不断深入和细化,发现除了动物生殖细胞外,体细胞中也存在piRNA合成途径,而且生殖细胞和体细胞中piRNA“乒乓”扩增过程存在差异。piRNA的体细胞途径可以直接依赖Piwi蛋白和piRNA簇Flamenco片段产生piRNA,以转座子为靶标形成piRNA,首先是piRNA前体与细胞器Yb小体(Yb body)结合,在Zuc及辅助因子(Vret、Mino、Gasz等)作用下可形成中间体piRNA的5′U位点,再产生piRNA次级结构,Zuc对Piwi-piRNA序列剪切形成3′末端,并通过外切酶trimmer、Papi作用切割Piwi-piRNA的成熟位点,接着发生Hen1介导的甲基化,最后成熟的Piwi-piRNA复合物进入细胞核起沉默作用,线粒体膜外因子在该途径中扮演重要角色[15]。生殖细胞和体细胞中piRNA生成途径的主要差异总结见表 1。

|
图 1 果蝇生殖细胞piRNA形成途径(乒乓循环)[16] Figure 1 piRNA biogenesis in Drosophila germ cells(Ping-Pong cycle)[16] |
|
|
表 1 生殖细胞和体细胞中piRNA生成途径的主要差异 Table 1 Major difference of the piRNA biogenesis in germ cells and somatic cells |
相比siRNA和miRNA,piRNA数量较多,约5×104种[5],其产生不依赖RNaseⅢ类核酸酶Dicer[17],长度略大于前两者,约在24~35 nt范围内[18],3′端2′-O-被甲基化修饰,5′端第一个核苷酸有尿嘧啶偏向性且第10位碱基有腺嘌呤偏向性[14]。小鼠piRNA主要分布在X染色体上,Y染色体上分布很少;而人体piRNA绝大多数分布在常染色体上,在性染色体上基本没有分布[3, 19]。果蝇中piRNA通常与Argonaute蛋白中Piwi亚家族成员(Piwi、Aub、Ago3) 特异性结合,分别形成Piwi-piRNA、Aub-piRNA、Ago3-piRNA 3类复合体。其中,Piwi-piRNA和Aub-piRNA中piRNA来源于反义链,5′端有尿嘧啶偏向性;而与Ago3结合的piRNA来源于正义链,5′端倾向于腺苷酸。因此,Piwi-piRNA或Aub-piRNA与Ago3-piRNA的5′端10 nt序列恰好互补配对。
1.3 piRNA预测和分析采用有效的方法预测和分析piRNA,对解读其“乒乓”机制,准确预测piRNA的功能具有重要意义。“piRNA Bank”数据库(http://pirnabank.ibab.ac.in/)中记录了人和一些模式生物(小鼠、大鼠、果蝇)的piRNA相关信息[19],可查询单个piRNA、piRNA簇、同源piRNAs,还可展示可视化的piRNA图谱。2011年,中国科学院动物研究所Y.Zhang等[20]提出了一种基于k-mer串频率的Fisher判别式,它是区分piRNA和其他非编码RNA的一种方法,能精确预测piRNA(精度大于90%)。在此基础上,研究者还开发了在线piRNA预测软件piRNApredictor(http://59.79.168.90/piRNA/index.php),可以预测非模式生物的piRNA序列,具有广泛的应用价值。近几年,科研工作者创建了“piBase”平台并进一步开发“PirTarget”程序,以利于通过piRNA “乒乓”循环机制去预测潜在的靶基因(如mRNA) [21-22]。笔者总结了几个piRNA预测和分析软件或程序的信息,如表 2所示。
|
|
表 2 预测和分析piRNA的相关软件或程序 Table 2 The related softwares or programs of predicting and analysing piRNA |
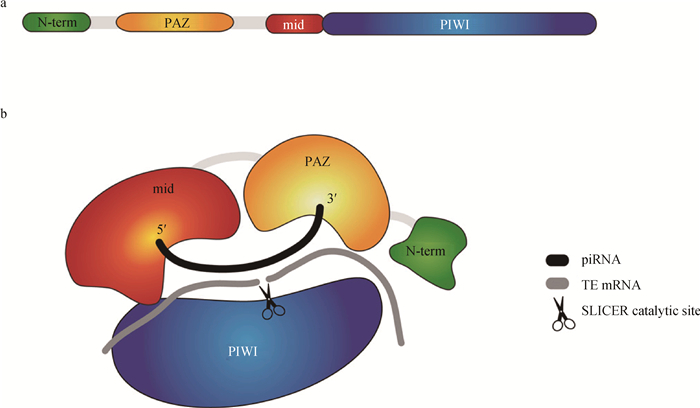
研究表明,果蝇卵巢中卵子的发生过程涉及2种特殊蛋白(Pumilio和Piwi),它们对维持生殖干细胞、促进配子不对称分裂等有重要的调控作用[23]。之后研究者在线虫、小鼠、大鼠、斑马鱼等多种试验动物及人类蛋白研究中相继发现了不同种类的Piwi亚家族蛋白因子,并且发现Piwi亚家族蛋白可特异性结合piRNA。果蝇中存在Piwi、Aub、Ago3 3类Piwi亚家族蛋白,且Ago3仅在雌性生殖细胞中表达[24];非洲爪蟾的Piwi亚家族蛋白包括XILI、XIWI、XIWI2 3种;在小鼠中发现了参与生殖细胞发育的3类Piwi亚家族蛋白:MIWI (Piwil1)、MILI (Piwil2) 和MIWI2(Piwil4)[25];灵长目动物包含Piwil1、Piwil2、Piwil3、Piwil4 4类Piwi亚家族蛋白[26],其中人的Piwi亚家族蛋白主要包括HIWI、HIWI2、Piwil3、HILI 4类[27]。大部分动物体的Piwi亚家族蛋白通常由N端结构域、PAZ结构域、mid结构域及Piwi结构域共四部分构成其保守结构(图 2a),piRNA的5′端和3′端分别与mid结构域和PAZ结构域结合,最终形成Piwi-piRNA沉默复合物(piRISC,图 2b),参与piRNA干扰途径[28]。
|
a.Piwi亚家族蛋白结构域由N端结构域、PAZ结构域、mid结构域及Piwi结构域共4部分构成,其中N端区域是富含精氨酸的概念域。b.Piwi亚家族蛋白域在空间构成上,其mid结构域锚定piRNA的5′端且PAZ结构域与piRNA的3′端相接;Piwi域是最大的一个结构域,它的催化位点可剪切与piRNA 5′端有关的10 nt长的靶向RNA[29] a. The domains of Piwi proteins are comprised of the N-terminal region, PAZ, mid and Piwi domains. The N-terminal region consists of a notional domain that is characterized by arginine rich motifs. b.The organization of the protein domains in space shows how the mid-domain anchors the piRNA at its 5′ end and the PAZ domain holds the 3′ end of the piRNA. Piwi is the largest domain and its catalytic site responsible for 'slicer' activity is positioned to cleave the backbone of annealed target RNA exactly 10 nt related to the 5′ end of the piRNA[29] 图 2 Piwi蛋白结构 Figure 2 Structure of Piwi protein |
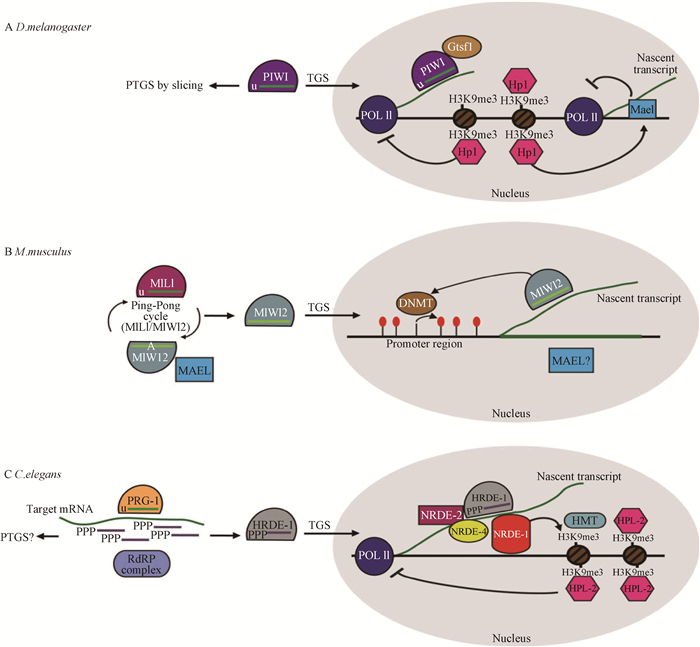
Piwi-piRNA复合体主要以转录水平调控和转录后调控2种方式抑制基因表达。其中,转录后调控具有序列特异性,依赖于有活性的催化酶。“乒乓”循环不仅利用转录后调控来破坏转座子mRNA,而且利用这种机制来扩增沉默活性元件的因子。相反,转录水平调控不依赖于靶RNA的催化,主要通过调控异染色质形成和介导基因组DNA甲基化修饰等方式调控基因的表达。一方面,Piwi亚家族蛋白与piRNA结合形成Piwi-piRNA复合体,可特异性作用于基因组序列或新生转录物上,引导对靶位点的表观遗传调控;另一方面,Piwi-piRNA的高表达导致DNA甲基化转移酶(DNMTs)表达异常,从而提高了DNA甲基化水平。不同生物体piRNA表观遗传调控的作用方式存在差异(图 3)。在黑腹果蝇中,Piwi定位于细胞核并启动组蛋白H3K9三甲基化,同时抑制RNA聚合酶Ⅱ的活性;锌指蛋白Gtsf1可能直接与Piwi互作,而异染色质蛋白Hp1与H3K9me3结合;Mael(Maelstrom spermatogenic transposon silencer)蛋白作用于H3K9me3甲基化的下游,抑制RNA聚合酶Ⅱ活性(图 3A)[30]。在小鼠中,MIWI2与MILI参与“乒乓”循环并转移至核内,通过DNA甲基转移酶(DNMT)作用启动转座子位点上游启动子元件的CpG甲基化(图 3B);在MIWI2位点的细胞质中存在鼠MAEL同源物,这种蛋白在核中的作用与此类似[31]。在线虫中,RNA依赖性RNA聚合酶(RdRP)使PRG-1、piRNA和靶RNA相互作用产生次级siRNA,这些siRNA结合于二级Ago3蛋白HRDE-1上,然后被转运至细胞核中,可能与前mRNA及核干扰因子(NRDE)协同作用,引发H3K9me3甲基化和RNA聚合酶Ⅱ停滞[32](图 3C)。
|
图 3 果蝇(A)、小鼠(B)及线虫(C)体内piRNA介导的转录沉默机制[14] Figure 3 piRNA mediated transcriptional silencing mechanism in D. melanogaster (A), M. musculus (B) and C. elegans (C) [14] |
最早在雄性小鼠生殖细胞中分离得到piRNA,之后发现果蝇[33]、爪蟾[34]及斑马鱼[35]的雌性配子中也存在piRNA。这意味着动物睾丸精原细胞、卵巢卵母细胞中均有piRNA分布。这些生殖细胞中由初级合成途径产生的piRNA分别与Piwi、Aub结合,之后通过“乒乓”循环产生次级piRNA与Ago3结合发挥作用。目前,很多学者报道了piRNA在雄性动物生殖细胞中的作用,但是对于piRNA在雌性动物中发挥的作用鲜有总结,因此,这里我们重点对不同物种雌性动物中piRNA及其作用进行阐述。
3.1 果蝇中的piRNA果蝇是生命科学研究领域最经典的模式生物之一,对果蝇piRNA的研究也最为深入,研究发现,雌性果蝇中的piRNA具有保证雌果蝇正常育性、防止DNA损伤、保证正常卵子形成与释放、保证卵巢中生殖细胞与体细胞之间的正常黏结等生物学功能。对果蝇胚胎发育时期的Piwi蛋白敲除后发现,卵巢中与piRNA簇转录作用有关的H3K9me3减少,piRNA形成受阻,转座子的沉默作用被解除,结果导致雌性不育[36]。果蝇卵巢体细胞和生殖细胞发育过程中piRNA表达有一定差异,体细胞中来源于flamenco(flam)单链簇的piRNA被加载到Piwi蛋白上形成Piwi-piRNA诱导沉默复合体(Piwi-piRISC),调节特异性反转录转座子gypsy的活性,使其维持在较低的表达水平,从而防止因gypsy过表达引起的病毒样颗粒感染邻近生殖细胞,保证雌果蝇正常生育;而生殖细胞中,piRNA簇则通常借助RNA聚合酶Ⅱ转录邻近基因的过程跨越终止信号继续转录产生piRNA,这些piRNA与Aub结合形成Aub-piRNA,通过slicer活性剪切mRNA,使其降解[37-41]。雌性果蝇生殖细胞中还存在一类特异性的反转录转座子I因子,其序列长约5.4 kb,主要作用是诱导雌性不育。piRNA可通过“乒乓”循环特异性抑制活跃的I因子,使其沉默[42]。Twin因子是CCR4-NOT脱腺苷酶复合体中CCR4的同源物,能够调控生殖干细胞(GSC)自我更新和分化。卵巢GSC的Twin可与Aub-piRNA及Ago3-piRNA互作来调节piRNA浓度,从而抑制GSC的转座子反转录活性,防止DNA损伤[43]。Parasitic-DNA是雌性果蝇的遗传转座子,染色体上不同位点Parasitic-DNA跳跃使基因失活,可能引起不育。性成熟的雌性果蝇可以产生piRNA靶向作用于Parasitic-DNA,经过Parasitic-DNA释放的转座子落入piRNA簇,piRNA簇产生反向piRNA,进而调控Parasitic-DNA,防止其跳跃,保证正常卵子形成与释放[44]。研究者发现,果蝇卵泡中的fascilins基因能够编码免疫球蛋白,这种免疫球蛋白有黏着作用,使细胞间黏结异常。piRNA对fascilins有沉默作用,抑制fascilins表达,使卵巢中生殖细胞与体细胞间正常黏结[45]。雌果蝇从胚胎后期至蛹期,其原始生殖细胞中piRNA均存在活跃的同源-依赖型反式沉默(Homology-dependent trans silencing)作用,细胞记忆机制使得piRNA抑制转座子活性的状态从胚胎期一直保持到成体阶段[46]。
3.2 其他模式生物中的piRNA除了果蝇外,小鼠、蟾蜍等其他模式生物的piRNA发挥作用的过程也有其自身的特点。研究表明,Piwi与piRNAs的结合在哺乳动物卵子发生过程中起重要作用[28],Astrin因子以蛋白的身份参与小鼠卵母细胞有丝分裂和减数分裂过程,尤其是对卵母细胞减数分裂过程中纺锤体装配及成熟有关键作用,piRNA可使Astrin功能缺失,起辅助干扰作用,导致卵母细胞纺锤体解体[47]。芳香烃受体(Aryl hydrocarbon receptor,AhR)是促进小鼠生殖细胞发育的因子之一,AhR缺失雌性小鼠的卵泡数减少,piRNA相关蛋白(MVH、MILI、MIWI)表达水平较低且转座子表达下降,暗示piRNA及相关蛋白、转座子表达与发育因子呈正相关,共同维持雌性生殖细胞的发育[48]。利用高通量测序研究非洲爪蟾生殖细胞及体细胞内的小RNAs表达情况,结果显示,piRNAs仅在蟾蜍的生殖细胞中参与表达调控,检测到piRNAs(piR-1和piR-2) 在蟾蜍卵细胞发育的Ⅰ和Ⅱ2个阶段均有表达[49],暗示,piRNAs主要在这2个阶段参与蟾蜍卵细胞发育。对非洲爪蟾卵母细胞Y12免疫沉淀物中piRNAs做深度测序,根据序列互补信息方法分析piRNAs的核苷酸组成,发现piRNA具有如下特征:核苷酸的第1位碱基富含尿嘧啶(U)且第10位碱基富含腺嘌呤(A),piRNAs的第10位核苷酸之间互补;piRNA与Piwi特异性结合,靶向性调控反转录转座子[37]。家蚕雌性染色体为ZW型,雄性为ZZ型。在家蚕的W染色体性别决定区域存在一种由Feminizer前体产生的特殊piRNA(Fem基因),这种piRNA对性别调控级联反应末端的Bmdsx基因进行雌性特异性剪切调控,最终促使个体发育为雌性[6, 50];当Piwi-piRNA复合体功能减弱时,Masc基因(一种CCCH型锌指蛋白)转录产生的mRNA增加,促进Bmdsx转座子趋向于雄性特异性剪切,成年雌性家蚕出现部分雄性分化,腹部呈雄性特征[51]。在此基础上,研究者分别构建了TG-W1、TG-W2和TG-Z 3个拷贝转基因家蚕新品系,研究家蚕piggyBac转座子与piRNA之间的关系,发现TG-W1、TG-W2插入位点两侧的piRNA表达量高于TG-Z插入位点附近的piRNA表达量,由此推测,外源piggyBac转座子在W、Z染色体上转座效率差异与插入位点附近的piRNA数量相关,证明piRNA可抑制家蚕卵巢中piggyBac转座子的过度活跃[52]。家蚕卵巢中表达的BmN4细胞的细胞核周围生殖云(一种细胞器)中,piRNA加载到Armitage及Yb蛋白上,在解旋酶BmVasa的作用下,能抑制转座子活性以防DNA损伤,保证BmN4细胞的正常功能[53]。Bdtud是桔小实蝇(一种果蔬害虫)卵巢中piRNA通路的相关基因之一,它可以作为桔小实蝇卵巢发育的标志性因子与piRNA协作,共同维持卵巢正常发育,这为研制高效杀虫剂以防治害虫产卵及交配提供了参考依据[54]。斑马鱼母系生殖细胞piRNA与Tudor域相关蛋白(Tdrds)特异性结合参与piRNA通路,在生殖细胞减数分裂粗线期的调控及干细胞维持方面有关键作用,Tdrd12突变的斑马鱼将发育为不育的雄性个体[55]。
3.3 家养动物中的piRNA除了模式生物,研究者还探究了piRNA在家养动物中的生物学作用。研究发现,鸡的卵巢及睾丸中均存在piRNA序列,暗示piRNA分子也参与了禽类生殖细胞的发育过程[56]。在鱼类中仅发现有2个Piwi旁系同源基因(Piwil1和Piwil2),通过比较不同多倍体鲫鲤性腺发育和配子发生过程中Piwi-piRNA信号通路表达的异同,发现piRNA的表达量与Piwi基因表达呈正相关,三倍体湘云鲫的Piwil1与Piwil2表达水平在繁殖和非繁殖季节均显著高于二倍体红鲫及异源四倍体鲫鲤;繁殖季节可育的鱼类下丘脑-垂体-性腺轴(HPG axis)相关因子表达增强,升高的雌激素和促黄体素可以抑制piRNA和Piwil基因的表达,从而解除了piRNA对其他性腺发育相关序列的抑制,最终促进排卵;在不育的雌性三倍体湘云鲫中,其卵巢中的促黄体素受体基因表达下调,引起Piwi-piRNA信号通路表达异常升高,三倍体湘云鲫最终表现为不育[57]。在猪发育的各个时期(初情前期、初情期、性成熟期),3种Piwi基因(Piwil1、Piwil2和Piwil4) 均在性腺中特异性表达,在初情前期和初情期时卵巢中上述3种Piwi表达量低于同期睾丸中的表达量,仔猪卵巢的Piwil2表达量高于其他2种Piwil基因,初情前期和初情期卵母细胞中Piwil1表达量高但Piwil4无表达,在性成熟的公、母猪生殖细胞中piRNA均有表达,但卵巢中piRNA长度比睾丸piRNA长2~3个核苷酸;这些Piwi蛋白能促进猪的生殖细胞发育,并与piRNA特异性结合共同抑制转座子活性[58]。
3.4 人的piRNA人胎儿卵巢的piRNA能够介导卵裂[59]。人配子体特异性因子1(Gametocyte-specific factor 1,GTSF1) 是piRNA途径中反转录转座子沉默的关键因子,研究者对女性生殖细胞中GTSF1基因表达进行追踪研究发现,GTSF1在性成熟和妊娠期第8~21周的卵母细胞中有较高的表达[7],暗示女性配子中存在piRNA,且对配子发育过程的反转录转座子有沉默作用。近几年来,科研工作者对肿瘤组织piRNAs表达的相关研究发现piRNA与肿瘤发生有关。乳腺癌是女性患者最常见的疾病之一,其病理病因有待进一步研究。piR-651是一种靶点piRNA,其在乳腺癌及宫颈癌细胞发育的G2/M阶段表达上调,当piR-651抑制剂转染至其癌组织后癌细胞生长受到抑制,说明此piRNA可促进乳腺癌及宫颈癌细胞的生长[60]。乳腺癌细胞中piR-4987、piR-20365、piR-20485和piR-20582这4种piRNA有明显上调趋势(P < 0.001)[9],提示piRNA可作为肿瘤标记之一,这对乳腺癌诊疗有重要意义。多发性骨髓瘤(Multiple myeloma, MM)是一种血液恶性肿瘤,为了验证多发性骨髓瘤(MM)组织中piRNA的表达情况,研究者分别选择了MM患者骨髓活检组织、骨髓CD138+细胞和正常的活检组织及CD138+细胞,发现MM患者骨髓活检组织和骨髓CD138+细胞中piRNA-823高表达;在此基础上,注入piRNA-823的抑制物antagomir-823,观察到piRNA-823表达下调后,加速了MM细胞的凋亡;检测DNMT3A和DNMT3B与piRNA-823之间的变化关系,发现piRNA-823高表达引起DNMT3A和DNMT3B的活性增强,基因组甲基化水平升高,肿瘤抑制基因的甲基化程度也升高,从而促进肿瘤发生。同时还验证了piRNA-823与MM血管新生作用的关系,发现piRNA-823表达与MM细胞的血管内皮细胞生长因子分泌呈正相关[61]。这些研究结果表明piRNA具有促进肿瘤发生的生物学作用。
4 展望piRNA作为一种内源性非编码小RNA,是近十年来小RNA领域关注的热点之一,近几年来该领域学者初步阐明了piRNA形成过程、piRNA与Piwi亚家族蛋白偶联发挥功能的方式,发现在雌、雄性动物体均存在Piwi-piRNA通路且作用分子机制类似,piRNA在雄性动物中保证精子正常生成和成熟,而在雌性动物中促进卵细胞正常发育和释放、使卵巢生殖细胞和体细胞正常黏结、保证个体正常生育等。piRNA的这些研究结果对于动物遗传育种和疾病诊治具有重要意义。但是,还有一些问题尚未解决,例如:(1) piRNA簇、piRNA前体区别于其他基因组的明显特征是什么?它们在产生piRNA过程中有哪些关键细节?(2) 新生piRNA输出细胞核过程中会有哪些信号?(3) piRNA与其他非编码小RNA(如miRNA和siRNA)是否存在生物关联、关联的机制是什么?(4) 与piRNA互作的因子还有哪些?它们通过什么互作机制和调控网络调节生殖细胞发育、配子形成等一系列复杂的生理学过程?(5) piRNA在雌、雄性动物中存在哪些表达差异和突变特性?这些问题均需要做更深入系统的研究。
| [1] | LEE R C, FEINBAUM R L, AMBROS V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843–854. DOI: 10.1016/0092-8674(93)90529-Y |
| [2] | ARAVIN A, GAIDATZIS D, PFEFFER S, et al. A novel class of small RNAs bind to MILI protein in mouse testes[J]. Nature, 2006, 442(7099): 203–207. |
| [3] | GRIVNA S T, BEYRET E, WANG Z, et al. A novel class of small RNAs in mouse spermatogenic cells[J]. Genes Dev, 2006, 20(13): 1709–1714. DOI: 10.1101/gad.1434406 |
| [4] | GONZALEZ J, QI H Y, LIU N, et al. Piwi is a key regulator of both somatic and germline stem cells in the Drosophila testis[J]. Cell Rep, 2015, 12(1): 150–161. DOI: 10.1016/j.celrep.2015.06.004 |
| [5] | HUANG Y, BAI J Y, REN H T. PiRNAs biogenesis and its functions[J]. Bioorg Khim, 2014, 40(3): 320–326. |
| [6] | KIUCHI T, KOGA H, KAWAMOTO M, et al. A single female-specific piRNA is the primary determiner of sex in the silkworm[J]. Nature, 2014, 509(7502): 633–636. DOI: 10.1038/nature13315 |
| [7] | HUNTRISS J, LU J P, HEMMINGS K, et al. Isolation and expression of the human gametocyte-specific factor 1 gene (GTSF1) in fetal ovary, oocytes, and preimplantation embryos[J]. J Assist Reprod Genet, 2017, 34(1): 23–31. DOI: 10.1007/s10815-016-0795-0 |
| [8] | DIETRICH I, SHI X H, MCFARLANE M, et al. The antiviral RNAi response in vector and non-vector cells against orthobunyaviruses[J]. PLoS Negl Trop Dis, 2017, 11: e0005272. DOI: 10.1371/journal.pntd.0005272 |
| [9] | HUANG G, HU H, XUE X, et al. Altered expression of piRNAs and their relation with clinicopathologic features of breast cancer[J]. Clin Transl Oncol, 2013, 15(7): 563–568. DOI: 10.1007/s12094-012-0966-0 |
| [10] | ASSUMPÇÃO C B, CALCAGNO D Q, ARAÚJO T M T, et al. The role of piRNA and its potential clinical implications in cancer[J]. Epigenomics, 2015, 7(6): 975–984. DOI: 10.2217/epi.15.37 |
| [11] | YAMANAKA S, SIOMI M C, SIOMI H. piRNA clusters and open chromatin structure[J]. Mob DNA, 2014, 5: 22. DOI: 10.1186/1759-8753-5-22 |
| [12] | MOYANO M, STEFANI G. piRNA involvement in genome stability and human cancer[J]. J Hematol Oncol, 2015, 8: 38. DOI: 10.1186/s13045-015-0133-5 |
| [13] | GIRARD A, HANNON G J. Conserved themes in small-RNA-mediated transposon control[J]. Trends Cell Biol, 2008, 18(3): 136–148. DOI: 10.1016/j.tcb.2008.01.004 |
| [14] | WEICK E M, MISKA E A. piRNAs: from biogenesis to function[J]. Development (Cambridge, England), 2014, 141(18): 3458–3471. DOI: 10.1242/dev.094037 |
| [15] | CZECH B, HANNON G J. One loop to rule them all: the Ping-Pong cycle and piRNA-guided silencing[J]. Trends Biochem Sci, 2016, 41(4): 324–337. DOI: 10.1016/j.tibs.2015.12.008 |
| [16] | LE THOMAS A, TÓTH K F, ARAVIN A A. To be or not to be a piRNA: genomic origin and processing of piRNAs[J]. Genome Biol, 2014, 15: 204. DOI: 10.1186/gb4154 |
| [17] | LUTEIJN M J, KETTING R F. Piwi-interacting RNAs: from generation to transgenerational epigenetics[J]. Nat Rev Genet, 2013, 14(8): 523–534. DOI: 10.1038/nrg3495 |
| [18] | SAITO K, ISHIZU H, KOMAI M, et al. Roles for the Yb body components Armitage and Yb in primary piRNA biogenesis in Drosophila[J]. Genes Dev, 2010, 24(22): 2493–2498. DOI: 10.1101/gad.1989510 |
| [19] | LAKSHMI S S, AGRAWAL S. piRNABank: a web resource on classified and clustered Piwi-interacting RNAs[J]. Nucleic Acids Res, 2008, 36(S1): D173–D177. |
| [20] | ZHANG Y, WANG X H, KANG L. A k-mer scheme to predict piRNAs and characterize locust piRNAs[J]. Bioinformatics, 2011, 27(6): 771–776. DOI: 10.1093/bioinformatics/btr016 |
| [21] | ZANG P, SI X H, SKOGERBØ G, et al. piRBase: a web resource assisting piRNA functional study[J]. Database, 2014, 2014: bau110. DOI: 10.1093/database/bau110 |
| [22] |
许甘霖, 齐绪峰, 蔡冬青. 依据"ping-pong"循环机制预测piRNA靶基因的搜索方法[J]. 基础医学与临床, 2017, 37(3): 399–401.
XU G L, QI X F, CAI D Q. The research method of predicting piRNA target genes by "ping-pong"mechanism[J]. Basic & Clinical Medicine, 2017, 37(3): 399–401. (in Chinese) |
| [23] | LIN H, SPRADLING A C. A novel group of pumilio mutations affects the asymmetric division of germline stem cells in the Drosophila ovary[J]. Development (Cambridge, England), 1997, 124(12): 2463–2476. |
| [24] | SAITO K, NISHIDA K M, MORI T, et al. Specific association of Piwi with rasiRNAs derived from retrotransposon and heterochromatic regions in the Drosophila genome[J]. Genes Dev, 2006, 20(16): 2214–2222. DOI: 10.1101/gad.1454806 |
| [25] | SAMJI T. Piwi, piRNAs, and germline stem cells: What's the link?[J]. Yale J Biol Med, 2009, 82(3): 121–124. |
| [26] | HAN B W, ZAMORE P D. PiRNAs[J]. Curr Biol, 2014, 24(16): R730–R733. DOI: 10.1016/j.cub.2014.07.037 |
| [27] | SASAKI T, SHIOHAMA A, MINOSHIMA S, et al. Identification of eight members of the Argonaute family in the human genome[J]. Genomics, 2003, 82(3): 323–330. DOI: 10.1016/S0888-7543(03)00129-0 |
| [28] | KLATTENHOFF C, THEURKAUF W. Biogenesis and germline functions of piRNAs[J]. Development (Cambridge, England), 2008, 135(1): 3–9. DOI: 10.1530/REP-07-0054 |
| [29] | TÓTH K F, PEZIC D, STUWE E, et al. The piRNA pathway guards the germline genome against transposable elements[C]//Advances in Experimental Medicine and Biology. Dordrecht: Springer, 2016, 886: 51-77. |
| [30] | LE THOMAS, ROGERS A K, WEBSTER A, et al. Piwi induces piRNA-guided transcriptional silencing and establishment of a repressive chromatin state[J]. Genes Dev, 2013, 27(4): 390–399. DOI: 10.1101/gad.209841.112 |
| [31] | DI GIACOMO M, COMAZZETTO S, SAINI H, et al. Multiple epigenetic mechanisms and the piRNA pathway enforce LINE1 silencing during adult spermatogenesis[J]. Mol Cell, 2013, 50(4): 601–608. DOI: 10.1016/j.molcel.2013.04.026 |
| [32] | BUCKLEY B A, BURKHART K B, GU S G, et al. A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality[J]. Nature, 2012, 489(7416): 447–451. DOI: 10.1038/nature11352 |
| [33] | COX D N, CHAO A, LIN H. Piwi encodes a nucleoplasmic factor whose activity modulates the number and division rate of germline stem cells[J]. Development (Cambridge, England), 2000, 127(3): 503–514. |
| [34] | WILCZYNSKA A, MINSHALL N, ARMISEN J, et al. Two Piwi proteins, Xiwi and Xili, are expressed in the Xenopus female germline[J]. RNA, 2009, 15(2): 337–345. DOI: 10.1261/rna.1422509 |
| [35] | HOUWING S, KAMMINGA L M, BEREZIKOV E, et al. A role for Piwi and piRNAs in germ cell maintenance and transposon silencing in Zebrafish[J]. Cell, 2007, 129(1): 69–82. DOI: 10.1016/j.cell.2007.03.026 |
| [36] | AKKOUCHE A, MUGAT B, BARCKMANN B, et al. Piwi is required during Drosophila embryogenesis to license dual-strand piRNA clusters for transposon repression in adult ovaries[J]. Mol Cell, 2017, 66(3): 411–419, e4. DOI: 10.1016/j.molcel.2017.03.017 |
| [37] | KIRINO Y, KIM N, DE PLANELL-SAGUER M, et al. Arginine methylation of Piwi proteins catalysed by dPRMT5 is required for Ago3 and Aub stability[J]. Nat Cell Biol, 2009, 11(5): 652–658. DOI: 10.1038/ncb1872 |
| [38] | SAITO K, SIOMI M C. Small RNA-mediated quiescence of transposable elements in animals[J]. Dev Cell, 2010, 19(5): 687–697. DOI: 10.1016/j.devcel.2010.10.011 |
| [39] | KLATTENHOFF C, XI H L, LI C J, et al. The Drosophila HP1 homolog Rhino is required for transposon silencing and piRNA production by dual-strand clusters[J]. Cell, 2009, 138(6): 1137–1149. DOI: 10.1016/j.cell.2009.07.014 |
| [40] | MALONE C D, BRENNECKE J, DUS M, et al. Specialized piRNA pathways act in germline and somatic tissues of the Drosophila ovary[J]. Cell, 2009, 137(3): 522–535. DOI: 10.1016/j.cell.2009.03.040 |
| [41] | MOHN F, SIENSKI G, HANDLER D, et al. The rhino-deadlock-cutoff complex licenses noncanonical transcription of dual-strand piRNA clusters in Drosophila[J]. Cell, 2014, 157(6): 1364–1379. DOI: 10.1016/j.cell.2014.04.031 |
| [42] | LUO S, LU J. Silencing of transposable elements by piRNAs in Drosophila: an evolutionary perspective[J]. Genomics Proteomics Bioinformatics, 2017, 15(3): 164–176. DOI: 10.1016/j.gpb.2017.01.006 |
| [43] |
傅子文. 果蝇基因Twin在卵巢生殖干细胞系的维持和分化中的作用[D]. 北京: 清华大学, 2015.
FU Z W. Twin gene functions in the maintenance and differentiation of the ovarian germline stem cell lineage in Drosophila[D]. Beijing: Tsinghua University, 2015. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10003-1016712221.htm |
| [44] | LESLIE M. The immune system's compact genomic counterpart[J]. Science, 2013, 339(6115): 25–27. DOI: 10.1126/science.339.6115.25 |
| [45] | GOU L T, DAI P, YANG J H, et al. Pachytene piRNAs instruct massive mRNA elimination during late spermiogenesis[J]. Cell Res, 2014, 24(6): 680–700. DOI: 10.1038/cr.2014.41 |
| [46] | MARIE P P, RONSSERAY S, BOIVIN A. From embryo to adult: piRNA-mediated silencing throughout germline development in Drosophila[J]. G3 (Bethesda), 2017, 7(2): 505–516. DOI: 10.1534/g3.116.037291 |
| [47] | YUAN J, LI M, WEI L, et al. Astrin regulates meiotic spindle organization, spindle pole tethering and cell cycle progression in mouse oocytes[J]. Cell Cycle, 2009, 8(20): 3384–3395. DOI: 10.4161/cc.8.20.9885 |
| [48] | RICO-LEO E M, MORENO-MARIN N, GONZÁLEZ-RICO F J, et al. piRNA-associated proteins and retrotransposons are differentially expressed in murine testis and ovary of aryl hydrocarbon receptor deficient mice[J]. Open Biol, 2016, 6(12): 160–186. |
| [49] | ARMISEN J, GILCHRIST M J, WILCZYNSKA A, et al. Abundant and dynamically expressed miRNAs, piRNAs, and other small RNAs in the vertebrate Xenopus tropicalis[J]. Genome Res, 2009, 19(10): 1766–1775. DOI: 10.1101/gr.093054.109 |
| [50] | XU J, CHEN S Q, ZENG B S, et al. Bombyx mori p-element somatic inhibitor (BmPSI) is a key auxiliary factor for silkworm male sex determination[J]. PLoS Genet, 2017, 13(1): e1006576. DOI: 10.1371/journal.pgen.1006576 |
| [51] | SAKAI H, SUMITANI M, CHIKAMI Y, et al. Transgenic expression of the piRNA-resistant Masculinizer gene induces female-specific lethality and partial female-to-male sex reversal in the silkworm, Bombyx mori[J]. PLoS Genet, 2016, 12(8): e1006203. DOI: 10.1371/journal.pgen.1006203 |
| [52] |
高杰. 家蚕内源性piRNA促进家蚕piggyBac转座子的稳定性[D]. 重庆: 西南大学, 2015.
GAO J. Endogenous piRNA promote the stability of piggy Bac transposon in Bombyx mori[D]. Chongqing: Southwest University, 2015.(in Chinese) |
| [53] | PATIL A A, TATSUKE T, MON H, et al. Characterization of Armitage and Yb containing granules and their relationship to nuage in ovary-derived cultured silkworm cell[J]. Biochem Biophys Res Commun, 2017, 490(2): 134–140. DOI: 10.1016/j.bbrc.2017.06.008 |
| [54] |
谢逸菲. 桔小实蝇piRNA通路相关基因tudor及海藻糖酶基因对卵巢发育影响的研究[D]. 重庆: 西南大学, 2015.
XIE Y F. The influence of piRNA pathway related gene tudor and trehalase gene on ovary development in Bactrocera dorsalis[D]. Chongqing: Southwest University, 2015. (in Chinese) http://d.wanfangdata.com.cn/Thesis/Y2876461 |
| [55] | DAI X Y, SHU Y Q, LOU Q Y, et al. Tdrd12 is essential for germ cell development and maintenance in Zebrafish[J]. Int J Mol Sci, 2017, 18(6): 1127. DOI: 10.3390/ijms18061127 |
| [56] |
陈蓉, 秦玉蓉, 陈国宏, 等. 地方鸡种小分子RNA在性腺中表达形式的初步研究[J]. 畜牧与兽医, 2009, 41(10): 30–33.
CHEN R, QIN Y R, CHEN G H, et al. The preliminry study of micromolecule RNA expression form in indigenous chicken breeds, gonads[J]. Animal Husbandry & Veterinary Medicine, 2009, 41(10): 30–33. (in Chinese) |
| [57] |
周毅. 多倍体鲫鲤Piwi和piRNA的表达与育性的相关性研究[D]. 长沙: 湖南师范大学, 2012.
ZHOU Y. Piwi and piRNA expression and their relationships with fertility of ploidy Cyprinids[D]. Changsha: Hunan Normal University, 2012. (in Chinese) http://www.wenkuxiazai.com/doc/da995b9976eeaeaad1f330ab.html |
| [58] | KOWALCZYKIEWICZ D, PAWLAK P, LECHNIAK D, et al. Altered expression of porcine Piwi genes and piRNA during development[J]. PLoS One, 2012, 7(8): e43816. DOI: 10.1371/journal.pone.0043816 |
| [59] | WILLIAMS Z, MOROZOV P, MIHAILOVIC A, et al. Discovery and characterization of piRNAs in the human fetal ovary[J]. Cell Rep, 2015, 13(4): 854–863. DOI: 10.1016/j.celrep.2015.09.030 |
| [60] | CHENG J, GUO J M, XIAO B X, et al. piRNA, the new non-coding RNA, is aberrantly expressed in human cancer cells[J]. Clin Chim Acta, 2011, 412(17-18): 1621–1625. DOI: 10.1016/j.cca.2011.05.015 |
| [61] | YAN H, WU Q L, SUN C Y, et al. piRNA-823 contributes to tumorigenesis by regulating de novo DNA methylation and angiogenesis in multiple myeloma[J]. Leukemia, 2015, 29(1): 196–206. DOI: 10.1038/leu.2014.135 |



