2. 农业农村部长江下游平原农业环境重点实验室江苏省农业科学院植物保护研究所,南京 210014
2. Key Laboratory of Agro-Environment in Dounstream of Yangze Plain, Ministry of Agriculture and Rural Affaris, Institute of Plant Protection,Jiangsu Academy of Agricultural Sciences,Nanjing 210014 )
植物病害严重威胁农林业生产,全球每年有40%的粮食作物遭受病虫的危害,由此造成的经济损失可达2 200亿美元[1]。长期以来,化学防治在植物病害的防控中担当了重要的角色,然而化学农药的滥用不但造成耐药病原物的产生,增大病害防治的难度,而且污染生态环境、危害人畜健康,严重阻碍农林业的可持续发展。利用环境友好的植物有益微生物(一类能够促进植物生长,防治植物病害的微生物)防治植物病害已成为降低化学农药使用量的重要选项之一[2-4],是可持续农林业的重要组成部分[5-8]。当前,已有大量的有益微生物菌株被用于防控植物病害的科学研究与实践中。相当数量的有益微生物在实验室与温室条件下表现出良好的防病效果,然而,在田间与野外环境中,其作用效果往往受到限制,具体表现为防效不稳定[9-10]。研究结果表明,在寄主植物和土壤中的定殖量不足与抑制病原物生长的效率不高,阻碍了生防微生物有益功能的高效发挥[10-11]。
根际是指能够被植物根系影响到的土壤空间[12],根际土壤中栖居着种类丰富的微生物,根际微生物群落的组装、动态变化及生物学功能与植物的健康密切相关[13-15]。根际生防微生物已被广泛应用于植物病害的防控中。构建与应用由两个或者多个根际微生物菌株组成的具有防治植物病害功能的根际生防菌群已被证明是改善根际生防微生物抑制植物病害的效率与稳定性的有效途径[10-11, 16-17]。相比于单一生防菌株,生防菌群可表现出更佳的防病效果(表 1,图 1),这主要是由于生防菌群防病机制的多样性更高[11, 18],在植物与土壤中的定殖更稳定[18-20],对防病相关基因的调控更强[21],拥有的抑菌谱更广[11, 20]。然而,对生防菌群增强防病效果机制的阐释尚不够深入、系统。现已证明,种间互作是复杂微生物群落形成的重要驱动力[22],生防菌群增强防病功能的效果可能与其中各成员的种间互作相关。本文将综述应用根际生防菌群防治植物病害的现状与根际生防菌群利用微生物的种间互作增强防病效果的潜在机制,并对未来根际生防菌群的研究方向与其构建与应用效率的提升策略进行了展望。

|
| 图 1 根际生防菌群提升植物病害防控效果的作用 |
目前,根际生防菌群已被应用于防控由真菌、卵菌、细菌、病毒以及线虫引起的植物病害(表 1)。根据菌群中成员的构成,可将生防菌群划分为以下3类:细菌生防菌群、细菌与真菌混合生防菌群以及真菌生防菌群。与单一生防菌株类似,生防菌群亦具有多种作用方式:竞争资源与生态位[23-25]、产生抑菌化合物[26-27]、诱导系统抗性以及调控微生物群落等[11, 28-31]。更为重要的是,相比于单一生防菌株,生防菌群可表现出更佳的防病效果(图 1)。Carrión等[32]发现由Chitionophaha sp. 94与黄杆菌(Flavobacterium sp.)98组成的根际菌群抑制立枯丝核菌(Rhizoctonia solani)侵染甜菜根系的效果优于单独使用上述两个菌株的处理。Niu等[33]构建了一个由分别属于寡养单胞菌属(Stenotrophomonas)、苍白杆菌属(Ochrobactrum)、短小杆菌属(Curtobacterium)、肠杆菌属(Enterobacter)、金黄杆菌属(Chryseobacterium)、草螺菌属(Herbaspirillum)以及假单胞菌属(Pseudomonas)的7株玉米根际细菌组成的高度简化且能够代表玉米根系微生物组的模式细菌群落,该群落能够抑制玉米苗枯病的发生,且防病效果优于各组成菌株。上述研究结果表明,应用根际生防菌群防治植物病害,将有助于改善单一生防菌株环境适应性不强、防效不稳定与抑菌谱较窄等不足[16],已成为植物病害生物防治研究的新方向和热点领域。
生防菌群增强防病效果的作用机制可能与以下因素有关:(1)生防菌群的防病机制更加多样[11, 18];(2)各成员占据不同的生态位点,避免了成员间的竞争,这将有助于生防菌群稳定的定殖于寄主植物与土壤[18-20];(3)生防菌群中微生物的种间互作可增强对防病相关基因的调控[21];(4)生防菌群具有更广的抑菌谱[18, 20]。随着对生防菌群研究的不断深入,越来越多的证据表明,种间互作是生防菌群中各成员提升其防病效果的重要策略[22]。生防菌群能够通过复杂的微生物种间互作网络,调控并增强防病关键性状,如在寄主植物和土壤中的定殖与抑制病原物的生长。
2 微生物种间互作促进根际生防微生物定殖高效的定殖于寄主植物与土壤是生防菌株保护寄主的基本前提。定殖量不足可能减弱生防菌株的有益功效,甚至导致病害防控的失败。研究发现,接种生防菌群能够显著改善生防微生物的定殖。Santhanam等[27]发现一个由5株细菌组成的生防菌群在烟草根表定殖的能力显著强于组成菌群的各菌株。Bardas等[34]发现,相比于各单一菌株,由两株假单胞菌(Pseudomonas)组成的具有防治炭疽病效果的菌群,在菜豆根尖的定殖量显著增加。以上结果表明,使用生防菌群能够促进生防微生物的定殖。这种定殖增效作用可能与菌群中微生物种间互作对微生物的生长、生物膜的形成以及运动性等定殖相关生物学过程的调控有关。
2.1 促进根际生防微生物生长快速、旺盛的生长是微生物定殖的关键。组成生防菌群的菌株能够通过互养共栖改善其在寄主生境的生长[35-36]。首先,菌群能够整合多种微生物的代谢能力,以利用无法被单一菌株代谢的基质(图 2)。例如,巴西固氮螺菌(Azospirillum brasilense)不能利用某些碳水化合物作为离体条件下生长所需的碳源,但A. brasilense 能够与某些特异降解上述碳水化合物的细菌形成菌群,此类细菌可将碳水化合物降解为A. brasilense能够利用的碳源,同时,A. brasilense通过固定大气中的氮气,向碳水化合物降解细菌提供氮源[37-38]。因而,上述协同代谢碳水化合物的过程可促进A. brasilense利用植物根系分泌物中的碳水化合物[39-40],提升其在植物根际的存活能力。其次,菌群中的成员能够清除植物生境中抑制微生物生长的物质(图 2)。甲烷氧化菌在植物根际的生长受到甲烷的抑制,而生丝微菌属(Hyphomicrobium spp.)细菌能够清除甲烷[40],且甲烷氧化菌能够通过与之形成菌群而生存于根际。因此,通过互养共栖实现的营养物质高效利用与有害物质清除,能够促进微生物在植物生境的生长。此外,组成菌群的菌株能够通过产生促生长物质促进其它成员的生长(图 2)。Peterson等[41]发现黄杆菌(Flavobacterium johnsoniae)C104与蜡样芽胞杆菌(Bacillus cereus)UW85共同培养5 d后,达到的种群数量比F. johesoniae C104单独培养相同时间后的种群数量高4至30倍,这主要是由于B. cereusUW85产生的肽聚糖能够促进F. johnsoniae C104的生长。
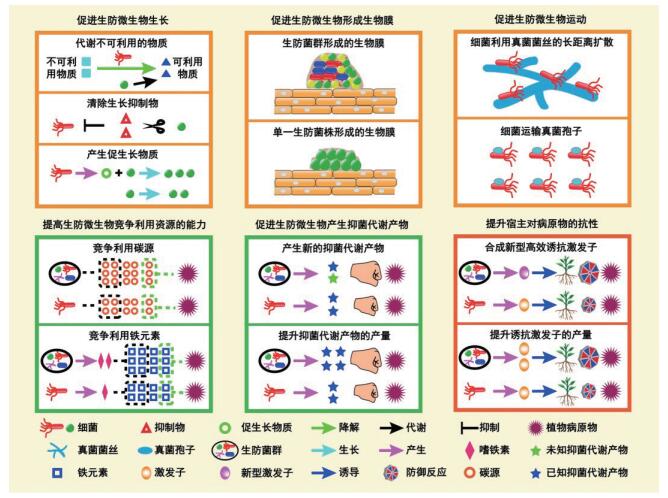
|
| 图 2 根际生防菌群提升植物病害防控效果的潜在机制 |
生物膜是包裹于由胞外多糖、蛋白质以及DNA组成的胞外基质中的,附着于固体或者液体表面的微生物群落[42]。生物膜的形成能够促进微生物在寄主植物与土壤中的定殖[43-44]。在实验室条件与自然状态下,属于不同分类单元的植物共生微生物能够形成多菌株生物膜,且微生物的种间互作能够促进此类生物膜的形成[27](图 2)。前期研究发现,一个由5个细菌菌株组成的生防菌群能够通过形成多菌株生物膜定殖于烟草的根系,且在离体与活体培养条件下,各菌株单独产生的生物膜显著少于菌群形成的生物膜[27]。与之类似,一个由3株分别属于黄单胞菌属(Xanthomonas)、Stenotrophomonas与微杆菌属(Microbacterium)的菌株组成的生防菌群,相比于各单一菌株,能够形成更多的生物膜[45]。Burm lle等[46]发现,将叶状微杆菌(Microbacterium phyllosphaerae)、日本乳杆菌(Shewanella japonica)、Dokdonia donghaensis和鲁氏不动杆菌(Acinetobacter lwoffii)混合培养24 h后形成的生物膜的量,比将上述菌株单独培养相同的时间后形成的生物膜的量增加了67%。目前,菌群提升生物膜产量的机制尚不明确,推测可能是由于其中的微生物种间互作进一步激活了与胞外基质积累相关的代谢途径与细胞间信号转导通路[27]。
2.3 促进根际生防微生物运动运动性是指在消耗能量的情况下,微生物移动或者做机械功[47],是关键的定殖相关生物学性状之一,包括swimming、swarming、gliding、twitching、sliding与darting[47]6种类型。Allard-Massicotte等[48]的研究结果表明,生防菌株在寄主植物上的早期定殖需要运动性参与。生防菌群中成员的运动能力可通过其中的微生物种间互作而增强。例如,真菌菌丝能够作为载体在寄主植物与土壤中分散细菌,被称为“真菌高速公路”[49-50](图 2)。Wick等[51]研究发现沙雷氏菌(Serratia)可以在毛霉菌(Mucor)与耐碱酵母菌(Galactomyces)形成的真菌菌丝网络上迅速扩散,相同时间内,其运动距离比无真菌菌丝网络时分别增加73%与79%。最近,Zhang等[52]发现根瘤菌可将枫香拟茎点霉(Phomopsis liquidambaris)作为网络运动至大豆根际,促进根瘤的形成。由菌根真菌福摩萨球囊菌(Glomus formosanum)CNPAB020形成的根外菌丝能够促进高效固氮慢生根瘤菌(Bradyrhizobium diazoefficiens)USDA 110在植物根际的转移[53]。细菌亦能够促进真菌孢子的扩散(图 2),例如,Paenibacillus vortex可将植物共生真菌烟曲霉(Aspergillus fumigatus)的分生孢子,从不利的生长环境中运出,同时,真菌菌丝可以作为P. vortex跨越空气间隙的桥梁[54]。此外,细菌之间也可通过种间互作增强运动能力。对氨苄青霉素敏感的P. vortex菌株能够通过携带具有氨苄青霉素抗性的大肠杆菌(Escherichia coli)共同swarming,定殖于含有氨苄青霉素的平板上,使两种细菌同时得到扩散[56-56]。因此,生防菌群中的微生物种间互作,可以促进生防菌株的迁移,进而提升其在寄主植物与土壤中的定殖效率。
综上,生防菌群中微生物的种间互作能够通过促进生长、生物膜形成与运动,有效提升生防菌株在寄主植物与土壤中的定殖能力(图 2)。因此,应用具有活跃微生物种间互作的生防菌群,能够改善防病微生物的存活与其对复杂且多变环境条件的适应,使得抑制植物病原物的有益作用更加稳定。
3 微生物种间互作增强根际生防微生物对病原物生长的抑制生防菌群抑制植物病原物生长的效果强于单一生防菌株(表 1,图 1)。如一个由两株枯草芽胞杆菌(Bacillus subtilis)S2BC-1与GIBC-Jamog组成的生防菌群比各单一菌株对番茄维管枯萎病病原菌,尖孢镰刀菌(Fusarium oxysporum f.sp. lycopersici)的抑制活性更高[29]。此外,荧光假单胞菌(Pseudomonas fluorescens)T5在离体条件下不能抑制立枯丝核菌(Rhizoctonia solani)的生长,然而,当该菌株与另外4株分离自酸豆(Tamarindus)的非拮抗细菌混合施用时,这个由5个菌株组成的菌群能够显著抑制R. solani的生长[57]。推测生防菌群中成员间的互作,可增强生防微生物竞争资源的能力,进一步激活抑菌物质的合成,增强其抑菌效果(图 2)。
3.1 提高根际生防微生物竞争利用资源的能力资源竞争是指有益微生物通过快速、高效的利用寄主植物附近有限的资源,限制或抑制病原物的生长[25],是生防微生物保护植物不受病原物侵染的一种重要策略。根际生防菌群中微生物的种间互作能够增强生防菌株竞争根系分泌物中营养物质的能力,例如,两个分别由8个Pseudomonas菌株与5个无毒Ralstonia菌株组成,能够防治R.solanacearum引起的番茄青枯病的生防菌群,对R. solanacearum种群密度的抑制效果显著强于菌群中的各单一菌株,这种增强的抑菌作用可归结于生防菌群与R. solanacearum之间更为显著的生态位重叠,主要体现在,生防菌群与R. solanacearum对番茄根系分泌物中48种碳源的代谢能力具有更高的相似度[24-25]。Irikiin等[58]研究发现,多样性高的生防菌群可利用碳源的种类更多,更有利于菌群中的生防微生物与病原物竞争利用碳源,从而抑制病原物的生长与病害的发生。此外,防病微生物与病原物能够竞争利用植物生境中有限的铁元素[59](图 2)。微生物对环境中铁元素的吸收一般是通过分泌结构多样、对铁元素具有高亲和度[60]的嗜铁素实现的。土壤微生物的种间互作能够调控嗜铁素的产生,如天蓝链霉菌(Streptomyces coelicolor)与另外5株土壤放线菌之间的互作可提升该放线菌群落产生嗜铁素的多样性,S. coelicolor中去铁胺的合成[61]能够被其它菌株产生的嗜铁素进一步激活。综上,根际生防菌群中微生物的种间互作能够增强生防微生物在植物生境中与病原物竞争利用资源的能力(图 2)。
3.2 促进根际生防微生物产生抑菌代谢产物微生物能够通过合成具有抑菌活性的代谢产物直接抑制或者杀死环境中包括植物病原物在内的竞争者[60, 62]。目前,相关研究主要聚焦于属于Bacillus、Pseudomonas与Trichoderma的生防微生物菌株,这些菌株能够产生脂肽、聚酮、细菌素、吩嗪、2,4-二乙酰基间苯三酚与几丁质酶等抑菌物质[62, 64]。研究发现,某些抑菌代谢产物在其来源菌株纯培养物中的浓度较低,或者其编码基因在纯培养过程中不表达,但将产生上述抑菌代谢产物的菌株与其它菌株混合培养后,抑菌代谢产物的产量与其编码基因的表达水平均显著提升[65, 67](图 2)。Lutz等[21]发现,P. fluorescens产生的2,4-二乙酰基间苯三酚促进了深绿木霉(Trichoderma atroviride)P1中几丁质酶基因nag1的表达,该结果表明将生防菌株混合可上调防病相关基因的表达。共培养内生真菌三隔镰刀菌(Fusarium tricinctum)与B.subtilis,可以将包括抑菌化合物在内的次生代谢产物的积累量提升78倍[68]。特异的微生物种间互作,不但能够促进已知抑菌化合物的产生,也可激活未知抑菌代谢产物的生物合成(图 2)。例如,新型抗生素amycomicin的产生依赖于两株土壤放线菌,拟无枝菌酸菌(Amycolatopsis sp.) AA4与S. coelicolor M145的种间互作, 菌株AA4是该抗生素的产生菌株,M145为诱导菌株[65]。由此可见,微生物的种间互作能够激活或者促进抑菌化合物的生物合成(图 2)。
综上所述,微生物的种间互作能够通过调控生防菌株竞争资源与产生抑菌化合物的能力,增强其抑制病原物与防控植物病害的效果(图 2)。因而,拥有活跃的微生物种间互作的根际生防菌群,能够增强其中防病菌株竞争必需资源和产生针对特定病原物的毒性化合物的能力,实现对植物病害更加高效的防治。
4 增强寄主对病原物的抗性诱导系统抗性(Induced systemic resistance, ISR)是指利用非病原物、共生微生物或某些化合物诱导寄主植物产生对病原物的抗性[69]。增强寄主植物对病原物的防御反应是生防菌群提升病害防控效果的重要途径之一(图 2)。Shanmugam等[29]研究发现,由B. subtilis GIBC-Jamog与B. subtilis S2BC-1组成的生防菌群处理番茄根组织后,其中的几丁质酶含量比各单一菌株处理分别提高了2.2倍与2.6倍, β-1,3-葡聚糖酶的含量分别提高了1.4倍与1.1倍。生防菌群可能通过激活多条不同的代谢途径与信号通路,增强寄主植物对病原物的ISR [11, 70-71]然而其中具体的作用机制仍有待深入阐释。生防菌群中的微生物种间互作可能促进了特异性诱导抗性激发子的产生或者新型高效诱抗激发子的合成,由此增强寄主植物对病原物的ISR(图 2)。
5 总结与展望综上所述,应用根际生防菌群是-种改善根际生防菌株防病功能的有效手段,已被成功应用于由真菌、细菌、病毒以及线虫引起的植物病害的防控中,并取得了良好的经济与生态效益,为进一步推动农林业的可持续发展提供了有力的技术保障。本文综述了应用根际生防菌群防治植物病害的现状,系统的总结并阐述了根际生防菌群可能利用其成员间的微生物种间互作,促进生防微生物在寄主植物生境的生长、生物膜形成与运动能力,增强生防微生物对生存资源的竞争与抑菌化合物的合成,同时,促进特异性诱导抗性激发子的产生或者新型高效诱抗激发子的合成,从而提升了根际生防微生物在寄主植物和土壤中的定殖能力与抑制病原物生长的效率,增强了寄主植物对病原物的免疫反应,实现根际生防微生物防病功能的强化。以上研究结果与理论知识,将为建立根际微生物种间互作模型,揭示根际生防菌群利用微生物种间互作增强防病效果的机制, 以,及开发与高效应用根际生防菌群产品提供重要的理论依据。基于对现有研究结果和理论知识的理解与分析,今后的根际生防菌群研究与应用中需重点关注以下几点。
5.1 进一步深入根际生防菌群的增效机制研究本文系统的阐述了根际生防菌群增强生防菌株防病效果的潜在作用机制,在此基础上,今后可通.过验证本文中提及的作用机制,对生防菌群增强防病效果的机理,特别是分子机理进行深入研究。应特别关注植物体多菌株生物膜形成的分子机制、生防菌群中多源复合途径抑菌代谢产物合成的分子机.制以及生防菌群增强寄主植物对病原物抗性的分子机制等科学问题。通过开展相关研究,建立生防菌群生物膜形成、运动性以及抑菌代谢产物的原位检测与分析体系,为最终揭示根际生防菌群增强防病效果的机制奠定方法学基础。此外,对于生防菌群增效机制的研究也将进一步深化我们对微生物种间互作机理的理解,拓宽我们对植物共生微生物群落的组装、群落中各成员的进化对植物共生体影响的认识[22]。
5.2 简化根际微生物组构建生防菌群长期以来,经验化的通过评价菌株间的相互兼容性(各成员之间互不抑制生长)与其多样性[20, 24-25, 72-73]将已有的根际防病微生物菌株混合, 是构建根际生防菌群的主要策略。然而,在某些情况下,由于菌株来源位点与病害发生位点各种因子的差异,将已有的防病微生物菌株混合施用后,并不能发挥出预期的防病效果; 直接分离发病位点附近的微生物,并从中筛选获得防病微生物菌株,需投入大量时间、物料与人力,极大的增加构建根际生防菌群的成本。近年来,随着微生物组学的快速发展与对植物微生物组功能认知的不断深人,通过简化根际微生物组构建生防菌群的策略已愈发受到重视[32-33,45, 74]该策略以寄主介导的植物微生物组选择为理论指导,通过采用宏基因组学技术对具有防病功能的微生物组进行解析,同时借助微生物分离培养技术,尽可能全面的捕捉微生物组中的成员,并基于对成员间互作关系与生物学功能的分析结果,确定有限数量的生防菌群候选菌株,混合制备多菌株微生物群落,检测其防病效果,最终将生防微生物组简化为保留了其中关键防病相关互作关系,由有限数量菌株组成,具有防治植物病害功能的菌群。该策略不但能够准确与快速的锁定候选菌株,简化生防菌群的构建过程,而且允许根据具体的防控需求,定制生防菌群,推动植物病害的精准防控。因此,未来获取生防菌群时,可将通过简化根际微生物组构建生防菌群的策略推广至更大范围。
5.3 根际生防菌群与单一生防微生物菌株搭配施用虽然根际生防菌群比单一生防菌株防治植物病害的效果更佳(图 1),但是在当前的病害绿色防控需求下,生防菌群并不能完全取代单一生防菌株,主要原因如下:(1)生防菌群是否具有强化防病功能的作用主要取决于其中各成员之间的兼容性[11, 75],获得多个兼容候选菌株的投入远高于获取单一生防菌株的成本,使得生防菌群产品的研发成本更高;(2)生防菌群产品的生产涉及多菌种混合发酵,其生产工艺的难度与复杂程度大于单一生防菌株制剂产品,增加了生防菌群产品的生产成本;(3)生防菌群产品的登记、审批较单一生防菌株产品复杂,且处理费用更高。因此,在植物病害绿色防控压力较大的现状下,将根际生防菌群与单一生防微生物菌株搭配施用,便于在短时间内实现生防制剂的大面积覆盖,同时将防控成本限制在可接受的范围内,有助于推动农林业的可持续发展。
| [1] |
联合国粮食及农业组织.粮农组织启动联合国“2020 国际植物健康年”. http://www.fao.org/news/story/zh/item/12536202/icode/. Food and Agriculture Organization of the United Nations. FAOlaunches 2020 as the UN's International Year of Plant Health.http://www.fao.org/news/story/zh/item/12536202/icode/. |
| [2] |
Velivelli SLS, Vos PD, Kromann P, et al. Biological control agents : from field to market, problems, and challenges[J]. Trends in Biotechnology, 2014, 32(10): 493-496. DOI:10.1016/j.tibtech.2014.07.002 |
| [3] |
Rahman SFSA, Singh E, Pieterse CMJ, et al. Emerging microbial biocontrol strategies for plant pathogens[J]. Plant Sci, 2018, 267: 102-111. DOI:10.1016/j.plantsci.2017.11.012 |
| [4] |
Fira D, Dimkic I, Beric T, et al. Biological control of plant pathogens by Bacillus species[J]. J Biotechnol, 2018, 285: 44-55. DOI:10.1016/j.jbiotec.2018.07.044 |
| [5] |
Alvarez B, Biosca EG. Bacteriophage-based bacterial wilt biocontrol for an environmentally sustainable agriculture[J]. Front Plant Sci, 2017, 8: 1218. DOI:10.3389/fpls.2017.01218 |
| [6] |
Busby PE, Soman C, Wagner MR, et al. Research priorities for harnessing plant microbiomes in sustainable agriculture[J]. PLoS Biol, 2017, 15(3:e2001793). |
| [7] |
Singh RL, Mondal S. Biotechnology for sustainable agriculture[M]. Sawston Cambridge : Woodhead Publishing, 2018.
|
| [8] |
Carzorla FM, Mercado-Blanco J. Biological control of tree and woody plant diseases :an impossible task?[J]. BioControl, 2016, 61: 233-242. DOI:10.1007/s10526-016-9737-0 |
| [9] |
Xu XM, Jeffries P, Pautasso M, et al. Combined use of biocontrol agents to manage plant diseases in theory and practice[J]. Phytopathology, 2011, 101(9): 1024-1031. DOI:10.1094/PHYTO-08-10-0216 |
| [10] |
Mazzola M, Freilich S. Prospects for biological soilborne disease control :application of indigenous versus synthetic microbiomes[J]. Phytopathology, 2017, 107(3): 256-263. DOI:10.1094/PHYTO-09-16-0330-RVW |
| [11] |
Sarma BK, Yadav SK, Singh S, et al. Microbial consortium-mediated plant defense against phytopathogens :readdressing for enhancing efficacy[J]. Soil Biol Biochem, 2015, 87: 25-33. DOI:10.1016/j.soilbio.2015.04.001 |
| [12] |
Lugtenberg B, Kamilova F. Plant-growth-promoting rhizobacteria[J]. Annu Rev Microbiol, 2009, 63: 541-556. DOI:10.1146/annurev.micro.62.081307.162918 |
| [13] |
Qu Q, Zhang Z, Peijnenburg WJGM, et al. Rhizosphere microbiome assembly and its impact on plant growth[J]. J Agric Food Chem, 2020, 68(18): 5024-5038. DOI:10.1021/acs.jafc.0c00073 |
| [14] |
Garcia J, Kao-Kniffin J. Microbial group dynamics in plant rhizospheres and their implications on nutrient cycling[J]. Front Microbiol, 2018, 9: 1516. DOI:10.3389/fmicb.2018.01516 |
| [15] |
Compant S, Samad A, Faist H, et al. A review on the plant microbiome :Ecology, functions, and emerging trends in microbial application[J]. J Adv Res, 2019, 19: 29-37. DOI:10.1016/j.jare.2019.03.004 |
| [16] |
Vorholt JA, Vogel C, Carlstrom CI, et al. Establishing causality:opportunities of synthetic communities for plant microbiome research[J]. Cell Host Microbe, 2017, 22(2): 142-155. DOI:10.1016/j.chom.2017.07.004 |
| [17] |
Woo SL, Pepe O. Microbial consortia :promising probiotics as plant biostimulants for sustainable agriculture[J]. Front Plant Sci, 2018, 9: 1801. DOI:10.3389/fpls.2018.01801 |
| [18] |
Pierson EA, Weller DM. Use of mixtures of fluorescent pseudomonads to suppress take-all and improve the growth of wheat[J]. Phytopathology, 1994, 84: 940-947. DOI:10.1094/Phyto-84-940 |
| [19] |
Pliego C, de Weert S, Lamers G, et al. Two similar enhanced root-colonizing Pseudomonas strains differ largely in their colonization strategies of avocado roots and Rosellinia necatrix hyphae[J]. Environ Microbiol, 2008, 10(12): 3295-3304. DOI:10.1111/j.1462-2920.2008.01721.x |
| [20] |
Thomloudi EE, Tsalgatidou PC, Douka D, et al. Multistrain versus single-strain plant growth promoting microbial inoculants - The compatibility issue[J]. Hell Plant Prot J, 2019, 12(2): 61-77. |
| [21] |
Lutz MP, Wenger S, Maurhofer M, et al. Signaling between bacterial and fungal biocontrol agents in a strain mixture[J]. FEMS Microbiol Ecol, 2004, 48(3): 447-455. DOI:10.1016/j.femsec.2004.03.002 |
| [22] |
Hassani MA, Duran P, Hacquard S. Microbial interactions within the plant holobiont[J]. Microbiome, 2018, 6(1): 58. DOI:10.1186/s40168-018-0445-0 |
| [23] |
McKellar ME, Nelson EB. Compost-induced suppression of Pythium damping-off is mediated by fatty-acid-metabolizing seed-colonizing microbial communities[J]. Appl Environ Microbiol, 2003, 69(1): 452-460. |
| [24] |
Hu J, Wei Z, Friman VP, et al. Probiotic diversity enhances rhizosphere microbiome function and plant disease suppression[J]. mBio, 2016, 7(6): e01790-16. DOI:10.1111/ele.12567 |
| [25] |
Wei Z, Yang T, Friman VP, et al. Trophic network architecture of root-associated bacterial communities determines pathogen invasion and plant health[J]. Nat Commun, 2015, 6: 8413. DOI:10.1038/ncomms9413 |
| [26] |
Thakkar A, Saraf M. Development of microbial consortia as a biocontrol agent for effective management of fungal diseases in Glycine maxL[J]. Arch Phytopathol Plant Prot, 2014, 48(6): 459-474. DOI:10.1080/03235408.2014.893638 |
| [27] |
Santhanam R, Menezes RC, Grabe V, et al. A suite of complementary biocontrol traits allows a native consortium of root-associated bacteria to protect their host plant from a fungal sudden-wilt disease[J]. Mol Ecol, 2019, 28(5): 1154-1169. DOI:10.1111/mec.15012 |
| [28] |
Solanki MK, Yandigeri MS, Kumar S, et al. Co-inoculation of different antagonists can enhance the biocontrol activity against Rhizoctonia solani in tomato[J]. Antonie Van Leeuwenhoek, 2019, 112(11): 1633-1644. DOI:10.1007/s10482-019-01290-8 |
| [29] |
Shanmugam V, Kanoujia N. Biological management of vascular wilt of tomato caused by Fusarium oxysporum f. sp. lycospersici by plant growth-promoting rhizobacterial mixture[J]. Biol Control, 2011, 57(2): 85-93. DOI:10.1016/j.biocontrol.2011.02.001 |
| [30] |
Georg S, Raupach K. Mixtures of plant growth-promoting rhizobacteria enhance biological control of multiple cucumber pathogens[J]. Phytopathology, 1998, 88(11): 1159-1164. DOI:10.1094/PHYTO.1998.88.11.1158 |
| [31] |
Zhang LN, Wang DC, et al. Consortium of plant growth-promoting rhizobacteria strains suppresses sweet pepper disease by altering the rhizosphere microbiota[J]. Front Microbiol, 2019, 10: 1668. DOI:10.3389/fmicb.2019.01668 |
| [32] |
Carrion VJ, Perez-Jaramillo J, Cordovez V, et al. Pathogen-induced activation of disease-suppressive functions in the endophytic root microbiome[J]. Science, 2019, 366(6465): 606-612. DOI:10.1126/science.aaw9285 |
| [33] |
Niu B, Paulson JN, Zheng X, et al. Simplified and representative bacterial community of maize roots[J]. Proc Natl Acad Sci USA, 2017, 114(12): E2450-E2459. DOI:10.1073/pnas.1616148114 |
| [34] |
Bardas GA, Lagopodi AL, Kadoglidou K, et al. Biological control of three Colletotrichum lindemuthianum races using Pseudomonas chlororaphis PCL1391 and Pseudomonas fluorescens WCS365[J]. Biol Control, 2009, 49(2): 139-145. DOI:10.1016/j.biocontrol.2009.01.012 |
| [35] |
Morris BE, Henneberger R, Huber H, et al. Microbial syntrophy : interaction for the common good[J]. FEMS Microbiol Rev, 2013, 37(3): 384-406. DOI:10.1111/1574-6976.12019 |
| [36] |
Mee MT, Collins JJ, Church GM, et al. Syntrophic exchange in synthetic microbial communities[J]. Proc Natl Acad Sci USA, 2014, 111(20): E2149-E2156. DOI:10.1073/pnas.1405641111 |
| [37] |
Bashan Y, Holguin G. Azospirillum-plant relationships : environmental and physiological advances[J]. Can J Microbiol, 1997, 43(2): 103-121. |
| [38] |
Bashan Y. Inoculants of plant growth-promoting bacteria for use in agriculture[J]. Biotechnol Adv, 1998, 16(4): 729-770. DOI:10.1016/S0734-9750(98)00003-2 |
| [39] |
Bais HP, Weir TL, Perry LG, et al. The role of root exudates in rhizosphere interactions with plants and other organisms[J]. Annu Rev Plant Biol, 2006, 57: 233-266. DOI:10.1146/annurev.arplant.57.032905.105159 |
| [40] |
Huang XF, Chaparro JM, Reardon KF, et al. Rhizosphere interactions :rootexudates , microbes , andmicrobial communities[J]. Botany, 2014, 92(4): 267-275. DOI:10.1139/cjb-2013-0225 |
| [41] |
Peterson SB, Dunn AK, Klimowicz AK, et al. Peptidoglycan from Bacillus cereus mediates commensalism with rhizosphere bacteria from the Cytophaga-Flavobacterium group[J]. Appl Environ Microbiol, 2006, 72(8): 5421-5427. DOI:10.1128/AEM.02928-05 |
| [42] |
Vlamakis H, Chai Y, Beauregard P, et al. Sticking together : building a biofilm the Bacillus subtilis way[J]. Nat Rev Microbiol, 2013, 11(3): 157-168. DOI:10.1038/nrmicro2960 |
| [43] |
Fan B, Chen XH, Budiharjo A, et al. Efficient colonization of plant roots by the plant growth promoting bacterium Bacillus amyloliquefaciens FZB42, engineered to express green fluorescent protein[J]. J Biotechnol, 2011, 151(4): 303-311. DOI:10.1016/j.jbiotec.2010.12.022 |
| [44] |
Beauregard PB, Chai Y, Vlamakis H, et al. Bacillus subtilis biofilm induction by plant polysaccharides[J]. Proc Natl Acad Sci USA, 2013, 110(17): E1621-1630. DOI:10.1073/pnas.1218984110 |
| [45] |
Berendsen RL, Vismans G, Yu K, et al. Disease-induced assemblage of a plant-beneficial bacterial consortium[J]. ISME J, 2018, 12(6): 1496-1507. DOI:10.1038/s41396-018-0093-1 |
| [46] |
Burmolle M, Webb JS, et al. Enhanced biofilm formation and incre-ased resistance to antimicrobial agents and bacterial invasion are caused by synergistic interactions in multispecies biofilms[J]. Appl Environ Microbiol, 2006, 72(6): 3916-3923. DOI:10.1128/AEM.03022-05 |
| [47] |
Harshey RM. Bacterial motility on a surface :many ways to a common goal[J]. Annu Rev Microbiol, 2003, 57: 249-273. DOI:10.1146/annurev.micro.57.030502.091014 |
| [48] |
Allard-Massicotte R, Tessier L, Lecuyer F, et al. Bacillus subtilis early colonization of Arabidopsis thaliana roots involves multiple chemotaxis receptors[J]. mBio, 2016, 7(6). DOI:10.1093/jac/41.suppl_3.13 |
| [49] |
Warmink JA, Nazir R, Corten B, et al. Hitchhikers on the fungal highway :The helper effect for bacterial migration via fungal hyphae[J]. Soil Biol Biochem, 2011, 43(4): 760-765. DOI:10.1016/j.soilbio.2010.12.009 |
| [50] |
Kohlmeier S, Smits THM, Ford RM, et al. Taking the fungal highway :mobilization of pollutant-degrading bacteria by fungi[J]. Environ Sci Technol, 2005, 39(12): 4640-4646. DOI:10.1021/es047979z |
| [51] |
Wick L, Remer R, Wurz B, et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil[J]. Environ Sci Technol, 2007, 41: 500-505. DOI:10.1021/es061407s |
| [52] |
Zhang W, Li XG, Sun K, et al. Mycelial network-mediated rhizobial dispersal enhances legume nodulation[J]. ISME J, 2020, 14(4): 1015-1029. DOI:10.1038/s41396-020-0587-5 |
| [53] |
de Novais CB, Sbrana C, da Conceicao Jesus E, et al. Mycorrhizal networks facilitate the colonization of legume roots by a symbiotic nitrogen-fixing bacterium[J]. Mycorrhiza, 2020, 30: 389-396. DOI:10.1007/s00572-020-00948-w |
| [54] |
Ingham CJ, Kalisman O, Finkelshtein A, et al. Mutually facilitated dispersal between the nonmotile fungus Aspergillus fumigatus and the swarming bacterium Paenibacillus vortex[J]. Proc Natl Acad Sci USA, 2011, 108(49): 19731-19736. DOI:10.1073/pnas.1102097108 |
| [55] |
Finkelshtein A, Roth D, Ben Jacob E, et al. Bacterial swarms recruit cargo bacteria to pave the way in toxic environments[J]. mBio, 2015, 6(3): e00074-15. DOI:10.1128/mBio.00074-15 |
| [56] |
Venieraki A, Tsalgatidou PC, et al. Swarming motility in plant-associated bacteria[J]. Hell Plant Prot J, 2016, 9(1): 16-27. DOI:10.1515/hppj-2016-0002 |
| [57] |
Kannan V, Sureendar R. Synergistic effect of beneficial rhizosphere microflora in biocontrol and plant growth promotion[J]. J Basic Microbiol, 2009, 49(2): 158-164. DOI:10.1002/jobm.200800011 |
| [58] |
Irikiin Y, Nishiyama M, Otsuka S, et al. Rhizobacterial community-level, sole carbon source utilization pattern affects the delay in the bacterial wilt of tomato grown in rhizobacterial community model system[J]. Applied Soil Ecology, 2006, 34(1): 27-32. DOI:10.1016/j.apsoil.2005.12.003 |
| [59] |
Gu S, Wei Z, Shao Z, et al. Competition for iron drives phytopathogen control by natural rhizosphere microbiomes[J]. Nat Microbiol, 2020, 5(8): 1002-1010. DOI:10.1038/s41564-020-0719-8 |
| [60] |
Traxler MF, Kolter R. Natural products in soil microbe interactions and evolution[J]. Nat Prod Rep, 2015, 32(7): 956-970. DOI:10.1039/C5NP00013K |
| [61] |
Traxler MF, Watrous JD, Alexandrov T, et al. Interspecies interactions stimulate diversification of the Streptomyces coelicolor secreted metabolome[J]. mBio, 2013, 4(4): e00459-13. DOI:10.1128/mBio.00459-13 |
| [62] |
Chen XH, Koumoutsi A, Scholz R, et al. Comparative analysis of the complete genome sequence of the plant growth-promoting bacterium Bacillus amyloliquefaciens FZB42[J]. Nat Biotechnol, 2007, 25(9): 1007-1014. DOI:10.1038/nbt1325 |
| [63] |
Haas D, Keel C. Regulation of antibiotic production in root-colonizing Peudomonas spp. and relevance for biological control of plant disease[J]. Annu Rev Phytopathol, 2003, 41: 117-153. DOI:10.1146/annurev.phyto.41.052002.095656 |
| [64] |
Ghishalberti EL, Sivasithamparam, K. Antifungal antibiotics produced by Trichoderma spp.[J]. Soil Biol Biochem, 1991, 23(11): 1011-1020. DOI:10.1016/0038-0717(91)90036-J |
| [65] |
Pishchany G, Mevers E, et al. Amycomicin is a potent and specific antibiotic discovered with a targeted interaction screen[J]. Proc Natl Acad Sci USA, 2018, 115(40): 10124-10129. DOI:10.1073/pnas.1807613115 |
| [66] |
Nutzmann HW, Reyes-Dominguez Y, Scherlach K, et al. Bacteria-induced natural product formation in the fungus Aspergillus nidulans requires Saga/Ada-mediated histone acetylation[J]. Proc Natl Acad Sci USA, 2011, 108(34): 14282-14287. DOI:10.1073/pnas.1103523108 |
| [67] |
Brakhage AA. Regulation of fungal secondary metabolism[J]. Nat Rev Microbiol, 2013, 11(1): 21-32. DOI:10.1038/nrmicro2916 |
| [68] |
Ola AR, Thomy D, Lai D, et al. Inducing secondary metabolite production by the endophytic fungus Fusarium tricinctum through coculture with Bacillus subtilis[J]. J Nat Prod, 2013, 76(11): 2094-2099. DOI:10.1021/np400589h |
| [69] |
Kloepper JW, Tuzun S, Kuć JA. Proposed definitions related to induced disease resistance[J]. Biocontrol Sci Tech, 1992, 2(4): 349-351. DOI:10.1080/09583159209355251 |
| [70] |
Jain A, Singh S, Kumar Sarma B, et al. Microbial consortium-mediated reprogramming of defence network in pea to enhance tolerance against Sclerotinia sclerotiorum[J]. J Appl Microbiol, 2012, 112(3): 537-550. DOI:10.1111/j.1365-2672.2011.05220.x |
| [71] |
Alizadeh H, Behboudi K, Ahmadzadeh M, et al. Induced systemic resistance in cucumber and Arabidopsis thaliana by the combination of Trichoderma harzianum Tr6 and Pseudomonas sp. Ps14[J]. Biol Control, 2013, 65(1): 14-23. DOI:10.1016/j.biocontrol.2013.01.009 |
| [72] |
Liu K, McInroy JA, Hu CH, et al. Mixtures of plant-growth-promoting rhizobacteria enhance biological control of multiple plant diseases and plant-growth promotion in the presence of pathogens[J]. Plant Dis, 2018, 102(1): 67-72. DOI:10.1094/PDIS-04-17-0478-RE |
| [73] |
Nicholas A. Lyons RK.. Bacillus subtilis protects public goods by extending kin discrimination to closely related species[J]. mBio, 2017, 8(4): e00723-17. DOI:10.1128/mBio.00723-17 |
| [74] |
Liu YX, Qin Y, Bai Y. Reductionist synthetic community approaches in root microbiome research[J]. Curr Opin Microbiol, 2019, 49: 97-102. DOI:10.1016/j.mib.2019.10.010 |
| [75] |
Haas D, Defago G. Biological control of soil-borne pathogens by fluorescent pseudomonads[J]. Nat Rev Microbiol, 2005, 3(4): 307-319. DOI:10.1038/nrmicro1129 |
| [76] |
Jain A, Singh A, Singh S, et al. Biological management of Sclerotinia sclerotiorum in pea using plant growth promoting microbial consortium[J]. J Basic Microbiol, 2015, 54: 1-12. DOI:10.1002/jobm.201400628 |
| [77] |
Jetiyanon K, Kloepper JW. Mixtures of plant growth-promoting rhizobacteria for induction of systemic resistance against multiple plant diseases[J]. Biol Control, 2002, 24(3): 285-291. DOI:10.1016/S1049-9644(02)00022-1 |
| [78] |
Srivastava R, Khalid A, Singh US, et al. Evaluation of arbuscular mycorrhizal fungus, fluorescent Pseudomonas and Trichoderma harzianum formulation against Fusarium oxysporum f. sp. lycopersici for the management of tomato wilt[J]. Biological Control, 2010, 53(1): 24-31. DOI:10.1016/j.biocontrol.2009.11.012 |
| [79] |
Zhou DM, Feng H, Schuelke T, et al. Rhizosphere microbiomes from root knot nematode non-infested plants suppress nematode infection[J]. Microb Ecol, 2019, 78(2): 470-481. |
| [80] |
Domenech J, Reddy MS, et al. Combined application of the biolo-gical product LS213 with Bacillus, Pseudomonas or Chryseobacte-rium for growth promotion and biological control of soil-borne dise-ases in pepper and tomato[J]. BioControl, 2006, 51: 245-258. DOI:10.1007/s10526-005-2940-z |
| [81] |
Chemeltorit PP, Mutaqin KH, Widodo W. Combining Trichoderma hamatum THSW13 and Pseudomonas aeruginosa BJ10-86 :a synergistic chili pepper seed treatment for Phytophthora capsici infested soil[J]. Eur J Plant Pathol, 2017, 147: 157-166. DOI:10.1007/s10658-016-0988-5 |
| [82] |
Raupach GS, Kloepper JW. Mixtures of plant growth-promoting rhizobacteria enhance biological control of multiple cucumber pathogens[J]. Phytopathology, 1998, 88(11): 1159-1164. DOI:10.1094/PHYTO.1998.88.11.1158 |
| [83] |
Yang W, Zheng L, Liu HX, et al. Evaluation of the effectiveness of a consortium of three plant-growth promoting rhizobacteria for biocontrol of cotton Verticillium wilt[J]. Biocontrol Sci Technol, 2014, 24(5): 489-502. DOI:10.1080/09583157.2013.873389 |
| [84] |
Thangavelu R, Gopi M. Combined application of native Trichoderma isolates possessing multiple functions for the control of Fusarium wilt disease in banana cv. Grand Naine[J]. Biocontrol Sci Technol, 2015, 25(10): 1147-1164. DOI:10.1080/09583157.2015.1036727 |





