合成生物学是将现代工程学原理和方法应用到生物学的一种新的研究领域[1-2]。合成生物学通过采用自下而上的研究方法实现了人工生物体系的建立[3-5],已经应用于药物合成[6-9]和个性化医疗[10-15]等多个领域。DNA组装是合成生物学的重要基础技术,是构建基因表达系统乃至整个染色体的关键环节[16-17]。传统的基于限制性消化和连接的克隆方法较为冗长耗时,而且不适合高通量DNA组装和多个DNA片段的同时组装,无法进行需要更高效率和保真度的复杂DNA结构设计[18-19]。近年来,为了适应快速发展的基因组设计,已经开发大量新的高效率、高保真度并且模块化的DNA组装方法[20-22]。
Golden Gate是一种利用ⅡS型限制性核酸内切酶介导DNA组装的新方法,可以实现多个DNA片段的高效“无缝”组装,已经被用于拥有重复的复杂二级结构的激活子的拼接和多重CRISPR-Cas9基因编辑等领域[16, 23-25]。Golden Gate的主要优点是可以将其模块化,而模块化是合成生物学的一个标志,但和许多方法一样Golden Gate在标准化和可重复性方面存在一定限制[26-27]。GoldenBraid(GB)是一种基于Golden Gate的标准化组装系统,可以在一个反应中实现多个标准化元件的无痕定向快速组装,并允许由标准化DNA片段组成的可重复使用的基因模块通过二进制组合无限生长[27]。GB系统不需要进行额外的PCR扩增和载体线性化的步骤,还允许在一步反应中进行限制消化-连接循环,使反应结束时只保留正确的组合,极大地提高了反应效率并节省了实验时间[24]。
GB是一种迭代组装方法,反应仅使用两种限制性内切酶,工具包较小,组装的转录单位更易于在其他组装中重复使用[26]。GB的简单高效、可重复性和可交换性的特性促进了实验之间元件的重复使用和研究小组之间的元件交换,成为植物分子生物学家构建复杂多基因结构的有效方法[26, 28],已经广泛应用于Cas9和Cas12a介导的植物基因编辑等领域,同时在酵母、真菌等生物上也已经得到应用[29-33]。
GB是一种基于ⅡS型限制性内切酶的限制连接反应,内切酶实现元件的释放,T4连接酶完成后续的元件定向组装,从而形成目标重组载体。GB标准方案采用ligase buffer,在进行较多DNA片段的组装时,LB平板上目标重组载体对应的白色菌落稀少,大多为空载体对应的蓝色菌落,我们推测很可能内切酶在ligase buffer中难以发挥完全的酶切功能导致酶切连接反应效率较低。除缓冲体系外,酶切连接反应效率还可能受到酶切时间、缓冲体系、ⅡS型限制性内切酶来源和底物配伍等多个因素影响,本研究通过对GB反应体系的参数进行系统优化,探讨酶切连接反应的最适条件,旨为今后应用GB系统进行更复杂的通路设计提供一种行之有效的组装方法。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒本研究所用大肠杆菌(Escherichia coli)DH5α、纯化DNA片段以及元件供体质粒(表 1)为实验室保存,pUPD2、pDGB3 α1/2和pDGB3 Ω1/2受体载体质粒来自Addgene提供的GoldenBraid 2.0 kit。
ⅡS限制性核酸内切酶:BsmBI/Esp3Ⅰ(ER0451)、FastDigest Esp3Ⅰ(FD0454)和FastDigest Eco31Ⅰ(FD0293),Thermo Fisher Scientific - CN产品,BsaI(#R0535),NEB(北京)产品;DNA连接酶:T4 DNA连接酶(M1804),Promega(北京)产品;ATP Solution(R0441),Thermo Fisher Scientific - CN产品。
1.2 方法 1.2.1 限制连接组装反应使用BsaI或BsmBI作为限制酶和T4连接酶建立限制连接反应,使DNA序列被驯化成标准化元件,并实现多个标准元件的组装和连接。限制连接反应的原始方案根据表 2搭建反应体系,反应在PCR仪中进行,热循环参数设置为37℃ 2 min,16℃ 5 min,25个循环。
将-80℃冰箱保存的E. coli DH5α感受态细胞放置于冰上解冻,取2 μL的GB反应产物加入50 μL的感受态细胞中,轻轻混匀,置于冰上孵育30 min,然后42℃水浴90 s,迅速放回冰上继续孵育2-5 min,在超净工作台中向离心管中分别加入1 mL LB培养基(不含抗生素)轻轻混匀,随后37℃水浴45 min。在超净工作台中取50 μL菌液涂布于含有IPTG(0.5 mmol/L)、X-gal(40 µg/mL)和受体载体对应抗生素的LB琼脂平板上,37℃过夜孵育以筛选阳性克隆。(pUPD2:CamR;pDGB3 α1/2:KanR;pDGB3 Ω1/2:SpeR)
1.2.3 GB反应评价方法统计每组3次重复实验中LB平板上包含目标重组载体的白色菌落和包含完整受体载体的蓝色菌落的数量,计算总菌落数。定义酶切连接效率作为GB反应的综合评价指标。
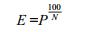
式中,E表示酶切连接效率(%),N表示有效克隆数量,即为白色菌落数量,P表示有效克隆比例(%),即白色菌落数量与总菌落数量的比例。当酶切连接效率达到97%以上时,由于接近理想极限,对体系优化不再敏感。
1.2.4 GB反应优化实验 1.2.4.1 酶切时间对GB反应的影响将酶切连接反应中37℃热孵育时间由原方案的2 min延长到4 min。观察统计总菌落数量和有效克隆数量,分析酶切时间对元件组装效率的影响。
1.2.4.2 缓冲体系对GB反应的影响将原方案中ligase buffer替换成FastDigest Buffer和FastDigest Buffer(ATP)预混液,预混液的配置方法为10 μL 10×FastDigest Buffer中加入1 μL ATP(100 mmol/L)取1.1 μL预混液加入10 μL反应体系,因此,反应体系中ATP终浓度为1 mmol/L。观察统计总菌落数量和有效克隆数量,考察缓冲体系对元件组装效率的影响。
1.2.4.3 限制性内切酶对GB反应的影响酶切连接反应中37℃热孵育时间设定4 min,缓冲体系使用FastDigest Buffer(ATP)预混液。将原方案中1 μL fermentas BsmBI/Esp3Ⅰ分别替换成0.5 μL fermentas BsmBI/Esp3Ⅰ和0.5 μL FastDigest Esp3Ⅰ,将1 μL NEB BsaI分别替换为0.5 μL NEB BsaI和0.5 μL FastDigest Eco31Ⅰ,观察统计总菌落数量和有效克隆数量,考察不同来源内切酶对元件组装效率的影响。
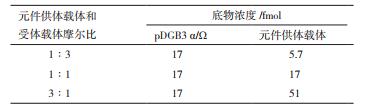
1.2.4.4 底物配伍对GB反应的影响酶切连接反应中37℃热孵育时间设定为4 min,使用FastDigest Buffer(ATP)预混液和0.5 μL FastDigest限制性内切酶。将原方案中按质量比计算各组分加入量更改为按摩尔比计算各组分加入量。针对不同的元件供体载体和受体载体摩尔比设置3个处理,处理Ⅰ元件供体载体和受体载体摩尔比为1:3;处理Ⅱ元件供体载体和受体载体摩尔比为1:1;处理Ⅲ元件供体载体和受体载体摩尔比为3:1。不同摩尔比的底物浓度见表 3。观察统计总菌落数量和有效克隆数量,考察底物配伍对元件组装效率的影响。可用以下公式计算DNA摩尔数[26]:
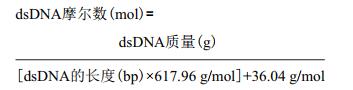
摩尔比=元件供体载体摩尔数:受体载体摩尔数。
2 结果 2.1 酶切时间对酶切连接效率的影响实验结果表明,当酶切时间为2 min(原方案)时,BsmBI-GB反应LB平板上的菌落基本上是蓝色,有效克隆比例只有8.08%;BsaI-GB反应LB平板上的菌落也以蓝色为主,有效克隆比例只有23.43%,对应酶切连接效率分别为0.3%和7.96%,均远远小于1。将酶切时间由2 min延长到4 min,发现有效克隆比例分别提升到23.66%(BsmBI)和72.59%(BsaI),比2 min实验提升了2倍左右。统计分析发现,不同酶切时间对应的有效克隆比例存在显著差异,P < 0.01(图 1),说明酶切时间从2 min延长到4 min,体系得到明显优化。考虑到酶切时间继续延长会导致实验进程过于冗长,所以我们建议在后续的实验中将每个循环的酶切时间设置为4 min。

|
| 以上误差线为标准误差;“*”表示P < 0.05水平差异显著 图 1 酶切时间对有效克隆比例的影响 |
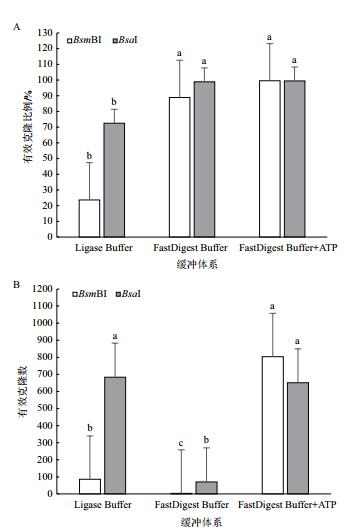
GB原方案采用ligase buffer作为整个反应的缓冲体系,在实验中发现即使在酶切时间延长到4 min后,LB平板上的菌落仍然以蓝色为主,特别是BsmBI-GB反应酶切连接效率只有18.71%,而选择更为合适的缓冲体系可能成为进一步提升GB反应效率的有效手段。实验表明,当缓冲体系替换为FastDigest Buffer(ATP)时,BsmBI-GB反应和BsaI-GB反应的LB平板上基本是白色菌落,有效克隆比例达到99.50%(图 2-A),有效克隆数量超过了600个/皿(图 2-B),酶切连接效率都达到了99.90%以上。统计分析发现,FastDigest Buffer(ATP)相比于ligase buffer,能够显著提升GB体系的有效克隆比例,P < 0.0001。所以我们建议在后续的实验中缓冲体系使用FastDigest Buffer(ATP)。
|
| A:缓冲体系对有效克隆比例的影响;B:缓冲体系对有效克隆数的影响;以上误差线为标准误差,不同小写字母表示差异显著P < 0.05 图 2 酶切时间对有效克隆比例的影响 |
限制性内切酶的用量决定了GB反应的成本,所以我们尝试在限制性内切酶减半的情况下,考察不同来源限制性内切酶的效果。首先,我们尝试在酶切时间延长到4 min并使用FastDigest Buffer(ATP)的基础上,将GB体系中fermentas BsmBI/Esp3Ⅰ和NEB BsaI的用量减半(0.5 μL)发现,LB平板上仍然有很多蓝色菌落,有效克隆比例分别是66.17%(BsmBI)和52.43%(BsaI),对应酶切连接效率分别为90.45%和87.38%。将上述方案中的fermentas和NEB核酸内切酶替换为FastDigest限制性内切酶,实验发现有效克隆比例分别提高到90.80%(FastDigest Esp3Ⅰ)和97.61%(FastDigest Eco31Ⅰ),是原内切酶方案的1.5倍左右,酶切连接效率都达到了98.00%以上。统计分析发现,FastDigest限制性内切酶相比于原方案内切酶,能够显著提高GB体系的有效克隆比例(P < 0.0001,图 3)。我们建议在后续的实验中使用0.5 μL FastDigest限制性内切酶以节省实验成本。

|
| 以上误差线为标准误差;“*”表示P < 0.05水平差异显著 图 3 限制性内切酶对有效克隆比例的影响 |
实验发现,当元件供体载体与受体载体摩尔比为1:3时,LB平板上大部分菌落是蓝色,有效克隆比例在40.36%左右,对应酶切连接效率为56.03%,因为体系中多余的受体载体会作为空载体转化大肠杆菌以蓝色菌落的形式存在,导致有效克隆比例下降;将元件供体载体与受体载体摩尔比提升到1:1时,发现有效克隆比例增加到90.80%,酶切连接效率提升到98.16%,达到可用水平,但LB平板上仍有小部分蓝色菌落;进一步将元件供体载体与受体载体摩尔比提高到3:1时发现,有效克隆比例在98.50%左右,LB平板上几乎没有蓝色菌落,酶切连接效率达到99.86%。统计分析发现,元件供体载体与受体载体摩尔比3:1时可以有效提高GB体系的有效克隆比例(P < 0.0001,图 4)。所以我们建议在后续的实验中元件供体载体和受体载体按3:1的摩尔比加入。

|
| 以上误差线为标准误差,不同小写字母表示差异显著(P < 0.05) 图 4 元件供体载体和受体载体摩尔比对有效克隆比例的影响 |
GoldenBraid(GB)标准化组装系统利用BsaI和BsmBI两种不同的ⅡS型内切酶可以实现连续的迭代组装以构建多基因表达载体,平台和工具包的不断改进使GB成为最快速简便的DNA组装方法之一[34]。Sarrion-Perdigones等[27]在一个反应中同时将5个标准化元件和一个受体载体组装在一起,通过二进制组装后得到14.5 kb的多基因结构。GB原则上允许多基因结构的无限增长,唯一的限制是载体能力和细菌的生物限制[28],但是随着元件的数量和大小的增加反应效率会降低,当GB反应中存在线性化片段时效率也会降低[26],因此多个DNA线性片段的组装较为困难,与此同时高昂的ⅡS限制性内切酶费用也使GB反应的成本一直居高不下[35]。提高GB反应的效率不仅可以解决DNA线性片段组装的问题,还可以减少内切酶的用量以节约成本。
作为一个限制-连接组装反应(图 5),限制性内切酶和T4 DNA连接酶的工作效率对GB反应是至关重要的。当限制性内切酶不能有效工作时,大部分底物不能被消化,导致用于连接反应的DNA线性片段的浓度较低,LB平板上总菌落数很多但有效克隆较少。当T4 DNA连接酶不能有效工作时,消化得到的线性片段无法连接在一起,LB平板上总菌落数较少。

|
| 3个标准化元件经过BsaI-限制连接反应组装到pDGB3 α1受体载体上。元件供体载体消化成元件和线性化pUPD2载体片段。然后元件和线性化pUPD2载体片段在T4 DNA连接酶作用下可以重新组装成元件供体载体。受体载体同上。在T4 DNA连接酶作用下将3个元件片段组装到受体载体中,生成目标表达载体。反应②、③同时进行 图 5 元件组装示意图 |
当限制性内切酶不能有效工作时,合适的酶切时间、缓冲体系和限制性内切酶可以使更多的底物被消化连接。适当的增加酶切时间是提高酶切效率最简便的方法[36]。酶切时间增加到4 min时,有更多的元件被释放出来连接成目标载体,有效克隆比例和酶切连接效率明显提高,有效克隆比例已经达到72.59%(BsaI),考虑到有效克隆比例的最高理论值为100%,继续延长时间无法再明显提升实验效果,反而会大幅度增加实验耗时,综上考虑采用4 min酶切时间,后续实验中再通过其他优化策略进一步提高反应效率。缓冲体系是决定酶是否发挥其最大催化效能的关键因素,选用合适的缓冲体系可以有效提升酶的催化能力。ligase buffer是T4 DNA连接酶最佳缓冲体系,但并不是内切酶的最优缓冲体系,可能会导致酶切不充分。FastDigest Buffer通过优化缓冲盐的配伍和牛血清蛋白(Bovine serum albumin,BSA)[36-39]的添加使内切酶和T4 DNA连接酶在其中可以同时保持几乎100%的活性,产品说明书建议使用T4 DNA连接酶时需要在反应体系中加入0.5 mmol/L ATP(https://www.thermofisher.com),为保险起见,本实验我们加入了1 mmol/L ATP以保证T4 DNA连接酶可以高效工作。此时反应酶切连接效率已经达到99%,为了节约成本,我们考虑将内切酶的用量减半。在相同用量条件下,FastDigest限制性内切酶进行底物DNA彻底酶切所需时间只有5-15 min,不到fermentas BsmBI和NEB BsaI的1/4[40-41],因此在用量减半(0.5 μL)的情况下,仍然可以实现高效酶切,达到保持较高酶切连接效率的同时降低实验成本的目的。
当限制性内切酶和T4 DNA连接酶都在有效工作时,GB反应中元件供体载体和受体载体之间的配伍成为影响酶切连接效率的一个重要因素[26]。此时底物基本被消化用于连接,目标重组载体产生速率r1和完整受体载体的产生速率r2可用公式表示:

二者的比值R代表了连接反应中目标重组载体的选择性,在充分酶切条件下,C元件浓度等于元件供体载体浓度,CLacZ浓度等于受体载体浓度,此时R可表示为:

反应中目标重组载体的选择性与底物的摩尔比有关,元件供体载体和受体载体摩尔比越大,目标重组载体的选择性就越高,获得的有效克隆比例也就越高,对应酶切连接效率也就越高。原方案中各种底物质粒的加入量都为75 ng,由于不同元件之间在DNA长度上存在较大差异,对应分子量不同,因此在各个组装实验中元件供体载体和受体载体的摩尔比各不相同,这可能成为组装效率不稳定的一个重要影响因素[26, 42]。因此建议在GB反应中使用摩尔比来计算底物配伍。优化后反应体系最终酶切连接效率已经接近100%,相比于初始反应体系酶切连接效率提高了10倍以上。通过本研究既提高了GB反应酶切连接效率,又节约了实验成本,为实现基因突变体元件的筛选建库提供了一种高效的工具。
4 结论本研究确定了更为高效的GB反应体系:FastDigest限制性内切酶0.5 μL,FastDigest Buffer 1 μL,ATP 1 mmol/L,元件供体载体和受体载体摩尔比设定为3:1,PCR仪器的热循环程序设定为37℃ 4 min,16℃ 5 min,25个循环。在此体系下最终酶切连接效率高达99.86%,接近100%,比优化前提高了10倍以上。为降低实验成本,在进行一些相对简单的合成生物学通路的构建时可以进一步缩减限制性内切酶的反应时间或者使用量。
| [1] |
Kiga D, Yamamura M. Synthetic biology[J]. New Generation Computing, 2008, 269(4): 347-364. |
| [2] |
Bartley BA, Kim K, Medley JK, et al. Synthetic biology:Engineering living systems from biophysical principles[J]. Biophys J, 2017, 112(6): 1050-1058. |
| [3] |
肖文海, 王颖, 元英进. 化学品绿色制造核心技术造核心技术1[J]. 化工学报, 2016, 67(1): 119-128. Xiao WH, Wang Y, Yuan JY. Core technology in chemicals green manufacturing:synthetic biology[J]. CIESC Journal, 2016, 67(1): 119-128. |
| [4] |
熊燕, 陈大明, 杨琛, 等. 合成生物学发展现状与前景[J]. 生命科学, 2011, 23(9): 826-837. Xiong Y, Chen DM, Yang C, et al. Progress and perspective of synthetic biology[J]. Chinese Bulletin of Life Sciences, 2011, 23(9): 826-837. |
| [5] |
朱星华, 李哲. 合成生物学的研究进展与应用[J]. 中国科技论坛, 2011(5): 143-148. Zhu XH, Li Z. The Research progress and future application of synthetic biology[J]. Forum on Science and Technology in China, 2011(5): 143-148. |
| [6] |
Medema MH, Breitling R, Bovenberg R, et al. Exploiting plug-and-play synthetic biology for drug discovery and production in microorganisms[J]. Nat Rev Microbiol, 2011, 9(2): 131-137. |
| [7] |
Breitling R, Takano E. Synthetic biology advances for pharmaceutical production[J]. Curr Opin Biotechnol, 2015, 35: 46-51. |
| [8] |
孔建强, 王伟, 程克棣, 等. 青蒿素的合成生物学研究进展[J]. 药学学报, 2013, 48(2): 193-205. Kong JQ, Wang W, Cheng KL, et al. Research progresses in synthetic biology of artemisinin[J]. Acta Pharmaceutica Sinica, 2013, 48(2): 193-205. |
| [9] |
蒙海林.基于系统—合成生物学的天然产物异源生物合成研究[D].广州: 华南理工大学, 2011. Meng HL. Natural products heterologous biosynthesis based on Systems-synthetic biology[D]. Guangzhou: South China University of Technology, 2011. |
| [10] |
Jain KK. Synthetic biology and personalized medicine[J]. Med Princ Pract, 2013, 22(3): 209-219. |
| [11] |
Ye H, Daoud-El Baba M, Peng RW, et al. A synthetic optogenetic transcription device enhances blood-glucose homeostasis in mice[J]. Science, 2011, 332(6037): 1565-1568. |
| [12] |
Weber W, Fussenegger M. Emerging biomedical applications of synthetic biology[J]. Nat Rev Gene, 2011, 13(1): 21-35. |
| [13] |
Kemmer C, Fluri DA, Witschi U, et al. A designer network coordinating bovine artificial insemination by ovulation-triggered release of implanted sperms[J]. J Control Release, 2011, 150(1): 23-29. |
| [14] |
Kemmer C, Gitzinger M, Daoud-El Baba M, et al. Self-sufficient control of urate homeostasis in mice by a synthetic circuit[J]. Nat Biotechnol, 2010, 28(4): 355-360. |
| [15] |
Deans TL, Grainger DW, Fussenegger M. Synthetic Biology:Innovative approaches for pharmaceutics and drug delivery[J]. Adv Drug Deliv Rev, 2016, 105: 1-2. |
| [16] |
Chao R, Yuan Y, Zhao H. Recent advances in DNA assembly technologies[J]. FEMS Yeast Res, 2015, 15(1): 1-9. |
| [17] |
Casini A, Storch M, Baldwin GS, et al. Bricks and blueprints:methods and standards for DNA assembly[J]. Nat Rev Mol Cell Biol, 2015, 16(9): 568-576. |
| [18] |
Cobb RE, Ning JC, Zhao H. DNA assembly techniques for next-generation combinatorial biosynthesis of natural products[J]. J Ind Microbiol Biotechnol, 2014, 41(2): 469-477. |
| [19] |
Engler C, Marillonnet S. Generation of families of construct variants using golden gate shuffling[J]. Methods Mol Biol, 2011, 729: 167-181. |
| [20] |
Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases[J]. Nat Methods, 2009, 6(5): 343-345. |
| [21] |
Ellis T, Adie T, Baldwin GS. DNA assembly for synthetic biology:from parts to pathways and beyond[J]. Integr Biol, 2011, 3(2): 109-118. |
| [22] |
晁然, 原永波, 赵惠民. 构建合成生物学制造厂[J]. 中国科学:生命科学, 2015, 45(10): 976-984. Chao R, Yuan YB, Zhao HM. Building biological foundries for next generation synthetic biology[J]. Scientia Sinica(Vitae), 2015, 45(10): 976-984. |
| [23] |
常汉臣, 王琛, 王培霞, 等. DNA组装技术[J]. 生物工程学报, 2019, 35(12): 2215-2226. Chang HC, Wang C, Wang PX, et al. DNA assembly technologies:a review[J]. Chinese Journal of Biotechnology, 2019, 35(12): 2215-2226. |
| [24] |
张阳璞, 杨淑慎. 几种新型植物基因表达载体的构建方法[J]. 生物工程学报, 2015, 31(3): 311-327. Zhang YP, Yang SS. Methods for construction of transgenic plant expression vector:a review[J]. Chinese Journal of Biotechnology, 2015, 31(3): 311-327. |
| [25] |
Sakuma T, Nishikawa A, Kume S, et al. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system[J]. Sci Rep, 2014, 4: 5400. |
| [26] |
Patron NJ. DNA assembly for plant biology[J]. Curr Protoc Plant Biol, 2016, 1(4): 604-616. |
| [27] |
Sarrion-Perdigones A, Falconi EE, Zandalinas SI, et al. GoldenBraid:an iterative cloning system for standardized assembly of reusable genetic modules[J]. PLoS One, 2011, 6(7): e21622. |
| [28] |
Sarrion-Perdigones A, Vazquez-Vilar M, Palací J, et al. GoldenBraid 2. 0:a comprehensive DNA assembly framework for plant synthetic biology[J]. Plant Physiol, 2013, 162(3): 1618-1631. |
| [29] |
Vazquez-Vilar M, Bernabé-Orts JM, Fernandez-Del-Carmen A, et al. A modular toolbox for gRNA-Cas9 genome engineering in plants based on the GoldenBraid standard[J]. Plant Methods, 2016, 12: 10. |
| [30] |
Bernabé-Orts JM, Casas-Rodrigo I, Minguet EG, et al. Assessment of Cas12a-mediated gene editing efficiency in plants[J]. Plant Biotechnol J, 2019, 17(10): 1971-1984. |
| [31] |
Vazquez-Vilar M, Gandía M, García-Carpintero V, et al. Multigene engineering by GoldenBraid cloning:From plants to filamentous fungi and beyond[J]. Curr Protoc Mol Biol, 2020, 130(1): e116. |
| [32] |
Pérez-González A, Kniewel R, Veldhuizen M, et al. Adaptation of the GoldenBraid modular cloning system and creation of a toolkit for the expression of heterologous proteins in yeast mitochondria[J]. BMC Biotechnol, 2017, 17(1): 80. |
| [33] |
Hernanz-Koers M, Gandia M, Garrigues S, et al. FungalBraid:a GoldenBraid-based modular cloning platform for the assembly and exchange of DNA elements tailored to fungal synthetic biology[J]. Fungal Genet Biol, 2018, 116: 51-61. |
| [34] |
Vazquez-Vilar M, Quijano-Rubio A, Fernandez-Del-Carmen A, et al. GB3. 0:a platform for plant bio-design that connects functional DNA elements with associated biological data[J]. Nucleic Acids Res, 2017, 45(4): 2196-2209. |
| [35] |
Sarrion-Perdigones A, Palaci J, Granell A, et al. Design and construction of multigenic constructs for plant biotechnology using the GoldenBraid cloning strategy[J]. Methods Mol Biol, 2014, 1116: 133-151. |
| [36] |
李建武, 李灏德, 文国宏, 等. 马铃薯EcoRⅠ/ MseⅠ内切酶组合AFLP反应体系的优化与引物筛选[J]. 江苏农业科学, 2017, 45(15): 25-29. Li JW, Li HD, Wen GH, et al. The potato EcoRⅠ/MseⅠ enzyme combination of AFLP reaction system optimization and primers screening[J]. Jiangsu Agricultural Sciences, 2017, 45(15): 25-29. |
| [37] |
Wu AZ, Lin CZ, Zhai YJ, et al. Investigation of the interaction between two phenylethanoid glycosides and bovine serum albumin by spectroscopic methods[J]. J Pharm Anal, 2013, 3(1): 61-65. |
| [38] |
朱杭飞, 姜晓明, 藏雨轩, 等. 限制性内切酶KpnⅠ和EcoRⅠ双酶切条件的优化[J]. 吉林医药学院学报, 2014, 35(4): 250-253. Zhu KF, Jiang XM, Zang XY, et al. The optimization of co-digestion conditions by KpnⅠ /EcoR Ⅰ[J]. Journal of Jilin Medical University, 2014, 35(4): 250-253. |
| [39] |
Plante D, Bélanger G, Leblanc D, et al. The use of bovine serum albumin to improve the RT-qPCR detection of foodborne viruses rinsed from vegetable[J]. Lett Appl Microbiol, 2011, 52(3): 239-244. |
| [40] |
Wang T, Wang D, Lyu Y, et al. Construction of a high-efficiency cloning system using the Golden Gate method and Ⅰ-SceⅠ endonuclease for targeted gene replacement in Bacillus anthracis[J]. J Biotechnol, 2018, 271: 8-16. |
| [41] |
Li B, Kaushik S, Kalinowski P, et al. Droplet digital PCR shows the D-Loop to be an error prone locus for mitochondrial DNA copy number determination[J]. Sci Rep, 2018, 8(1): 11392. |
| [42] |
付峰.鲤春病毒血症病毒(SVCV)糖蛋白基因分析、改造及其克隆与表达[D].上海: 上海海洋大学, 2005. Fu F. Bioinformatic analysis, modification, cloning, and expression of the glycoprotein gene of spring viremia of carpvirus(SVCV)[D]. Shanghai: Shanghai Ocean University, 2005. |