金黄色葡萄球菌(Staphylococcus aureus)是一种重要的病原菌,可引起人和动物化脓感染、肺炎、肠炎、心内膜炎、败血症及脓毒症等[1-2]。近年来由于抗生素的滥用,导致金黄色葡萄球菌耐药性产生,以耐甲氧西林金黄色葡萄球菌(MRSA)最为常见,由此导致的疾病逐渐增多,治疗效果越来越不显著。因此,迫切需要寻找新的抗金黄色葡萄球菌的药物靶点和策略来控制病原菌的感染。表面蛋白是革兰氏阳性菌的一种重要毒力因子,在细胞粘附、生物被膜形成、抵抗吞噬及入侵宿主细胞等方面发挥着重要的作用[3]。分选酶(srt)广泛的存在于革兰氏阳性菌中,它可将携带有LPXTG基序分选信号的表面蛋白锚定到细菌细胞壁肽聚糖上。金黄色葡萄球菌分选酶A(srtA)是细菌将表面致病性蛋白锚定到细胞壁上的关键酶[4]。研究表明通过抑制金黄色葡萄球菌Newman菌株srtA基因可降低金黄色葡萄球菌对机体的感染能力[5]。因此srtA是一个有潜在应用价值的药物治疗靶标[6]。
CRISPR/Cas9(Clustered regulatory interspaced short palindromic repeats/CRISPR-associated protein 9)是一种编辑效率较高的新型基因组靶向修饰技术。该技术仅通过一段sgRNA来识别靶位点,利用Cas9蛋白进行切割,产生双链DNA断裂(Double-strand breaks,DSBs),真核生物可通过非同源末端连接(Non-homologous end joining,NHEJ)修复方式和同源重组方式(Homology-directed repair,HDR)进行自我修复从而达到高效的基因编辑效果,目前应用已趋于成熟。原核生物除变形菌门、厚壁菌门、放线菌门等少数菌外,大多数细菌中并没有非同源重组修复机制,因此,CRISPR/Cas9系统在原核生物中运用并不广泛[7-10]。但随着研究者不断地优化,该系统在原核生物中也取得一定的进步。目前,已经在大肠杆菌[11],枯草芽孢杆菌[12]、耻垢分枝杆菌[13]、肺炎双球菌[14]、乳酸菌[15]、巴斯德梭菌[16]及放线菌[17]等细菌中成功的进行了基因编辑,可实现特定位点的突变、剪切、删除、插入、替换及多个基因同时编辑等一系列编辑方式[18]。
本研究利用CRISPR/Cas9技术构建了耐甲氧西林金黄色葡萄球菌USA300 srtA基因缺失株,为srtA的深入研究和抗金黄色葡萄球菌新药物研发提供了理论基础和数据支撑,为CRISPR/Cas9基因编辑技术在原核生物基因敲除和筛选等方面提供技术参考。
1 材料与方法 1.1 材料 1.1.1 实验动物、菌株与质粒实验动物:昆明小鼠(20±2g)购于中国农业科学院兰州兽医研究所动物实验中心。
菌株:金黄色葡萄球菌USA300-TCH1516菌株由甘肃农业大学微生物与免疫学实验室保存。大肠杆菌感受态细胞Top10、大肠杆菌感受态细胞DH5α购自北京全式金生物有限公司。金黄色葡萄球菌RN4220菌株(金黄色葡萄球菌RN4220菌株为NCTC8325菌株经紫外线和化学等方法诱导产生的限制性内切酶缺陷型菌株,其基因型为mec、rsbU、agr阴性。RN4220菌株能够接受来源于外部的其他物种的DNA质粒,同时,可以将外源质粒DNA进行修饰,修饰后的质粒可以被野生型金黄色葡萄球菌接受而不被降解)由中国科学技术大学孙宝林老师馈赠。
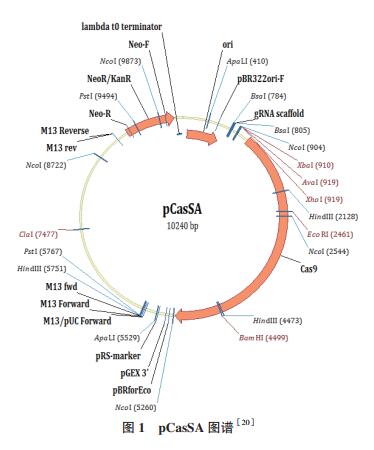
质粒:pCasSA质粒(金黄色葡萄球菌和大肠杆菌温度敏感性质粒)由上海科技大学季泉江老师馈赠。pLI50质粒(金黄色葡萄球菌单拷贝整合质粒)购自湖南丰晖生物科技有限公司。
1.1.2 主要试剂和培养基试剂:细菌基因组DNA提取试剂盒、DL2000 Marker、DL15000 Marker、琼脂糖、DNA纯化回收试剂盒、质粒小提试剂盒购自天根生化科技(北京)有限公司。普通TaqDNA聚合酶、DL5000 Marker、T4DNA连接酶、PrimeSTAR HS(PREMIX)、QuickCut TM EcoR Ⅰ、QuickCutTM BamH Ⅰ购自大连宝生物(TaKaRa)公司。Xho Ⅰ、Xho Ⅰ和Xba Ⅰ限制性内切酶购自NEB(北京)有限公司。Ⅰ型核酸染料、卡那霉素、氨苄霉素、氯霉素购自北京索莱宝科技有限公司。溶葡球菌酶购自上海生工生物工程有限公司。
培养基:LB固体和LB液体培养基用于培养大肠杆菌,TSB液体培养基和TSA固体培养基用于培养金黄色葡萄球菌。
1.2 方法 1.2.1 引物设计利用NCBI上公布的USA300-TCH1516基因组序列,使用在线软件CCTop-CRISPR/Cas9 target online predictor在srtA基因的CDS区设计3对sgRNA分别为:sgRNA1,sgRNA2,sgRNA3。使用软件primer5.0和DNASTAR分别设计srtA基因左右同源臂扩增引物srtA-L-F和srtA-L-R,srtA-R-F和srtA-R-R;缺失回补基因扩增引物HB-srtA-F和HB-srtA-R;sgRNA检测引物(位于sgRNA两侧)srtA-sgRNA-F和srtA-sgRNA-R;srtA基因缺失鉴定引物WJD-srtA-F和WJD-srtA-R,NJD-srtA-F和NJD-srtA-R。引物均由苏州金唯智生物科技有限公司合成(表 1)。
参照Zhao等[19]金黄色葡萄球菌感受态细胞制作方法。分别挑取金黄色葡萄球菌RN4220和USA300菌株单个菌落于5 mL的LB液体培养基中过夜培养,然后按照1:1 000的比例接种到100 mL的LB培养基中,220 r/min摇菌培养至OD600值为0.6为止。无菌条件下将培养的细菌移至冰冷的50 mL灭菌离心管中,冰浴10 min后,4℃,5 000 r/min离心10 min。弃上清,加入50 mL的冰冷的0.5 mol/L的蔗糖溶液冰浴15 min后,4℃,5 000 r/min离心10 min。重复上述步骤,依次用1/2,1/4体积的蔗糖溶液重悬,最后弃去上清,加入5 mL冰冷的0.5 mol/L的蔗糖溶液重悬,分装于1.5 mL冰冷的无菌离心管中,每管100 μL,-80℃冷冻保存,备用。
1.2.3 pCasSA-sgRNA质粒的构建使用在线软件设计3对sgRNA。即选取srtA基因的CDS区中某一NGG(N为任意碱基)序列前面20个碱基的DNA片段,定义为sgRNA上游序列(sgRNA-F),将这段序列反向互补,定义为sgRNA下游序列(sgRNA-R),并分别在sgRNA上下游序列5′ 端添加“AAAC”和“GAAA”即为限制性内切酶 Bsa Ⅰ酶切位点。对sgRNA上下游序列通过碱基配对原则退火形成双链sgRNA序列,退火体系为:sgRNA上下游序列(100 μmol/L)各2 μL,ddH2O 21 μL。反应程序为:98℃到25℃每30 s降低1℃,4℃放置8 h。
质粒pCasSA为金黄色葡萄球菌和大肠杆菌温度敏感性质粒,用于金黄色葡萄球菌的基因编辑(图谱见图 1)。该质粒可以利用Bsa Ⅰ位点插入sgRNA片段,靶向目标基因,利用Xho Ⅰ和Xba Ⅰ位点插入目标基因左右同源臂序列,进行同源修复,进而达到金黄色葡萄球菌基因敲除的目的。
构建pCasSA-sgRNA质粒。使用限制性内切酶Bsa Ⅰ对pCasSA质粒上的Bsa Ⅰ位点进行特异性酶切,酶切体系为:pCasSA 30 μL、Xho Ⅰ 2 μL、10×NEBuffer 5 μL、ddH2O 13 μL。使用天根生化科技(北京)有限公司DNA纯化回收试剂盒进行酶切质粒的纯化回收。酶切回收的pCasSA质粒与退火后的双链sgRNA序列经T4 DNA连接酶16℃连接3 h。连接体系为:T4 DNA Ligase 1 μL、10×T4 DNA Ligase buffer 2 μL、退火后的双链sgRNA序列12 μL,酶切回收后的pCasSA质粒1 μL、ddH2O 4 μL。连接产物经热激转化至大肠杆菌感受态细胞Top10中。用50 mg/μL的卡那霉素筛选阳性克隆,30℃增菌后用,用sgRNA上游序列(sgRNA1-F,sgRNA2-F,sgRNA3-F)和sgRNA检测下游引物srtA-sgRNA-R进行PCR扩增。将获得的PCR产物用1%的琼脂糖凝胶电泳检测,阳性pCasSA-sgRNA质粒(pCasSA-sgRNA1,pCasSA-sgRNA2,pCasSA-sgRNA3)送公司测序。测序结果使用MegAlign软件进行分析比对。
1.2.4 pCasSA-sgRNA质粒切割效率检测将pCasSA-sgRNA质粒(pCasSA-sgRNA1,pCasSA-sgRNA2,pCasSA-sgRNA3)电击转入缺陷型金黄色葡萄球菌RN4220菌株中修饰(电击条件:21 kV/cm,100 Ω,25 μF),提取质粒(提前使用溶葡球菌酶进行破壁处理)并转入金黄色葡萄球菌USA300菌株中(电击条件:21 kV/cm,100 Ω,25 μF),在TSA固体培养基上30℃倒置培养2 d后,观察平板上菌落的形态及数量。
1.2.5 pCasSA-sgRNA-srtA缺失质粒的构建以金黄色葡萄球菌USA300菌株基因组为模板,用引物srtA-L-F和srtA-L-R,srtA-R-F和srtA-R-R分别扩增srtA基因左右同源臂srtA-L和srtA-R序列,反应体系为:PrimeSTAR HS 25 μL,上下游引物各2 μL,DNA模板1 μL,ddH2O 20 μL。反应程序为:98℃ 10 s,55℃ 5 s,72℃ 1 min,35个循环。使用融合PCR,将纯化回收后的srtA-L和srtA-R两段序列融合,融合体系为:PrimeSTAR HS 25 μL,srtA-L、srtA-R序列各2 μL,ddH2O 21 μL。反应程序为:98℃ 10 s,55℃ 5 s,72℃ 1 min,35个循环,获得srtA-L+R序列。融合后的srtA-L+R序列浓度较低,需要将srtA-L+R序列进行再扩增。扩增体系为:PrimeSTAR HS 12.5 μL,srtA-L-F、srtA-R-R引物各1 µL,融合后的srtA-L+R序列25 μL,ddH2O 10.5 μL。反应程序为:98℃ 10 s,55℃ 15 s,72℃ 2.2 min,35个循环。使用限制性内切酶Xho Ⅰ和Xba Ⅰ将纯化后srtA-L+R序列和有切割效率的pCasSA-sgRNA质粒进行双酶切。纯化回收,T4 DNA连接酶过夜连接,连接体系为:T4 DNA Ligase 1 μL、10×T4 DNA Ligase buffer 2 μL、srtA-L+R 9 μL,酶切后的pCasSA-sgRNA质粒1 μL、ddH2O 7 μL。连接产物通过热激转化至大肠杆菌Top10中,用50 mg/μL卡那霉素筛选阳性克隆,30℃增菌后,用引物srtA-L-F和srtA-R-R进行PCR检测并测序,正确的质粒命名为pCasSA-sgRNA-srtA。
1.2.6 金黄色葡萄球菌USA300 srtA基因缺失株的构建及其鉴定将pCasSA-sgRNA-srtA质粒电击转入金黄色葡萄球菌RN4220菌株经修饰后,转入金黄色葡萄球菌USA300菌株中,用15 mg/μL氯霉素TSA固体培养基筛选阳性克隆。随机挑取10个阳性菌落,编号为:1-10号。30℃增菌后,提取基因组,用引物WJD-srtA-F,WJD-srtA-R,进行PCR初步筛选,扩增体系为:PrimeSTAR HS 12.5 μL,WJD-srtA-F、WJD-srtA-R引物各1 µL,模板1 μL,ddH2O 9.5 μL。反应程序为:98℃ 10 s,55℃ 15 s,72℃ 3 min,30个循环。对于疑似敲除的菌株,经测序检测结果。同时,使用引物NJD-srtA-F,NJD-srtA-R检测srtA基因是否存在。扩增体系为:PrimeSTAR HS 12.5 μL,NJD-srtA-F,NJD-srtA-R引物各1 µL,模板1 μL,ddH2O 9.5 μL。反应程序为:98℃ 10 s,55℃ 5 s,72℃ 30 s,30个循环。
1.2.7 重组质粒消除为消除重组质粒对金黄色葡萄球菌USA300的影响,需要消除质粒pCasSA-sgRNA-srtA。具体方法参照Chen等[20]质粒消除方法。选择一个金黄色葡萄球菌srtA基因缺失(ΔsrtA USA300)菌落。30℃过夜培养,然后按照1:1 000的比例接种到5 mL的不含有抗生素TSB液体培养基中,42℃,220 r/min培养,直到培养菌液出现混浊。取适量菌液划线于TSB固体平板上,在37℃过夜培养。从平板上随机挑取几个菌落,接种于新鲜液体培养基中,直到培养菌液出现混浊,取适量分别涂布于含有5 mg/μL氯霉素或不含氯霉素的TSA固体培养基平板上。如果菌落在不含抗生素的培养基平板上生长,而在含有抗生素的培养基平板上不生长,证实重组质粒消除。
1.2.8 金黄色葡萄球菌USA300 srtA基因缺失对细菌生长的影响将菌株接种于TSB液体培养基中,37℃摇床220 r/min培养24 h后,按1:1 000的比例转接到新鲜的TSB液体培养基中,37℃摇床220 r/min培养,每隔0.5 h测一次OD600,测定10次,最后根据OD600值绘制出细菌的生长曲线。
1.2.9 金黄色葡萄球菌USA300 srtA基因缺失对小鼠生存率的影响挑取金黄色葡萄球菌单个菌落于新鲜的TSB液体培养基中,37℃,200 r/min过夜培养。然后按照1:1 000的比例转接到100 mL的TSB培养基中,37℃,220 r/min培养至OD600值为2.0。使用PBS溶液稀释洗涤3次,调整各菌株菌量至同一水平,并稀释至2×108 CFU/mL的细菌浓度。将小鼠随机分为3组,分别为正常对照组(生理盐水)、野生型菌株组(WT USA300)和基因缺失菌株组(ΔsrtA USA300),每组15只小鼠。除正常对照组小鼠尾静脉注射生理盐水,其余组别小鼠分别注射相应的2×108 CFU/mL浓度的菌液,接种剂量为:每20 g小鼠接种0.2 mL。分别于0-14 d每天观察小鼠死亡情况,并绘制小鼠生存率曲线。
1.2.10 金黄色葡萄球菌USA300 srtA基因缺失对器官组织内细菌数量的影响使用1×108 CFU/mL浓度的菌液尾静脉注射小鼠,接种剂量为:每20 g小鼠接种0.1 mL。选用感染1 d、3 d、5 d后的小鼠眼球采血,颈椎脱臼处死小鼠,取心脏、肾脏、肝脏和脾脏,进行脏器菌落计数。具体方法如下:无菌条件下,取出小鼠心脏、肾脏、肝脏和脾脏称其重量;向组织样本中加入1 mL PBS溶液后,置于高通量组织研磨仪中充分研磨成匀浆液;将匀浆液10倍梯度稀释后,取200 µL样品稀释液涂布于TSB培养平板上,37℃倒置培养24 h。菌落数在30-300之间视为计数有效。计算器官组织内的载菌量(logCFU/g),公式为:CFU/g=[3个平板的CFU的平均数×5×匀浆液体积(mL)×稀释倍数]/组织重量(g)。
为了鉴定感染小鼠器官组织中的细菌为金黄色葡萄球菌,需要对平板菌落进行鉴定。具体方法如下:挑取单个菌落,增菌后,进行革兰氏染色,同时提取基因组,使用引物23S rRNA-F/R进行PCR鉴定。PCR产物寄送公司测序,测序结果提交GenBank数据库,进行BLAST比对分析。
1.2.11 金黄色葡萄球菌USA300 srtA基因缺失对小鼠肾脏组织病理变化的影响选用步骤1.13中感染1 d后的小鼠,眼球采血,颈椎脱臼处死小鼠,取肾脏用10%的中性甲醛溶液固定7 d,参考陈福广[5]方法进行石蜡切片制作与HE染色,光学显微镜观察小鼠肾脏病理变化。
1.2.12 金黄色葡萄球菌USA300菌株srtA基因缺失回补株的构建以金黄色葡萄球菌USA300菌株基因组为模板,HB-srtA-F,HB-srtA-R为引物扩增srtA基因及其启动子片段(HB-srtA基因),反应体系为:PrimeSTAR HS 25 μL,HB-srtA-F、HB-srtA-R各2 μL,DNA模板2 μL,ddH2O 19 μL。反应程序为:98℃ 10 s,55℃ 5 s,72℃ 1 min,35个循环。待纯化回收后,用限制性内切酶EcoR Ⅰ和BamH Ⅰ将pLI50质粒和HB-srtA进行双酶切,酶切体系为:QuickCutTM EcoR Ⅰ 2 μL,QuickCutTM BamH Ⅰ 2 µL,QuickCut Green Buffer 5 µL,PLI50/回补基因srtA 1 µg ddH2O补充至50 µL。30 min后纯化回收,用T4连接酶过夜连接,热激转化至DH5α菌株,37℃增菌后提取质粒,用双酶切和测序分别检测回补质粒PLI50-srtA是否构建成功,将构建成功的回补质粒经修饰后转入ΔsrtA USA300中,构建ΔsrtA USA300回补株(Δ∷srtA USA300)。回补株构建成功后,需进一步进行功能验证,检测srtA基因缺失后其功能是否恢复。即观察srtA基因回补后,菌株生长状况,感染后小鼠生存率、器官组织载菌量及肾脏病理组织学变化。
1.2.13 统计学分析应用GrphPad Prism7.0数据处理软件对数据进行统计学分析,结果以Mean±SD表示。应用SPSS17.0分析数据统计学差异。P < 0.05视为差异性显著,P < 0.01视为差异性极显著。
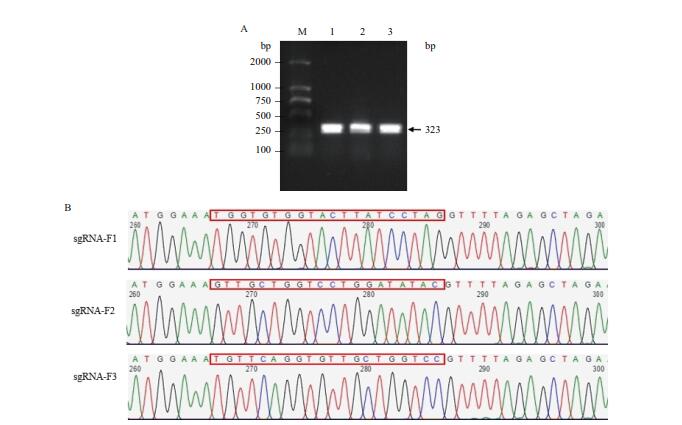
2 结果 2.1 pCasSA-sgRNA质粒的构建及鉴定将退火后的双链sgRNA序列与酶切后的pCasSA质粒连接后,用sgRNA上游序列(sgRNA1-F,sgRNA2-F,sgRNA3-F)和sgRNA检测引物srtA-sgRNA-R进行PCR鉴定,检测结果如图 2-A所示。将验证的pCasSA-sgRNA质粒送公司测序,如图 2-B所示,测序结果正确。证明pCasSA-sgRNA质粒构建成功。
|
| A:pCasSA-sgRNA PCR鉴定结果,M:DL2000 marker,1:pCasSA-sgRNA1,2:pCasSA-sgRNA2,3:pCasSA-sgRNA3;B:pCasSA-sgRNA质粒测序结果 图 2 pCasSA-sgRNA质粒的构建及鉴定 |
将修饰后的pCasSA-sgRNA质粒电击转入金黄色葡萄球菌USA300菌株中,30℃培养2 d后观察。与对照组pCasSA质粒菌落(图 3-A)相比,存在pCasSA-sgRNA2质粒的菌落在TSA固体平板上仅有几个生长(图 3-D),存在pCasSA-sgRNA1,pCasSA-sgRNA3质粒的菌落几乎长满整个平板(图 3-B,3-C)。这说明pCasSA-sgRNA2具有较高的切割效率。选用pCasSA-sgRNA2质粒进行下一步实验。

|
| A:pCasSA;B:pCasSA-sgRNA1;C:pCasSA-sgRNA3;3:pCasSA-sgRNA2 图 3 pCasSA-sgRNA质粒切割效率检测 |
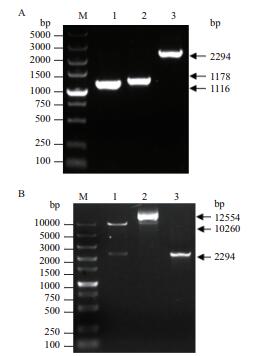
以金黄色葡萄球菌USA300菌株基因组为模板,分别扩增获得大小为1 116 bp和1 178 bp的srtA基因左右同源臂srtA-L和srtA-R序列,用融合PCR将srtA-L和srtA-R序列融合后获得2 294 bp的srtA-L+R序列,琼脂糖凝胶电泳结果显示与预期结果一致。(图 4-A)。将srtA-L+R序列构建到pCasSA-sgRNA2质粒中,经限制性内切酶Xho Ⅰ和Xba Ⅰ双酶切验证,如图 4-B所示,获得10 260 bp和2 294 bp的2条特异性条带,均与预期大小一致。对重组好的质粒送测序鉴定,结果与原序列一致。证明pCasSA-srtA-sgRNA2质粒构建成功。
|
| A:srtA基因左右同源臂构建,M:DL5000 marker,1:srtA基因左臂(srtA-L)序列PCR扩增结果,2:srtA基因右臂(srtA-R)序列PCR扩增结果,3:srtA基因左右同源臂融合(srtA-L+R)序列PCR扩增结果;B:pCasSA-srtAsgRNA2双酶切验证结果,M:DL10000 marker,1:pCasSA-srtA-sgRNA2双酶切产物,2:pCasSA-srtA-sgRNA2,3:srtA-L+R序列PCR扩增结果 图 4 pCasSA-srtA-sgRNA质粒构建及鉴定 |
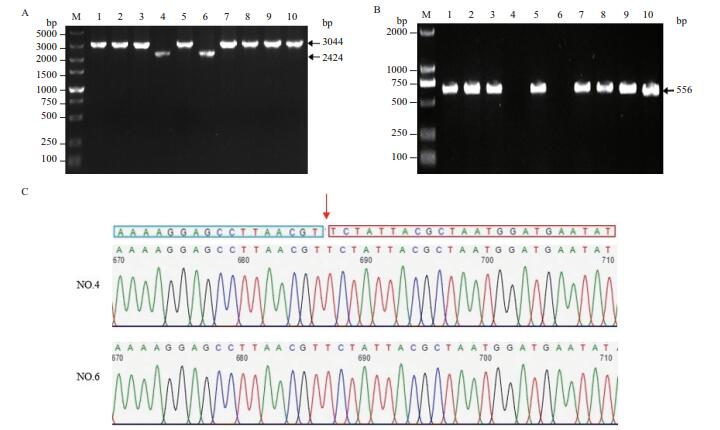
将修饰后的pCasSA-srtA-sgRNA2质粒转入USA300菌株中,30℃培养2 d,TSA平板长出菌落后,随机挑取10个单个菌落,30℃增菌后,提取基因组,用引物WJD-srtA-F,WJD-srtA-R进行PCR扩增,鉴定srtA基因敲除情况,如图 5-A所示,srtA基因没有被敲除修复的菌,PCR显示为3 044 bp,反之出现2 424 bp大小的条带。为了进一步检测srtA基因是否被敲除,用引物NJD-srtA-F,NJD-srtA-R进行PCR扩增,如图 5-B所示,srtA基因没有被敲除的菌,PCR将会扩增出556 bp大小的条带,反之不能扩增出条带。如图 5-C所示,测序结果与PCR验证结果一致。证明ΔsrtA USA300构建成功。
|
| A:ΔsrtA USA300菌株PCR鉴定结果,M:DL5000 marker,4、6:ΔsrtA USA300,5、7-10:USA300菌株;B:M:DL2000 marker,4、6:ΔsrtA USA300菌株,5、7-10:USA300菌株;C:ΔsrtA USA300菌株测序鉴定结果(NO.4,NO.6) 图 5 Δ srtA USA300菌株的筛选及鉴定 |
pCasSA-srtA-sgRNA2质粒经30℃,42℃,37℃培养后。由于pCasSA质粒中含有温度敏感的repF复制子,使得该质粒在42℃下无法复制,从而在细菌中消除。如图 6所示,在含有氯霉素的TSA平板上无菌落生长(图 6-A),在不含有氯霉素的TSA平板长出菌落(图 6-B),证明重组质粒pCasSA-srtA-sgRNA2被消除,可进行下一步实验。

|
| A:含有氯霉素的TSA培养板;B:不含有氯霉素的TSA培养板 图 6 质粒消除鉴定 |
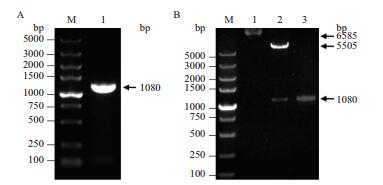
回补基因HB-srtA经PCR扩增后获得大小为1 080 bp的条带(图 7-A),与质粒pLI50(5 505 bp)连接后获得pLI50-srtA质粒,经限制性内切酶EcoR Ⅰ和BamH Ⅰ双酶切验证获得5 505 bp和1 080 bp大小的2条特异性条带(图 7-B)。pLI50-srtA质粒经测序结果正确,证明pLI50-srtA质粒构建成功。将修饰后的pLI50-srtA回补质粒导入ΔsrtA USA300中,成功构建了Δ∷srtA USA300。
|
| A:HB-srtA基因PCR扩增结果;B:pLI50-srtA质粒鉴定结果,M:DL5000 marker,1:pLI50-srtA,2:pLI50-srtA双酶切产物,3:HB-srtA基因PCR扩增结果. 图 7 HB-srtA基因PCR扩增及pLI50-srtA质粒鉴定 |
如图 8所示,srtA基因缺失后,前2 h细菌生长缓慢,2 h后进入生长对数期初期。与WT USA300相比,ΔsrtA USA300生长状态相似。srtA基因回补后,Δ∷srtA USA300生长与WT USA300生长状态基本一致,证明srtA基因缺失并不会影响金黄色葡萄球菌的生长。说明该突变是一种非致死性突变。

|
| 图 8 WT USA300,Δ srtA USA300和Δ ∷ srtA USA300生长曲线 |
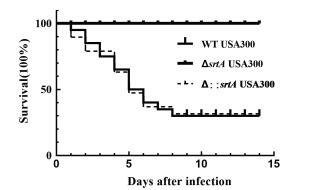
小鼠经尾静脉接种2×108 CFU/mL的金黄色葡萄球菌后,对小鼠的生存率进行分析,结果如图 9所示。WT USA300显著的引起了小鼠的死亡。srtA缺失后小鼠无死亡。srtA基因回补后,感染小鼠与WT USA300组的小鼠死亡情况基本一致,证明srtA基因回补后其功能基本恢复。
|
| 图 9 srtA基因缺失对小鼠生存率的影响 |
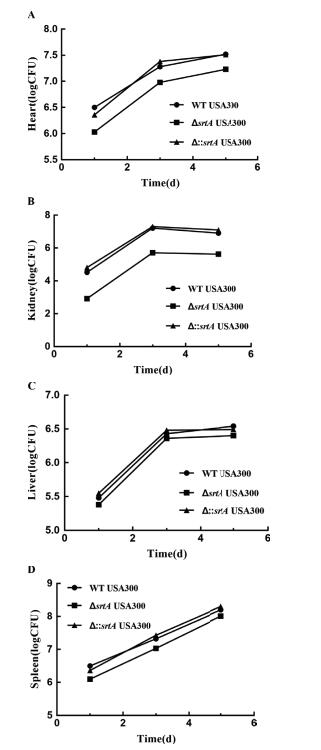
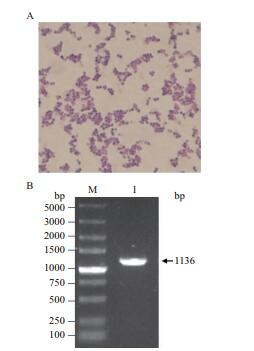
分别对感染1 d、3 d、5 d后的小鼠器官组织进行平板计数,统计心脏、肾脏、肝脏和脾脏中的细菌数量。如图 10所示,与WT USA300组相比,ΔsrtA USA300组在小鼠心脏中的细菌数量(图 10-A)显著减少(P < 0.05),在小鼠肾脏中的细菌数量(图 10-B)极显著减少(P < 0.01),但WT USA300组与ΔsrtA USA300组小鼠在肝脏和脾脏内细菌数量(图 10-C,10-D)无显著性差异。srtA基因回补后,Δ∷srtA USA300组小鼠载菌量基本恢复至野生型水平。如图 11所示,平板菌落经革兰氏染色,油镜下观察呈单个、短链、葡萄状的蓝紫色菌落(图 11-A)。经过PCR扩增,核酸琼脂糖凝胶电泳获得大小为1 136 bp的条带(图 11-B),公司测序,最终鉴定感染小鼠组织中的细菌为金黄色葡萄球菌。
|
| A:心脏;B:肾脏;C:肝脏;D:脾脏 图 10 srtA基因缺失对小鼠器官内细菌数量的影响 |
|
| A:革兰氏染色结果;B:23S rRNA PCR扩增结果 图 11 细菌的分离与鉴定 |
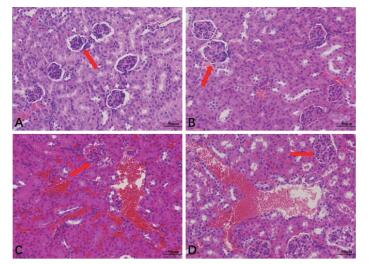
光学显微镜下观察肾脏病理变化,结果如图 12所示,正常对照组(图 12-A),细胞排列规则,肾小管和肾小球结构完整。WT USA300组(图 12-C)肾小球变性、严重坏死,肾小管上皮细胞肿胀,管腔变小或消失。肾间质充血、水肿,并可见较大面积的弥漫性出血。ΔsrtA USA300组(图 12-B)肾小球体积增大,肾小球毛细血管内皮细胞增生、肿大,肾小球囊腔狭窄,并可见局灶性出血,整体病变明显减轻。srtA基因回补后,菌株对小鼠肾脏损伤程度基本恢复至与WT USA300组同等水平(图 12-D)。
|
| A:正常对照组;B:ΔsrtA USA300组;C:WT USA300组;D:Δ∷ srtA USA300组;红色箭头所指为肾小球 图 12 srtA基因缺失对小鼠肾脏病理学变化的影响 |
光学显微镜下观察肾脏病理变化,结果如图 12所示,正常对照组(图 12-A),细胞排列规则,肾小管和肾小球结构完整。WT USA300组(图 12-C)肾小球变性、严重坏死,肾小管上皮细胞肿胀,管腔变小或消失。肾间质充血、水肿,并可见较大面积的弥漫性出血。ΔsrtA USA300组(图 12-B)肾小球体积增大,肾小球毛细血管内皮细胞增生、肿大,肾小球囊腔狭窄,并可见局灶性出血,整体病变明显减轻。srtA基因回补后,菌株对小鼠肾脏损伤程度基本恢复至与WT USA300组同等水平(图 12-D)。
3 讨论随着耐甲氧西林、耐万古霉素等金黄色葡萄球菌的出现和抗生素不良反应的增加,使得防治金黄色葡萄球菌感染的难度加大。因此寻求新型药物靶标和开发治疗金黄色葡萄球菌感染的新药物,已成为众人近年来关注的焦点问题之一。新型药物靶标的发现离不开基因组层面功能基因的筛选以及后续基因功能的验证。金黄色葡萄球菌中用于功能基因筛选的技术主要为转座子插入突变的方法。这种方法筛选过程十分繁琐,费时费力,并且转座子插入位置的随机性,使得该技术不能应用于特定的基因群[21]。后续基因功能的验证也离不开该基因缺失株的构建。以往金黄色葡萄球菌用于构建基因缺失株的方法主要为基于同源重组的方法。以pBT2、pMAD、pKOR1和pIMAY质粒最为常见。但大多操作繁琐,需要正向或者反向的筛选,效率较低。张晓静[22]使用pBT2穿梭质粒构建OatA基因敲除株,挑取1 440个单克隆,仅获得3个阳性克隆,效率不足1%。限制了高效基因功能的研究。CRISPR/Cas9系统作为一种新型基因编辑技术,可以精确、高效的对基因组进行敲除或者修饰,可在短时间内获得基因敲除突变体,从而更好地完成基因功能的研究[23]。
基于CRISPR/Cas9系统,本研究以pCasSA为载体,人工引入srtA基因左右同源臂,构建了耐甲氧西林金黄色葡萄球菌USA300 srtA基因缺失株。具有效率高,操作简单的特点。挑取的10个菌株中,获得2个基因缺失株。CRISPR/Cas9系统中,sgRNA可以靶向目标基因,Cas9蛋白可以对基因组进行特异性切割,获得DSBs,由于非同源重组末端修复的缺乏,导致细菌死亡。但金黄色葡萄球菌存在逃逸机制,仅有极少数菌不被切割存活了下来[24]。因此本研究根据平板上菌落的多少,筛选获得可能有切割效率的sgRNA。在筛选srtA基因缺失株时,往往敲除质粒中同源臂的存在可能会影响检测结果,因此本研究设计srtA基因外部和内部鉴定引物,外部鉴定引物WJD-srtA-F,WJD-srtA-R位于同源臂两端,能消除质粒对检测结果的影响。内部鉴定引物NJD-srtA-F,NJD-srtA-R位于srtA基因内部,保证了检测的准确性。这样可以直观的通过观察琼脂糖凝胶电泳条带大小达到初步筛选的效果。为避免pCasSA-srtA-sgRNA质粒存在对金黄色葡萄球菌的影响,本研究利用pCasSA质粒在42℃不能复制的原理,进行质粒的消除。最终获得不含有质粒的srtA基因缺失株。经生长曲线测定显示srtA基因缺失不会影响金黄色葡萄球菌USA300菌株的生长。因此,抑制srtA的活性不会对病原菌带来生存压力,可以有效的减少细菌耐药性的发生,有利于金黄色葡萄球菌药物抑制剂筛选,以弥补目前针对耐药性菌株感染的抗生素治疗不足[25]。
金黄色葡球菌USA300菌株属于MRSA,具有很强的毒力和致病性。据报道,MRSA菌血症的死亡率可高达80%[26]。MRSA主要以皮肤软组织和呼吸道感染为主,属于多位点序列ST8型,通常包含SCCmecIVa型元件、arcA基因、编码杀白细胞素(PVL)基因的噬菌体ФSA2USA,精氨酸分解代谢移动遗传元件(Arginine catabolic mobile Bsaed element,ACME)和SpeG基因[27]。这些基因被认为是USA300菌株得以生存和发挥毒力作用的重要因素。srtA作为金黄色葡萄球菌最重要的转肽酶,负责将20多种表面毒力因子蛋白锚定到细菌的细胞壁上,抑制后细菌感染小鼠的毒力显著降低。USA300菌株强的致病性与毒力是否与srtA基因相关,有待进一步研究。因此,本研究构建了耐甲氧西林金黄色葡萄球菌USA300 srtA基因缺失株,尾静脉接种金黄色葡萄球菌,建立了小鼠菌血症模型,探究USA300菌株srtA基因缺失对小鼠致病能力的影响。结果显示,与WT USA300相比,ΔsrtA USA300显著降低了小鼠的死亡率。在小鼠接种1×108 CFU/mL浓度的金黄色葡萄球菌1 d、3 d、5 d后,与WT USA300组小鼠相比,感染ΔsrtA USA300小鼠心脏中的细菌数量显著性减少,肾脏中的细菌数量极显著性减少,而肝脏和脾脏中的细菌量未见明显降低,这与张奇文等[28]构建单增李斯特菌srtA基因缺失株后肝脏和脾脏中载菌量显著降低有所不同,这可能与不同种属细菌与机体的相互作用机制不同有关。光学显微镜下观察感染1 d后的小鼠肾脏组织病理变化,发现与WT USA300组小鼠相比,ΔsrtA USA300组小鼠肾脏组织病变明显减轻,表明srtA基因的缺失降低了细菌对肾脏的损伤,这可能与srtA基因的缺失后减少了某些炎性因子的含量或参与调节炎症的某种信号通路有关。此外,MRSA对多种抗生素产生耐药,有研究表明生物膜形成与耐药性产生有关[29],而srtA锚定的多种表面蛋白参与细菌生物膜的黏附机制,是否srtA基因的缺失与细菌的耐药性有直接或者间接的关系,需要进一步探究。
本研究结果表明,srtA基因缺失并没有使USA300菌株完全丧失对小鼠的致病性,可见srtA分选的表面蛋白只部分参与了细菌的致病力。尽管srtA参与了众多C端含有分选信号的表面毒力蛋白锚定到细胞表面的过程,可能致病菌本身还存在其他作用机制或其他毒力因子作用,或存在类似蛋白分担srtA的作用,srtA基因缺失后激活了相似蛋白发挥补偿作用。这方面的研究需要进一步去探索。
4 结论本研究成功构建了耐甲氧西林金黄色葡萄球菌USA300菌株srtA基因缺失株和回补株,为深入研究耐甲氧西林金黄色葡萄球菌srtA基因提供实验菌株,为进一步阐明金黄色葡萄球菌毒力因子的致病机制提供理论依据。
| [1] |
Lowy FD. Staphylococcus aureus infections[J]. New England Journal of Medicine, 1998, 339(8): 520-532. |
| [2] |
Smyth DS, Kafer JM, Wasserman GA, et al. Nasal carriage as a source of Staphylococcus aureus bacteremia[J]. New England Journal of Medicine, 2001, 344(1): 11-16. |
| [3] |
Hou X, Wang M, Wen Y, et al. Quinone skeleton as a new class of irreversible inhibitors against Staphylococcus aureus sortase A[J]. Bioorg Med Chem Lett, 2018, 28(10): 1864-1869. |
| [4] |
Wardenburg JB, Patel RJ, Schneewind O. Surface proteins and exotoxins are required for the pathogenesis of Staphylococcus aureus pneumonia[J]. Infect Immun, 2007, 75(2): 1040-1044. |
| [5] |
陈福广.分选酶A在金黄色葡萄球菌引起的乳腺炎、菌血症和肺炎致病过程中的作用[D].长春: 吉林大学, 2014. Chen FG. Role of sorter A in the pathogenesis of mastitis, bacteremia and pneumonia caused by Staphylococcus aureus[D]. Changchun: Jilin University, 2014. |
| [6] |
Cossart P, Jonquieres R. Sortase, a universal target for therapeutic agents against Gram-positive bacteria[J]. Proc Natl Acad Sci USA, 2000, 97(10): 5013-5015. |
| [7] |
Bowater R, Doherty AJ. Making ends meet:repairing breaks in bacterial DNA by non-homologous end-joing[J]. PLoS Genetics, 2006, 2(2): 93-99. |
| [8] |
Spoto M, Guan C, Fleming E, et al. A universal, genomewide guidefinder for CRISPR/Cas9 targeting in microbial genomes[J]. mSphere, 2020, 5(1): 10-20. |
| [9] |
Mougiakos I, Bosma EF, de Vos WM, et al. Next generation prokaryotic engineering:the CRISPR-Cas toolkit[J]. Trends in Biotechnology, 2016, 34(7): 575-587. |
| [10] |
Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. PNAS, 2012, 109(39): 2579-2586. |
| [11] |
Xia J, Wang L, Zhu JB, et al. Expression of She-wanella frigidimarina fatty acid metabolic genes in E. coli by CRISPR/cas9-coupled lambda Red recombineering[J]. Biotechnology Letters, 2016, 38(1): 117-122. |
| [12] |
Hong KQ, Liu DY, Chen T, et al. Recent advances in CRISPR/Cas9 mediated genome editing in Bacillus subtilis[J]. World Journal of Microbiology and Biotechnology, 2018, 34(10): 151-155. |
| [13] |
邢述永, 路志群, 夏海洋, 等. 构建CRISPR-Cas9介导的耻垢分枝杆菌基因组高效删除系统[J]. 微生物学通报, 2018, 45(12): 2738-2750. Xing SY, Lu ZQ, Xia HY, et al. CRISPR-Cas9-assisting efficient and sequential genome mutants in Mycobacterium smegmatis[J]. Microbiology China, 2018, 45(12): 2738-2750. |
| [14] |
Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2016, 31(3): 233-239. |
| [15] |
Oh JH, Van Pijkeren JP. CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri[J]. Nucleic Acids Res, 2014, 15(17): 17-21. |
| [16] |
Michael EP, Mark RB, et al. Harnessing heterologous and endoge-nous CRISPR-Cas machineries for efficient markerless genome editing in Clostridium[J]. Sci Rep, 2016, 6(5): 125-129. |
| [17] |
Cobb RE, Wang J, Zhao HM. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J]. ACS Synthetic Biology, 2015, 4(6): 723-728. |
| [18] |
Khanzadi MN, Khan AA. CRISPR/Cas9:Nature's gift to prokaryotes and an auspicious tool in genome editing[J]. Journal of Bsaic Microbiology, 2019, 10(6): 1002-1005. |
| [19] |
Zhao C, Shu X, Sun B. Construction of a dCas9-bsaed gene knockdown system in Staphylococcus aureus[J]. Applied and Environmental Microbiology, 2017, 83(12): 12-17. |
| [20] |
Chen W, Zhang Y, Yeo WS, et al. Rapid and efficient genome editing in Staphylococcus aureus by using an engineered CRISPR/Cas9 system[J]. Journal of the American Chemical Society, 2017, 139(10): 3790-3795. |
| [21] |
Penewit K, Holmes EA, McLean K, et al. Efficient and scalable precision genome editing in Staphylococcus aureus through conditional recombineering and CRISPR/Cas9-mediated counterselection[J]. Mbio, 2018, 9(5): 7-18. |
| [22] |
张晓静, 冯世源, 杜崇涛, 等. 金黄色葡萄球菌OatA基因敲除菌株及其回补菌株的构建[J]. 现代生物医学进展, 2015, 4(15): 25-29. Zheng XJ, Feng SY, Du CT, et al. Construction of OatA mutant mutant and complementation of Staphylococcus aureus[J]. Progress in Modern Biomedicine, 2015, 4(15): 25-29. |
| [23] |
Horvath PM, Kavalali ET, Monteggia LM. CRISPR/Cas9 system-mediated impairment of synaptobrevin/VAMP function in postmitotic hippocampal neurons[J]. Journal of Neuroscience Methods, 2016, 278(15): 57-64. |
| [24] |
郑小梅, 张晓立, 于建东, 等. CRISPR-Cas9介导的基因组编辑技术的研究进展[J]. 生物技术进展, 2015, 11(1): 1-9. Zheng XM, Zhang XL, Yu JD, et al. Advance in and application of CRISPR-Cas9 technology in bacteria[J]. Current Biotechnology, 2015, 11(1): 1-9. |
| [25] |
Liu Y, Shi D, et al. Dracorhodin Perochlorate attenuates Staphylo-coccus aureus USA300 virulence by decreasing α-toxin expression[J]. World J Microbiol Biotechnol, 2017, 33(1): 117-121. |
| [26] |
Hornak JP, Anjum S, Reynoso D, et al. Adjunctive ceftaroline in combination with daptomycin or vancomycin for complicated methicillin-resistant bacteremia after monotherapy failure[J]. Ther Adv Infect Dis, 2019, 10(6): 117-128. |
| [27] |
Martens, Ev an, Demain, et al. The antibiotic resistance crisis, with a focus on the United States[J]. The Journal of Antibiotics:An International Journal, 2017, 70(5): 1030-1039. |
| [28] |
张奇文, 凌晨, 吴学林, 等. 单增李斯特菌srtA基因缺失株的构建及srtA基因对其毒力的影响研究[J]. 中国动物传染病学报, 2019, 27(4): 23-31. Zhang QW, Ling C, Wu XL, et al. Construction and virulence evaluation of the srtA gene deletion strain of Listeria monocytogenes[J]. Chinese Journal of Animal Infectious Diseases, 2019, 27(4): 23-31. |
| [29] |
张静玲, 李国明, 邱景富. 金黄色葡萄球菌生物膜形成能力及其耐甲氧西林相关性[J]. 中国消毒学杂志, 2018, 35(1): 14-16. Zhang JL, Li GM, Qiu JF. Research on relationship between methicillin resistance of Staphylococcus aureus clinical isolates and its biofilm forming ability[J]. Chinese Journal of Antisepsis, 2018, 35(1): 14-16. |