草酰乙酸水解酶是产生草酸的关键性酶[1]。在草酸合成的最后一步中,该酶能够将草酰乙酸水解,直接形成草酸[2],是真菌中特有的酶。在真菌的草酸代谢研究中,草酰乙酸水解酶也是研究的必要性酶。在核盘菌中,将草酰乙酸水解酶的基因敲除,草酸不再分泌,可以降低核盘菌对植物的侵害能力[3]。在黑曲霉中将该酶的基因敲除,可以避免草酸的产生,从而提高了生产中柠檬酸的纯度[4]。而相反,在黑曲霉中将草酰乙酸水解酶的基因过量表达,可以形成产草酸的独特菌株[5]。
真菌中草酸的形成主要是在细胞质中,利用细胞中的载体蛋白将草酸转运至胞外,继而在细胞外发挥作用[6]。而细胞质中草酰乙酸的来源可分为3类,一类是来自于细胞质中丙酮酸形成的草酰乙酸[7];一类是苹果酸在苹果酸脱氢酶形成的草酰乙酸[8];还有一类是线粒体中形成的柠檬酸经过穿梭,在柠檬酸裂解酶的作用下,形成的草酰乙酸[9]。而在真菌中,对于草酸的形成途径也有所不同,大致可分为两类:一类以黑曲霉中草酰乙酸水解酶单独作用形成的草酸[10];还有一类是以褐腐菌中草酰乙酸水解酶和乙醛酸脱氢酶或者是乙醛酸氧化酶一起作用形成的草酸[8, 11]。总的来说,草酰乙酸水解酶在真菌的草酸合成中起关键性作用。
香菇属于大型真菌,在中国食用菌中产量长期位居第一。属于偏酸性食用菌,木腐菌中属于白腐菌[6],在生长过程中,会产生一定量的草酸对环境pH进行调节[12]。而草酸在真菌生长过程中有很多作用,因此研究草酰乙酸水解酶对于了解香菇生长及育种有很大的帮助。在病原菌中对草酰乙酸水解酶的报道很多,而在食用菌中的报道中却没有。
本研究中克隆了香菇LeOAH1基因,分析其结构、序列特征、系统进化关系等基本生物学信息,并利用Real-time PCR方法检测LeOAH1基因在不同pH条件下的表达情况,并在体外进行了原核表达,初步探讨了该酶LeOAH1在香菇菌丝生长中的作用,旨在为进一步研究该基因的生物学功能奠定基础。
1 材料与方法 1.1 材料 1.1.1 供试菌种香菇(L.edodes)菌株为L808,25℃活化培养。大肠杆菌为Trans1-T1和BL21(DE3);质粒为pET-28a。
1.1.2 主要试剂与设备Difco TM标准PDA培养基(美国Becton公司)、LB Broth Powder(生工生物工程(上海)股份有限公司)、DNA提取试剂盒(DP305)(天根生物科技(北京)有限公司)、E.Z.N.A. Plant RNA Kit(美国Omega公司)、HiScript Ⅱ 1st Strand cDNA Synthesis Kit和ChamQ Universal SYBR qPCR Master Mix(南京诺唯赞公司)、Premix TaqTM(日本TaKaRa公司)、96孔反应板和QuantStudioTM 6 Flex Real-Time PCR(美国ABI公司)、核酸检测仪(Biodropsis BD-1000)(北京五洲东方科技发展有限公司)、高效液相色谱仪(Agilent 1100)。
1.2 方法 1.2.1 总DNA、RNA提取及cDNA合成使用DNA提取试剂盒提取菌丝的总DNA,使用E.Z.N.A. Plant RNA Kit提取菌丝的总RNA,依说明书进行操作。使用1%非变性琼脂糖凝胶电泳和核酸检测仪分别检测DNA、RNA的完整性和浓度。使用HiScript Ⅱ 1st Strand cDNA Synthesis Kit进行RNA的反转录,依说明书进行操作。提取后的DNA和反转录后的cDNA分别放于-20℃,保存待用。
1.2.2 LeOAH1的基因克隆通过同源序列比对,在转录组中找到了香菇中草酰乙酸水解酶的序列LE01Gene02176,命名为LeOAH1,并在NCBI中blastprimer进行引物设计(表 1)。根据所设计的引物,以cDNA和gDNA为模板进行扩增,克隆获得oah1基因。PCR扩增条件:95℃预变性3 min;94℃变性30 s,60℃退火40 s,72℃延伸100 s,34个循环;72℃延伸10 min。PCR扩增后,经1%的琼脂糖凝胶电泳分离,分别回收PCR产物,将回收产物连入pEASY-T1克隆载体,导入Trans1-T1克隆感受态细胞,挑选阳性菌落,由上海生工生物工程有限公司进行测序。
利用ExPAsy-ProtParam(https://web.expasy.org/protparam/)进行等电点和蛋白分子量预测;利用GSDS 2.0(http://gsds.cbi.pku.edu.cn/)进行基因结构分析;利用Bioedit软件进行氨基酸序列比对;利用Mega7软件中邻接法(Neighbor-Joining method)建立系统发育树;利用Wolfpsort(https://wolfpsort.hgc.jp/)和Cello(http://cello.life.nctu.edu.tw)两个网站进行亚细胞定位预测;利用SWISS-MODEL(https://swissmodel.expasy.org/)进行蛋白结构的预测[13]。
1.2.4 不同pH条件下,草酸含量测定和LeOAH1基因表达分析利用25 mmol/L磷酸盐缓冲液,使PDA培养基形成pH 2-8梯度。通过香菇菌丝在pH梯度培养基上的生长情况,还有对pH梯度和磷酸盐的双重考虑,选择了pH3、CK、pH6作为测定对象,分别测定3种pH梯度条件下香菇菌丝分泌胞外的草酸含量和oah1基因表达量。
草酸提取方法:取生长8 d的培养基,去掉玻璃纸,将菌丝与培养基分开,在培养基中心取直径为1 cm左右的琼脂块,将琼脂块捣碎,加入8 mL 0.2%稀硫酸,摇床25℃140 r/min,提取2 h,然后用滤纸进行过滤,离心管收集[14]。
草酸含量测定:采用高效液相的方法测定,有机酸分析柱:SB-Aq,4.6×150 mm,5 um;流动相:2 mmol/L硫酸(pH1.96)和纯甲醇(色谱级);配比:9﹕1;柱温:30℃,流速:0.3 mL/min,检测波长:210 nm。利用标准品配置梯度浓度,绘制标准曲线,测定草酸含量。
oah1基因表达量的测定:收集pH3、CK、pH6三个处理的菌丝,提取RNA,反转录后形成cDNA,放于-20℃,保存待用。RT-PCR操作按照ChamQ Universal SYBR qPCR Master Mix说明书进行,以β-actin作为内参基因,引物见表 1,反应体系:cDNA模板2 μL,上下游引物各0.4 μL(10 μmol/L),SYBR qPCR Master Mix 10 μL,ddH2O 7.2 μL。qPCR反应程序:95℃预变性3 min;95℃变性3 s,60℃退火32 s,40个循环;72℃延伸30 s,每个基因3次重复。RT-PCR结果参考2-ΔΔCt法进行分析[15]。
1.2.5 蛋白的原核表达及SDS-PAGE分析选择EcoRⅠ和NotⅠ作为质粒构建中的酶切位点,对表达载体pET-28a进行双酶切,纯化回收pET-28a载体片段和oah1基因片段,进行同源重组连接,获得重组原核表达载体pET-28a-oah1,转化BL21(DE3)感受态细胞。涂布至卡那霉素(100 μg/mL)抗性平板;将抗性平板上长出的单菌落挑取至含有卡那霉素(100 μg/mL)的LB液体培养基中,37℃、180 r/min震荡培养过夜;按照1.5%接种量将过夜培养物转接至含有卡那霉素的LB液体培养基中,37℃、180 r/min震荡3 h,至OD600值达到0.6-0.7左右;加入1 mmol/L IPTG,180 r/min过夜诱导,选择了16℃、20℃进行诱导,筛选合适的诱导温度,离心收集菌体。利用PBS缓冲液重悬菌体后,通过超声破碎法(超声1 s,停3 s,共10 min)破碎菌体,然后12 000 r/min离心分离上清和沉淀。通过SDS-PAGE检测LeOAH1蛋白表达情况。
1.2.6数据分析实验数据重复均在3次以上,利用Graphpad Prism 6软件进行数据分析
2 结果 2.1 香菇LeOAH1基因克隆及基因结构分析收集菌丝提取总RNA,采用核酸检测仪对其质量、纯度与浓度进行检测,1%的琼脂糖凝胶电泳检测其完整性。测序结果显示LeOAH1的gDNA长度为1 906 bp;cDNA长度为1 356 bp(图 1-A);含有8个内含子,9个外显子(图 1-B)。
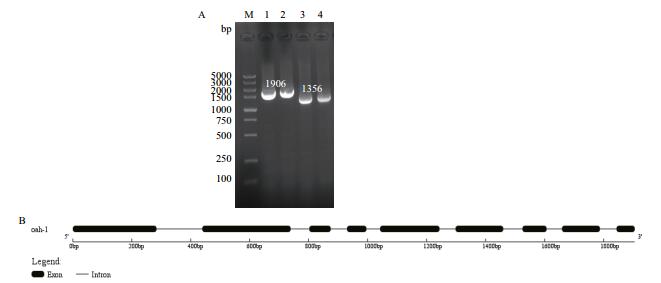
|
| A:1-2:gDNA;3-4:cDNA 图 1 香菇LeOAH1基因扩增结果(A)和结构分析(B) |
通过SMART氨基酸序列分析,在图 2中213-361aa之间含有超家族所共有的保守结构域。在NCBI中检索BLAST,获得与香菇LeOAH1同源的11种真菌OAH的氨基酸序列,通过氨基酸序列比对分析,与NCBI中日本香菇菌株NBRC 111202相似度最高(82%),其次为灰光柄菇,相似度为64%,且发现OAH结构上在真菌中还是较为保守的(图 3)。
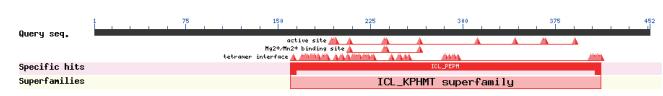
|
| 红色为保守结构域 图 2 LeOAH1蛋白序列的结构分析 |

|
| 红色为保守序列;黄色为相似序列 图 3 12种真菌OAH氨基酸序列比对 |
利用Mega7软件构建OAH的系统发育进化树(图 4),确认了该序列为草酰乙酸水解酶基因。由于香菇属于白腐菌,与褐腐菌同属于担子菌,通过系统发育树分析,与白腐菌中的灰光柄菇、牛排菌和褐腐菌中癞拟层孔菌的亲缘关系较近;而与子囊菌中黑曲霉、核盘菌和灰霉菌的亲缘关系较远。
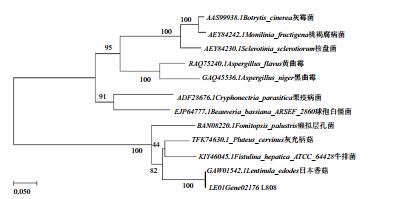
|
| 图 4 LeOAH1蛋白的同源性分析 |
LeOAH1的ORF区为1 356 bp,编码氨基酸为451aa,预测了LeOAH1等电点是6.80;预测蛋白分子量是48.903 46 kD。OAH属于异柠檬酸裂解酶/磷酸烯醇丙酮酸变位酶超家族成员,OAH蛋白为同源四聚体结构,氨基酸序列具Mg2+/Mn2+的结合位点(图 5)。通过Wolfpsort和Cello进行预测,该蛋白的亚细胞定位在细胞质中。分子式为C2156H3449N605O635S28,其中丙氨酸含量最高11.3%。蛋白二级结构主要由4种构象组成,分别是α-螺旋(Hh)、无规则卷曲(Cc)、延伸链(Ee)、β-折叠(Tt),其中,α-螺旋占比例最高为40.08%,含有184个氨基酸,无规则卷曲占比例34.59%,含有156个氨基酸,延伸链占比例17.52%,含有79个氨基酸,β-折叠占比例最低为7.10%,含有32个氨基酸

|
| 图 5 LeOAH1蛋白的三维结构预测 |
对不同pH条件下香菇菌丝的生长进行观察,发现当培养基pH为4时,香菇菌丝生长速度最快;其次是CK、pH5和pH3,而pH6的菌丝生长速度最慢,pH较低(pH2)与较高(pH7和pH8)时均不能生长。
综合考虑,对能够生长的5个处理进行草酸含量分析发现,草酸含量随着pH的上升而不断增加;从中选择了pH3、pH6作为处理组,CK作为对照组,其pH在5左右;LeOAH1的基因表达量也随着pH的上升而增多(图 6)。
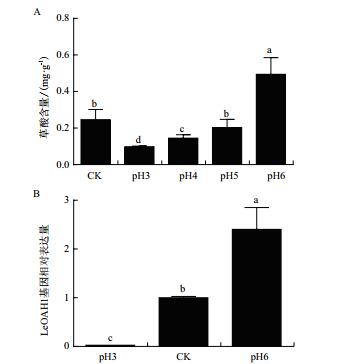
|
| 基因表达量的计算以无任何处理的菌丝CK作为对照,平均值±SD,不同小写字母代表显著性差异(P < 0.05,n=3) 图 6 不同pH条件下香菇菌丝的草酸含量(A)和LeOAH1的基因表达量(B) |
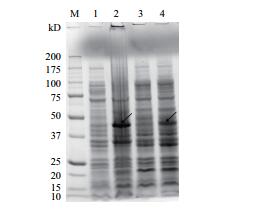
转入重组质粒pET28a-oah1的菌体,用IPTG(最终浓度1mmol/L)诱导重组蛋白表达,经SDS-PAGE电泳分析,诱导12-16 h后,在20和16℃均出现1条与预期大小48.9 kD相符的蛋白(图 7)。说明基因可体外诱导表达,比较发现20℃更适合蛋白诱导。后期研究其酶活和理化性质需要进一步纯化。
|
| 1,3:未诱导;2:20℃诱导;4:16℃诱导,箭头:诱导的目的蛋白 图 7 LeOAH1基因诱导表达产物SDS-PAGE分析 |
在真菌中草酸合成的来源,分别是草酰乙酸[16]和乙醛酸[17],而形成草酸的主要是草酰乙酸[18]。在对灰霉菌中草酸代谢途径的研究中,对草酰乙酸水解酶的进行基因敲除,草酸不再分泌,明确了草酸在灰霉菌中是由草酰乙酸产生的唯一代谢途径[19]。而在产黄青霉中报道,将oah基因敲除则草酸合成受到抑制,菌株不再产生草酸,而如果将该基因进行回补,则草酸又能顺利产生,明确了草酸在产黄青霉中合成及来源的基本路径[20]。因此,为了研究草酸在香菇中的代谢途径,后期可能需要对该基因进行敲除或者干扰,确定其基本的代谢途径。由于在褐腐菌中报道了草酸的来源主要是草酰乙酸和乙醛酸,而乙醛酸是在乙醛酸脱氢酶的作用下形成草酸[21]。文献中已报道乙醛酸脱氢酶与细胞色素结合,才可对乙醛酸发生催化反应[22],在香菇基因组中未发现乙醛酸脱氢酶,但是有类似的细胞色素,推测该细胞色素可能会催化该反应。因此干扰该基因对于研究香菇中草酸代谢途径会有很大的帮助。
本实验采用了基因克隆的手段,获得了香菇LeOAH1基因,明确了该基因的基本结构和氨基酸序列。通过生物信息学的分析,发现该蛋白属于异柠檬酸裂解酶/磷酸烯醇丙酮酸变位酶超家族成员[23]。该家族主要催化P-C或C-C键的形成/裂解;主要成员有磷酸烯醇丙酮酸突变酶(Phosphoenolpyruvate mutase,PEPM),磷酸壬酸丙酮酸水解酶(Phosphonopyruvate hydrolase,PPH),羧基PEP突变酶(CarboxyPEP mutase,CPEP mutase),草酰乙酸水解酶(Oxaloacetate hydrolase,OAH),异柠檬酸裂合酶(Isocitrate lyase,ICL)和2-甲基异柠檬酸裂合酶(2-methylisocitrate lyase,MICL)[24]。而该LeOAH1蛋白具有Mg2+/Mn2+的结合位点,属于不稳定性蛋白,结构为同源四聚体。同源序列比对发现,除与日本香菇品种NBRC111202的蛋白序列相似度高达(82%)外,还与灰光柄菇的蛋白序列相似度较高(64%)。而与病原菌中核盘菌和灰霉菌的相似度不高,这可能是由于香菇与灰光柄菇同属于担子菌的关系,与子囊菌的亲缘关系较远,因此子囊菌与担子菌之间的OAH蛋白差异较为明显。通过高效液相和荧光定量PCR分析,不同pH条件下香菇菌丝草酸分泌量和oah的基因表达量成正相关,也确定了香菇菌丝草酸分泌随pH变化的关系。在实验中发现当pH为3时,基因的相对表达量接近于零,而此时草酸的含量也非常低,与黑曲霉中报道当pH大于等于4时LeOAH1才能进行基因表达相一致[25]。这也间接说明oah的调控可能受到环境pH的影响,这与灰霉菌中报道oah基因的调控可能跟响应pH的转录因子pacc有很大关系相一致[26]。
草酸在真菌中的作用各不相同,可以调节环境pH[27],溶解金属氧化物且具有解毒的作用[28],促进木质纤维素的降解,特别是促进锰过氧化物酶的活力[29],病原菌还能利用草酸侵染及破坏植物组织[30]。研究香菇草酰乙酸水解酶,对于研究其草酸代谢及调控的是必不可少的。LeOAH1基因表达量与草酸含量是成正比关系,香菇中分泌草酸调节环境pH,当oah基因过量表达时,草酸分泌量就会过量增多。本实验对该酶的基因进行克隆,并且分析了解草酰乙酸水解酶与草酸之间的关系,为研究香菇菌丝生长及生理功能研究奠定基础。
4 结论克隆了香菇中LeOAH1基因,确定了该基因的gDNA、cDNA长度和氨基酸数量,生物信息学分析了该基因在不同pH条件下的表达量及与草酸分泌量的密切关系,并对该基因进行原核表达,确认其能够进行体外诱导,最适诱导温度为20℃。
| [1] |
Jarosz-Wilkolazka A, Gadd GM. Oxalate production by wood-rotting fungi growing in toxic metal-amended medium[J]. Chemosphere, 2003, 52(3): 541-547. |
| [2] |
Dutton MV, Evans CS. Oxalate production by fungi:Its role in pathogenicity and ecology in the soil environment[J]. Canadian Journal of Microbiology, 1996, 42(9): 881-895. |
| [3] |
Liang X, Liberti D, Moyi LI, et al. Oxaloacetate acetylhydrolase gene mutants of Sclerotinia sclerotiorum do not accumulate oxalic acid, but do produce limited lesions on host plants[J]. Molecular Plant Pathology, 2015, 16(6): 559-571. |
| [4] |
Pedersen H, Christensen B, Hjort C, et al. Construction and characterization of an oxalic acid nonproducing strain of Aspergillus niger[J]. Metabolic Engineering, 2000, 2(1): 34-41. |
| [5] |
Kobayashi K, et al. Oxalic acid production by citric acid-producing Aspergillus niger overexpressing the oxaloacetate hydrolase gene oahA[J]. J Ind Microbiol Biotechnol, 2014, 41(5): 749-756. |
| [6] |
Watanabe T, et al. Oxalate efflux transporter from the brown rot fungus Fomitopsis palustris[J]. Appl Environ Microbiol, 2010, 76(23): 7683-7690. |
| [7] |
周桥胜.球孢白僵菌pH响应转录因子BbPacC的功能研究[D].重庆: 西南大学, 2015. Zhou QS. Characterization of pH-responsive transcription factor, BbPacC, in the entomopathogenic fungus Beauveria bassiana[D]. Chongqing: Southwest University, 2015. |
| [8] |
Munir E, et al. A physiological role for oxalic acid biosynthesis in the wood-rotting basidiomycete Fomitopsis palustris[J]. Proc Natl Acad Sci USA, 2001, 98(20): 11126-11130. |
| [9] |
伏贝贝.改造酿酒酵母乙酰辅酶A合成途径及其在香叶醇生产中的应用[D].济南: 齐鲁工业大学, 2018. Fu BB. Engineering acetyl-CoA synthetic pathway and its application in geraniol production in Saccharomyces cerevisiae[D]. Jinan: Qilu University of Technology, 2018. |
| [10] |
Kubicek CP, Schreferl-Kunar G, Wöhrer W. Evidence for a cytoplasmic pathway of oxalate biosynthesis in Aspergillus niger[J]. Appl Environ Microbiol, 1988, 54(3): 633-637. |
| [11] |
Akamatsu Y, Shimada M. Partial purification and characterization of glyoxylate oxidase from the brown-rot basidiomycete Tyromyces palustris[J]. Phytochemistry, 1994, 37(3): 649-653. |
| [12] |
段应策, 胡姿仪, 张瑞颖.草酸是香菇调控环境pH促进菌丝生长的重要因素[C].多彩菌物美丽中国——中国菌物学会2019年学术年会论文摘要, 2019. Duan YC, Hu ZY, Zhang RY. The oxalate is involved in regulating ambient pH to improve mycelial growth for Lentinula edodes[C].Colorful Fungi Beautiful China-Abstract of papers presented at the 2019 Academic Annual Meeting of Mycological Society of China, 2019. |
| [13] |
王竹青, 陈云, 杨玉婷. 甘蔗抗坏血酸过氧化物酶基因(ScAPX)的克隆及表达分析[J]. 农业生物技术学报, 2015, 23(2): 170-180. Wang ZQ, Chen Y, Yang YT. Cloning and expression analysis of peroxidase gene(ScAPX1)from sugarcane[J]. Journal of Agricultural Biotechnology, 2015, 23(2): 170-180. |
| [14] |
Guggiari M, Bloque R, Aragno M. Experimental calcium-oxalate crystal production and dissolution by selected wood-rot fungi[J]. Int Biodeterior Biodegradation, 2011, 65(6): 803-809. |
| [15] |
侯志浩, 赵梦然, 邬向丽. 热胁迫下糙皮侧耳实时荧光定量PCR内参基因的选择[J]. 食用菌学报, 2019(3): 11-18. Hou ZH, Zhao MR, Wu XL. Selection of reference genes for real-time quantitative PCR of Pleurotus ostreatus under heat stress[J]. Acta Edulis Fungi, 2019(3): 11-18. |
| [16] |
Lenz H, et al. Partial purification and some properties of oxalacetase from Aspergillus niger[J]. Eurjbiochem, 1976, 1: 225-236. |
| [17] |
Balmforth AJ, Thomson A. Isolation and characterization of glyoxylate dehydrogenase from the fungus Sclerotium rolfsii[J]. Biochemical Journal, 1984, 218(1): 113-118. |
| [18] |
Mäkelä M, Galkin S, Hatakka A. Production of organic acids and oxalate decarboxylase in lignin-degrading white rot fungi[J]. Enzyme and Microbial Technology, 2002, 30(4): 542-549. |
| [19] |
Han Y, Joosten HJ, Niu W. Oxaloacetate Hydrolase, the C-C bond lyase of oxalate secreting fungi[J]. Journal of Biological Chemistry, 2007, 282(13): 9581-9590. |
| [20] |
Gombert AK, Veiga T, Puig-Martinez M. Functional characterization of the oxaloacetase encoding gene and elimination of oxalate formation in the β-lactam producer Penicillium chrysogenum[J]. Fungal Genet Biol, 2011, 48(8): 831-839. |
| [21] |
Munir E, Yoon JJ, Tokimatsu T. New role for glyoxylate cycle enzymes in wood-rotting basidiomycetes in relation to biosynthesis of oxalic acid[J]. J Wood Sci, 2001, 47(5): 368-373. |
| [22] |
Tokimatsu T, Nagai Y, Hattori T. Purification and characteristics of a novel cytochrome c dependent glyoxylate dehydrogenase from a wood-destroying fungus Tyromyces palustris[J]. FEBS Letters, 1998, 437(1-2): 117-121. |
| [23] |
Narayanan B, Niu W, Joosten HJ. Structure and function of 2, 3-dimethylmalate lyase, a PEP mutase/isocitrate lyase superfamily member[J]. J Mol Biol, 2009, 386(2): 486-503. |
| [24] |
Lu Z, Feng X, Song L, et al. Diversity of function in the isocitrate lyase enzyme superfamily:The Dianthus caryophyllus petal death protein cleaves α-keto and α-hydroxycarboxylic acids[J]. Biochemistry, 2005, 44(50): 16365-16376. |
| [25] |
Pedersen H, Hjort C, Nielsen J. Cloning and characterization of oah, the gene encoding oxaloacetate hydrolase in Aspergillus niger[J]. Molecular and General Genetics, 2000, 263(2): 281-286. |
| [26] |
Rascle C, Dieryckx C, Dupuy JW, et al. The pH regulator PacC:a host-dependent virulence factor in Botrytis cinerea[J]. Environmental Microbiology Reports, 2018, 10(5): 555-568. |
| [27] |
Alkan N, et al. Virulence regulation of phytopathogenic fungi by pH[J]. Antioxid Redox Signal, 2013, 9: 1012-1025. |
| [28] |
Jarosz-Wilkoazka A, Marcin G. Organic acids production by white rot basidiomycetes in the presence of metallic oxides[J]. Canadian Journal of Microbiology, 2006, 52(8): 779-785. |
| [29] |
Kuan IC, Tien M. Stimulation of Mn peroxidase activity:A poss-ible role for oxalate in lignin biodegradation[J]. Proc National Acad Sci USA, 1993, 90(4): 1242-1246. |
| [30] |
Guimarães RL, Stotz HU. Oxalate Production by Sclerotinia sclerotiorum deregulates guard cells during infection[J]. Plant Physiology, 2004, 136(3): 3703-3711. |





