2. 中国科学院大学,北京101408
2. University of the Chinese Academy of Sciences, Beijing 101408
滇牡丹(Paeonia delavayi)属芍药科芍药属(Paeonia)牡丹组(Sect. Moutan DC.)植物,是中国西南地区特有的野生资源植物,是芍药属分布最南的一个牡丹类群[1]。作为一种民族药材,其根皮入药称为丹皮,用于镇痛、镇静、抗炎、退热、止血、降糖,具有很好的药用价值[2]。同时滇牡丹是油用牡丹新品种培育的重要种质资源,也是开发食用油的潜在木本油料植物资源之一。牡丹籽油作为一种多功能食用油,含有至少14种脂肪酸成分,包括油酸、亚油酸、亚麻酸、棕榈酸、硬脂酸等,其中亚麻酸的含量极高[3],油脂品质优良,营养丰富。目前,关于滇牡丹的研究主要集中在栽培学[4]、孢粉学[5]、病害防治[6]及遗传多样性[7-8]方面,关于滇牡丹的分子生物学研究的报道较少。目前将分子生物学技术应用于植物基因功能的研究较为普遍,而研究植物生长发育过程中关键基因的功能是开发利用这些基因的前提。功能基因表达量及表达模式是确定基因功能的重要指标,内参基因是检测基因表达量的必要参照,在检测目标基因表达水平变化中起到校正作用,筛选合适的内参基因是研究基因表达的关键,能够减少实验误差,提高结果可靠性[9]。因此,筛选相对稳定的内参是研究基因表达分析的前提[10]。
实时荧光定量PCR技术具有准确性强、操作简单、结果可靠且成本低廉的特点。目前已经被广泛应用于分子生物学研究的多个领域。聚合酶链式反应(Polymerase chain reaction,PCR)自从被发现后[11],就迅速成为科学研究的热点,并被进一步应用于医学研究。目前在植物学研究中的应用也越来越广泛,常见于植物抗逆机理研究、病原菌的检测、植物与微生物互作机理研究、植物抗病性检测、信号转导、环境对植物基因表达的影响等方面[12],解决了许多植物病理学和植物育种中的热点和焦点问题。大量文献表明荧光定量PCR技术已应用于主要作物如缺素条件的水稻[13],转基因的玉米[14]、大豆[15]等,近年来在林木如杉木[16]和黄梁木[17]甚至中药中如川续断[18]、黄精[19]和穿心莲[20]也都采用了实时荧光定量PCR技术展开相关研究。
在植物的荧光定量PCR实验中,常用的内参基因主要包括肌动蛋白基因(ACT)、3-磷酸甘油醛脱氢酶基因(GAPDH)、转录延伸因子基因(EF1α)及β微管蛋白基因(TUB)等,这些常用的内参基因或参与细胞的基本代谢,或是作为细胞的组成成分,根据实验条件和材料的特点选择相应合适的内参进行标准化是得到准确荧光定量分析结果的关键[21]。基于滇牡丹种子发育过程中的基因转录组数据并参考相关文献,本研究选择了8个看家基因(Housekeeping genes,HKGs)即GAPC、PEPC、CYC、ACT、EF2α、TUB、ACP1和RPL1作为候选内参基因,选择Ge Norm等3个程序评估了8个候选内参基因在滇牡丹不同发育时期的种子中荧光定量PCR检测结果的稳定性,为检测滇牡丹不同发育时期种子中功能基因的表达分析提供参考。
1 材料与方法 1.1 材料采集于云南省玉溪市澄江县梁王山(东经102°52' 45" ,北纬24°43' 57" )野生分布的滇牡丹种子,牡丹开花后每隔15 d取一次样,分别取开花后25、40、55、70和85 d的种子。将所取的滇牡丹种子样品置于冷冻管中,放于液氮中速冻,冻存后备用。
1.2 方法 1.2.1 RNA的提取不同发育时期的滇牡丹种子样品RNA提取采用TIANGEN公司的多糖多酚总RNA提取试剂盒(DP441),按照说明书进行操作。获得样品总RNA。
1.2.2 总RNA的检测获得总RNA后,用1%的琼脂糖电泳和紫外分光光度计检测RNA样品质量。测定总RNA的浓度和纯度,A260/280≈2.0-2.1,A260/230≈2.0,电泳检测结果显示总RNA完整性较好,条带清晰,无DNA和其他杂质污染,28S与18S条带比值约为2:1,适合用作进一步反应。然后将提取的RNA样品置于-80℃储藏备用。
1.2.3 反转录cDNA样品反转录采用TIANGEN公司的FastKing gDNA Dispelling RT SuperMix(KR118)试剂盒按说明书反转录合成cDNA第一链。反应体系如下:总反应体系20 µL,5×FastKing-RT SuperMix 4 µL,总RNA 1 µg,加RNA-free纯水补足到20 µL。反应程序为42℃、15 min:95℃、3 min。反应的第一步为去除基因组并进行反转录反应,第二步进行了酶灭活,完成cDNA第一链的合成。将反转录产物即cDNA置于-20℃下保存,以备进行后续PCR反应。
1.2.4 内参基因的选择和引物设计根据滇牡丹种子全长转录组数据库(未发表)选择了GAPC、PEPC、CYC、ACT、EF2α、TUB、ACP1和RPL1共8个候选基因作为滇牡丹种子不同发育时期的内参基因。利用Primer Premier 5.0软件进行特异性引物的设计,标准如下:引物Tm 55±5℃,碱基长度18-22 bp,GC含量介于40%-60%之间。扩增产物长度在200-300 bp之间。由擎科生物科技有限公司(昆明)负责合成,引物信息见表 1、2。对设计的引物进行PCR扩增,通过凝胶电泳验证其特异性。
本实验采用QIAGEN公司的QuantiNova SYBR Green PCR Kit试剂盒,在384孔板的LightCycler 384 System上进行点样,在罗氏LightCycler480 II实时荧光定量PCR仪器上设置参数,进行荧光定量PCR反应,每个样品设置3个重复。反应体系为:总体系10 µL。包含上下游引物各1 µL,1 µL cDNA模板,5 µL 2× SYBR Green PCR Master Mix,1 µL QN ROX Reference Dye,1 µL RNase-free water。反应条件:预变性95℃、2 min;变性95℃、5 s;退火60℃、10 s,共40个循环。延伸阶段收集荧光信号,反应结束后获得溶解曲线。
1.2.6 数据分析一般认为扩增效率E应介于90%-110%之间。采用GeNorm等3个软件对候选内参基因的表达稳定性进行分析。首先计算ΔCt,根据公式E=2-ΔCt,将数据输入Excel表格,导入分析软件进行分析,获得候选内参基因的表达值,判定出稳定表达的内参基因。
2 结果 2.1 RNA质量检测选择开花后25、40、55、70、85 d的种子,利用TIANGEN公司的多糖多酚总RNA提取试剂盒(DP441)方法提取上述样本的总RNA,并对获得的RNA的浓度和质量进行检测,获得的RNA条带(图 1),质量完好可用做下一步反应。
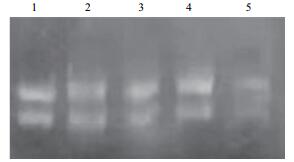
|
| 1-5:滇牡丹开花后25、40、55、70和85 d的种子提取的RNA 图 1 滇牡丹总RNA样品的电泳检测结果 |
为了保证荧光定量分析的准确性,本实验用1.0%的琼脂糖凝胶电泳和测序对8个候选内参基因的引物特异性进行验证,结果显示扩增产物段大小符合预计(图 2),无引物二聚体和非特异扩增片段。
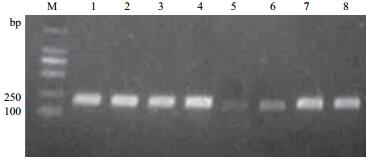
|
| M:DL2501 Marker;1-8:8个候选内参基因GAPC、PEPC、CYC、TUB、EF2α、ACP、ACT、RPL1的PCR产物 图 2 滇牡丹8个候选内参基因PCR产物的电泳检测结果 |
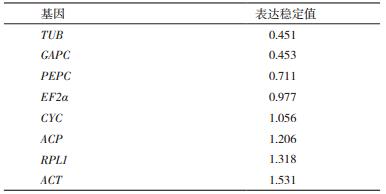
基因的表达丰度用Ct表示,对各基因的表达丰度进行统计发现(图 3),在滇牡丹种子不同发育时期,8个内参基因的表达水平存在一定变化,其中TUB基因的表达丰度相对较高,而CYC和EF2α内参基因的表达丰度较低。

|
| 图 3 八个内参基因在滇牡丹种子不同发育时期的Ct值变化 |
为了筛选在种子不同发育时期均能稳定表达的内参基因,本实验利用GeNorm和Normfinder和BestKeeper软件分别对开花后25、40、55、70和85 d的种子中的8个候选内参基因的稳定性进行了分析。
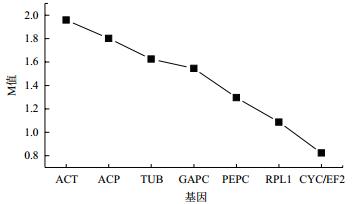
2.3.1 GeNorm分析结果GeNorm软件用M值评估各表达稳定性,分析结果显示,8个候选内参基因在种子不同发育时期中M值有所差异,根据表达稳定性大小排序(M值越小越稳定),由高到低依次为CYC/EF2(0.824)>RPL1(1.087)>PEPC(1.296)>GAPC(1.545)>TUB(1.625)>ACP(1.801)>ACT(1.958)(图 4)。
|
| 图 4 GeNorm软件分析滇牡丹种子不同发育时期内参基因的表达稳定值排序 |
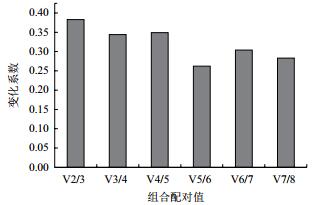
GeNorm软件生成内参基因的配对变异数(Vn/n+1)柱形图,可以根据柱形图选择的最适合内参基因数目。GeNorm软件可以通过分析计算所得的配对差异值Vn/n+1来确定该实验条件下所需内参基因的最适个数,以减少使用单一内参基因所引起的偏差和波动。选择阈值为0.15,当Vn/n+1>0.15,表明最适合的内参基因个数是n+1个;Vn/n+1 < 0.15,表明最适合的内参基因有n个。GeNorm柱形图分析结果显示当选择V2/3值(0.383)大于程序所推荐值0.15(图 5),最合适的内参组合基因数目是3个。滇牡丹种子不同发育时期表达最稳定的基因组合是RPL1,EF2和CYC,最不稳定的基因是ACT。
|
| 图 5 GeNorm软件分析确定滇牡丹种子不同发育时期的最适内参基因的数目 |
Normfinder是基于方差分析对内参基因的表达稳定性直接进行评价,通过程序计算获得稳定值(Stability,S),稳定值越低代表基因的表达稳定性越高,但该软件只能选择一个合适的内参基因作标准。分析结果显示,TUB内参基因最稳定,而ACT基因最不稳定(表 3)。该软件的结果与GeNorm得出的结果相比稍有不同,这可能与2种软件的计算方法的差异有关。但关于ACT基因在8个基因中表达最不稳定的结果。两个软件的结果是一致的。
BestKeeper软件主要通过该程序计算可以获得标准变异系数(SD)和变异相关系数(CV)以及每个基因之间产生配对的相关系数(r),以这3个参数来判断内参基因的稳定性。判定原则为相关系数越大、标准偏差和变异系数越小,内参基因稳定性越好,反之,稳定性越差;其中SD值的默认阈值为1.0,低于该值即认为表达稳定;当SD>1时,则该内参基因表达不稳定。BestKeeper分析结果(表 4)显示只有CYC、EF2α基因的SD值小于1,根据r值排序,EF2α>CYC,综合结果发现EF2α和CYC基因表达最稳定,而ACT基因在8个内参基因中表达稳定性最差。
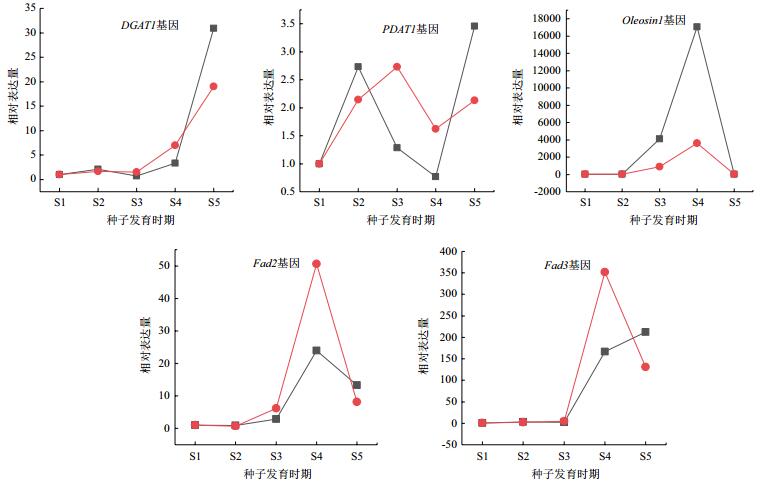
以通过稳定性综合评价得到的表现最稳定的CYC和EF2α作为内参基因,选择滇牡丹种子中油脂合成途经相关基因(表 5),设计引物(表 6),对其表达量进行RT-qPCR分析,结果表明,以不同基因作为内参,5个油脂合成途径相关基因在RT-qPCR分析中的表达趋势基本一致,相对表达量结果相近(图 6)。
|
| 红色:CYC做内参时各基因表达量;黑色:EF2α做内参时各基因表达量 图 6 滇牡丹种子中油脂合成相关基因的荧光定量PCR分析结果(分别以CYC和EF2α为参照) |
目前,应用最为广泛的基因表达分析手段是荧光定量PCR技术,但是RNA不完整、污染以及反转录效率低等因素常影响其准确性。因此,选用合适的内参基因是保证荧光定量PCR分析结果可靠性的重要前提[22]。为挖掘适用于滇牡丹种子不同发育时期基因表达分析的内参基因,本研究以滇牡丹不同发育阶段的种子为材料,选择8个候选内参基因,利用Genorm、Normfinder和BestKeeper软件分析了它们在滇牡丹成种子不同发育时期的表达稳定性。根据各软件评价标准[23-25],综合3个软件分析的结果,成功筛选出了符合要求的内参基因。3个软件分析的结果表明,在种子的不同发育时期中能够稳定表达的基因是CYC基因和EF2α基因,3个软件分析结果都显示ACT基因的稳定性较差,说明该基因不适合作为滇牡丹种子的内参基因。
肌动蛋白ACT基因是植物中被广泛应用的内参基因。在“金魁”猕猴桃(Actinidia deliciosa)中表达最为稳定[26],在不同发育时期的银杏(Ginkgo biloba)雌株叶片中也是表达最稳定[27],该结论同样也在4种木本油料种子油茶(Camellia oleifera)、沙棘(Hippophae rhamnoides)、文冠果(Xanthoceras sorbifolia)和油用牡丹(Paeonia suffruticosa)中得到了验证[28],而在本实验中ACT基因的表达极不稳定,这进一步验证了同一内参基因在不同实验材料中表达稳定性不一定相同的结论。
CYC基因(亲环素),又称环孢素A结合蛋白,也写作CYP等,具有多种生物学活性,存在于细胞质和各个细胞器内。CYC基因在甜樱桃的营养器官中是最适宜的内参基因[28],在大豆种子发育过程中也是表达最稳定的[15],这都与本研究的结果一致。而EF2α基因(Translation elongation factor 2),即翻译延伸因子2,也写作EF2,是编码蛋白质合成过程中一类蛋白质因子的基因。因为表达量恒定,所以是荧光定量PCR中常用的内参基因之一。之前有关于萝卜的研究发现,在不同品种、组织类型、光周期和春化处理及不同发育阶段验证8个候选内参基因表达稳定性时,EF2在不同的实验条件下均能稳定表达[29]。本实验中,EF2同样在滇牡丹种子的不同发育时期稳定表达。
已有研究报道了牡丹组中不同物种基因表达分析的最适内参基因,王彦杰等[30]以牡丹(Paeonia suffruticosa Andr.)的不同组织(根、茎、叶和花瓣)为材料,利用荧光定量PCR技术探讨了5种常用看家基因的表达情况,认为各实验条件下使用UBQ和GAPDH两个表达最稳定的基因组合。评估牡丹的10个内参基因发现GAPDH和UBC被认为是在“凤丹”(Paeonia ostii)和“西施”(Paeonia ostii)品种中最稳定的两个参考基因[31]。张莞晨等[27]研究油用牡丹(Paeonia suffruticosa)种子的7个候选内参基因的稳定性,发现表达最稳定的基因是ACT。这些与本实验获得的结果有很大的差异,有可能是因为实验所用的材料不同,说明滇牡丹与油用牡丹虽然同属于芍药属植物,但二者之间存在一定的差异性。本研究以筛选到的表现最稳定的CYC和EF2α基因作为内参,对滇牡丹转录组测序数据中筛选到的油脂合成途径中的部分相关基因表达量进行荧光定量PCR验证,发现5个油脂合成相关的基因的荧光定量PCR结果的表达趋势基本一致,表达量相近,表明本研究筛选出的内参基因可用作滇牡丹之间油脂合成相关基因表达分析的内参基因,也为牡丹组其他植物油脂合成相关基因表达量分析时内参基因的筛选提供了参考。
4 结论本实验通过3个软件评估了8个传统内参基因在滇牡丹种子中表达的稳定性,获得了两个最适合滇牡丹种子的内参基因CYC和EF2。
| [1] |
潘温文. 中国西南地区特有野生花卉——滇牡丹[J]. 园林, 2015(12): 56-58. Pan WW. Paeonia delavayi—endemic wild flowers in southwest China[J]. Landscape Architecture, 2015(12): 56-58. |
| [2] |
吴少华, 吴大刚, 陈有为, 等. 紫牡丹的化学成分研究[J]. 中草药, 2005(5): 648-651. Wu SH, Wu DG, Chen YW, et al. Chemical constituents of Paeonia delavayi[J]. Chinese Traditional and Herbal Drugs, 2005(5): 648-651. |
| [3] |
张水滔, 徐娟, 高明, 等. 滇牡丹籽油的脂肪酸组成及不同天然抗氧化剂对其货架期的影响[J]. 中国油脂, 2018, 43(3): 124-126. Zhang ST, Xu J, Gao M, et al. Fatty acid composition of peony seed oil and itsshelf life affected by different natural antioxidants[J]. China Oils and Fats, 2018, 43(3): 124-126. |
| [4] |
潘温文, 李伟, 谭芮, 等. 不同处理对滇牡丹种子萌发及幼苗生长的影响[J]. 西部林业科学, 2014, 43(2): 48-52. Pan WW, Li W, Tan R, et al. Effects of different treatments on seed germination and seedling growth of Paeonia delavayi[J]. Journal of West China Forestry Science, 2014, 43(2): 48-52. |
| [5] |
张艳丽.滇牡丹花色类群遗传背景分析[D].北京: 中国林业科学研究院, 2011. Zhang YL. Analysis genetic backgroud to flower color groups in Paeonia delavayi Franch. [D]. Beijing: Chinese Academy of Forestry, 2011. |
| [6] |
缪福俊, 安曼云, 华梅, 等. 诱抗剂BTH对滇牡丹3种主要病害的诱抗研究[J]. 西部林业科学, 2017, 46(3): 116-120. Miao FJ, An MY, Hua M, et al. The Study of the inducer BTH on resistance to the three main disease of Paeonia delavayi[J]. Journal of West China Forestry Science, 2017, 46(3): 116-120. |
| [7] |
李奎.滇牡丹保护生物学与遗传多样性研究[D].北京: 中国林业科学研究院, 2013. Li K. Research on conservation biology and genetic diversity of Paeonia delavayi complex(Paeoniaceae)[D]. Beijing: Chinese Academy of Forestry, 2013. |
| [8] |
任秀霞, 张盈, 薛璟祺, 等. 滇牡丹天然居群的遗传多样性分析[J]. 植物遗传资源学报, 2015, 16(4): 772-780. Ren XX, Zhang Y, Xue JQ, et al. Genetic diversity analysis of natural populations in Paeonia delavayi[J]. Journal of Plant Genetic Resources, 2015, 16(4): 772-780. |
| [9] |
Yoo WG, Kim TI, Li S, et al. Reference genes for quantitative analysis on Clonorchis sinensisgene expression by real-time PCR[J]. Parasitology Research, 2009, 104(2): 321-328. DOI:10.1007/s00436-008-1195-x |
| [10] |
杨晶, 卢玉彬, 迟孟涵, 等. 红花种子不同发育时期内参基因表达稳定性分析[J]. 中草药, 2017, 48(9): 1845-1850. Yang J, Lu YB, Chi MH, et al. Analysis on stability of reference genes in different developmental stages of seeds from Carthamus tinctorius[J]. Chinese Traditional and Herbal Drugs, 2017, 48(9): 1845-1850. |
| [11] |
Saiki R, Scharf S, Faloona F, et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia[J]. 1985, 230(4732): 1350-1354.
|
| [12] |
胡丹丹, 顾金刚, 姜瑞波, 等. 定量RT-PCR及其在植物学研究中的应用[J]. 植物营养与肥料学报, 2007(3): 520-525. Hu DD, Gu JG, Jiang RB, et al. Quantitative RT-PCR and its application in botany research[J]. Journal of Plant Nutrition and Fertilizers, 2007(3): 520-525. |
| [13] |
魏毅东, 陈玉, 郭海萍, 等. 水稻缺素胁迫下实时荧光定量RT-PCR的内参基因的选择[J]. 农业生物技术学报, 2013, 21(11): 1302-1312. Wei YD, Cheng Y, Guo HP, et al. Selection of reference genes for real-time quantitative RT- PCR in rice(Oryza sativa L. ssp. japonica)under nutrient deficiency[J]. Journal of Agricultural Biotechnology, 2013, 21(11): 1302-1312. |
| [14] |
常丽娟, 宋君, 张富丽, 等. 实时荧光定量PCR方法检测转基因玉米MIR604[J]. 浙江农业学报, 2017, 29(11): 1769-1774. Chang LJ, Song J, Zhang FL, et al. A real-time fluorescent quantitative PCR for detection of genetically modified maize line MIR604[J]. Acta Agriculturae Zhejiangensis, 2017, 29(11): 1769-1774. |
| [15] |
王骁.基于实时荧光定量PCR的大豆内参基因筛选[D].晋中: 山西农业大学, 2016. Wang X. Selection of candidate reference genes for gene expression studies by RT-qRCR in soybean[D]. Jinzhong: Shanxi Agricultural University, 2016. |
| [16] |
张颖, 陈婉婷, 陈冉红, 等. 杉木实时荧光定量PCR分析中内参基因的选择[J]. 林业科学研究, 2019, 32(2): 65-72. Zhang Y, Chen WT, Chen RH, et al. Quantitative real-time PCRanalysis of Cunninghamia lanceolata in the selection of the reference genes[J]. Forest Research, 2019, 32(2): 65-72. |
| [17] |
张登, 李景剑, 张梦洁, 等. 黄梁木实时荧光定量PCR分析中内参基因的选择[J]. 植物学报, 2018, 53(6): 829-839. Zhang D, Li JJ, Zhang MJ, et al. Selection and validation of reference genes for quantitative RT-PCR analysis in Neolamarckia cadamba[J]. Chinese Bulletin of Botany, 2018, 53(6): 829-839. |
| [18] |
晋海军, 王海霞, 刘绍红, 等. 川续断根实时荧光定量PCR内参基因的筛选[J]. 分子植物育种, 2018, 16(24): 7998-8004. Jin HJ, Wang HX, Liu SH, et al. Selection of reference genes for quantitative real-time PCR in Dipsacu asperides roots[J]. Molecular Plant Breeding, 2018, 16(24): 7998-8004. |
| [19] |
王世强, 党凯凯, 牛俊峰, 等. 黄精实时荧光定量PCR内参基因的筛选[J]. 基因组学与应用生物学, 2017, 36(11): 4770-4777. Wang SQ, Dang KK, Niu JF, et al. Screening of reference genes based on quantitative real-time PCR analysis in Polygonatum sibiricum[J]. Genomics and Applied Biology, 2017, 36(11): 4770-4777. |
| [20] |
李竹君, 郭健雄, 李永文, 等. 穿心莲实时定量PCR分析中内参基因的选择[J]. 广州中医药大学学报, 2013, 30(2): 240-244, 286. Li ZJ, Guo JX, Li YW, et al. Selection of reference genes for real-time quantitative PCR in Andrographis paniculata(Burm. f)Nees[J]. Journal of Guangzhou University of Traditional Chinese Medicine, 2013, 30(2): 240-244, 286. |
| [21] |
王旭, 敖妍, 刘阳, 等. 文冠果实时荧光定量PCR内参基因的筛选[J]. 分子植物育种, 2020, 18(9): 2977-2986. Wang X, Ao Y, Liu Y, et al. Selection of reference genes for quantitative real-time PCR in Xanthoceras sorbifolium Bunge[J]. Molecular Plant Breeding, 2020, 18(9): 2977-2986. |
| [22] |
Andersen CL, Jensen JL, Orntoft TF. Normalizationof Real-time quantitative reverse transcription-PCR data:A model-based variance estimation approach to identify genes suited for normalization, approach to identify genes suited for normalization, applied to bladderand colon cancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250. DOI:10.1158/0008-5472.CAN-04-0496 |
| [23] |
吴建阳, 何冰, 杜玉洁, 等. 利用geNorm、NormFinder和BestKeeper软件进行内参基因稳定性分析的方法[J]. 现代农业科技, 2017(5): 278-281. Wu JY, He B, Du YJ, et al. Analysis method of systematically evaluating stability of reference genes using geNorm, NormFinder and BestKeeper[J]. Modern Agricultural Science and Technology, 2017(5): 278-281. |
| [24] |
张贤, 王建红, 李增强, 等. 紫云英实时荧光定量PCR分析中内参基因的选择[J]. 农业生物技术学报, 2017, 25(8): 1356-1365. Zhang X, Wang JH, Li ZQ, et al. Reference gene selection for real-time quantitative PCR normalization in Astragalus sinicus[J]. Journal of Agricultural Biotechnology, 2017, 25(8): 1356-1365. |
| [25] |
张计育, 黄胜男, 王涛, 等. 金魁猕猴桃RT-qPCR内参基因的筛选[J]. 上海农业学报, 2018, 34(1): 84-88. Zhang JY, Huang SN, Wang T, et al. Screening of reference genes for reverse transcription quantitative real-time PCR in Actinidia deliciosa[J]. Acta Agriculturae Shanghai, 2018, 34(1): 84-88. |
| [26] |
苏西娅, 石元豹, 杨晓明, 等. 银杏实时荧光定量PCR分析中内参基因的选择与验证[J]. 植物生理学报, 2019, 55(6): 875-882. Su XY, Shi YB, Yang XM, et al. Selection and validation of reference genes for quantitative real-time PCR analysis in Ginkgo biloba[J]. Plant Physiology Journal, 2019, 55(6): 875-882. |
| [27] |
张莞晨, 阮成江, 李景滨, 等. 四种木本油料内参基因筛选及Actin基因时空表达分析[J]. 分子植物育种, 2018, 16(14): 4576-4582. Zhang WC, Ruan CJ, Li JB, et al. Screening of reference genes in four woody-oil trees and spatio-temporal expression analysis of Actin gene[J]. Molecular Plant Breeding, 2018, 16(14): 4576-4582. |
| [28] |
张芳明.甜樱桃内参基因的筛选[D].郑州: 河南农业大学, 2013. Zhang FM. Selection of reference genes in sweet cherry[D]. Zhengzhou: Henan Agricultural University, 2013. |
| [29] |
Xu Y, Zhu X, Gong Y, et al. Evaluation of reference genes for gene expression studies in radish(Raphanus sativus L.)using quantitative real-time PCR[J]. Biochemical and Biophysical Research Communications, 2012, 424(3): 398-403.
|
| [30] |
王彦杰, 董丽, 张超, 等. 牡丹实时定量PCR分析中内参基因的选择[J]. 农业生物技术学报, 2012, 20(5): 521-528. Wang YJ, Dong L, Zhang C, et al. Reference gene selection for real-time quantitative PCR normalization in tree peony(Paeonia suffruticosa Andr.)[J]. Journal of Agricultural Biotechnology, 2012, 20(5): 521-528. |
| [31] |
Li J, Han J, Yang J, et al. Selection of reference genes for quantitative real-time PCR during flower development in tree peony(Paeonia suffruticosa andr.)[J]. Frontiers in Plant Science, 2016, 7: 516. |