根际是指植物根周围数毫米范围内的微域环境, 是植物与土壤进行物质和能量交换的活跃中心,在植物与植物,植物与土壤生物以及土壤环境相互作用中起着至关重要的作用。自1904年德国科学家Lorenz Hitner提出“根际”概念以来,根际研究迄今已有100多年的时间,国内外研究人员已在根际.研究方法、根际养分特征、根系分泌物鉴定与调控机制等方面取得了长足的进步[1-3]。随着近年来根际微生物组研究的蓬勃发展[4-6]以及质谱鉴定和组学技术的日新月异,植物与微生物之间的根际化学对话研究受到科学家的高度重视,并迅速成为植物学与生物学领域的研究前沿与热点。根际强化调控需要我们明确植物-微生物之间是通过何种“语言”进行对话的[7]。
几乎所有涉及植物与相关微生物的相互作用都依赖于合适的化学信号。目前,已有很多研究明确了植物或微生物产生的低分子量信号物质的主要功能,包括根际微生物组的招募、塑造和活性调控[8];诱导植物免疫系统而抑制病原菌[6];诱导细菌趋化,维持植物生长,养分吸收利用和胁迫耐受性[9-10];调节地下到地上的互作[11]。可见,根际信号物质在植物抗病、抗逆以及养分吸收利用中均有重要作用,对于它们的研究将有助于提高作物产量,减少过量农药和高量氮肥使用,为环境友好的绿色农业发展作出贡献[12]。然而,养分转化与高效利用是植物生长和抵抗逆境胁迫的基础,本文主要围绕根际化学信号物质在氮、磷、铁养分转化中的最新研究进展进行综述。
1 根际化学信号物质概念植物根系能释放一系列初级代谢物(如糖类、氨基酸和有机酸)和次级代谢物(如酚类、黄酮类化合物、萜类化合物)来塑造和影响根际微生物菌群,并与其交流,最终达到吸引和促进有益微生物,同时抵抗病原菌或有害微生物的目的。根际微生物能拓宽植物适应环境的能力,而根际微生物的影响力主要依赖于化学分泌物,这些化学分泌物,有些是作为满足微生物生长需要的能量起作用,而有些则可能是作为信号物质调控着根际的相互作用。除了豆科植物与根瘤菌共生,菌根真菌共生等经典例子[13-14],大量关于根际化学分泌物调控非共生微生物群落的信号类型与作用机制仍有待深入。在根际领域,通常用以下术语指代这方面的研究,例如“根际对话”、“根际化学通讯、“根际信号交流”、“地下相互作用”、“复杂的植物-微生物互作”等[7, 15]。
植物首先会通过根际沉积物和根系分泌物影响根系周围的微生物繁殖。这种从土著微生物中招募和塑造根际微生物群落是一种植物主动调控的方式,涉及维持、限制或终止微生物生长和活性的过程。严格意义上说,化学物质作为营养和底物,在确定根际微生物种类中的重要作用不能被视为信号[16]。另外,根际微生物也会迅速感应和响应植物或微生物分泌的特定非营养性化学物质,这个过程中特定物质将引起细胞内一系列的信号级联反应,最终导致基因座的转录,这才能定义为根际化学信号物质[16-17]。这些信号物质主要产生于下列生物体之间的交互作用:(1)植物-植物,(2)植物-微生物,(3)微生物-微生物(种内和种间)。值得注意的是,一些根际化学分泌物在发挥信号功能调控基因表达的同时,也能作为碳源促进微生物的生长[10, 18]。当物质作为“信号”发挥作用时,剂量浓度一般较低,主要在纳摩尔至低微摩尔水平的较低浓度范围内[19]。
2 根际化学信号物质的类型一系列植物源和微生物源的根际化学信号物质已被鉴定(表 1),从植物-植物之间的化感作用,到植物-微生物互作中豆科植物-根瘤菌共生体、菌根真菌和根际促生菌,再到微生物-微生物之间的群体感应过程,它们在典型的根际信号互作过程中起到了关键的调控作用。按物质类型分,可分为低分子量的糖类(如果糖、D-半乳糖)、有机酸(如苹果酸、水杨酸)、酚类化合物(如儿茶酚、黄酮)。另外,还有一些高分子量的脂肪酸及其衍生物(如茉莉酸、芥酸酰胺、1,9-癸二醇)、萜类化合物(如独脚金内酯)、内酯类化合物(如N-酰基高丝酸氨内酯、香豆素)、杂环类化合物(哌嗪二酮、喹诺酮)、多糖(如脂壳聚糖、几丁质)、蛋白(如鞭毛蛋白)等。这些化学物质具有刺激植物-微生物共生、诱导细菌群体感应、调控养分吸收转化以及驱动微生物组等信号功能。然而大多数情况下,仅从植物或微生物单方面解析了上述化学信号物质的作用,对它们与互作对象之间的信号互馈关系及机制则知之甚少。
土壤氮循环主要包括生物固氮作用、氨化作用、硝化作用、反硝化作用、厌氧氨氧化作用等过程。尽管植物不能直接固氮或进行硝化作用,但能吸收硝态氮(NO3-)和铵态氮(NH4+),且在不同的基因型和环境中对氮形态有不同的偏好[20]。越来越多的研究表明,植物根系释放根系分泌物来控制根际微生物群落从而催化氮的转化[21]。多种化合物被认为是植物与其他植物或其他类型生物之间在地下对话的“语言”(图 1)。本节讨论调控固氮、硝化与反硝化过程的根际信号物质类型及其作用机制。
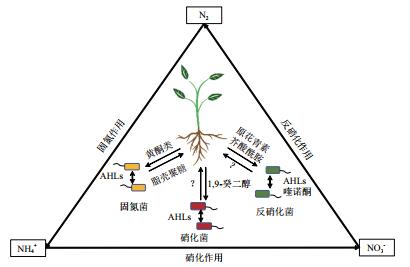
|
| 图 1 已知根际化学信号物质对氮转化过程的调控 |
豆科植物与根瘤菌之间的共生关系网络是根际化学信号物质调控氮素转化的最经典例子。研究者发现的第一个豆科植物与根瘤菌交流信号物质是植物分泌的黄酮类化合物(2-phenyl-1, 4-benzopyrone derivatives),能诱导根瘤菌结瘤基因(nod gene)的表达[13]。这些基因随后合成并分泌脂壳聚糖(Lipo-chiooligosaccharides, LCOs),也称为结瘤因子(Nod factor),为宿主-微生物特异性和结节形成奠定了基础[22]。。宿主植物随后通过位于根表皮细胞质膜上的受体激酶来感知这些LCOs,激活Ca2+依赖的信号级联反应,从而导致结节形成[23]。除了黄酮类化合物与LCOs的根际信号通讯,研究人员还发现植物分泌的酚类化合物如香兰醇、对羟基苯甲酸和3,4-二羟基苯甲酸等[24],以及根瘤菌分泌的蛋白(如鞭毛蛋白)也参与了植物与根瘤菌共生体信号交流过程[25]。
豆科作物除了与微生物发生根际对话,也会与相邻植物发生相互作用。在一项禾本科作物玉米与豆科作物蚕豆间作的研究中,证实玉米根系分泌物在提高豆科作物结瘤和固氮中的重要作用,这是由玉米分泌的黄酮类信号物质激发的[26]。豆科植物感应到玉米分泌物后,根内染料木素(Genistein,也是豆科作物和根瘤菌对话的关键信号物质)分泌量增加了近两倍,并上调了参与黄酮类化合物合成的查尔酮黄烷酮异构酶基因表达,以及介导结瘤和生长素合成基因的表达[26]。这项研究解析了中国特色间套作系统中资源高效利用的根际互作机制,是植物与植物通过根系间相互作用提高土壤养分利用效率的典型例证。
根瘤菌与根瘤菌之间也会发生化学信号交流,N-酰基高丝氨酸内酯类化合物(N-acyl homoserine lactones,AHLs)是根瘤菌群体感应(QS)的一类重要信号分子,在调节豆科植物-根瘤菌共生的频率和结瘤率中发挥重要作用。Veliz-Vallejos等[27]发现添加1 μmol/L苜蓿中华根瘤菌(Sinorhizobium meliloti)分泌的3-oxo-C14-HSL,能促进豆科模式植物蒺藜苜蓿根系的根瘤数量。Palmer等[28]通过对群体感应信号物质酰基高丝酸氨内酯(AHLs)文库的筛选,发现根瘤菌中含有较长酰基链(> 12个碳)的AHLs会增加结节数量,且AHLs对结瘤的影响不依赖于内酯环的完整性,也可能取决于酰胺酶的活性。最近在一项木豆的研究中,Gosai等[29]发现3-O-C12-HSL和3-OH-C16-HSL能诱导根瘤菌鞭毛运动和趋化等基因表达。更重要的是,根瘤菌分泌的AHLs也会被豆科植物根系识别与响应,激活植物体内的响应机制并向细菌提供反馈信息[30]。
根瘤菌等微生物产生的化学信号合成系统还能成功转移到植物上,这种信号物质必须具备以下条件:(1)能进行植物基因工程;(2)引起细菌响应;(3)分泌至根际;(4)通常不是植物产生的信号。一类被称为“rhizopine”的物质被认为是理想的信号,早期研究就发现了其在植物-微生物共生与竞争中的作用[31]。Geddes等[32]最近发现根瘤菌产生rhizopine(Scylloinosamine和3-O-methyl-scyllo-inosamine)的天然生物合成路径,并通过合成生物学技术将微生物scylloinosamine的合成系统转移到豆科模式植物蒺藜苜蓿和谷物大麦上。Scylloinosamine能在苜蓿和大麦体内合成,分泌至体外后能被根际细菌迅速感应进行化学通讯。这项合成信号研究提供了植物与根际微生物之间“跨界信号”的应用案例,为小麦和玉米等非豆类作物的固氮途径提供了重要启示,在利用增强根系微生物菌群提高贫瘠土壤上作物产量方面具有广阔的应用前景。
3.1.2 硝化作用植物根系能产生和分泌的抑制微生物硝化作用的物质,被称为生物硝化抑制剂(Biological nitrification inhibitors,BNIs)。BNIs具有减少农田氮素流失和提高作物氮素利用率的潜能,有望成为农业生产中化学合成硝化抑制剂的替代措施之一。迄今为止,从植物根系分泌物中分离和鉴定出5种BNIs物质:高粱中的高粱酮(在疏水性组分中占主导作用的苯醌类化合物)、樱花素(亲水性黄酮类化合物)和对羟基苯丙酸甲酯(亲水性苯丙酸类化合物)[33-34];湿身臂形草中的臂形草内酯(环状二萜)[35]和水稻中的1,9-癸二醇(脂肪醇)[36]。BNIs很有可能作为根系特定化学信号与硝化细菌等微生物进行交流。以水稻为例,除了受NH4+等环境因子诱导,根际硝化细菌的存在能使水稻BNIs1,9-癸二醇的分泌量增强两倍以上,但反硝化细菌却没有此诱导效应[37]。而硝化菌是通过何种“语言”与水稻进行对话,具体的信号互作机制尚不清楚。大多数BNIs能同时抑制硝化菌两个关键酶氨单加氧酶(AMO)和羟胺氧化酶(HAO)的活性。而且,1,9-癸二醇和对羟基苯丙酸甲酯等BNIs物质单独添加至土壤后,能同时抑制氨氧化细菌(AOB)和氨氧化古菌(AOA)的丰度[38-39],这是BNIs相比于化学合成硝化抑制剂的优势之一,如目前在农牧业中普遍应用的硝化抑制剂双氰胺主要调控AOB。未来对于植物根内调控BNI分泌基因,BNIs与根际微生物的信号互作机制,以及BNIs的农业应用途径的研究,将有助于BNIs等根际化学信号从理论研究真正走向生产实践。
此外,硝化菌之间也存在信号通讯,AHLs是硝化菌群体感应系统的典型化学信号物质。Mellbye等[40]利用UPLC-MS对氨氧化细菌(AOB)中的亚硝化螺菌属(Nitrosospira),以及亚硝酸盐氧化菌(NOB)中硝化杆菌属(Nitrobacter)和硝化螺菌属(Nitrospira)分泌的AHLs类型进行了调查发现,Nitrosospira multiformis,Nitrosospira briensis以及Nit-robacter vulgaris,Nitrospira moscoviensis分别能产生C10-HSL、3-OH-C14-HSL、C10:1-HSL和C8-HSL。Shen等[41]从维氏硝化杆菌(Nitrobacter winogradskyi)中鉴定了一种新的AHLs信号分子7,8-反-N-(癸酰基)高丝氨酸内酯,它能增强亚硝酸盐氧化还原酶NXR和硝酸盐/亚硝酸盐转运蛋白NarK的活性从而促进亚硝酸盐转化。但是,硝化微生物分泌的AHLs信号被植物识别与响应的分子机制还缺乏了解。
正如上文中提到非固氮植物玉米可以促进相邻豆科植物结瘤和固氮,植物通过根际互作影响其它植物生长不仅能表现在当季作物或间套作系统中,也能表现在轮作系统中。Karwat等[42]通过两年田间试验发现与高BNI活性的牧草B. humidicola轮作后,显著提高了后茬玉米的氮素利用率和产量,这主要归因于牧草特定根系分泌物或提取物在土壤中残留的BNI活性。此外,与BNI效应强的高粱轮作后,蔬菜作物的氮素利用率也能提高6.7%,产量增加10.3 t/(hm2·a)[43]。上述研究指出了根系分泌物在作物轮作系统中的重要性,为根际化学信号物质在农业生产上的实际应用途径提供了新的思路。然而,具体起作用的BNIs物质类型与调控机制,以及间作或轮作植物品种搭配有待进一步探究。
3.1.3 反硝化作用植物能产生和分泌有机化合物作为碳源或信号物质调控微生物反硝化过程。但与固氮与硝化作用相比,植物源调控微生物反硝化的物质研究相对匮乏。Lu等[44]利用根系分泌物原位连续收集系统探讨了水生植物浮萍-反硝化细菌的根际对话过程,发现非营养性功能物质芥酸酰胺与顺-7-十六烯酸甲酯能促进典型反硝化荧光假单胞菌Pseudomonas fluorescens的活性,并在50 μmol/L浓度下能促进硝酸盐还原酶(NAR)与亚硝酸盐还原酶(NIR)的活性,芥酸酰胺的分泌还受高NO3-浓度的诱导[45]。该研究首次揭示了脂肪酸酰胺类化合物和脂肪酸甲酯类化合物在调控反硝化方面的非营养性信号作用。除了促进反硝化,植物也能抑制反硝化过程。例如,Bardon等[46]首次在低氮环境生长的侵入性杂草何首乌属(Fallopia spp.)提取物中发现了生物反硝化抑制剂(Biological denitrification inhibitors,BDIs)原花青素(黄酮类化合物),主要通过影响细菌细胞膜的稳定性,特异性抑制Pseudomonas brassicacearum NFM 421的硝酸盐还原酶变构作用[47]。植物根系分泌特定反硝化调控剂可能与BNIs类似,是植物适应特定氮营养生境的适应机制,未来研究有必要进一步探讨植物调控根际反硝化过程的信号机制与生态意义。
在反硝化微生物之间的互作中,细菌分泌的QS信号同样能影响反硝化。Toyofuku等[48]发现铜绿假单胞菌(Pseudomonas aeruginosa)的反硝化作用受AHLs介导的群体感应系统抑制,信号分子C4-HSL和3-OXO-C12-HSL调控了转录因子RhlR或LasR蛋白从而影响反硝化基因表达。除了AHLs信号,一个喹诺酮类物质——2-庚基-3-羟基-4-喹诺酮被证明不依赖于调节蛋白PqsR,主要通过Fe螯合途径抑制了P. aeruginosa的反硝化活性[49]。类似地,在一项反硝化脱氮副球菌(Paracoccus denitrificans)的研究中,Zhang等[50]通过报告菌株检测和高效液相色谱-质谱联用检测方法发现P. denitrificans群体感应信号分子类型为C16-HSL,能与QS转录因子PdeR蛋白结合进行种群内与种群间的“通讯”,主要通过调控铁转运方式,从主动运输变为自由扩散,维持细胞内铁浓度的稳定,达到调控反硝化和适应环境的目的。与硝化微生物一样,目前反硝化微生物的QS信号研究仅停留在微生物本身,QS信号物质如何影响植物,植物又是如何反馈的有待深入。
综上,根际化学信号物质在固氮、硝化与反硝化等氮转化过程中发挥了重要作用,然而氮转化途径是紧密连锁的,大多数研究目前仅关注了信号物质对单个过程的调控,未来研究需要评价根际化学信号物质对其他氮循环过程(如厌氧氨氧化过程)以及整个根际微生物菌群的影响,并试图阐明其内在的因果关系。上述研究也发现,QS信号物质AHLs能同时介导固氮菌、硝化菌和反硝化菌种内的信号交流,但影响不同氮循环菌群种间交流的信号物质类型和互作机制还缺乏研究。
3.2 磷转化磷是植物必需的大量元素,在维持植物生命活动中发挥着不可替代的作用。植物自身除了调节根系形态与生理特征外,还能与丛枝菌根(Arbuscular mycorrhizal,AM)真菌等土壤微生物形成共生关系,通过微生物的帮助来活化与吸收磷。植物与AM真菌的共生关系正始于彼此间的化学信号交换。独脚金内酯(Strigolactones,SLs)是植物分泌的经典信号物质。早期研究发现在缺磷条件下,SLs能促进根寄生植物独脚金属(Striga spp.)和列当属(Orobanche spp.)种子萌发[14]。随后,SLs还被证实能作为激素控制植物分枝与根构型[51],同时塑造作物水稻的根际微生物组[52]。2005年,日本科学家Akiyama等[14]首次从百脉根根系分泌物中分离得到5-脱氧独脚金醇(5-deoxy-strigol),并发现该物质在诱导真菌分枝、改变真菌生理和线粒体活性中起着重要作用。随后,研究还发现了结构型角质单体[53]与几丁质相关分子[54]能作为特定的植物信号刺激菌根真菌分枝。在AM真菌方面,Maillet等[55]从菌根真菌分泌物中鉴定了菌根因子(Myc factors)——脂质几丁寡糖(Mycorrhizal lipochitinoligosaccharides,Myc-LCOs),它与根瘤菌分泌的结瘤因子功能相似。此外,AM分泌物能诱导植物根内淀粉积累、糖代谢、磷吸收利用以及信号传导等基因表达[56]。尽管SLs与Myc-LCOs在识别和共生过程中起重要作用,但目前尚未阐明信号物质调控植物磷营养的分子机制,而且植物与真菌信号受体仍然未知。
除了与植物共生,AM真菌还能与菌丝周围伴生的解磷细菌之间进行互作。当菌根形成后,根际的内涵不仅包括受根系分泌物影响的土壤区域,也包括根外菌丝延伸生长的土壤区域,即“菌丝际”(Hyphosphere)。解磷细菌可以在菌根真菌的菌丝际定殖,并利用菌丝分泌物作为“碳源”促进其生长和代谢活性,活化土壤难溶性磷[57]。Zhang等[10]进一步利用室内分隔培养皿体系,发现菌丝分泌物中的果糖也可作为“信号”在短时间内刺激解磷细菌磷酸酶蛋白基因表达,加强有机磷的活化和吸收。最近在土壤体系下的研究发现,高浓度的果糖添加到玉米菌丝际后改变了土壤细菌群落组成,刺激细菌磷酸酶活性和有机磷矿化,从而提高了土壤中有机形态累积的磷的利用[58]。上述研究揭示了果糖在调控微生物磷吸收转化中“碳源”与“信号”的双重功能,并为通过微生物互作(真菌与细菌)强化菌丝际效应来提高作物养分效率提供了新思路。
植物与植物之间的互作也能影响养分磷的吸收利用。在我国北方典型的玉米/蚕豆间作体系中,除了前面提到的玉米能促进豆科植物固氮,蚕豆相反也能增强玉米对磷的吸收,这是由于蚕豆根系分泌的有机酸可以活化土壤中难溶性磷,从而促进玉米对磷的吸收利用[59],这一发现解释了农业生产上和玉米间作能大量增产的原因。在花生-木薯这一南方红壤区重要间作系统中,发现木薯根系能释放氰化物信号被相邻花生接收,释放气态信号物质乙烯,乙烯能作为“信号”调控花生根际微生物组中的核心物种放线菌门Catenulispora sp.的丰度,重塑微生物的网络结构。新构建的微生物组能加速根际有机氮磷矿化,为花生地下部提供可利用的氮磷养分,从而提高了花生果实的饱满程度和产量[60]。该研究推进了植物-植物联合根际微生物之间的根际化学信号在调控养分利用方面的认识。
3.3 铁吸收铁是植物生长和发育的必需营养元素,也是许多酶必不可少的辅助因子。尽管土壤中的总铁含量远高于植物所需的铁含量,但由于铁主要以不溶性三价铁(Fe3+)形式存在,在大多数土壤中的生物有效性较低。根系分泌物在植物在响应缺铁环境中具有重要作用,最典型可能就是禾本科植物分泌的麦根酸类物质,又称为植物铁载体(Phytosiderophore),通过化学络合作用,提高根际土壤铁的有效性[61-63],但是麦根酸类物质并不是根际化学信号物质的途径起作用的。因此,近年来研究者开始更多关注根系分泌物中可能作为根际化学信号的物质在铁营养中的作用。例如,Stringlis等[9]研究表明,缺铁条件下植物根部特异性转录因子MYB72可以促进以荧光酚类化合物香豆素(东莨菪亭)等根系分泌物的合成与分泌,且东莨菪亭能选择性地改变植物根系的菌群群落,抑制植物病原菌尖孢镰刀菌及黄萎病菌,但对促植物生长的植物益生菌无影响[9]。Voges等[64]随后利用拟南芥简化合成微生物群落方法,将根系分泌的香豆素与根际细菌之间建立了机理联系。可见,根系分泌香豆素是植物适应缺铁胁迫的一种有效策略。那么,根际微生物在植物缺铁条件下的作用如何?Jin等[65]研究发现缺铁条件下,根际微生物在感应到红三叶草分泌的酚类化合物后,一方面分泌铁载体(Siderophore)促进红三叶草对铁的利用,另外还能产生生长素类物质,提高三价铁还原能力。因此,植物源酚类化合物和微生物源铁载体等可能是根际对话的关键化学信号。
此外,铁载体也介导了根际菌群之间竞争核心稀缺资源铁的信号途径。Butaite等[66]在土壤和淡水环境中筛选了300多种假单胞菌菌株,通过菌群互作实验证实了荧光铁载体(Pyoverdine)能调控自然假单胞菌属之间对铁的螯合和竞争力,驱动细菌菌群之间的共进化与多样性。在单一菌群互作基础上,Gu等[67]研究证明铁载体也是根际细菌与病原菌之间争夺根际铁素的工具,决定了根际细菌与青枯菌的铁素竞争力和作物健康。分泌抑制型铁载体的根际细菌通常能抑制体外和自然土壤中病原菌,保护番茄植物免受感染;相反,具有促进型铁载体的根际细菌通常在竞争中处于劣势,并促进病原体对植物的感染。由于铁载体是一类结构不同的化学信号,每类铁载体都依赖于其相容的受体来进行铁吸收,而抑制病原菌的根际微生物组会产生病原菌无法利用的铁载体。这项铁载体的研究为利用根际有益菌解决土传青枯病问题、开拓植物健康的“靶向疗法”提供了新思路。
除了铁载体,微生物分泌的其他特定信号可能会发挥类似的作用。例如,许多细菌能分泌小分子的AHLs信号分子,与铁载体一样,AHLs化学结构多样并存在于多个微生物物种中。因此,铁载体介导的复杂微生物菌群互作机制很可能也适用于基于AHLs的通信系统,值得探索。尽管上述研究从微生物-微生物菌群互作水平上建立了根际微生物组对铁竞争与植物保护之间的因果关系,但主要是通过大规模的表型分析获得的,未来需要进一步研究铁载体调控根际微生物菌群互作的分子机制。
3.4 其他植物通过在根系招募大量种属特异的微生物,进化出了强大的环境适应能力,这些微生物参与了植物养分吸收和抵抗胁迫等重要生理过程。植物-根际微生物组之间的信号互作不仅影响土壤养分的利用,还在植物生长和土壤健康方面发挥重要作用。Lebeis等[8]通过比较拟南芥野生型和突变体的根际细菌群落组成,首次将植物免疫系统激素信号水杨酸与根特定的微生物组关联起来。而且,水杨酸对根际微生物组发挥了不同的作用,能提高一些有益微生物丰度,并降低一些病原菌的丰度。除了激素信号,Huang等[68]发现植物特异代谢物三萜类化合物在组装和调控根际微生物方面的重要作用。在鉴定拟南芥三种不同根系三萜类化合物(拟南芥宁素、拟南芥宁基中链脂肪酸酯和拟南芥啶素)的生物合成途径基础上,分离培养了自然土壤生长拟南芥根际细菌的资源库,并在体外测试了纯化的三萜类化合物(或混合物)对拟南芥根系分离的19个分类多样菌株生长活性的影响,发现三萜类化合物能直接调控特异的根系细菌种类,如促进Arthrobacter sp.和抑制Arenimonas sp.,该调节作用与拟南芥野生型和三萜突变体根中不同菌种的相对丰度差异相关。这为利用植物激素或天然产物作为潜在信号促进根系益生菌在绿色农业中的应用提供了理论依据。
此外,根际微生物组能系统地诱导根系代谢物分泌(SIREM)。Korenblum等[11]通过水培分根实验,将番茄一半根系置于富含自然土壤微生物的收集液中,而另一半则在无菌条件下培育,发现“局部”番茄根际微生物组可以诱导复杂的根-根信号,从而介导“系统”根内代谢物和根系分泌物的产生。例如,芽孢杆菌(Bacillales)可以利用SIREM过程触发整个根系中酰基糖的分泌,而假单胞菌(Pseudomonadales)可诱导根分泌阿魏酸己糖。叶和根代谢组和转录组的结果表明,糖基化壬二酸是一种潜在的微生物组诱导的植物源信号物质,随后将以游离壬二酸形式渗出,该信号在根局部产生后,可能会到达地上部叶片,随后下降至另外一端根部,最终介导根际相互作用。该研究系统全面地揭示了根际微生物组局部定殖后植物整个根系统代谢物的变化,微生物重新编程的SIREM可能会影响植物生长与土壤健康。以上研究挖掘了一些潜在的根际信号物质类型与功能,明确了它们在影响植物生长方面的潜力,然而它们与养分转化之间的联系还有待深入。
4 总结与展望根际化学信号物质对于养分转化与高效利用具有重要作用。本文主要从植物-植物,植物-微生物,微生物-微生物之间的交互作用,讨论了调控氮、磷、铁养分转化的根际化学信号物质类型与调控机制。除了共生微生物与植物信号交流,根际定殖的非共生微生物(如假单胞菌属、芽孢杆菌属)也发挥着关键作用。一些根际化学分泌物同时发挥着“信号”与“营养”的双重功能,通过塑造特定根际微生物组,调控养分转化与植物生长;微生物分泌的QS信号物质AHLs还能介导多种养分转化过程。大量未知的根际信号物质类型与已知信号物质的调控机理有待深入。
未来植物与根际微生物在根-土-微生物互作过程中发生的信号共享可能成为许多研究的主题,不仅局限于单一方向的信号调控,将拓展到双向通信,再延伸到不同生物体之间的跨界信号互馈(图 2)。对于植物和微生物分泌物及其调控基因的检测与鉴定,将有助于我们理解根际信号的调控途径与网络。最新在化学检测方面的进展将大大加强这些研究,特别是气相色谱-质谱(GC-MS),液相色谱-质谱(LC-MS)等。这些技术采用无目标的定性和定量技术可以对根系分泌物或微生物分泌物进行大规模的分析,称为代谢组学。如果联合养分吸收利用差异的品种与其他生物特性(如BNI活性),以及活性组分极性分离技术,将更好地确定影响养分转化的根际化学信号类型,还能与全基因组关联分析与转录组学结合,试图揭示参与根际信号物质合成与分泌的植物基因。此外,宏基因组测序是解析微生物群落组成的强大工具,随着微生物高通量筛选和重组菌群体系新技术的应用[5],已将发现关联性的目标成功推进到建立菌群与植物养分利用等表型之间的因果关系,为根际化学信号物质调控微生物组的机制研究提供了前所未有的机遇。然后,可以将其与宏转录组学和宏蛋白组学结合,有望洞悉根:际信号调控的特定基因位点和蛋白靶点。这些方法的集成将产生大量的信息,为全面揭示根际化学信号物质的调控养分转化的分子机制奠定基础,未来的挑战将是独特的实验设计以及对组学数据的合理验证。
迄今为止,大量的研究已经确立了根际微生物组在养分利用与植物生长中的重要性,并且植物能够控制根际微生物组的组成,以选择特定的微生物功能。最近一项研究确定了水稻氮转运和感知基因NRTI.IB是影响籼稻根际富集细菌组成的驱动因素之一[69],而根际化学信号的研究似乎是沟通植物基因与根际微生物之间联系的桥梁(图 2),能为养分高效利用开辟新的途径,更好地提高农作物生产力并减少农用化学品的使用。在未来农业应用中,一方面可以通过基因工程方法改变根际信号物质的植物代谢或分泌途径,将其作为调控根际微生物成员以有利于植物养分高效利用的一种方式[68]; 另一方面,特定的根际信号物质也可以直接作为化学指示.物,促进植物生长或有益根际微生物组,提高养分利用。通过设计这些解决方案,能有助于丰富有益根际微生物组,从而提高植物养分吸收利用,以及对生物和非生物胁迫的抗性,实现绿色农业的可持续发展。
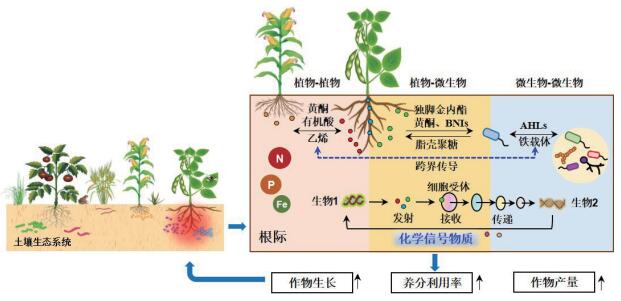
|
| 在土壤生态系统中,不同生物体之间的交互作用发生在地上与地下,在多个生物类别之内或跨越多个生物类别,同时发生也可能 发生在不同时间。根际是植物与根际生物体交流的中心,化学信号物质调控植物-植物,植物-微生物及微生物-微生物之间的 互作。例如,植物能分泌黄酮类化合物和有机酸等物质为相邻植物识别,还能分泌独脚金内酯和BNIs等招募和调控根际微生物; 微生物反过来释放脂壳聚糖等影响植物生长,同时通过分泌AHLs和铁载体等化学信号调控种内或种间的交流,甚至发生跨界传导。在根际生物体内(如生物1 )相关基因受诱导后,能发射特定化学信号被其他生物接收(如生物2),并通过一系列信号级联反应 启动关键基因表达,再释放其他小分子物质影响根际生物。通过上述信号互作,能提高根际氨磷铁等养分利用率,促进作物生长 与增加作物产量,维持土壤生态系统健康 图 2 根际化学信号物质在土壤生态系统不同生物体之间调控养分高效利用示意图 |
| [1] |
刘芷宇, 李良谟, 施卫明. 根际研究法[M]. 南京: 江苏科学迄今为止, 大量的研究已经确立了根际微生物技术出版社, 1997. Liu ZY, Li LM, Shi WM. Method of rhizosphere research[M]. Nanjing: Jiangsu Science and Technology Press, 1997. |
| [2] |
王占义, 潘宁, 罗茜, 等. 一种新型根系分泌物收集装置与收集方法的介绍[J]. 土壤学报, 2010, 47(4): 747-752. Wang ZY, Pan N, Luo Q, et al. A new type of device and method for collecting plant root exudates[J]. Acta Pedologica Sinica, 2010, 47(4): 747-752. |
| [3] |
Shen JB, Li CJ, Mi GH, et al. Maximizing root/rhizosphere efficiency to improve crop productivity and nutrient use efficiency in intensive agriculture of China[J]. J Exp Bot, 2013, 64(5): 1181-1192. DOI:10.1093/jxb/ers342 |
| [4] |
Lundberg DS, Lebeis SL, Paredes SH, et al. Defining the core Arab-idopsis thaliana root microbiome[J]. Nature, 2012, 488(7409): 86-90. DOI:10.1038/nature11237 |
| [5] |
Bai Y, Muller DB, Srinivas G, et al. Functional overlap of the Arabidopsis leaf and root microbiota[J]. Nature, 2015, 528(7582): 364-369. DOI:10.1038/nature16192 |
| [6] |
Castrillo G, Teixeira PJPL, Paredes SH, et al. Root microbiota drive direct integration of phosphate stress and immunity[J]. Nature, 2017, 543(7646): 513-538. DOI:10.1038/nature21417 |
| [7] |
李春俭, 马玮, 张福锁. 根际对话及其对植物生长的影响[J]. 植物营养与肥料学报, 2008(1): 178-183. Li CJ, Ma W, Zhang FS. Rhizosphere talk and its impacts on plant growth[J]. Journal of Plant Nutrition and Fertilizers, 2008(1): 178-183. |
| [8] |
Lebeis SL, Paredes SH, Lundberg DS, et al. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa[J]. Science, 2015, 349(6250): 860-864. DOI:10.1126/science.aaa8764 |
| [9] |
Stringlis IA, et al. MYB72-dependent coumarin exudation shapes root microbiome assembly to promote plant health[J]. Proc Natl Acad Sci USA, 2018, 115(22): E5213-E5222. DOI:10.1073/pnas.1722335115 |
| [10] |
Zhang L, Feng G, Declerck S. Signal beyond nutrient, fructose, exuded by an arbuscular mycorrhizal fungus triggers phytate mineralization by a phosphate solubilizing bacterium[J]. ISME Journal, 2018, 12(10): 2339-2351. DOI:10.1038/s41396-018-0171-4 |
| [11] |
Korenblum E, Dong YH, Szymanski J, et al. Rhizosphere microbiome mediates systemic root metabolite exudation by root-to-root signaling[J]. Proc Natl Acad Sci USA, 2020, 117(7): 3874-3883. DOI:10.1073/pnas.1912130117 |
| [12] |
Zhang RF, Vivanco JM, Shen QR. The unseen rhizosphere root-soil-microbe interactions for crop production[J]. Current Opinion in Microbiology, 2017, 37: 8-14. DOI:10.1016/j.mib.2017.03.008 |
| [13] |
Redmond JW, Batley M, Djordjevic MA, et al. Flavones induce expression of nodulation genes in Rhizobium[J]. Nature, 1986, 323(6089): 632-635. DOI:10.1038/323632a0 |
| [14] |
Akiyama K, Matsuzaki K, Hayashi H. Plant sesquiterpenes induce hyphal branching in arbuscular mycorrhizal fungi[J]. Nature, 2005, 435(7043): 824-827. DOI:10.1038/nature03608 |
| [15] |
孙波, 廖红, 苏彦华, 等. 土壤-根系-微生物系统中影响氮磷利用的一些关键协同机制的研究进展[J]. 土壤, 2015, 47(2): 210-219. Sun B, Liao H, Su YH, et al. Advances in key coordinative mechanisms in soil-root-microbe systems to affect nitrogen and phosphorus utilization[J]. Soils, 2015, 47(2): 210-219. |
| [16] |
Venturi V, Keel C. Signaling in the Rhizosphere[J]. Trends Plant Sci, 2016, 21(3): 187-198. DOI:10.1016/j.tplants.2016.01.005 |
| [17] |
Chagas FO, Pessotti RD, Caraballo-Rodriguez AM, et al. Chemical signaling involved in plant-microbe interactions[J]. Chemical Society Reviews, 2018, 47(5): 1652-1704. DOI:10.1039/C7CS00343A |
| [18] |
Jiang YN, Wang WX, Xie QJ, et al. Plants transfer lipids to sustain colonization by mutualistic mycorrhizal and parasitic fungi[J]. Science, 2017, 356(6343): 1172-1175. DOI:10.1126/science.aam9970 |
| [19] |
Hassan, S, Mathesius U. The role of flavonoids in root-rhizosphere signalling :opportunities and challenges for improving plant-microbe interactions[J]. J Exp Bot, 2012, 63(9): 3429-3444. DOI:10.1093/jxb/err430 |
| [20] |
Britto DT, Kronzucker HJ. Ecological significance and complexity of N-source preference in plants[J]. Annals of Botany, 2013, 112(6): 957-963. DOI:10.1093/aob/mct157 |
| [21] |
Coskun D, Britto DT, Shi WM, et al. How plant root exudates shape the nitrogen cycle[J]. Trends Plant Sci, 2017, 22(8): 661-673. DOI:10.1016/j.tplants.2017.05.004 |
| [22] |
Lerouge P, Roche P, Faucher C, et al. Symbiotic host-specificity of Rhizobium meliloti is determined by a sulfated and acylated glucosamine oligosaccharide signal[J]. Nature, 1990, 344(6268): 781-784. DOI:10.1038/344781a0 |
| [23] |
Oldroyd GED. Speak, friend, and enter :signalling systems that promote beneficial symbiotic associations in plants[J]. Nature Reviews Microbiology, 2013, 11: 252-263. DOI:10.1038/nrmicro2990 |
| [24] |
Mandal SM, Chakraborty D, Dey S. Phenolic acids act as signaling molecules in plant-microbe symbioses[J]. Plant Signaling & Behavior, 2010, 5(4): 359-368. DOI:10.4161/psb.5.4.10871 |
| [25] |
Krehenbrink M, Downie JA. Identification of protein secretion systems and novel secreted proteins in Rhizobium leguminosarum bv. viciae[J]. BMC Genomics, 2008, 9-55. DOI:10.1186/1471-2164-9-55 |
| [26] |
Li B, Li YY, Wu HM, et al. Root exudates drive interspecific facilitation by enhancing nodulation and N2 fixation[J]. Proc Natl Acad Sci USA, 2016, 113(23): 6496-6501. DOI:10.1073/pnas.1523580113 |
| [27] |
Veliz-Vallejos DF, van Noorden GE, Yuan MQ, et al. A Sinorhizobium meliloti-specific N-acyl homoserine lactone quorum-sensing signal increases nodule numbers in Medicago truncatula independent of autoregulation[J]. Frontiers in Plant Science, 2014, 5: 551. |
| [28] |
Palmer AG, Mukherjee A, Stacy DM, et al. Interkingdom responses to bacterial quorum sensing signals regulate frequency and rate of nodulation in Legume-Rhizobia symbiosis[J]. Chembiochem, 2016, 17(22): 2199-2205. DOI:10.1002/cbic.201600373 |
| [29] |
Gosai J, Anandhan S, Bhattacharjee A, et al. Elucidation of quorum sensing components and their role in regulation of symbiotically important traits in Ensifer nodulating pigeon pea[J]. Microb Res, 2020, 231: 126354. DOI:10.1016/j.micres.2019.126354 |
| [30] |
Gonzalez JE, Marketon MM. Quorum sensing in nitrogen-fixing rhizobia[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 574-592. DOI:10.1128/MMBR.67.4.574-592.2003 |
| [31] |
Murphy PJ, Wexler W, Grzemski W, et al. Rhizopines-Their role in symbiosis and competition[J]. Soil Biology & Biochemistry, 1995, 27(4-5): 525-529. DOI:10.1016/0038-0717(95)98627-Z |
| [32] |
Geddes BA, Paramasivan P, Joffrin A, et al. Engineering transkingdom signalling in plants to control gene expression in rhizosphere bacteria[J]. Nature Communications, 2019, 10: 11. DOI:10.1038/s41467-018-07826-2 |
| [33] |
Zakir HAKM, Subbarao GV, Pearse SJ, et al. Detection, isolation and characterization of a root-exuded mcompound, methyl3-(4-hydroxyphenyl)propionate, responsible for biological nitrification inhibition by Sorghum(Sorghum bicolor)[J]. New Phytol, 2008, 180(2): 442-451. DOI:10.1111/j.1469-8137.2008.02576.x |
| [34] |
Subbarao GV, Nakahara K, Ishikawa T, et al. Biological nitrification inhibition(BNI)activity in sorghum and its characterization[J]. Plant Soil, 2013, 366(1/2): 243-259. |
| [35] |
Subbarao GV, Nakahara K, Hurtado MP, et al. Evidence for biological nitrification inhibition in Brachiaria pastures[J]. Proc Natl Acad Sci USA, 2009, 106(41): 17302-17307. DOI:10.1073/pnas.0903694106 |
| [36] |
Sun L, Lu YF, Yu FW, et al. Biological nitrification inhibition by rice root exudates and its relationship with nitrogen-use efficiency[J]. New Phytol, 2016, 212(3): 646-656. DOI:10.1111/nph.14057 |
| [37] |
Zhang XN, Lu YF, Yang T, et al. Factors influencing the release of the biological nitrification inhibitor 1, 9-decanediol from rice(Oryza sativa L.)roots[J]. Plant Soil, 2019, 436(1/2): 253-265. DOI:10.1007/s11104-019-03933-1 |
| [38] |
Nardi P, Akutsu M, Pariasca-Tanaka J, et al. Effect of methyl 3-4-hydroxyphenyl propionate, a Sorghum root exudate, on N dynamic, potential nitrification activity and abundance of ammonia- oxidizing bacteria and archaea[J]. Plant Soil, 2013, 367(1/2): 627-637. |
| [39] |
Lu YF, Zhang XN, Jiang JF, et al. Effects of the biological nitrification inhibitor 1, 9-decanediol on nitrification and ammonia oxidizers in three agricultural soils[J]. Soil Biology and Biochemistry, 2019, 129: 48-59. DOI:10.1016/j.soilbio.2018.11.008 |
| [40] |
Mellbye BL, Spieck E, Bottomley PJ, et al. Acyl-Homoserine lactone production in nitrifying bacteria of the genera Nitrosospira, Nitrobacter, and Nitrospira identified via a survey of putative quorum-sensing genes[J]. Applied and Environmental Microbiology, 2017, 83(22:e01540-17). |
| [41] |
Shen QX, Gao J, Liu J, et al. A new acyl-homoserine lactone molecule generated by Nitrobacter winogradskyi[J]. Scientific Reports, 2016, 6: 22903. DOI:10.1038/srep22903 |
| [42] |
Karwat H, Moreta D, Arango J, et al. Residual effect of BNI by Brachiaria humidicola pasture on nitrogen recovery and grain yield of subsequent maize[J]. Plant Soil, 2017, 420(1/2): 389-406. |
| [43] |
Zhang M, Fan C H, Li QL, et al. A 2-yr field assessment of the effects of chemical and biological nitrification inhibitors on nitrous oxide emissions and nitrogen use efficiency in an intensively managed vegetable cropping system[J]. Agriculture Ecosystems & Environment, 2015, 201: 43-50. |
| [44] |
Lu YF, Zhou YR, Nakai S, et al. Stimulation of nitrogen removal in the rhizosphere of aquatic duckweed by root exudate components[J]. Planta, 2014, 239(3): 591-603. DOI:10.1007/s00425-013-1998-6 |
| [45] |
Sun L, Lu YF, Kronzucker HJ, et al. Quantification and enzyme targets of fatty acid amides from duckweed root exudates involved in the stimulation of denitrification[J]. Journal of Plant Physiology, 2016, 198: 81-88. DOI:10.1016/j.jplph.2016.04.010 |
| [46] |
Bardon C, Piola F, Bellvert F, et al. Evidence for biological denitr ification inhibiton(B D I) by plant secondary metabolites[J]. New Phytol, 2014, 204: 620-630. DOI:10.1111/nph.12944 |
| [47] |
Bardon C, Poly F, Piola F, et al. Mechanism of biological denitrific-ation inhibition :procyanidins induce an allosteric transition of the membrane-bound nitrate reductase through membrane alteration[J]. FEMS Microbiol Ecol, 2016, 92(5): 11. DOI:10.1093/femsec/fiw034 |
| [48] |
Toyofuku M, Nomura N, Fujii T, et al. Quorum sensing regulates denitrification in Pseudomonas aeruginosa PAO1[J]. Journal of Bacteriology, 2007, 189(13): 4969-4972. DOI:10.1128/JB.00289-07 |
| [49] |
Toyofuku M, Nomura N, Kuno E, et al. Influence of the Pseudomonas quinolone signal on denitrification in Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2008, 190(24): 7947-7956. DOI:10.1128/JB.00968-08 |
| [50] |
Zhang Y, Gao J, Wang LS, et al. Environmental adaptability and quorum sensing :Iron uptake regulation during biofilm formation by Paracoccus denitrificans[J]. Applied and Environmental Microbiology, 2018, 84(14): 15. |
| [51] |
Gomez-Roldan V, Fermas S, et al. Strigolactone inhibition of shoot branching[J]. Nature, 2008, 455(7210): 189-194. DOI:10.1038/nature07271 |
| [52] |
Nasir F, Shi SH, Tian L, et al. Strigolactones shape the rhizomicrobiome in rice(Oryza sativa)[J]. Plant Science, 2019, 286: 118-133. DOI:10.1016/j.plantsci.2019.05.016 |
| [53] |
Wang ET, Schornack S, Marsh JF, et al. A common signaling process that promotes mycorrhizal and Oomycete colonization of plants[J]. Current Biology, 2012, 22(23): 2242-2246. DOI:10.1016/j.cub.2012.09.043 |
| [54] |
Genre A, Chabaud M, et al. Short-chain chitin oligomers from arbuscular mycorrhizal fungi trigger nuclear Ca2+ spiking in Medicago truncatula roots and their production is enhanced by strigolactone[J]. New Phytol, 2013, 198(1): 179-189. DOI:10.1111/nph.12120 |
| [55] |
Maillet F, Poinsot V, Andre O, et al. Fungal lipochitooligosaccharide symbiotic signals in arbuscular mycorrhiza[J]. Nature, 2011, 469(7328): 58-63. DOI:10.1038/nature09622 |
| [56] |
徐丽娇, 姜雪莲, 郝志鹏, 等. 丛枝菌根通过调节碳磷代谢相关基因的表达增强植物对低磷胁迫的适应性[J]. 植物生态学报, 2017, 41(8): 815-825. Xu LJ, Jiang XL, Hao ZP, et al. Arbuscular mycorrhiza improves plant adaptation to phosphorus deficiency through regulating the expression of genes relevant to carbon and phosphorus metabolism[J]. Chinese Journal of Plant Ecology, 2017, 41(8): 815-825. |
| [57] |
Zhang L, Feng G, Declerck S., et al. Signal beyond nutrient, fructose, exuded by an arbuscular mycorrhizal fungus triggers phytate mineralization by a phosphate solubilizing bacterium[J]. ISME Journal, 2018, 12(10): 2339-2351. DOI:10.1038/s41396-018-0171-4 |
| [58] |
Zhang L, Peng Y, Zhou JC, et al. Addition of fructose to the maize hyphosphere increases phosphatase activity by changing bacterial community structure[J]. Soil Biol Biochem, 2020, 142: 9. DOI:10.1016/j.soilbio.2020.107724 |
| [59] |
Li L, Li SM, Sun JH, et al. Diversity enhances agricultural productivity via rhizosphere phosphorus facilitation on phosphorus-deficient soils[J]. Proc Natl Acad Sci USA, 2007, 104(27): 11192-11196. DOI:10.1073/pnas.0704591104 |
| [60] |
Chen Y, Bonkowski M, Shen Y, et al. Root ethylene mediates rhizosphere microbial community reconstruction when chemically detecting cyanide produced by neighbouring plants[J]. Microbiome, 2020, 8(1): 17. DOI:10.1186/s40168-020-0795-2 |
| [61] |
Takagi S, Nomoto K, Takemoto T. Physiological aspect of mugineic acid, a possible phytosiderophore of graminaceous plants[J]. Journal of Plant Nutrition, 1984, 7(1-5): 469-477. DOI:10.1080/01904168409363213 |
| [62] |
Shi WM, Chino M, Youssef RA, et al. The occurrence of mugineic acid in the rhizosphere soil of barley plant[J]. Soil Science and Plant Nutrition, 1988, 34(4): 585-592. DOI:10.1080/00380768.1988.10416473 |
| [63] |
Zhang FS, Treeby M, Romheld V, et al. 1991. Mobilization of iron by phytosiderophores as affected by other micronutrients[J]. Plant Soil, 1991, 130(1/2): 173-178. DOI:10.1007/BF00011872 |
| [64] |
Voges M, Bai Y, Schulze-Lefertet P, et al. Plant-derived coumarins shape the composition of an Arabidopsis synthetic root microbiome[J]. Proc Natl Acad Sci USA, 2019, 116(25): 12558-12565. DOI:10.1073/pnas.1820691116 |
| [65] |
Jin CW, He YF, Tang CX, et al. Mechanisms of microbially enhanced Fe acquisition in red clover(Trifolium pratense L.)[J]. Plant Cell and Environment, 2006, 29(5): 888-897. DOI:10.1111/j.1365-3040.2005.01468.x |
| [66] |
Butaite E, Baumgartner M, Wyder S, et al. Siderophore cheating and cheating resistance shape competition for iron in soil and freshwater Pseudomonas communities[J]. Nat Commun, 2017, 8: 12. DOI:10.1038/s41467-017-00025-5 |
| [67] |
Gu SH, Wei Z, Shao ZY, et al. Competition for iron drives phytopathogen control by natural rhizosphere microbiomes[J]. Nature Microbiology, 2020. DOI:10.1038/s41564-020-0719-8 |
| [68] |
Huang ACC, Jiang T, Liu YX, et al. A specialized metabolic network selectively modulates Arabidopsis root microbiota[J]. Science, 2019, 364(6440:eaau6389). |
| [69] |
Zhang JY, Liu YX, Zhang N, et al. NRT1. 1B is associated with root microbiota composition and nitrogen use in field-grown rice[J]. Nature Biotechnology, 2019, 37(6): 676-684. DOI:10.1038/s41587-019-0104-4 |





