2. 棉花生物学国家重点实验室 中国农业科学院棉花研究所,安阳 455000
2. State Key Laboratory of Cotton Biology, Institute of Cotton Research of Chinese Academy of Agricultural Sciences, Anyang 455000
土壤是植物生存和发展的基础。微生物作为土壤中最活跃的组成部分,由细菌、古菌、真菌等组成,数量庞大且种类多样,是土壤有机质和养分循环转化的主要推动力[1]。不同的种植制度会改变土壤微生物组成,进而影响作物的生长发育。作物连作使土壤病原微生物富集,有益微生物减少,导致土壤微生态失衡,土传病害发生严重[2-6]。轮作能够促进有益微生物活性,抑制有害微生物,在一定程度上减少连作障碍的发生,从而提高作物的产量和品质[7-9]。
细菌是土壤微生物中数量最多、分布最广的组成部分[10],其群落结构与组成不仅能够反映土壤性状的变化趋势,而且能直接影响土壤生态功能的发挥[11-12]。吕雅悠等[13]研究发现根际细菌A21-4可以通过促进辣椒土壤酶的活性,从而提高土壤速效氮磷钾的含量。王娜[14]的研究表明枯草芽孢杆菌DP10和链霉菌DK56对棉花土壤中大丽轮枝菌的抑制性最强,生防效果显著。目前,关于土壤微生物的研究已从传统的平板培养转变为分子生物学方法,尤其是高通量测序技术,其具有数据通量高、微生物检测结果多样丰富等优势而被迅速应用[15]。近年来,该技术被广泛应用于植物土壤微生物的研究,但关于轮作下棉花根际土壤细菌变化的研究还鲜有报道。本研究利用高通量测序技术,通过分析花生、谷子轮作后棉花根际土壤细菌群落结构的演替规律,对选择合理种植措施和改善棉田土壤生态功能具有重要意义。
1 材料与方法 1.1 材料棉花品种为新陆早61,谷子品种为豫18,花生品种为中花413。
1.2 方法 1.2.1 试验设计试验地点位于新疆昌吉回族自治州的中国农业科学院西部农业研究中心。于2017年和2018年开展棉田轮作试验,设置棉花-棉花连作、花生-棉花轮作、谷子-棉花轮作、谷子-谷子连作、花生-花生连作共5种处理方式,每个处理3次重复,采用随机区组排列。各作物覆膜种植,一膜三行,水肥以膜下滴灌的方式施入。
1.2.2 棉花产量测定2018年棉花吐絮后实收测产。产量数据通过SPSS(24.0)进行统计分析。
1.2.3 土壤样品采集2018年9月10日,在各试验小区采用S形随机取样的方式,每小区取8个点混样。利用抖根法取20 cm深的粘附于作物根系的土壤鲜样,标记并立即置于冰上保存,带回实验室进行后续处理。
1.2.4 测序样品制备采用CTAB方法提取土壤样品总DNA,利用细菌检测通用引物16S V4区引物(515F/806R)进行PCR扩增,产物经琼脂糖凝胶电泳检测并回收后进行浓度测定和等量混样,充分混匀后的产物再经2%琼脂糖胶凝胶电泳纯化,委托北京诺禾致源股份有限公司进行文库构建及上机测序。
1.2.5 测序数据分析按照97%的一致性(Identity),利用Uprase软件将所有样品的Clean Reads聚类成OTUs(Operational Taxonomic Units),通过软件TBtools绘制韦恩图。使用Qiime软件计算Shannon指数、Simpson指数、Chao1指数、ACE指数等微生物α多样性指数;计算各样品间的Unifrac距离,构建UPGMA(Unweighted Pair-group Method with Arithmetic Mean)聚类树。通过稀释曲线绘制和主成分分析(PCA,Principal Component Analysis)对测序数据进行评价。
2 结果 2.1 不同轮作处理对棉花产量的影响与棉花连作相比,花生-棉花轮作使棉花单铃重提高了17.2%,籽棉产量增加了32.19%;谷子-棉花轮作使棉花单铃重提高了14.5%,籽棉产量增加了18.13%(表 1),表明轮作显著提高了棉花铃重和籽棉产量。
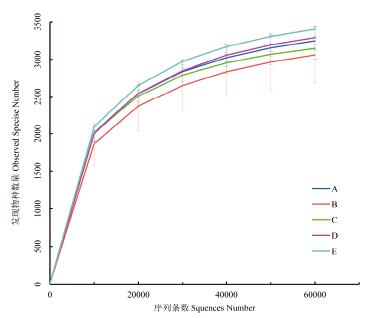
试验中所有样品经过质控平均得到80,167条有效数据(表 2),质控有效率达93.85%。随着测序量的不断增加,各处理稀释曲线趋向平坦(图 1),表明样品文库测序数据量合理,测序深度已基本覆盖到样品中的所有细菌。
|
| A:棉花连作;B:花生-棉花轮作;C:谷子-棉花轮作;D:谷子连作;E:花生连作 图 1 不同种植方式下根际土壤细菌稀释曲线 |
以97%作为一致性标准,将所有序列聚类成为OTUs,5个处理共产生6 174个OTUs。各处理OTUs数量由表 3可知,轮作组(处理B和处理C)OUTs数量低于连作组(处理A、处理D、处理E),表明轮作处理降低了根际土壤细菌物种数量。
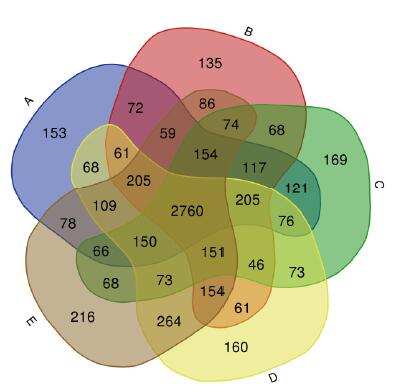
OTUs分布如图 2所示,所有处理共有的OTUs为2 760个,占全部OTUs的44.7%。3个连作处理的特有OTUs分布中,处理A与处理D之间,A特有OTUs为820个,D特有OTUs为982个;处理A与处理E之间,A特有OTUs为873个,E特有OTUs为1 086个。花生连作和谷子连作中特有OUTs数量明显多于棉花连作。2个轮作处理与棉花连作的特有OTUs分布中,处理A与处理B之间,A特有OTUs为821个,B特有OTUs为775个;处理A与处理C之间,A特有OTUs为805个,C特有OTUs为722个。同棉花连作相比,轮作处理降低了棉花根际土壤细菌中特有的OTUs数量。
|
| A:棉花连作;B:花生-棉花轮作;C:谷子-棉花轮作;D:谷子连作;E:花生连作 图 2 不同种植方式下的根际土壤细菌OTUs分布韦恩图 |
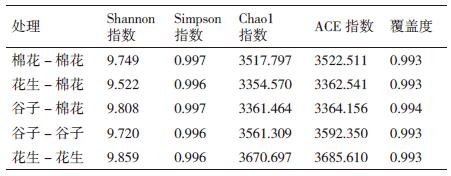
为进一步分析不同种植方式下根际土壤细菌的多样性和丰富度,采用α多样性分析方法,分析结果见表 4,反映物种多样性的Shannon指数、Simpson指数显示,花生-棉花轮作的Shannon指数低于棉花连作,Simpson指数无差异;谷子-棉花轮作的Shannon指数高于棉花连作,Simpson指数无差异。反映物种丰富度的Chao1指数、ACE指数显示,2个轮作处理分别与棉花连作相比,其各项指数均降低;花生-棉花轮作与花生连作相比,其各项指数均显著降低;谷子-棉花轮作与谷子连作相比,其各项指数均显著降低。同连作处理相比,轮作处理降低了根际土壤细菌丰富度,物种数量减少。
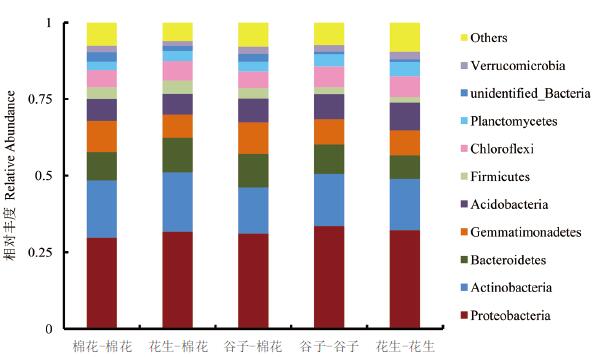
在门的水平上(图 3),5种种植方式共发现10个主要菌门(> 1%),分别是变形菌门(Proteobacteria,29.69%-33.38%)、放线菌门(Actinobacteria,15.09%-19.39%)、拟杆菌门(Bacteroidetes,7.63%- 11.31%)、芽单胞菌门(Gemmatimonadetes,7.70%- 10.23%)、酸杆菌门(Acidobacteria,6.70%-9.03%)、厚壁菌门(Firmicutes,1.84%-4.55%)、绿弯菌门(Choroflexi,5.54%-6.96%)、浮霉菌门(Planctomycetes,2.83%-4.75%)、未辨识菌门(unidentified- Bacteria,0.78%-2.94%)、疣微菌门(Verrucomicrobia,1.61%-2.43%)。这些优势菌门中的变形菌门在谷子连作中占比33.38%,花生连作中占比32.15%,棉花连作中占比29.69%,同棉花连作相比,轮作处理提高了变形菌门的相对丰度,花生-棉花轮作提高了1.58%,谷子-棉花轮作提高了1.29%;浮霉菌门在棉花连作中相对丰度为2.83%,是5个处理中丰度最低的,但在谷子连作中的相对丰度高达3.97%,花生连作中的相对丰度高达4.74%,同棉花连作相比,花生-棉花和谷子-棉花两个轮作处理的浮霉菌门相对丰度分别提高了18.2%和13.7%。拟杆菌门在3个连作处理(棉花连作、谷子连作、花生连作)中的相对丰度分别9.19%、9.54%、7.63%,在2个轮作处理(花生-棉花轮作和谷子-棉花轮作)中的相对丰度分别为11.31%和11.07%。与连作处理相比,轮作处理提高了拟杆菌门的相对丰度。
|
| 图 3 不同种植方式下根际土壤细菌在门水平上的相对丰度 |
在属的水平上(图 4),所有处理相对丰度排名前十的菌属分别有鞘氨醇单胞菌属(Sphingomonas,2.98%-3.95%)、未知细菌属(unidentified_Bacteria,0.64%-2.67%)、未知酸杆菌属(unidentified_ Acidobacteria,1.79%-2.82%)、Pontibacter(0.13%- 1.83%)、溶杆菌属(Lysobacter,0.62%-1.93%)、拟杆菌属(Bacteroides,0.01%-0.70%)、莱茵海默氏菌属(Rheinheimera,0.007%-0.88%)、Haliangium (1.02%-1.54%)、类诺卡氏菌属(Nocardioides,1.05%-1.81%)、小梨形菌属(Pirellula,0.59%- 1.32%)。其中鞘氨醇单胞菌属(Sphingomonas)在5个处理中的相对丰度最大(≥ 3%),该菌属在花生连作和谷子连作中相对丰度最高,分别为3.55%、3.96%,在棉花连作中丰度最低为2.98%,通过轮作处理后,该菌属在棉花根际土壤细菌中的相对丰度均提高,在花生-棉花轮作中的丰度提高到3.93%,在谷子-棉花轮作中的丰度提高到3.30%。该结果表明轮作处理提高了棉花根际土壤中鞘氨醇单胞菌属的相对丰度。
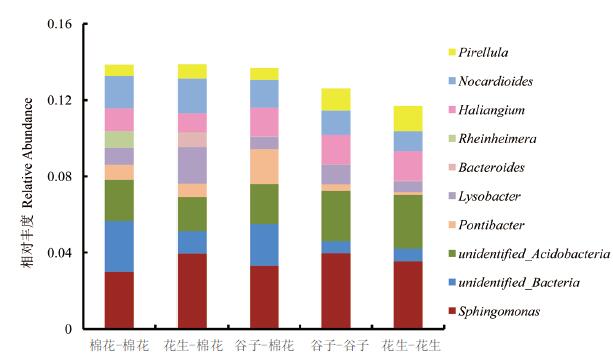
|
| 图 4 不同种植方式下根际土壤细菌在属水平上的相对丰度 |
基于OTU水平的PCA分析结果如图 5所示,主成分1(PCA1)和主成分2(PCA2)对样品的贡献值分别为42.69%和21.41%。处理A(棉花-棉花)与处理B(花生-棉花)、处理C(谷子-棉花)两个处理的距离较近,而与处理D(谷子-谷子)、处理E(花生-花生)两个处理的距离较远。
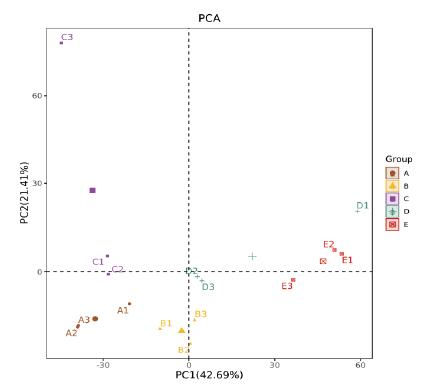
|
| 图 5 各处理根际土壤细菌群落的PCA 分析 |
UPGMA聚类分析如图 6所示,棉花-棉花、花生-棉花和谷子-棉花3个处理因其根际细菌群落结构相似度高被聚类在一起,谷子-谷子、花生-花生2个处理因菌群结构相似被聚类为一个分支,这与PCA分析的结果一致。

|
| 图 6 门水平上的根际土壤细菌UPGMA 聚类树 |
连作导致土壤病原菌富集,土壤质量恶化,从而使得作物产量降低。研究表明,轮作能协调作物与土壤之间的关系,改善根际微生态环境,有助于提高作物产量,是防治作物发生连作障碍的有效途径之一[3, 16-18]。本研究中对比棉花连作、轮作处理中棉花的单铃重和籽棉产量均显著提高,表明轮作处理对棉花产量有积极作用。
植物根际为微生物提供能源物质,有研究证明轮作制度通过改变作物的种类和生物量的方式增加了土壤微生物的食物来源,进而导致土壤微生物多样性的改变[19]。本研究通过高通量测序技术以97%的一致性将序列聚类出共6 174个OTUs,根据各处理OTUs分布绘制韦恩图,用来分析不同样本(组)之间共有、特有的OTUs,其中棉花连作、谷子连作和花生连作3个连作处理中共有OTUs为3 224个,棉花连作中特有OTUs为463个;棉花参与的处理(棉花连作、花生-棉花轮作和谷子-棉花轮作)中共有OTUs为3 236个,棉花连作中特有OTUs为408个,这表明轮作处理后的棉花根际土壤中共有细菌OUTs增多,特有OTUs减少。α多样性指数用来解释物种的多样性和丰富度,细菌α多样性指数显示,代表根际土壤细菌多样性的Shannon指数和Simpson指数无显著差异,但代表样品群落丰富度的ACE指数和Chao1指数显示,轮作处理的数值低于连作处理。通过对本试验根际土壤细菌OTUs分布和细菌α多样性指数的分析可知,轮作处理使棉花根际土壤细菌物种数量减少,丰富度明显降低。有研究表明连作可以导致病原微生物富集,降低土壤微生物活性及数量[20-22],所以本试验存在轮作措施促进了棉花根际土壤中某些有益细菌的活性,例如拟杆菌门、变形菌门、浮霉菌门和鞘氨醇单胞菌属的富集,从而抑制了病原菌存活的现象。
土壤中庞大的微生物数量意味着其群落结构的复杂性,群落结构的变化趋势以群落组分变化为基础[23],不同的作物类型,对应其土壤细菌群落的组成成分不同,这就意味着生长作物的变化为某些特异性细菌提供了生存环境。为此,菌群针对作物的改变重新进行了选择性适应。王悦等[24]通过研究不同种植模式下丹参根际土壤微生物群落结构发现轮作模式下芽单胞菌门相对丰度低于连作和套作,轮作和套作时浮霉菌门和拟杆菌门的相对丰度则高于连作。试验通过分析不同种植方式下的根际土壤细菌群落组成及相对丰度发现,门水平上的变形菌门、浮霉菌门和属水平上的鞘氨醇单胞菌属,其相对丰度在花生连作和谷子连作中高,而在棉花连作中低,通过轮作处理后,其菌门(属)在棉花根际土壤细菌中的相对丰度均明显提高;另有拟杆菌门在轮作中的相对丰度均高于连作。这表明了轮作处理中前茬作物的存在给予了棉花根际土壤中这些菌门(属)适宜的生存环境,促进了它们的活性。土壤细菌在植物生长过程中发挥着重要作用,孙建光等[25]从玉米根际土壤中分离出鞘氨醇单胞菌属菌株GD542,该菌株具有较高的固氮酶活性,可以提高土壤中的全氮含量;王秀红等[26]研究发现变形菌门、浮霉菌门、拟杆菌门等是好氧堆肥过程中共享的优势菌门,它们的存在提高了植物对土壤碳源的利用效率。通过前人的研究成果可以推断,试验中受轮作处理影响的细菌菌门(属)可以为棉花的生长带来积极地影响。
主成分分析[27]是一种基于欧式距离的应用 方差分解,对多维数据进行降维,从而提取出数据中最主要的元素和结构的方法。各样品在PCA图中的距离越接近,则它们的群落组成越相似。UPGMA[28]是为了研究不同样本间相似性的聚类分析方法。基于OTU水平的PCA分析和UPGMA聚类分析发现,花生-棉花轮作处理、谷子-棉花轮作处理和棉花连作处理的根际土壤细菌组成相似性更强,这表明虽然棉花不同的种植方式会影响其菌群相对丰度的高低,但其群落结构组成总体上不会发生显著变化。这与Li等[29]发现根系分泌物虽可改变细菌群落中某些主要种群的丰富度,但是不足以改变细菌的群落结构的研究结论一致。
本研究使用的高通量测序技术仅为二代测序技术,不能精确到种的水平,而且试验只检测了棉花根际土壤中细菌的变化。未来应使用精细到种水平上的三代测序技术,来监测棉田根际土壤中细菌、真菌的群落结构变化,这对进一步探究棉花根际土壤微生态功能具有重要意义。
4 结论本研究发现对棉田进行花生-棉花轮作处理可以使棉花的增产效果更显著。通过高通量测序技术,检测各处理的根际土壤细菌发现,轮作处理中的前茬作物(花生、谷子)影响了棉田根际土壤菌群的丰度,它们的存在提高了棉田中拟杆菌门、变形菌门、浮霉菌门、鞘氨醇单胞菌属的相对丰度,这些菌门(属)的含量与棉花的生长发育密切相关。
| [1] |
林先贵, 胡君利. 土壤微生物多样性的科学内涵及其生态服务功能[J]. 土壤学报, 2008, 45(5): 892-900. Lin XG, Hu JL. Scientific connotation and ecological service function of soil microbial diversity[J]. Acta Pedologica Sinica, 2008, 45(5): 892-900. |
| [2] |
薛超, 黄启为, 凌宁, 等. 连作土壤微生物区系分析、调控及高通量研究方法[J]. 土壤学报, 2011, 48(3): 612-618. Xue C, Huang QW, Ling N, et al. Analysis, regulation and highthroughput sequencing of soil microflora in mono-cropping system[J]. Acta Pedologica Sinica, 2011, 48(3): 612-618. |
| [3] |
刘军, 唐志敏, 刘建国, 等. 长期连作及秸秆还田对棉田土壤微生物量及种群结构的影响[J]. 生态环境学报, 2012, 21(8): 1418-1422. Liu J, Tang ZM, Liu JG, et al. Effects of cotton continuous cropping and returning stalks to soil on the quantities and community structure of soil microbes[J]. Ecology and Environmental Sciences, 2012, 21(8): 1418-1422. |
| [4] |
张晓玲, 潘振刚, 周晓锋, 等. 自毒作用与连作障碍[J]. 土壤通报, 2007(4): 781-784. Zhang XL, Pan ZG, Zhou XF, et al. Autotoxicity and continuous cropping obstacles :A review[J]. Chinese Journal of Soil Science, 2007(4): 781-784. |
| [5] |
郑良永, 胡剑非, 林昌华, 等. 作物连作障碍的产生及防治[J]. 热带农业科学, 2005(2): 58-62. Zheng LY, Hu JF, Lin CH, et al. The production of succession cropping obstacles and its prevention and cure steps[J]. Chinese Journal of Tropical Agriculture, 2005(2): 58-62. |
| [6] |
刘金波, 许艳丽. 我国连作大豆土壤微生物研究现状[J]. 中国油料作物学报, 2008(1): 132-136. Liu JB, Xu YL. Current research of soil microbial of successive soybean cropping in China[J]. Chinese Journal of Oil Crop Science, 2008(1): 132-136. |
| [7] |
李文娇, 杨殿林, 赵建宁, 等. 长期连作和轮作对农田土壤生物学特性的影响研究进展[J]. 中国农学通报, 2015(3): 173-178. Li WJ, Yang DL, Zhao JN, et al. Progress on effects of long-term continuous cropping and rotation systems on farmland soil biological characteristics in farmland[J]. Chinese Agricultural Science Bulletin, 2015(3): 173-178. |
| [8] |
祁虹, 王树林, 王燕, 等. 粮棉轮作与土壤深翻对棉花生育性状及产量的影响[J]. 天津农业科学, 2016, 22(8): 113-116. Qi H, Wang SL, Wang Y, et al. Effects of rotation and soil deep ploughing on cotton development traits and yield[J]. Tianjin Agricultural Sciences, 2016, 22(8): 113-116. |
| [9] |
姚钦 . 黑土区轮作方式下土壤微生物多样性研究[D]. 哈尔滨:东北林业大学 , 2012. Yao Q. Microorganism diversity of mollisols in different rotation systems[D]. Harbin :Northeast Forestry University, 2012. |
| [10] |
John D, Barns SM, Ticknor LO, et al. Empirical and theoretical bacterial diversity in four Arizona soils[J]. Applied and Environmental Microbiology, 2002, 68(6): 3035-3045. DOI:10.1128/AEM.68.6.3035-3045.2002 |
| [11] |
蒋永梅, 师尚礼, 田永亮, 等. 高寒草地不同退化程度下土壤微生物及土壤酶活性变化特征[J]. 水土保持学报, 2017, 31(3): 244-249. Jiang YM, Shi SL, Tian YL, et al. Characteristics of soil microorganism and soil enzyme activities in alpine meadows under different degrees of degradation[J]. Journal of Soil and Water Conservation, 2017, 31(3): 244-249. |
| [12] |
Delgado-Baquerizo M, Maestre FT, Reich PB, et al. Carbon content and climate variability drive global soil bacterial diversity patterns[J]. Ecological Monographs, 2016, 86(3): 373-390. DOI:10.1002/ecm.1216 |
| [13] |
吕雅悠, 于迪, 丁方丽, 等. 促植物生长根际细菌 A21-4 对田间辣椒生长及根际土壤微生态环境的影响[J]. 中国生物防治学报, 2016, 32(1): 86-92. Lu YY, Yu D, Ding FL, et al. Effects of PGPR strain A21-4 on growth and rhizosphere soil charactors of pepper in field[J]. Chinese Journal of Biological Control, 2016, 32(1): 86-92. |
| [14] |
王娜 . 植物内生细菌的定殖动态及其对辣椒疫病和棉花黄萎病的防治效果研究[D]. 南京 :南京师范大学 , 2015. Wang N. Colonization dynamics of endophytic bacteria and its effect on control of pepper blight and cotton verticillium wilt[D]. Nanjing :Nanjing Normal University, 2015. |
| [15] |
Schuster SC. Next-generation sequencing transforms today’s biology[J]. Nature Methods, 2008, 5(1): 16-18. |
| [16] |
吴艳飞, 张雪艳, 李元, 等. 轮作对黄瓜连作土壤环境和产量的影响[J]. 园艺学报, 2008(3): 357-362. Wu YF, Zhang XY, Li Y, et al. Influence of rotation on continuous cropping soil environment and cucumber yield[J]. Acta Horticulturae Sinica, 2008(3): 357-362. |
| [17] |
Stark C, Condron LM, Stewart A, et al. Effects of past and current crop management on soil microbial biomass and cativity[J]. Biology and Fertility of Soils, 2007, 43(5): 531-540. DOI:10.1007/s00374-006-0132-3 |
| [18] |
徐文修, 罗明, 李银平, 等. 作物茬口对连作棉田土壤环境及棉花产量的影响[J]. 农业工程学报, 2011, 27(3): 271-275. Xu WX, Luo M, Li YP, et al. Effects of crop stubbles on cotton yield and soil environment in continuously cropped cotton field[J]. Transactions of the Chinese Society of Agricultural Engineering, 2011, 27(3): 271-275. |
| [19] |
Coskun D, Britto DT, Shi W, et al. How plant root exudates shape the nitrogen cycle[J]. Trends Plant Sci, 2017, 22: 661-673. DOI:10.1016/j.tplants.2017.05.004 |
| [20] |
吴凤芝, 赵凤艳, 刘元英. 设施蔬菜连作障碍原因综合分析与防治措施[J]. 东北农业大学学报, 2000(3): 241-247. Wu FZ, Zhao FY, Liu YY. On the reasons of continuous cropping obstacles in vegetable facility gardening[J]. Journal of Northeast Agricultural University, 2000(3): 241-247. |
| [21] |
高旭梅, 刘娟, 张前兵, 等. 耕作措施对新疆绿洲长期连作棉田土壤微生物、酶活性的影响[J]. 石河子大学学报 :自然科学版, 2011, 29(2): 145-152. Gao XM, Liu J, Zhang QB, et al. Effects of tillage practices on soil microbial and enzyme activity in long-term continuous cotton of xinjiang oasis[J]. Journal of Shihezi University :Natural Science Edition, 2011, 29(2): 145-152. |
| [22] |
徐瑞富, 王小龙. 花生连作田土壤微生物群落动态与土壤养分关系研究[J]. 花生学报, 2003(3): 19-24. Xu RF, Wang XL. Relation of microbial population dynamics and nutrient in soil of continuous cropping with peanut[J]. Journal of Peanut Science, 2003(3): 19-24. |
| [23] |
苗祯, 杜宗军, 李会荣, 等. 5 株北极微藻藻际环境的细菌多样性[J]. 生态学报, 2015, 35(5): 1587-1600. Miao Z, Du ZJ, Li HR, et al. Analysis of bacterial diversity in the phycosphere of five Arctic microalgae[J]. Acta Ecologica Sinica, 2015, 35(5): 1587-1600. |
| [24] |
王悦, 杨贝贝, 王浩, 等. 不同种植模式下丹参根际土壤微生物群落结构变化[J]. 生态学报, 2019, 39(13): 4832-4843. Wang Y, Yang BB, Wang H, et al. Variation in microbial community structure in the rhizosphere soil of Salvia miltiorrhiza Bunge under three cropping modes[J]. Acta Ecologica Sinica, 2019, 39(13): 4832-4843. |
| [25] |
孙 建 光, 张 燕 春, 徐晶, 等. 玉 米 根 际 高 效 固 氮 菌Sphingomonas sp[J]. 中国生态农业学报, 2010, 18(1): 89-93. Sun JG, Zhang YC, Xu J, et al. Isolation, identification and inoculation effect of nitrogen-fixing bacteria Sphingomonas GD542 from maize rhizosphere[J]. Chinese Journal of Eco-Agriculture, 2010, 18(1): 89-93. |
| [26] |
王秀红, 李欣欣, 史向远, 等. 好氧堆肥微生物代谢多样性及其细菌群落结构[J]. 环境科学研究, 2008, 31(8): 1457-1463. Wang XH, Li XX, Shi XY, et al. Microbial metabolism diversity and bacterial flora structure during aerobic composting[J]. Research of Environmental Sciences, 2008, 31(8): 1457-1463. |
| [27] |
Jolliffe IT. Principal component analysis[J]. Journal of Marketing Research, 1986, 87(100): 513. |
| [28] |
Li Y, Xu L. An improvement for unweighted pair group method with arithmetic mean and its application[J]. Journal of Beijing University of Technology, 2007, 33(12): 1333-1339. |
| [29] |
Li XG, Ding CF, Hua K, et al. Soil sickness of peanuts is attributable to modifications in soil microbes induced by peanut root exudates rather than to direct allelopathy[J]. Soil Biology and Biochemistry, 2014, 78: 148-159. |