2. 天水师范学院生物工程与技术学院, 天水 741001
2. School of Bioengineering and Biotechnology, Tianshui Normal University, Tianshui 741001
淀粉是普遍存在于植物体内, 用于植物贮存能量的一种多糖[1]。目前生产淀粉的原料主要是玉米、小麦、薯类、稻谷等作物[2-3]。我国作为农业大国, 根据中国农业统计资料显示截止到2017年, 上述四类作物的产量已占我国粮食总产量的90%以上[4]。淀粉是以葡萄糖为单位通过α-1, 4糖苷键连接的高分子碳水化合物。依据单糖连接方式, 淀粉可分为直链淀粉和支链淀粉[5]。淀粉是人类的重要食物来源, 也是多种工业生产的原料[6]。淀粉加工工艺主要有酸法、酶酸法、全酶法和双酶法等。其中, 酶法在国内外加工淀粉中已广泛应用[7]。α-淀粉酶(EC 3.2.1.1)是指通过水解淀粉分子内部中的α-1, 4-糖苷键, 将淀粉水解为糊精、单糖、少量麦芽糖和葡萄糖的一类酶[8]。来源于细菌和真菌的α-淀粉酶被广泛应用于食品、生物燃料、造纸、医药、纺织、洗涤剂、动物饲料等行业, 占整个酶制剂市场的25%-30%[9-10]。在面团发酵过程中, 淀粉酶通过对淀粉的水解作用提高面团的糖含量, 进而增大面包的体积, 改善色泽[11-12]。在超声波作用下对织物进行酶退浆, 不仅可以提高退浆效率, 还缩短退浆时间[13]。在生物乙醇的生产过程中, 淀粉酶发挥着将原料醪液液化的作用[14]。淀粉酶还可以将含淀粉的食物颗粒水解成小分子的水溶性寡糖, 用水冲洗后可去除污渍。当前90%的液体除垢剂中含有α-淀粉酶[15]。将淀粉酶添加到动物饲料中, 可有效提高饲料中淀粉的消化率, 有利于动物的生长发育[16]。
在不同的应用领域, 对淀粉酶性能的要求不同。特别是工业化生产中的高温、低温、高盐、碱性、酸性等苛刻条件制约着淀粉酶产业的发展[17-18]。因此, 随着全球资源环境压力的持续增大和绿色合成产业的需求, 酶的应用和需求量迅速增加, 酶基因资源的挖掘和开发显得非常迫切。目前, 热稳定性的α-淀粉酶主要来源于细菌(特别是芽孢杆菌), 而放线菌和真菌来源的报道较少[19]。高温放线菌类群是一类能够在极端环境下生长代谢的革兰氏阳性、化能异养菌。微生物学界认为它是介于细菌和放线菌之间的一类微生物。高温放线菌具有代谢产物丰富, 很多具有重要的工业价值, 其中蛋白酶、淀粉酶、脂酶、纤维素酶等应用广泛[20]。2005年, Yoon等[21]报道高温放线菌-莱斯氏属(Laceyella)。其主要分布于大曲[22-23]、温泉[24]、海洋[25]、土壤[26]、堆肥[27]等。朱国东等[28]从云南腾冲热泉中筛选到一株嗜热放线菌Laceyella sp.RHA1, 淀粉酶的最适温度为60℃。来源于Laceyella sacchari5-氨基乙酰丙酸合成酶LS-ALAS不仅具有良好的热稳定性, 还有较高的酶活力[29]。研究还发现, 高温放线菌Laceyella sacchari有一定的降解纤维素的能力[30]。Singh[31]研究了Laceyella sacchari B42的木聚糖酶活性, 其研究表明, 最适反应温度和pH分别为9.0和60℃。在60℃处理6 h后还保存90%的活性。Dolashki等[32]研究了Laceyella sacchari酪氨酸酶的性质。Laceyella sacchari嗜热蛋白酶在乳酸乳球菌表达系统中成功表达, 且重组酶的热稳定性好, 还在pH 6.0-11.0范围内具有较高的活性[33]。来源于Laceyella putida葡聚糖酶LpGluA的最适反应pH和最适反应温度分别为4.2和60℃[34]。Laceyella sacchari亮氨酸脱氢酶Ls-LeuDH也具有较好的热稳定性[25]。在50℃处理72 h时, Laceyella sacchari LP175菌株聚乙丙交酯降解酶的粗酶对PLLA的分解效率达到91%[26]。另外, 其还具有高效的生淀粉降解能力, 生物乙醇的产量高达90.9 g/L[35]。Laceyella sp. DS3 α-淀粉酶AmyLa最适反应温度和pH分别为55℃和7.0, 其固定化酶的最适反应温度没有变化, 但是提高了在pH4.0-7.0范围内的酶活性[36-37]。来源于Laceyella sacchari TSI-2的α-淀粉酶在70℃和pH7.0时表现出最高活性, 能够高效去除棉布上的淀粉污渍, 其固定化酶的稳定性增强[19, 38]。四甲基吡嗪作为Laceyella sacchari产生的大量代谢物, 还被认为是酱香白酒风味的主体成分之一[23]。因此, 积极开展嗜热放线菌Laceyella sp.的相关研究十分重要。
本研究从安阳面粉厂附近土壤里分离筛选产α-淀粉酶的Laceyella sp.菌株, 采用同源克隆技术获取α-淀粉酶基因。其次, 在大肠杆菌表达系统中进行功能研究。最后, 基于同源建模方法的蛋白质三维结构, 分析差异氨基酸位点与酶学性质上的差异, 为进一步的分子改良提供基础。
1 材料与方法 1.1 材料 1.1.1 样品、菌株及质粒样品:采集自安阳市面粉厂附近的土壤。
菌株: E.coli Trans1-T1、E.coli BL21(DE3)购自北京全式金生物技术有限公司。
质粒: pET-22b(+)表达载体由本实验室保存。
1.1.2 培养基固体淀粉筛选培养基(g/L):可溶性淀粉5 g, 牛肉膏3 g, 蛋白胨10 g, NaCl 5 g, 琼脂20 g。
高氏一号培养基(g/L):可溶性淀粉20 g, KNO3 1 g, K2HPO4 0.5 g, MgSO4·7H2O 0.5 g, NaCl 0.5 g, FeSO4·7H2O 0.01 g, 琼脂20 g。
1.1.3 主要仪器设备PCR仪:伯乐生命医学产品有限公司; HVE-50高压蒸汽灭菌锅:日本HIRAYA-MA公司; SPECOOD S600分光光度计:德国耶拿分析仪器有限公司; HNY-ⅢB摇床:天津欧诺仪器股份有限公司; CF16RN高速离心机:株式会社日立制作所; 电泳仪:伯乐生命医学产品(上海)有限公司; ÄKTA蛋白纯化仪:通用电气医疗集团。
1.2 方法 1.2.1 产淀粉酶菌株的筛选及鉴定取适量土样加入到50 mL无菌水中制成土壤悬液, 37℃, 180 r/min培养20 min, 作为原液。取原液进行梯度稀释, 分别取10-4、10-5、10-6的稀释菌悬液100 μL涂布于固体淀粉筛选培养基上, 37℃培养12 h。在固体淀粉筛选培养基上滴加碘液, 选取能产生无色透明圈的菌落, 且透明圈直径与菌落直径之比较大者进行菌株鉴定。
依据细菌基因组提取试剂盒说明书进行提取DNA操作。以细菌通用鉴定引物27F: 5’-AGAGTTTGATCCTGGCTCAG-3’、1492R: 5’-GGTTACCTTGTTACGACTT-3’, 扩增菌株的16S rDNA序列。PCR扩增完成后, 取适量产物于1%琼脂糖凝胶进行电泳检测, 以TAE缓冲液作为电泳缓冲液, 电压150 V, 20 min。条带大小验证正确后, 取PCR扩增产物送至公司测序。在NCBI(https: //www.ncbi.nlm.nih.gov/)网站上, 选用BLASTn程序对菌株的16S rDNA序列与GenBank数据库进行比对分析。最后, 依据LPSN(http://www.bacterio.net/)公布的Laceyella sp.属标准菌株, 通过MEGA7.0软件[39]的邻位相连法[40], 构建系统发育树。
1.2.2 α-淀粉酶基因AmyL的克隆以Laceyella sp.DS3(GenBank号: LN901326)基因组序列中的α-淀粉酶基因序列为依据, 设计正向引物MF-8-1 F: 5’-GAAGTGACCCAGCTACGAGATGAGC-3’以及反向引物MF-8-1 R: 5’-GACCCCAACTATATCAAGCCAGAATTCGG-3’, 通过PCR扩增得到α-淀粉酶基因AmyL全长。
1.2.3 pET-22b-AmyL重组质粒的构建以重叠延伸PCR[41](Overlap-PCR)构建重组表达载体。根据α-淀粉酶基因AmyL以及质粒pET-22b(+)的序列设计引物Amy MF-8-1 F1: 5’-GGATATCGGAATTAATTCGGATCCGAATTCGCTTTCTCCTGCGGATTGGCAAG-3’、Amy MF-8-1 R1: 5’-GTGGTGGTGGTGGTGCTCGAGTTTGGTGAATATCTTTACTTCTTTGGGGC-3’、Amy MF-8-1 F2: 5’-GCCCCAAAGAAGTAAAGATATTCACCAAACTCGAGCACCACCACCACCAC-3’、Amy MF-8-1 R2: 5’-CTTGCCAATCCGCAGGAGAAAGCGAATTCGGATCCGAATTAATTCCGATATCC-3’。其中Amy MF-8-1 F1与Amy MF-8-1 R2反向互补, Amy MF-8-1 R1与Amy MF-8-1 F2反向互补。
通过上述引物对基因以及质粒进行PCR扩增, 引入同源臂。再以质粒扩增产物为模板、基因扩增产物为大引物, 两者按照质量比为1:10构建反应体系进行PCR扩增, 吸取部分PCR产物进行琼脂糖凝胶电泳检测。将经由PCR扩增后构建成功的重组表达载体命名为pET22b-AmyL, 并转化至E.coli Trans1-T1, 同时选取菌液PCR验证正确的单克隆进行测序验证。
1.2.4 重组α-淀粉酶AmyL的表达及纯化将测序正确的重组表达载体转化到E.coli BL21(DE3)中表达, 然后以1%的接菌量转接于200 mL LB液体培养基(氨苄霉素浓度: 100 μg/mL)中, 37℃、180 r/min培养2 h至OD600达到0.6-0.8之间, 加入400 μL 0.5 mol/L IPTG, 30℃、180 r/min诱导4 h。
通过ÄKTA蛋白纯化系统纯化胞内总蛋白溶液[42]: (1)将胞内总蛋白溶液经0.22 μm滤膜过滤; (2)用结合缓冲液平衡层析柱; (3)取经过滤膜过滤的样品上样; (4)用洗脱缓冲液进行线性洗脱; (5)收集洗脱峰进行酶活性的测定。对纯化后的蛋白质进行SDS-PAGE以及HPLC-MS/MS质谱分析鉴定。
1.2.5 重组α-淀粉酶AmyL活性测定及性质研究采用二硝基水杨酸(DNS)法[43]进行α-淀粉酶的酶活力测定。将900 μL 1%淀粉酶底物在45℃预热5 min, 加入100 μL稀释的酶液反应10 min后, 用DNS溶液终止反应, 沸水浴5 min。反应液冷却至室温后, 测定540 nm的吸光值。淀粉酶的酶活单位定义为:在最适温度和最适pH下, 每毫升反应体系每分钟分解淀粉生成1 μmol还原糖所需的酶量为一个酶活单位。
1.2.5.1 最适反应温度的测定在pH6.0反应体系下, 分别测定温度在25-60℃时的酶活力, 反应时间为10 min。酶活力最高点即为酶的最适反应温度, 同时计算不同温度下的相对酶活力。
1.2.5.2 最适反应pH的测定在45℃反应条件下, 以柠檬酸缓冲液和磷酸缓冲液配制, 分别测定pH为4.8、5.2、5.6、6.0、6.4、6.8、7.2、7.6、8.0时的酶活力, 反应时间为10 min。酶活力最高点即为酶的最适反应pH, 同时计算其他pH下的相对酶活力。
1.2.5.3 温度稳定性的测定取适当酶液, 分别放置在40、45、50、60℃下水浴保温10、20、30、50 min, 测定残余的酶活力, 以未处理的酶液作对照, 计算处理不同时间后残余的相对酶活力。
1.2.5.4 pH稳定性的测定取适当酶液在温度为45℃下, 分别在pH为5.0、6.0、7.0、8.0、9.0、10.0下保温1 h后, 以未处理的酶液作对照, 测定不同pH下处理后残余的相对酶活力。
1.2.5.5 酶促反应动力学研究以经过蛋白质纯化后的目的蛋白, 对淀粉酶的动力学参数进行测定。配制不同浓度的淀粉底物, 基于淀粉酶的最适反应温度、最适反应pH, 测定酶的活力, 同时测定蛋白浓度, 计算出酶的比活力。比活力定义:每毫克酶蛋白所含的酶活力单位。以1/[S]为横坐标, 1/比活为纵坐标, 作Lineweaver-Burk双倒数图[44]。
1.2.5.6 金属离子及化学试剂对酶活力的影响在酶学性质测定反应体系中加入终浓度分别为1、5和10 mmol/L的不同离子及化学试剂, 测定酶活力, 以不加化学试剂的反应体系作为对照。探究不同金属离子及化学试剂对淀粉酶活力的影响。
1.2.5.7 底物特异性研究将可溶性淀粉、支链淀粉、马铃薯淀粉、普鲁兰多糖、α-环糊精、β-环糊精、γ-环糊精等底物分别溶解于0.1 mmol/L pH 6.0的柠檬酸-磷酸氢二钠缓冲溶液, 终浓度为5 mg/mL。加入适当稀释倍数的重组淀粉酶AmyL, 在45℃下反应10 min。以可溶性淀粉为底物时的酶活力为100%, 分别计算其他底物的相对酶活力。
1.2.6 重组酶的同源建模从PDB数据库中检索与AmyL序列相似且功能已知的α-淀粉酶, 以来源于Anoxybacillus的α-淀粉酶(PDB ID号: 5A2B;序列一致性: 56%)为模板, 利用Discovery Studio 2018软件对淀粉酶AmyL进行三维结构的建模。然后使用相关模块对构建好的模型进行能量最小化和结构优化, 并采用Ramanchandran Plot和Profile-3D评估模型。Deepview软件也应用于部分结构功能分析。
2 结果 2.1 产淀粉酶菌株的筛选及鉴定土壤样品经稀释涂布后, 在固体淀粉筛选培养基上, 选取具有较大透明圈的菌落作为候选菌株。在多次分离、纯化的基础上, 获得目的菌株, 编号为MF-8-1。菌株MF-8-1在固体淀粉筛选培养基上的菌落特征为纯白色、干燥、多皱、边缘不规则, 能产生明显的透明圈。
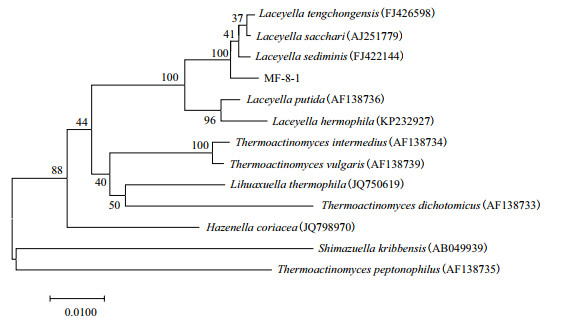
提取MF-8-1的基因组, 利用引物27F和1492R扩增其16S rDNA序列后进行测序。经序列比对后发现其与Laceyella sp.属的16S rDNA序列(GenBank号: EU430566.1)的一致性最高, 序列相似性为99%。依据高温放线菌科及Laceyella sp.属的不同种16S rDNA序列, 构建系统发育树(图 1)。结果表明MF-8-1与Laceyella sediminis(GenBank号: FJ422144)的进化距离最小。
|
| 图 1 菌株MF-8-1的16S rDNA系统发育树 |
以MF-8-1基因组DNA为模板, MF-8-1 F、MF-8-1 R为引物, 在KOD-Plus-Neo高保真聚合酶的作用下, 扩增出一条2.2 Kb左右的片段, 与预期DNA片段大小符合。测序结果表明该基因全长为1467 bp, 编码488个氨基酸和一个终止密码子, 将该基因命名为AmyL。利用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)对AmyL编码蛋白进行信号肽预测。结果表明AmyL编码蛋白具有一个由35个氨基酸残基组成的信号肽, 剪切位点介于Leu35和Ser36之间。
2.3 重组α-淀粉酶AmyL的诱导表达及纯化将构建的重组表达载体转化到BL21, 通过PCR方法验证, 获得重组表达菌株。经IPTG诱导后, 重组菌的细胞破碎液具有很高的淀粉酶活性, 而空白对照无法检测出淀粉酶活性。同时, 对培养两种重组菌的上清液进行淀粉酶活性的检测后, 结果显示上清液均无活性, 表明淀粉酶基因AmyL在周质中表达。
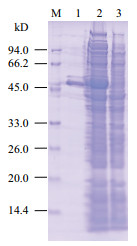
对重组菌的胞内总蛋白进行蛋白纯化, 并对纯化后蛋白进行SDS-PAGE电泳检测。结果表明AmyL基因在E.coli BL21(DE3)中得到了较好的表达(图 2), 在55 kD附近出现了较为明显的条带, 且与预期大小相符。经质谱鉴定, 有32个肽段与理论上的肽段相匹配, 其肽段覆盖率为73%, 说明重组子表达的蛋白质为AmyL。
|
| M :蛋白分子量标准; 1 : E.coli BL21(pET-22b-AmyL)纯化后的蛋白; 2 :E.coli BL21(pET-22b-AmyL)胞内总蛋白; 3:空白对照E.coli BL21(pET-22b)胞内总蛋白 图 2 重组α-淀粉酶AmyL的SDS-PAGE分析 |
重组α-淀粉酶AmyL的最适反应温度为45℃, 在40-50℃下可保持较高酶活力, 在60℃下酶活力全部损失(图 3)。在40-50℃下, 酶的稳定性较好, 保温50 min后酶活力仍能维持在80%左右。但在60℃下处理50 min后, 酶活力全部损失(图 4)。
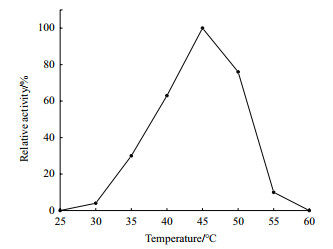
|
| 图 3 温度对重组α-淀粉酶AmyL酶活力的影响 |
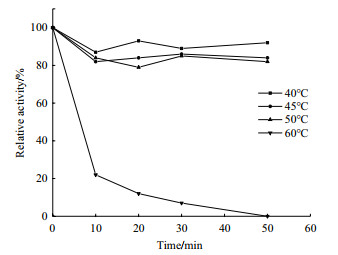
|
| 图 4 重组α-淀粉酶AmyL的温度稳定性 |
重组α-淀粉酶AmyL的最适反应pH值为6.0, 在pH 5.6-6.8下具有较高的酶活性, 其相对酶活在80%以上。pH < 5.6后, 酶活力急剧下降。在pH 4.8、8.0处, 酶活力完全损失(图 5)。在pH 6.0-10.0时, 酶活力均无下降且有些许上升, 说明该酶能耐受偏碱性环境(图 6)。
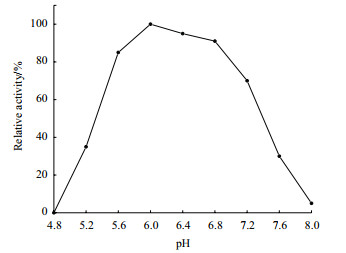
|
| 图 5 pH对重组α-淀粉酶AmyL酶活力的影响 |

|
| 图 6 重组α-淀粉酶AmyL的pH稳定性 |
以0.1、0.4、0.7、1.0、2.0、3.0和5.0 mg/mL浓度的可溶性淀粉作底物, 测定酶的活力; 做Lineweaver-Burk双倒数图(图 7), 数据分析后得出酶的比活、Km和Vmax分别为185.39 U/mg、0.95 mg/mL、343.12 μmol/(min·mg)。进一步计算得出, AmyL的Kcat为315.28 s-1, 催化效率Kcat/Km为331.13 mL/(s·mg)。
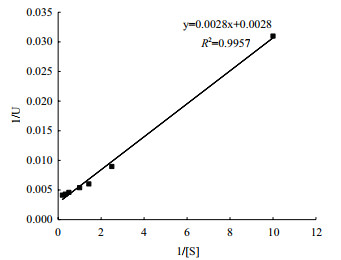
|
| 图 7 重组α-淀粉酶AmyL的Lineweaver-Burk双倒数图 |
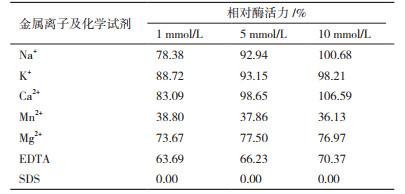
以未加化学试剂的反应体系作为对照, 得到不同浓度的金属离子及化学试剂对重组酶活力的影响如表 1所示。当体系中Na+、K+、Ca2+浓度达到10 mmol/L浓度时, 对酶活力有一定的保护作用, 而较低浓度下会使酶活力损失; Mn2+、Mg2+、EDTA对重组α-淀粉酶AmyL均存在抑制作用, 但Mn2+的抑制作用比Mg2+、EDTA略强; SDS对重组α-淀粉酶AmyL具有强抑制作用, 即使只有1 mmol/L, 也能使酶活力全部丧失, 说明重组α-淀粉酶AmyL对SDS敏感。
重组α-淀粉酶AmyL不仅可以高效地催化可溶性淀粉的水解, 对其他底物也表现出较高的水解活性。将重组α-淀粉酶AmyL对可溶性淀粉的酶活定义为100%时, AmyL对于支链淀粉和马铃薯淀粉的相对酶活力分别为180.3%和111.6%, 对β-环糊精和普鲁兰多糖的相对酶活力分别为2.5%和0.8%, 而以α-环糊精和γ-环糊精为底物时, 未检测到活性。
2.5 α-淀粉酶AmyL同源建模及结构分析AmyL与来源于Laceyella sacchari的α-淀粉酶(TCW41560)的序列一致性为99%, 与晶体结构已知的Anoxybacillus α-淀粉酶5A2B的一致性为56%。以5A2B作为模板, 对AmyL进行同源建模。从结构上可以看出(图 8), AmyL由3个结构域组成, 即结构域A、B、C。结构域A由8个α-螺旋和8个β-折叠交替组成的TIM桶构成; 结构域B是TIM桶的第3个α-螺旋和β-折叠之间形成的不规则β-like结构; 结构域C由位于蛋白质C端的8个反平行β-折叠以及一些无规则卷曲构成。通过分析发现, 结构域A的β3、β4、β5和β7折叠C端附近存在GH13家族的4个CSR: CSR I(152DVVANH157)、CSR II(228DGLRVDTVKHVPKWFWREF246)、CSR III(256GEVFHG261)和CSR IV(317FIDNHD322)。

|
| 图 8 AmyL的三维结构 |
淀粉酶广泛存在于动植物及微生物中, 其中微生物来源的淀粉酶具有各种不同的性质, 在淀粉酶的开发研究中具有相当重要的地位。国内外报道产淀粉酶的微生物[29-35]主要有:枯草芽孢杆菌(Ba-cillus subtilis)[45]、地衣芽孢杆菌(Bacillus licheni-formis)[46]、蜡状芽孢杆菌(Bacillus cereus)[47]、巨大芽孢杆菌(Bacillus megaterium)[48]、多粘类芽孢杆菌(Bacillus polymyxa)[49]、高温放线菌(Therm-oactinomyces)[50]等。
本研究根据Laceyella sp.DS3基因组序列中的α-淀粉酶基因序列, 采用同源克隆从Laceyella sp.中克隆出α-淀粉酶基因AmyL并在大肠杆菌中表达。其最适反应温度为45℃, 最适反应pH值为6.0, 高浓度的Na+、K+、Ca2+能提高该淀粉酶的稳定性。朱国东等[28]从云南腾冲热泉中筛选到一株嗜热放线菌Laceyella sp.RHA1, 经过硫酸铵沉淀和分子筛层析, 获得低分子量的淀粉酶(11.2 kD)。其最适温度为60℃, 最适pH值为6.0, Ca2+能够提高野生酶的最适反应温度。Lomthong等[35]利用发酵条件优化方法, 研究了Laceyella sacchari LP175的分解酶对木薯生淀粉的降解能力; 且生物乙醇的产量高达90.9 g/L(理论产量的88%)。来源于温泉的Laceyella sacchari TSI-2α-淀粉酶的最适反应温度为70℃, 在37-100℃都具有酶活性, 100℃处理1 h还保留70%以上的活性。其最适反应pH值为7.0, 在pH 4.0-9.0内有酶活性; 在pH 4.0条件下处理3 h, 还保留60%以上的相对酶活力; 在pH9.0条件下处理6 h, 还保留70%以上的相对酶活力; 固定化酶提高了在有机溶剂中的稳定性; 表面活性剂还可提高相对酶活力; 能够高效去除棉布上的淀粉污渍[19, 38]。嗜热微生物Thermomyces dupontii> L18的TdAmy A的最适反应温度和pH值分别为50℃和6.5[51]。
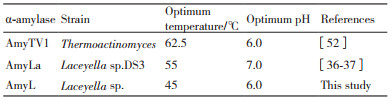
Laceyella sp.DS3的α-淀粉酶的野生酶的最适温度为50℃, 在E.coli中表达的重组AmyLa最适温度为55℃, 最适pH为7.0;游离酶在pH3-5时的相对酶活力小于20%, 在pH 6-7时的相对酶活力在80%以上; 而固定化酶在pH3-8时相对酶活力保持在80%以上[36-37]。本研究的AmyL与α-淀粉酶AmyLa以及来源于Thermoactinomyces的α-淀粉酶AmyTV1[52]的酶学性质存在较大差异(表 2)。但通过氨基酸序列比对发现三者的序列一致性极高, 只存在4个位点的差异, 分别是261、342、361、420位氨基酸。
为了从分子层面探究其酶学性质产生差异的原因, 通过Discovery Studio 2018软件构建了AmyL、AmyLa、AmyTV1的三维结构, 发现三者的整体三维结构基本保持一致, 其中261、361位点位于结构域A的α-螺旋上、342位点位于结构域A的loop结构上、420位点位于结构域C的反平行β-折叠上(图 8)。以AmyL作为参照, 利用Deepview软件分析AmyLa、AmyTV1在上述4个氨基酸位点的氢键数目变化。结果显示, 当361位氨基酸由组氨酸(AmyL)变为酪氨酸(AmyLa、AmyTV1)时, Tyr361与Thr365之间会形成一个新的氢键, 该位点的氢键由4个变为5个。在蛋白质内部形成一个氢键所获得平均能量约为0.6 kcal/mol[53]。Suvd等[54]通过比较来源于嗜中温微生物Bacillus licheniformis的α-淀粉酶(Bacillus licheniformisα-amylase, BLA)以及来源于嗜热微生物Bacillus stearothermophilus的α-淀粉酶(Bacillus stearothermophilusα-amylase, BSTA)的结构发现, BSTA较BLA在三维结构上增加了9个氢键, 使得BSTA获得了更好的热稳定性。因此, 氢键数量是重组酶AmyL的最适反应温度低于AmyLa、AmyTV1的最适反应温度的因素之一。此外, α-淀粉酶AmyL具有典型的(α/β)8桶状结构(TIM桶), 该结构是α-淀粉酶具有催化活性的关键因素[55]。α-淀粉酶AmyL的361位点位于TIM桶的第8个α-螺旋C端上, 氢键数量的增加使其在折叠成螺旋结构时易产生较大的构象张力[56], 进而引起熵变的增加使得AmyL的最适温度较AmyLa、AmyTV1有所下降。因此, 361位点对AmyL的最适反应温度及最适反应pH具有重要影响。
4 结论本研究从安阳面粉厂附近土壤中筛选到一株具有淀粉酶活性的菌株, 经过分子生物学鉴定, 确定为Laceyella sp.。将其α-淀粉酶基因克隆, 并在E.coli BL21(DE3)中成功表达。α-淀粉酶基因AmyL全长1 467 bp, 编码488个氨基酸和一个终止密码子, 在Leu35和Ser36之间存在信号肽酶切割位点。重组酶AmyL的最适反应温度为45℃, 最适反应pH值为6.0, 在温度为40-50℃、pH值为6.0-10.0的范围内具有较稳定的酶活力, 其Km和Vmax分别为0.95 mg/mL和343.12 μmol/(min·mg), 高浓度的Na+、K+、Ca2+对重组酶AmyL酶活力有保护作用。
| [1] |
赵国华, 张蕴玉, 雷琳, 等. 淀粉对微生物的结合作用及应用研究进展[J]. 中国食品学报, 2019, 3: 1-12. Zhao GH, Zhang YY, Lei L, et al. Research advances in the binding mechanism of starch to microorganisms and its applications[J]. Journal of Chinese Institute of Food Science and Technology, 2019, 3: 1-12. |
| [2] |
Sajilata MG, Singhal RS, Kulkarni PR. Resistant starch-a review[J]. Comprehensive Reviews in Food Science and Food Safety, 2006, 5(1): 1-17. DOI:10.1111/j.1541-4337.2006.tb00076.x |
| [3] |
Gupta R, Gigras P, Mohapatra H, et al. Microbial α-amylases: a biotechnological perspective[J]. Process Biochemistry, 2003, 38(11): 1599-1616. DOI:10.1016/S0032-9592(03)00053-0 |
| [4] |
中华人民共和国农业部. 中国农业统计资料[M]. 北京: 中国农业出版社, 2018.
|
| [5] |
左丽君, 张爱忠, 姜宁. 直链支链淀粉比对动物肠道内消化、肠道菌群和肠道组织形态影响的研究进展[J]. 饲料博览, 2018(1): 1-3. Zuo LJ, Zhang AZ, Jiang N. Research progress on the effect of the proportion of amylose and amylopectin on starch digestion, intestinal microflora and intestinal tissue morphology in the intestines of animals[J]. Feed Review, 2018(1): 1-3. DOI:10.3969/j.issn.1001-0084.2018.01.001 |
| [6] |
杨力权, 杨国保, 陈贵元, 等. 一株高温酸性淀粉酶产生菌的分离、筛选、鉴定及其酶学性质研究[J]. 云南大学学报:自然科学版, 2019, 41(5): 1047-1054. Yang LQ, Yang BG, Chen GY, et al. Isolation, screening and identification of a thermostable acidic amylase-producing strain and its enzymatic properties[J]. Journal of Yunnan University: Natural Sciences Edition, 2019, 41(5): 1047-1054. |
| [7] |
王培力.高温α-淀粉酶耐热嗜酸的分子改良及其高效表达的研究[D].北京: 中国农业科学院, 2016. Wang PL. Improvement of the thermal and acidic stability and efficient expression of the alpha amylase BLA[D]. Beijing: Chinese Academy of Agriculture Science, 2016. |
| [8] |
Shah IJ, Gami PN, Shukla RM, et al. Optimization for α-amylase production by Aspergillus oryzae using submerge fermentation technology[J]. Basic Research Journal Microbiology, 2014, 1: 1-10. |
| [9] |
Wang X, Kan G, Shi C, et al. Purification and characterization of a novel wild-type α-amylase from Antarctic sea ice bacterium Pseudoalteromonas sp. M175[J]. Protein Expression and Purification, 2019, 164: 105444. DOI:10.1016/j.pep.2019.06.004 |
| [10] |
Gopinath SCB, Aubu P, Arshad MKM, et al. Biotechnological processes in microbial amylase production[J]. BioMed Research International, 2017, 1272193. |
| [11] |
Struyf N, Verspreet J, Verstrepen KJ, et al. Investigating the impact of α-amylase, α-glucosidase and glucoamylase action on yeast-mediated bread dough fermentation and bread sugar levels[J]. Journal of Cereal Science, 2017, 75: 35-44. DOI:10.1016/j.jcs.2017.03.013 |
| [12] |
Amel EO, Gagaoua M, Bourekoua H, et al. Improving bread quality with the application of a newly purified thermostable α-amylase from Rhizopus Oryzae FSIS4[J]. Foods, 2017, 6(1): 1. DOI:10.3390/foods6010001 |
| [13] |
朱蓓蓓, 周杰, 朱娣, 等. 超声波辅助退浆节能强化技术[J]. 印染, 2016, 42(21): 19-22. Zhu BB, Zhou J, Zhu D, et al. Ultrasonic assisted desizing technology for energy saving[J]. Dyeing and Finishing, 2016, 42(21): 19-22. |
| [14] |
Morais RR, Pascoat AM, Pereira-Júnior MA, et al. Bioethanol production from Solanum lycocarpum starch: A sustainable non-food energy source for biofuels[J]. Renewable Energy, 2019, 140. |
| [15] |
李文钊, 臧传刚, 李义, 等. α-淀粉酶的研究与应用进展[J]. 当代化工, 2017, 46(11): 2292-2299. Li WZ, Zang CG, Li Y, et al. Research and application progress of α-Amylase[J]. Contemporary Chemical Industry, 2017, 46(11): 2292-2299. DOI:10.3969/j.issn.1671-0460.2017.11.031 |
| [16] |
张余慧, 程晓芳, 袁丹丹, 等. 浅谈淀粉酶在动物生产中的应用研究进展[J]. 广东饲料, 2018, 27(3): 33-35. Zhang YH, Cheng XF, Yuan DD, et al. Application of amylase in animal towel production[J]. Guangdong Feed, 2018, 27(3): 33-35. DOI:10.3969/j.issn.1005-8613.2018.03.011 |
| [17] |
Sindhu R, Binod P, Madhavan A, et al. Molecular improvements in microbial α-amylases for enhanced stability and catalytic efficiency[J]. Bioresource Technology, 2017, 245: 1740-1748. DOI:10.1016/j.biortech.2017.04.098 |
| [18] |
Dey TB, Kumar A, Banerjee R, et al. Improvement of microbial α-amylase stability: strategic approaches[J]. Process Biochemistry, 2016, 51(10): 1380-1390. DOI:10.1016/j.procbio.2016.06.021 |
| [19] |
Shukla RJ, Singh SP. Characteristics and thermodynamics of α-amylase from thermophilic actinobacterium, Laceyella sacchari TSI-2[J]. Process Biochemistry, 2015, 50(12): 2128-2136. DOI:10.1016/j.procbio.2015.10.013 |
| [20] |
Jiang Z. The systematics of family thermoactinomycetaceae and the multi-omics research for its hyphae differentiation[J]. Kunming: Yunnan University, 2018. |
| [21] |
Yoon JH, Kim IG, Shin YK, et al. Proposal of the genus Thermoactinomyces sesu strict and three new genera, Laceyella, Thermoflavimicrobium and Seinonella, on the basis of phenotypic, phylogenetic and chemotaxonomic analyses[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55: 395-400. DOI:10.1099/ijs.0.63203-0 |
| [22] |
Li D, Huang W, Wang CX, et al. The complete genome sequence of the thermophilic bacterium Laceyella sacchari FBKL4. 010 reveals the basis for tetramethylpyrazine biosynthesis in Moutai-flavor Daqu[J]. Microbiologyopen, 2019, e922. |
| [23] |
李豆南, 黄魏, 王晓丹, 等. 酱香型大曲中高温放线菌的筛选及风味成分分析[J]. 食品科学, 2018, 39(6): 171-176. Li DN, Huang W, Wang XD, et al. Identification and flavor profile of a thermoactinomycetaceae strain separated from moutai-flavor Daqu[J]. Food Science, 2018, 39(6): 171-176. |
| [24] |
Ming H, Ji WL, Li S, et al. Laceyella thermophila sp. nov., a thermophilic bacterium isolated from a hot spring[J]. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(8): 2953-2958. DOI:10.1099/ijsem.0.002057 |
| [25] |
Zhu WJ, Li Y, Jia HH, et al. Expression, purification and characterization of a thermostable leucine dehydrogenase from the halophilic thermophile Laceyella sacchari[J]. Biotechnology Letters, 2016, 38(5): 855-861. DOI:10.1007/s10529-016-2053-z |
| [26] |
Lomthong T, Hanphakphoom S, Kongsaeree P, et al. Enhancement of poly(L-lactide)-degrading enzyme production by Laceyella sacchari LP175 using agricultural crops as substrates and its degradation of poly(L-lactide)polymer[J]. Polymer Degradation and Stability, 2017, 143: 64-73. DOI:10.1016/j.polymdegradstab.2017.06.017 |
| [27] |
施庆珊, 梁文涛, 疏秀林, 等. 一株高温放线菌及其在造纸污泥堆肥过程中的应用[J]. 农业环境科学学报, 2008(1): 368-371. Shi QS, Liang WT, Shu XL, et al. A Thermophylic actinomycete strain and its application on composting of pulp and paper mill sludges[J]. Journal of Agro-Environment Science, 2008(1): 368-371. DOI:10.3321/j.issn:1672-2043.2008.01.068 |
| [28] |
朱国东, 陈波, 张兰兰, 等. 高温放线菌莱斯氏属RHA1菌株产低分子量α-淀粉酶的初步研究[J]. 生物技术通报, 2011(10): 199-205. Zhu GD, Chen B, Zhang LL, et al. Purification and characterization of a novel low molecular weight α-amylase from thermophilic actinomycete strain Laceyella sp.RHA1[J]. Biotechnology Bulletin, 2011(10): 199-205. |
| [29] |
孟庆龙.热稳定5-氨基乙酰丙酸合成酶的筛选、表达、酶学性质研究及其应用[D].天津: 天津大学, 2016. Meng QL. Screening, expression, characterization and application of thermostable 5-aminolevulinlic acid synthase[D]. Tianjin: Tianjin University, 2016. |
| [30] |
Xiao Y, Zeng GM, Yang ZH, et al. Changes in the acrinomycetal communities during continuous thermophilic composing as revealed by denaturing gradient gel electrophoresis and quantitative PCR[J]. Bioresource Technology, 2011, 102(2): 1383-1388. DOI:10.1016/j.biortech.2010.09.034 |
| [31] |
Singh V, Pandey VC, Agrawal S. Potential of Laceyella sacchari strain B42 crude xylanase in biobleaching of kraft pulp[J]. African Journal of Biotechnology, 2013, 12: 570-579. |
| [32] |
Dolashki A, Voelter W, Gushterova A, et al. Isolation and characterization of novel tyrosinase from Laceyella sacchari[J]. Protein Pept Lett, 2012, 19(5): 538-543. DOI:10.2174/092986612800191035 |
| [33] |
Jørgensen CM, Madsen SM, Vrang A, et al. Recombinant expression of Laceyella sacchari thermitase in Lactococcus lactis[J]. Protein Expression and Purification, 2013, 92(2): 148-155. DOI:10.1016/j.pep.2013.09.009 |
| [34] |
Kobayashi T, Uchimura K, Kubota T, et al. Biochemical and genetic characterization of beta-1, 3 glucanase from a deep subseafloor Laceyella putida[J]. Appl Microbiol Biotechnol, 2016, 100(1): 203-214. DOI:10.1007/s00253-015-6983-5 |
| [35] |
Lomthong T, Lertwattanasakul N, Kitpreechavanich V. Production of raw starch degrading enzyme by the thermophilic filamentous bacterium Laceyella sacchari LP175 and its application for ethanol production from dried cassava chips[J]. Starch-Starke, 2016, 68. |
| [36] |
Ahmed KA, Mohamed I, Noha FO. Heterologous expression, purification, immobilization and characterization of recombinant α-amylase AmyLa from Laceyella sp. DS3[J]. International Journal of Biological Macromolecules, 2019, 132: 1274-1281. DOI:10.1016/j.ijbiomac.2019.04.010 |
| [37] |
El-Sayed AKA, Abou-Dobara MI, El-Fallal AA, et al. Gene sequence, modelling and enzymatic characterization of α-amylase AmyLa from the Thermophile Laceyella sp. DS3[J]. Starch-St?rke, 2016, 69. DOI:10.1002/star.201600255 |
| [38] |
Shukla RJ, Singh SP. Structural and catalytic properties of immobilized α-amylase from Laceyella sacchari TSI-2[J]. International Journal of Biological Macromolecules, 2016, 85: 208-216. DOI:10.1016/j.ijbiomac.2015.12.079 |
| [39] |
Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7. 0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [40] |
Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4): 406-425. |
| [41] |
Tuzlakoglu MO, Akbulut N, Issever SO, et al. Ligase-independent cloning of amylase gene from a local Bacillus subtilis isolate and biochemical characterization of the purified enzyme[J]. New Biotechnology, 2013, 171(2): 263-278. |
| [42] |
Zakalskiy AE, Zakalska OM, Rzhepetskyy YA, et al. Overexpression of(His)6-tagged human arginase I in Saccharomyces cerevisiae and enzyme purification using metal affinity chromatography[J]. Protein Expression and Purification, 2012, 81(1): 63-68. DOI:10.1016/j.pep.2011.09.001 |
| [43] |
Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar[J]. Analytical Biochemistry, 1959, 31(3): 426-428. |
| [44] |
Lineweaver H, Burk D. The determination of enzyme dissociation constants[J]. Journal of America Chemistry Society, 1934, 56(3): 658-666. DOI:10.1021/ja01318a036 |
| [45] |
Takenaka S, Miyatake A, Tanaka K, et al. Characterization of the native form and the carboxy-terminally truncated halotolerant form of α-amylases from Bacillus subtilis strain FP-133[J]. Journal of Basic Microbiology, 2015, 55(6): 780-789. DOI:10.1002/jobm.201400813 |
| [46] |
Vrsalović PA, Findrik BZ, Durđa VR. Mathematical modeling of maize starch liquefaction catalyzed by α-amylases from Bacillus licheniformis: effect of calcium, pH and temperature[J]. Bioprocess and Biosystems Engineering, 2013, 36(1): 117-126. DOI:10.1007/s00449-012-0767-4 |
| [47] |
Priyadarshini S, Pradhan SK, Ray P. Production, characterization and application of thermostable, alkaline alpha-amylase(AA11)from Bacillus cereus strain SP-CH11 isolated from Chilika Lake[J]. International Journal of Biological Macromolecules, 2020, 145: 804-812. DOI:10.1016/j.ijbiomac.2019.11.149 |
| [48] |
Gutiérrez-García AK, Alvarez-Guzmán CL, Leon-Rodriguez Ade. Autodisplay of alpha amylase from Bacillus megaterium in E. coli for the bioconversion of starch into hydrogen, ethanol and succinic acid[J]. Enzyme and Microbial Technology, 2020, 134: 109477. DOI:10.1016/j.enzmictec.2019.109477 |
| [49] |
何江红, 王荣钰, 舒小芳, 等. 川西藏区牦牛酸醡肉中产蛋白酶和淀粉酶菌株的分离鉴定及其生理特性分析[J]. 中国调味品, 2018, 43(12): 60-65. He JH, Wang RY, Shu XF, et al. Isolation and identification of protease and amylase producing strains in Sour Yak Meat from western Sichuan Tibetan areas and analysis of their physiological characteristics[J]. China Condiment, 2018, 43(12): 60-65. DOI:10.3969/j.issn.1000-9973.2018.12.011 |
| [50] |
El-Sayed AK1, Abou Dobara MI, El-Fallal AA, et al. Purification, sequencing, and biochemical characterization of a novel calcium-independent α-amylase AmyTVE from Thermoactinomyces vulgaris[J]. Applied Biochemistry and Biotechnology, 2013, 170(3): 483-497. DOI:10.1007/s12010-013-0201-7 |
| [51] |
Wang YC, Zhao N, Ma JW, et al. High-level expression of a novel α-amylase from Thermomyces dupontiiin Pichia pastoris and its application in maltose syrup production[J]. International Journal of Biological Macromolecules, 2019, 127: 683-692. DOI:10.1016/j.ijbiomac.2019.01.162 |
| [52] |
Hofemeister B, Konig S, Hoang V, et al. The gene amyE(TV1)codes for a nonglucogenic α-amylase from Thermoactinomyces vulgaris 94-2A in Bacillus subtilis[J]. Applied and Environmental Microbiology, 1994, 60: 3381-3389. DOI:10.1128/AEM.60.9.3381-3389.1994 |
| [53] |
Han YQ. Increasing the thermostability of CRL1 by protein semi-rational design[J]. Changchun: Jilin University, 2011. |
| [54] |
Suvd D, Fujimoto Z, Takase K, et al. Crystal structure of Bacillus stearothermophilus alpha-amylase: possible factors determining the thermostability[J]. The Journal of Biochemistry, 2001, 129(3): 461-468. |
| [55] |
高艳云.分子改造提高酸性α-淀粉酶热稳定性[D].上海: 华东理工大学, 2016. Gao YY. Improving thermostability of acidic α-amylase through molecular modification[D]. Shanghai: East China University of Science and Technology, 2016. |
| [56] |
Mabrouk SB, Aghajari N, Ali MB, et al. Enhancement of the thermostability of the maltogenic amylase MAUS149by Gly312Ala and Lys436Arg substitutions[J]. Bioresource Technology, 2011, 102: 1740-1746. DOI:10.1016/j.biortech.2010.08.082 |