2. 中国农业大学食品科学与营养工程学院 北京食品营养与人类健康高精尖创新中心农业部农业转基因生物安全评价(食用)重点实验室,北京 100083
2. Beijing Advanced Innovation Center for Food Nutrition and Human Health, College of Food Science and Nutritional Engineering, China Agricultural University, Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety), Ministry of Agriculture, Beijing 100083
随着市场经济的迅猛发展,人们生活水平的日益提高,全民健康意识也逐步提高。为了保障人民衣食住行各方面的安全,进一步发展检测分析技术显得尤为重要。现代社会的快节奏在一定程度上促使检测技术的革新,建立精准快速的方法检测对人类健康和生态系统造成有害影响的污染物,已成为了各领域关注的热点[1]。适配体(Aptamer)是一类由几十个核苷酸组成单链短序列,与传统的抗体相比适配体对靶标物质除了具有更高的敏感性和选择性外,还存在能大规模生产、批次间变异小、货架期长等诸多优点。适配体经修饰可进一步增强其稳定性和靶向性,可作为抗体替代品用于检测分析领域[2]。
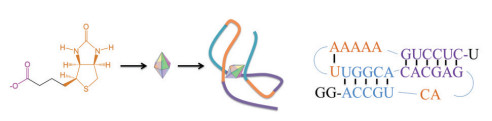
适配体主要有单链脱氧核糖核酸(ssDNA)和核糖核酸(RNA)两类[3]。适配体可通过链内碱基间的氢键相互作用折叠形成诸如茎环、凸环、口袋和假结等二级结构,而富G序列还可通过鸟嘌呤间的氢键相互作用,片层堆叠形成G-四链体和G-三链体结构[4],这些二级结构可集合形成独特的三级结构对目标分子进行特异性识别。与抗原抗体结合的方式类似,适配体可结合金属离子、化合物、蛋白质、病毒、生物细胞和细菌等多种靶标物质[5]。
小分子靶标物质的分子量不超过500 Da,包括兽药、农药、生物标志物、环境污染物、内源性代谢物及生物毒素等。与大分子物质不同,分子量小的目标物与寡核苷酸适配体的尺寸差异较大,使得小分子靶标的适配体筛选分离的难度更大[6]。小分子靶标物质的痕量检测在食品安全、医学诊断、环境监测等领域均具有重要的意义,尤其是检测重金属、毒素和抗生素等可能对人体造成有害影响的小分子具有实际应用价值。目前,小分子靶标适配体的筛选总体成功率并不高[5],故有必要根据小分子与适配体的相互作用规律,针对目标物设计合理有效的检测进程,建立快速准确检测分析小分子靶标的方法。因此,本文从靶标分子的结构性质出发,梳理了近年来各类小分子靶标与适配体的作用规律及作用方式,旨在为完善小分子分析检测体系提供有益参考。
1 小分子物质的分类及性质 1.1 单质单质的性质往往与其元素的性质相关,由金属键结合而成的固态金属晶体内部存在游离电子,而原子则采取密堆积形式以保证晶体的稳定。有研究表明金属较易失去电子而带正电荷,如镉[7]、锌[8]等。而非金属单质则往往带负电,如砷(As)与带负电的DNA之间存在一定程度的静电排斥作用,砷的适配体(Ars-3)对As(III)的识别过程存在争议[9]。
1.2 化合物化合物可分为无机和有机两大类。无机化合物大致可分为氧化物、盐、酸、碱等,往往通过电离得到的离子与其他物质相互作用,如氧化锌通过Zn2+与DNA相互作用形成复合结构[10]。根据有机化合物的碳骨架结构差异可分为链状化合物和环状化合物。有机化合物结构中含有-OH、-CONH2的部分往往具有亲水性,通常含有-NH2的结构常带正电,含基团-COOH的部分则带负电。这些结构基团共同影响着链状化合物的性质,如链状结构的L-瓜氨酸和L-精氨酸,其性质由带正电的氨基和带负电的羧基共同决定[11];而链状化合物乙醇胺的功能特性,也受其结构中羟基和氨基的影响[12]。
环状化合物可分为碳环化合物和杂环化合物,其性质往往受到带电基团及其所含环状结构的影响。根据结构中是否含有芳香环,碳环化合物可分为芳香族和脂环族,杂环化合物可包括芳杂环和脂杂环。与碳环化合物不同,杂环化合物往往包含由碳原子和其他原子(如氧、氮、硫等)形成环状结构。杂环化合物是迄今为止数目最庞大的一类有机化合物,这些杂环结构广泛存在于各种具有生物活性的天然产物、有机材料、农药和药品中[13]。常见的脂杂环小分子化合物有:麦芽糖、鸟嘌呤、茶碱和链霉素等。芳杂环类有多巴胺、可卡因、氯霉素和四环素等[11]。
2 小分子与适配体的作用规律适配体能在识别靶标的同时鉴别这些小分子间极其微小的结构差异,这种高特异性识别过程与适配体和靶标的相互作用形式有关,包括带电基团和芳香环结构等。例如,L-酪氨酸的RNA适配体(dopa2)能分辨靶标带电荷基团的差异,而对芳香环结构的识别没有特异性,故dopa2对L-芳香环结构与L-酪氨酸相近)有高亲和力,但对缺少羧基的多巴胺的亲和降低力约10倍[14]。当靶标分子既没有平面芳香环、也没有带电荷的功能基团时,可能会产生一种与常见内部凸环和发卡环结构不同的结构,如与生物素结合的适配体具有独特的假结折叠结构(图 1),这种新型识别方式可能是由靶标的自然属性所致[15]。
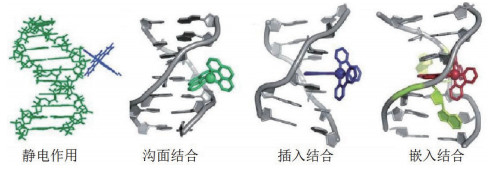
小分子往往通过疏水和静电相互作用、氢键和碱基堆积等作用力结合适配体[16]。金属类小分子靶标往往会“镶嵌”在适配体链上,而小分子化合物则会被“条网状”的适配体链段兜住。小分子与适配体的键合方式主要是非共价结合,包括静电结合、沟面结合、插入作用和嵌入作用[17]。如图 2所示,静电作用是指小分子物质中带正电的部分通过静电相互作用与核酸分子骨架中带负电的基团键合的过程。沟面结合是指分子与DNA双螺旋结构中小沟或大沟的碱基对边缘发生相互作用的过程,构型体积较小的化合物主要沿着小沟方向与核酸结合,氢键以及碱基的疏水性均能推动两者的键合。插入结合是指小分子的共轭芳香环平面插入核酸双链(RNA局部双链或DNA双螺旋)碱基对的过程,相邻碱基间的范德华力、疏水作用和π-π电子堆积均能稳定结合构象。嵌入结合是指小分子借助氢键和范德华力替代DNA小沟处的错配碱基并与相应的碱基产生π-π堆积作用的过程。总之,与静电结合与沟面结合相比,插入结合和嵌入结合的键合方式相对较强;嵌入结互作用更强。
靶标分子往往存在多个与之特异性结合的适配体,而适配体序列长短及碱基组成均决定了其间的结构差异。经SELEX技术筛选得到的适配体往往含有部分冗长序列,可结合定点突变技术,在保留适配体与靶标结合区域完整的同时截断多余冗长序列。有研究表明,适配体识别靶标的区域具有较为复杂的结构,如发夹、三通连接和G-四链体结构等[19]。对于某些由嘌呤和嘧啶碱基交替排列呈茎环状发夹结构的适配体而言,茎部序列在某种程度上能对环状结构的稳定起到一定的作用,有研究成功通过延长茎部序列长度稳定核酸构象[20]。三通连接结构的适配体则是通过碱基互补配对并折叠形成合适的二级结构,在三通连接处形成“空腔”利用疏水作用与靶标(如可卡因、胆酸等)结合,而非借助极性功能基团识别靶标[21]。G-四链体结构由4条富含鸟嘌呤的链段形成的支架和几个在靶标识别过程中起作用的环状基序组成,该结构与靶标间存在较强的疏水结合能力,故OTA、核仁素、氯化血红素和荧光素等靶标物质的适配体往往呈G-四链体结构[22-23]。
3 小分子与适配体相互作用的分析方法研究靶标小分子与核酸相互作用的方法主要包括两大类,一类是较为直观地分析微观结构变化,如透射电子显微镜、选区电子衍射、X-射线晶体衍射、扫描隧道显微镜、原子力显微镜等[24-25];另一类方法在相互作用的研究过程中更为普遍,主要是借助图谱、物质性质变化来间接分析体系的相互用。寡核苷酸适配体作为核酸的一部分,亦可借鉴这些方法分析其与小分子的相互作用。
3.1 借助图谱的分析常见的用于分析相互作用的图谱有紫外-可见光吸收光谱、荧光发射光谱、拉曼散射光谱、圆二色谱和核磁共振波谱。借助吸收光谱和发射光谱可以推断小分子靶标与核酸适配体间的相互作用模式以及结合强度。两者的相互作用会促使靶标紫外-可见光吸收光谱的波长和强度发生变化,且作用力往往与吸收光谱的红移程度和减色率呈正比[26],若靶标分子与核酸以相对较弱的静电结合方式键合,吸收光谱的特征峰则无明显变化。而一些有荧光特性的染料小分子通过沟面或插入结合适配体,在荧光发射光谱中会出现荧光增强的现象[27]。借助拉曼散射光谱则能得到分子结构转变(包括振动和转动)的相关信息,据此有研究发现高浓度的Cd2+会促使DNA双螺旋结构中的部分碱基堆叠坍塌、氢键断裂[28]。借助圆二色谱并结合透析实验可推断小分子物质与核酸的键合方式,通过对比复合结构对偏振光的吸收程度可判断小分子对DNA螺旋轴的取向及其立体选择性。借助核磁共振波谱可分析小分子物质与核酸的结合区域,由于不同位置的氢原子在共振状态中所吸收的电磁波频率不同,故小分子的氢化学位移与核酸碱基中的质子化学位移值重叠处即是两者的亲和位点[26]。
3.2 借助性质的改变当晶体结构等参数缺乏、紫外吸收光谱和荧光光谱的变化均不明显时,可借助凝胶电泳、流体力学、解旋温度差异以及分子的电活性等,分析推断小分子化合物与核酸的相互作用模式与结合强度。琼脂糖凝胶电泳的条带位置反映了核酸片段的大小差异,借此能初步判断小分子与其是否存在一定的相互作用:若两者间仅是静电结合,那么电泳迁移率的变化不大;而插入结合的模式则会使得复合结构的电泳迁移率减慢。流体力学中复合体系黏度的变化反映了核酸片段长度的差异:若小分子以插入结合的方式与核酸作用,整个片段长度增加体系黏度增大[29];若是不完全插入结合小分子的部分嵌入会使原有DNA双螺旋结构更加扭结,复合体系黏度减小;而非插入结合(如静电结合、沟面结合)的模式则对体系的黏度的影响不大[30]。还可借助DNA双链的解旋温度判断小分子与DNA的相互作用模式:小分子的插入结合模式会在一定程度上抑制DNA双链的解旋,从而导致解旋温度升高;分子的嵌入结合模式会引起碱基翻转错位,致使原有核酸结构的稳定性降低,解旋温度降低。伏安特性的变化也能反映核酸的构象,且电势差与半波电位的差异则能体现小分子物质与核酸的结合方式[31]。
4 小分子靶标与适配体相互作用的类型理论上,任一小分子靶标物质均能从RNA和DNA文库中筛选分离得到适配体。RNA具有的模块化性质使其能够形成独立的结构域,并自动折叠呈“口袋”,同时保持对小分子靶标的结合特性。由于DNA缺少RNA所具有的2'-羟基,筛选出的RNA和DNA适配体往往具有明显不同的序列和折叠结构[11]。但也存在与靶标分子结合的DNA和RNA适配体具有相同序列(碱基T在RNA序列中替换为U)的报道:这两条适配体均形成G-四链体结构,并以较低的亲和力与核黄素结合[32]。
4.1 金属类小分子靶标 4.1.1 与核酸嘧啶基团作用带正电的金属小分子往往与其适配体的嘧啶基团中带负电的部分结合。不同金属的适配体往往具有不同的结构,如Cd(Ⅱ)、Zn2+和Pd2+的适配体呈茎环状结构,靶标分子通过配位键结合适配体环状部分,而茎状部分碱基对则利用氢键起到一定的稳定作用[7, 33]。金属氧化物ZnO与适配体Poly-d(T)30的键合过程是:Zn2+利用氢键和静电相互作用规则性的结合于DNA分子胸腺嘧啶碱基间,依次堆积逐渐形成晶体状ZnO纳米壳层[25]。
4.1.2 与核酸嘌呤基团作用在靶标与其适配体的结合过程常会受到体系中金属离子的影响,如Mg2+的存在可增强赭曲霉毒素A与其适配体间的相互作用,由于赭曲霉毒素A的适配体富含鸟嘌呤,K+也可通过与鸟嘌呤O6的静电作用,进一步诱导其G-四链体结构的形成并起到一定的稳定作用[34]。而Ag+则能螯合鸟嘌呤碱基中的N7和C6O基团,破坏原有的G-四链体结构[35]。
4.2 化合物类小分子靶标 4.2.1 由靶标的带电基团主导带电基团在一定程度上会影响适配体与靶标间的相互作用,氨基酸类小分子主要借助氨基和羧基与适配体结合,羟基起到一定的辅助作用[11]。RNA适配体通常围绕氨基酸折叠,通过氢键识别靶标分子[36],如谷氨酸尿素部分带负电荷的氧原子与尿嘧啶的N3之间通过氢键作用;而精氨酸带正电的氨基,则作为氢键的供体与胞嘧啶作用[37]。
糖类小分子与适配体的相互作用主要依赖于氢键和疏水作用,其中氢键由糖苷类化合物(如麦芽糖、阿拉伯糖等)吡喃环中的氧与羟基形成,由于带电基团和芳香环结构的缺乏,以羟基结构为主的糖类小分子与其适配体的亲和力并不太强[11]。Masud等[38]研究表明在适配体胸腺嘧啶核苷C5处修饰质子化的氨基阳离子,可增强其与唾液酸化乳糖(含有带负电的羧基)的亲和力,并以此推测该适配体为三通连接结构,其茎部区域含有经修饰的胸腺嘧啶核苷。不同的适配体的特异性结合区域存在差异,Yang等[39]测试了DNA适配体对寡糖基序的选择性识别能力,发现其能够辨别大分子表面糖蛋白修饰的微小差异;而Srisawat等[40]筛选出葡聚糖G-100的RNA适配体,推测其最小识别位点是由4个以上葡萄糖残基通过α-1,6-糖苷键相连而成。
还有部分天然产物也同样借助氢键与疏水作用结合适配体。Zimmermann等[41]利用核磁共振波谱推断了茶碱-RNA适配体的复合结构,结果表明适配体利用复杂的氢键网络和堆叠相互作用“包裹”茶碱,两者的特异性识别取决于多个接触位点,咖啡因N7位置比茶碱多一个甲基,该甲基所致的空间排斥在一定程度上会削弱氢键作用,这也是茶碱适配体对咖啡因亲和性极低的原因。Stojanovic等[42]筛选了可卡因的DNA适配体并对其二级结构进行推测,结果表明在其三通连接处具有亲脂性。Kato等[43]则发现胆酸的单链DNA适配体也表现为三通连接结构,其形成的“空腔”可经疏水作用结合靶标。有研究表明甾族化合物(又称类固醇,如胆酸)的适配体不是通过极性功能基团特异性识别靶标,而是通过折叠成合适的二级结构来识别大小形状不同的靶标分子[21]。
4.2.2 由靶标的环状结构主导适配体主要通过腺苷和核糖结构识别核酸类小分子,其特异性受空间作用的影响[11]。例如,最初筛得ATP的DNA适配体主要识别靶标的碱基和糖,5'-磷酸基团不参与结合;随着筛选思路地转换,也得到了强烈作用于ATP的β-和γ-磷酸基团的新型RNA适配体[44]。部分辅助因子类小分子与适配体的结合也与环状结构有关,如S-腺苷蛋氨酸、S-腺苷同型半胱氨酸、辅酶A的腺苷部分与其RNA适配体结合[11],黄素嘌呤二核苷酸(Flavin adenine dinucleotide,FAD)则是通过其结构中的异咯嗪母核与其适配体作用[45]。
染料类小分子噻唑橙可与G-四链体结构的DNA适配体形成复合物并产生强烈荧光,而溴化乙锭的三环平面基团亦可插入结合于DNA互补配对结构的堆积碱基中产生荧光。RNA适配体往往以“结合口袋”的形式识别染料小分子,识别过程主要受到碱基堆积和形态互补的推动。孔雀石绿的适配体借助A30碱基卡住MG的外苯环以形成稳定的复合结构,其结合位点呈不对称内部凸环,两侧经互补配对形成双链RNA结构[46-47]。荧光染料DFHBI结合于RNA适配体的G4-链体结构中,未成对的鸟嘌呤残基将DFHBI卡住,使其两个芳香环在同一个平面形成荧光构象[48]。
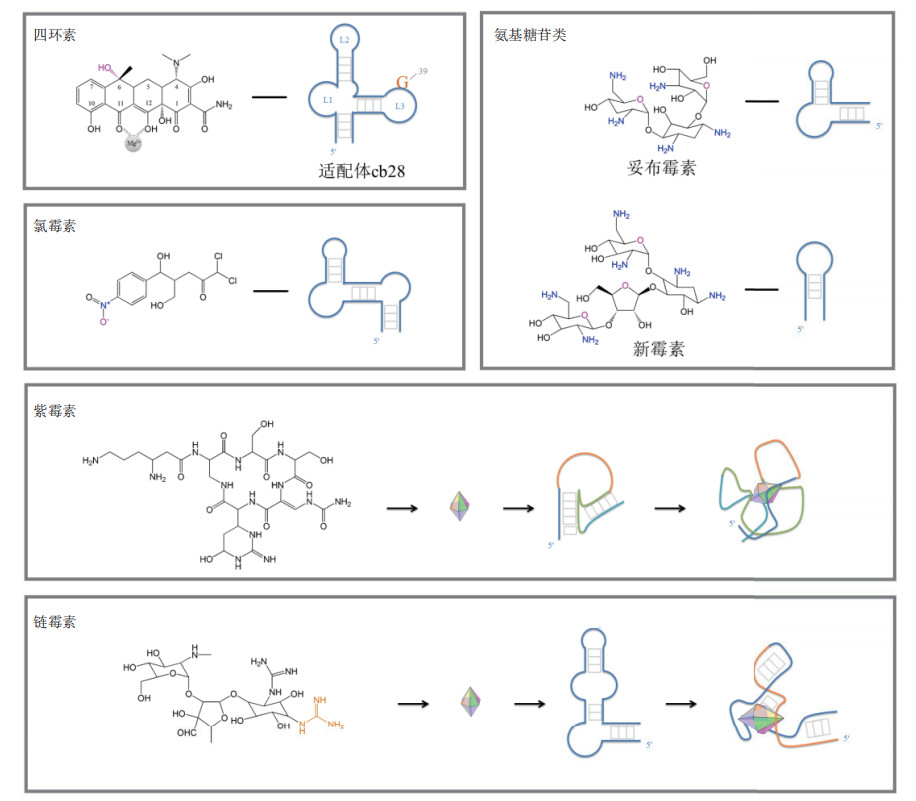
4.2.3 多种相互作用共同决定如图 3所示,还有一些小分子化合物与适配体的相互作用由带电基团和环状结构共同介导,包括四环素、链霉素、氯霉素及紫霉素等抗生素类靶标。由4个环组成的四环素平面结构,一侧具有的亲水功能基团能形成离子相互作用;另一侧则作为疏水面存在非极性和堆积作用。Berens等[49]通过铅剪切和硫酸二甲酯(Dimethylsulfate,DMS)修饰实验分析了RNA适配体与四环素结合后的二级结构变化,并构建四环素最小适配体cb28的最简二级结构图,推断出cb28结构中L3的G39可能是两者结合位点,而四环素的6位羟基是两者相互识别的关键。氨基糖苷类抗生素的基本构件是2-脱氧链霉胺,其多样性取决于糖环上的羟基、甲基和羟甲基,其适配体呈茎环状结构,茎部常存在小的内环或单个核苷酸的凸起,主要带负电荷的环状结构则作为结合“口袋”结合带正电荷的氨基糖苷类化合物[11]。氯霉素由芳香族氨基酸(苯丙氨酸等)的生物合成途径中衍生而来的,其RNA适配体由3个螺旋组成,中间被两个不对称的凸环(一侧是单个的腺苷酸,另一侧含4-6个腺嘌呤)隔断[50]。链霉素是含有胍基的氨基环醇糖苷类抗生素,Tereshko等[51]通过对链霉素与适配体的复合物进行X射线晶体结构的高分辨解析,发现适配体呈L型结构,L型的每个臂上的两个环形成一个口袋将链霉素夹在其中,并通过碱基边缘与其接触。紫霉素是由6个氨基酸组成的碱性肽,其核心结构是一个十六元环,Wallis等[52]发现筛选得到的紫霉素RNA适配体大部分含有由14个核苷酸组成的高度保守区,茎环状的适配体通过“假结”(环部序列与其邻近序列的碱基配对形成类似“打结”形状)识别紫霉素。
5 应用所谓“磨刀不误砍柴工”,小分子靶标与适配体相互作用的研究必不可少。这一阶段的准确探索奠定了后续改性修饰适配体并将其应用于实际的基础,同时又印证了前期适配体筛选过程的准确性和有效性。
5.1 适配体的筛选了解小分子靶标与适配体的相互作用形式及其结合位点,能在一定程度上使得核酸文库序列设计更有效,同时还可根据已知结合位点推测靶标结构类似物的适配体序列,而进一步比对不同基团间的相互作用,明确适配体的特异性,避免该适配体在后续应用中出现的交叉反应。金属类小分子在适配体筛选过程中往往能起到一定的辅助作用,有研究采用NaCl浓度梯度富集精氨酸适配体,而筛选L-异亮氨酸适配体的过程也需要Zn2+参与,这可能是因为异亮氨酸会与Zn2+结合或者适配体含有“Zn2+的RNA结构” [11]。缓冲液体系中的金属离子种类不同,筛得适配体也不同,在筛选维生素B12的适配体时选用含有Li+和Mg2+的缓冲液,所得的适配体依赖于高浓度的Li+,但结合时不需要Mg2+存在,当用Na+替代Li+用于筛选时,所得的适配体不再依赖Li+,而是强烈依赖Mg2+[11]。
5.2 生物传感器的搭建了解小分子靶标与适配体的相互作用形式及作用位点,有利于搭建精准高效的生物传感器,实现对物质进行定性定量检测,而借助生物传感器的信号变化亦能推测核酸自身的碱基错配及靶标结合位点。有学者基于适配体结构转换功能搭建了3类荧光生物传感器,包括分子信标、分裂适配体传感器、应变位移传感器[53]。这些传感器均涉及到明确靶标与适配体的结合位点,使得两者在结合态和游离态之间处于最佳热力学平衡。而确定最佳位点及作用碱基数,往往需要通过大量的序列设计和重复试验。而复合体系中的金属离子也会影响适配体与小分子靶标的结合,如Mg2+与四环素的酮和烯醇基团配位是其与cb28适配体结合的关键[49];而在链霉素适配体也需在Mg2+存在下才能结合链霉素[51]。故通过一定的技术手段准确了解小分子与适配体之间的作用方式就显得至关重要,同时也为上述生物传感器的顺利搭建起到了一定的积极意义。
6 总结与展望小分子与核酸的键合方式主要有共价结合和非共价结合,与DNA双螺旋结构不同,单链适配体与小分子的相互作用几乎是非共价结合,且两者间“沟面结合”较少。适配体倾向于经碱基互补配对折叠形成茎环状结构,并缠绕靶标小分子,使其以“插入结合”和“嵌入结合”的形式嵌入适配体结构中。小分子化合物往往通过其带电的基团与适配体形成氢键,且两者间还存在疏水和静电相互作用。当小分子结构中含有环状结构时,除了带电基团的影响,由于环状结构自身的空间位阻作用,使得体系中往往存在碱基堆积效应。带正电的金属离子通常与适配体嘧啶基团上带负电的结构作用,金属离子在一定程度上能调控适配体的构象控制G-四链体结构的形成与坍塌,亦能调控靶标与适配体的结合能力,如Mg2+ 能控制四环素和链霉素结合其RNA适配体。总之,小分子物质的自身结构特性是影响适配体与其结合的关键,链状适配体通常围绕靶标小分子缠绕折叠,结合位点附近碱基的带电基团使其稳定,非结合位点的序列则往往利用碱基互补配对呈“茎秆”状稳定整个复合结构。
结构决定性质,物质的性质又影响其功能特性,准确把握小分子物质与适配体的键合方式与结合位点,探究小分子物质与适配体的复合结构是适配体应用与发展的基础。但影响适配体折叠的因素有很多,包括pH值、离子强度、阳离子种类等。目前缺少非常灵敏的监测核酸适配体结构变化的手段,适配体结构的柔性使得其与靶标间较难形成晶体结构,使得X-ray晶体衍射的检测适配体结构信息变得困难,而基础研究数据的缺乏,往往使得从结构和功能的角度分析问题的过程变得困难[54]。近年来,研究者们主要利用小分子物质与适配体性质的改变,结合各类图谱分析,推断两者间可能存在的结合方式。分类整合已有小分子物质与适配体的复合结构信息,针对可能存在的性质变化拟定标准化测定分析过程是未来的发展趋势。随着生物技术的飞速发展,新型检测模式的深入研究,结合各项新技术建立新型适配体结构功能分析体系指日可待。
| [1] |
Dorst BV, Mehta J, Bekaert K, et al. Recent advances in recognition elements of food and environmental biosensors :A review[J]. Biosensors and Bioelectronics, 2010, 26(4): 1178-1194. DOI:10.1016/j.bios.2010.07.033 |
| [2] |
Sekhon SS, Park GY, Park DY, et al. Aptasensors for pesticide detection[J]. Toxicology and Environmental Health Sciences, 2018, 10(5): 229-236. DOI:10.1007/s13530-018-0370-4 |
| [3] |
Song KM, Lee S, Ban C. Aptamers and their biological applications[J]. Sensors, 2012, 12(12): 612-631. |
| [4] |
Limongelli V, De TS, Cerofolini L, et al. The G-triplex DNA[J]. Angewandte Chemie International Edition, 2013, 52(8): 2269-2273. DOI:10.1002/anie.201206522 |
| [5] |
Wang T, Chen C, Larcher LM, et al. Three decades of nucleic acid aptamer technologies :lessons learned, progress and opportunities on aptamer development[J]. Biotechnology Advances, 2019, 37(8): 28-50. |
| [6] |
Mcconnell EM. In vitro selection and characterization of DNA aptamers to a small molecule target[J]. Current Protocols in Chemical Biology, 2017, 9(4): 233. |
| [7] |
Wu Y, Zhan S, Wang L, et al. Selection of a DNA aptamer for cadmium detection based on cationic polymer mediated aggregation of gold nanoparticles[J]. The Analyst, 2014, 139(6): 1550-1561. DOI:10.1039/C3AN02117C |
| [8] |
Rajendran M, Ellington AD. Selection of fluorescent aptamer beacons that light up in the presence of zinc[J]. Analytical and Bioanalytical Chemistry, 2008, 390(4): 1067-1075. |
| [9] |
Kim M, Um HJ, Bang S, et al. Arsenic removal from vietnamese groundwater using the Arsenic-Binding DNA Aptamer[J]. Environmental Science & Technology, 2009, 43(24): 9335-9340. |
| [10] |
Bawazer LA, Newman AM, Gu Q, et al. Efficient selection of biomineralizing DNA aptamers using deep sequencing and population clustering[J]. ACS Nano, 2013, 8(1): 387-395. |
| [11] |
Klussmann S. The aptamer handbook :functional oligonucleotides and their applications[J]. Wiley-VCH, 2006. |
| [12] |
Reinemann C, Stoltenburg R, Strehlitz B. Investigations on the specificity of DNA aptamers binding to ethanolamine[J]. Analytical Chemistry, 2009, 81(10): 3973-3978. DOI:10.1021/ac900305y |
| [13] |
Jiang Y, Xu K, Zeng C. Use of electrochemistry in the synthesis of heterocyclic structures[J]. Chemical Reviews, 2018, 118(9): 4485-4540. DOI:10.1021/acs.chemrev.7b00271 |
| [14] |
Mannironi C, Di Nardo A, Fruscoloni P, et al. In vitro selection of dopamine RNA ligands[J]. Biochemistry, 1997, 36(32): 9726-9734. DOI:10.1021/bi9700633 |
| [15] |
Nix J, Sussman D, Wilson C. The 1.3 crystal structure of a biotin-binding pseudoknot and the basis for RNA molecular recognition[J]. Journal of Molecular Biology, 2000, 296(5): 1235-1244. DOI:10.1006/jmbi.2000.3539 |
| [16] |
Zhou J, Rossi J. Aptamers as targeted therapeutics :current potential and challenges[J]. Nature Reviews Drug Discovery, 2017, 16(3): 181. DOI:10.1038/nrd.2016.199 |
| [17] |
Pheeney CG, Arnold AR, Grodick MA, et al. Multiplexed electrochemistry of DNA-Bound metalloproteins[J]. Journal of the American Chemical Society, 2013, 135(32): 11869-11878. DOI:10.1021/ja4041779 |
| [18] |
何晓俊.化学小分子与RNA的键合作用及其对RNA稳定性调控研究[D].湘潭: 湘潭大学, 2015. He XJ. The RNA-binding and RNA stability studies of regulation by chemical small molecules[D]. Xiangtan : Xiangtan University, 2015. |
| [19] |
Xiao X, Zhu L, He W, et al. Functional nucleic acids tailoring and its application[J]. TrAC Trends in Analytical Chemistry, 2019, 118: 138-157. DOI:10.1016/j.trac.2019.05.027 |
| [20] |
Adamala K, Engelhart AE, Szostak JW. Generation of functional RNAs from inactive oligonucleotide complexes by non-enzymatic primer extension[J]. Journal of the American Chemical Society, 2015, 137(1): 483-489. DOI:10.1021/ja511564d |
| [21] |
Kato T, Yano K, Ikebukuro K, et al. Interaction of three-way DNA junctions with steroids[J]. Nucleic Acids Research, 2000, 28(9): 1963. DOI:10.1093/nar/28.9.1963 |
| [22] |
Tahiri-Alaoui A, Frigotto L, Manville N, et al. High affinity nucleic acid aptamers for streptavidin incorporated into bi-specific capture ligands[J]. Nucleic Acids Research, 2002, 30(10): e45. DOI:10.1093/nar/30.10.e45 |
| [23] |
Warner KD, Chen MC, Song W, et al. Structural basis for activity of highly efficient RNA mimics of green fluorescent protein[J]. Nature Structural & Molecular Biology, 2014, 21(8): 658. DOI:10.1038/nsmb.2865 |
| [24] |
Cassina V, Seruggia D, Beretta GL, et al. Atomic force microscopy study of DNA conformation in the presence of drugs[J]. European Biophysics Journal, 2011, 40(1): 59-68. DOI:10.1007/s00249-010-0627-6 |
| [25] |
Bawazer LA, Newman AM, Gu Q, et al. Efficient selection of biomineralizing DNA aptamers using deep sequencing and population clustering[J]. ACS Nano, 2013, 8(1): 387-395. DOI:10.1021/nn404448s |
| [26] |
李蓓蕾.钌(Ⅱ)/钴(Ⅲ)配合物与DNA/RNA作用机理及抗肿瘤活性研究[D].湘潭: 湘潭大学, 2012. Li BL. DNA-, RNA-binding behaviors and cytotoxic properties of Ru(Ⅱ)and Co(Ⅲ)Complexes[D]. Xiangtan: Xiangtan University, 2012. |
| [27] |
Wu FY, Xiang YL, Wu YM, et al. Study of interaction of a fluorescent probe with DNA[J]. Journal of Luminescence, 2009, 129(11): 1286-1291. DOI:10.1016/j.jlumin.2009.06.010 |
| [28] |
Deng H, Bloomfield VA, Benevides JM, et al. Dependence of the raman signature of genomic B-DNA on nucleotide base sequence[J]. Biopolymers :Original Research on Biomolecules, 1999, 50(6): 656-666. DOI:10.1002/(SICI)1097-0282(199911)50:63.0.CO;2-9 |
| [29] |
Gao EJ, Liu L, Zhu MC, et al. Synthesis, characterization, interaction with DNA, and cytotoxic effect in vitro of new monoand dinuclear Pd(II)and Pt(II)complexes with benzo[d] thiazol-2-amine as the primary ligand[J]. Inorganic Chemistry, 2011, 50(11): 4732-4741.
|
| [30] |
Hirohama T, Kuranuki Y, Ebina E, et al. Copper(II)complexes of 1, 10-phenanthroline-derived ligands :studies on DNA binding properties and nuclease activity[J]. Journal of Inorganic Biochemistry, 2005, 99(5): 1205-1219. DOI:10.1016/j.jinorgbio.2005.02.020 |
| [31] |
王君, 王琦, 陈丹丹, 等. DNA与小分子化合物相互作用的研究进展与展望[J]. 辽宁大学学报:自然科学版, 2013, 40(4): 289-300. Wang J, Wang Q, Chen DD, et al. The research progress and prospect of interaction of DNA with small molecule compounds[J]. Journal of Liaoning University: Natural Sciences Edition, 2013, 40(4): 289-300. DOI:10.3969/j.issn.1000-5846.2013.04.001 |
| [32] |
Lauhon CT, Szostak JW. RNA aptamers that bind flavin and nicotinamide redox cofactors[J]. Journal of the American Chemical Society, 1995, 117(4): 1246-1257. DOI:10.1021/ja00109a008 |
| [33] |
Rajendran M, Ellington AD. Selection of fluorescent aptamer beacons that light up in the presence of zinc[J]. Analytical & Bioanalytical Chemistry, 2008, 390(4): 1067-1075. |
| [34] |
Shen P, Li W, Liu Y, et al. High-throughput low-background g-quadruplex aptamer chemiluminescence assay for ochratoxin A using a single photonic crystal microsphere[J]. Analytical Chemistry, 2017, 89(21): 11862. DOI:10.1021/acs.analchem.7b03592 |
| [35] |
Lu YJ, Ma N, Li YJ, et al. Styryl quinolinium/G-quadruplex complex for dual-channel fluorescent sensing of Ag + and cysteine[J]. Sensors and Actuators B :Chemical, 2012, 173: 295-299. DOI:10.1016/j.snb.2012.07.007 |
| [36] |
Yang Y, Kochoyan M, Burgstaller P, et al. Structural basis of ligand discrimination by two related RNA aptamers resolved by NMR spectroscopy[J]. Science, 1996, 272(5266): 1343-1347. DOI:10.1126/science.272.5266.1343 |
| [37] |
Burgstaller P, Kochoyan M, Famulok M. Structural probing and damage selection of citrullineand arginine-specific RNA aptamers identify base positions required for binding[J]. Nucleic Acids Research, 1995, 23(23): 4769-4776. DOI:10.1093/nar/23.23.4769 |
| [38] |
Masud MM, Kuwahara M, Ozaki H, et al. Sialyllactose-binding modified DNA aptamer bearing additional functionality by SELEX[J]. Bioorg Med Chem, 2004, 12(5): 1111-1120. DOI:10.1016/j.bmc.2003.12.009 |
| [39] |
Yang Q, Goldstein IJ, Mei HY, et al. DNA ligands that bind tightly and selectively to cellobiose[J]. Proceedings of the National Academy of Sciences, 1998, 95(10): 5462-5467. DOI:10.1073/pnas.95.10.5462 |
| [40] |
Srisawat C, Goldstein IJ, Engelke DR. Sephadex-binding RNA ligands :rapid affinity purification of RNA from complex RNA mixtures[J]. Nucleic Acids Res, 2001, 29: 5. DOI:10.1093/nar/29.2.e5 |
| [41] |
Zimmermann GR, Jenison RD, Wick CL, et al. Interlocking structural motifs mediate molecular discrimination by a theophylline-binding RNA[J]. Nature Structural Biology, 1997, 4(8): 644-649. DOI:10.1038/nsb0897-644 |
| [42] |
Stojanovic MN, Prada PD, Landry DW. Fluorescent sensors based on aptamer self-assembly[J]. Journal of the American Chemical Society, 2000, 122(46): 11547-11548. DOI:10.1021/ja0022223 |
| [43] |
Kato T, Takemura T, Yano K, et al. In vitro selection of DNA aptamers which bind to cholic acid[J]. Biochimica et Biophysica Acta, 2000, 1493(1-2): 12-18. DOI:10.1016/S0167-4781(00)00080-4 |
| [44] |
Sazani PL, Larralde R, Szostak JW. A small aptamer with strong and specific recognition of the triphosphate of ATP[J]. Journal of the American Chemical Society, 2004, 126(27): 8370-8371. DOI:10.1021/ja049171k |
| [45] |
Roychowdhury-Saha M, Lato SM, Shank ED, et al. Flavin recognition by an RNA aptamer targeted toward FAD[J]. Biochemistry, 2002, 41(8): 2492-2499. DOI:10.1021/bi015719d |
| [46] |
Grate D, Wilson C. Laser-mediated, site-specific inactivation of RNA transcripts[J]. Proceedings of the National Academy of Sciences, 1999, 96(11): 6131-6136. DOI:10.1073/pnas.96.11.6131 |
| [47] |
Flinders J, Defina SC, Brackett DM, et al. Recognition of planar and nonplanar ligands in the malachite green-RNA aptamer complex[J]. Chembiochem, 2010, 5(1): 62-72. |
| [48] |
Huang H, Suslov NB, Li NS, et al. A G-quadruplex-containing RNA activates fluorescence in a GFP-like fluorophore[J]. Nature Chemical Biology, 2014, 10(8): 686-691. DOI:10.1038/nchembio.1561 |
| [49] |
Berens C, Thain A, Schroeder R. A tetracycline-binding RNA aptamer[J]. Bioorganic & Medicinal Chemistry, 2001, 9(10): 2549-2556. DOI:10.1016/S0968-0896(01)00063-3 |
| [50] |
Burke DH, Hoffman DC, Brown A, et al. RNA aptamers to the peptidyl transferase inhibitor chloramphenicol[J]. Chemistry and Biology, 1997, 4(11): 833-843. DOI:10.1016/S1074-5521(97)90116-2 |
| [51] |
Tereshko V, Skripkin E, Patel DJ. Encapsulating streptomycin within a small 40-mer RNA[J]. Chemistry & Biology, 2003, 10(2): 175-187. DOI:10.1016/S1074-5521(03)00024-3 |
| [52] |
Wallis MG, Schroeder R. The binding of antibiotics to RNA[J]. Progress in Biophysics & Molecular Biology, 1997, 67(23): 141. DOI:10.1016/S0079-6107(97)00011-4 |
| [53] |
Feagin TA, Maganzini N, Soh HT. Strategies for creating structureswitching aptamers[J]. ACS Sensors, 2018, 3(9): 1611-1615. DOI:10.1021/acssensors.8b00516 |
| [54] |
Luo Z, He L, Wang J, et al. Developing a combined strategy for monitoring the progress of aptamer selection[J]. Analyst, 2017, 142(17): 3136-3139. DOI:10.1039/C7AN01131H |