2. 中国科学院遗传与发育生物学研究所,北京 100101
2. Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101
选育早熟高产的新品种是作物遗传育种研究的主攻方向之一。氮素(Nitrogen)既是生物大分子如核酸、脂类和蛋白质等的组成成分,也是植物激素、叶绿素等生长发育必需元素的构成组分。氮素是植物生长发育不可或缺的大量元素,也是调控植物开花时间和种子产量最为重要的营养元素。
在合适的外界环境和内部因子的共同作用下,植物茎顶端的分生组织(Shoot apical meristem,SAM)由营养期转入生殖期,分化形成花芽,随后花器官发育、配子体发生、授粉受精,形成种子和果实。作物的生殖生长时期如抽穗、开花以及成熟期与作物产量密切相关。氮素不足迫使作物早衰,产量较低;而在农业生产实践中大量施用氮肥会导致作物“贪青晚熟”即生殖生长延后,开花成熟推迟。特别是在高纬度地区,生育后期如温度过低会影响灌浆从而导致作物大量减产。氮素的积累也是促进种子发育和提高作物产量的重要因素,取决于植物的氮素利用效率NUE(Nitrogen use efficiency),主要由氮素摄取效率NUpE(Nitrogen uptake efficiency)和氮素同化效率NUtE(Nitrogen utilization efficiency)共同决定[1]。
硝酸根(NO3-)是植物吸收利用的主要氮素来源,也是植物生长过程中的重要信号分子,在调节植物发育如种子萌发、根系形态建成、植株分支、叶片伸展、成花、种子发育以及植株衰老等过程中发挥重要作用[2]。其中,植物开花和产量与硝酸根转运、代谢与信号转导途径等多方面密切相关。基于此,本文对近年来模式植物拟南芥、水稻和其他主要农作物中硝酸根调控植物开花时间和种子产量的分子机制研究进展进行综述,重点介绍参与其中的关键基因及作用机理,以期为合理利用氮肥、提高氮素利用效率、培育早熟高产的作物新品种提供较为全面的理论参考。
1 硝酸根调控植物开花的分子机制开花是植物从营养生长到生殖生长过渡的关键阶段。高等植物开花包括开花诱导、花原基形成和花器官发育3个阶段,其进程受到复杂基因网络的调控。外界环境中不同浓度的硝酸根会对植物开花时间产生不同程度的影响。对模式植物拟南芥的研究表明,硝酸根浓度与开花时间呈U形曲线关系[3]。拟南芥在土培、水培和固体培养基条件下,硝酸根或氮素最适浓度分别为4 mmol/L、1 mmol/L和3-9mmol/L,此时植株体内硝酸根含量和信号转导水平最适宜植物开花,因此开花时间最早。浓度高于最适浓度时促进营养生长,延迟开花;而浓度低于最适浓度时,植株营养不足生长迟缓,也会导致开花延迟。目前,已鉴定得到部分参与硝酸根调控开花途径的关键基因,初步解析了植物感知并响应外界不同浓度硝酸根从而调控开花时间的分子机制。但是,所涉及的研究大多没有覆盖整个U型曲线,这些调控因子只在某一范围的硝酸根浓度下发挥作用。
1.1 硝酸根信号通路中参与开花调控的关键基因AtNRT1.1(Nitrate transporter 1.1)定位于根系表皮和皮层细胞,是拟南芥双亲和性硝酸根转运蛋白及感受器,在根系硝酸根摄取和植株初级硝酸根反应中发挥主导作用[4]。与野生型拟南芥植株相比,AtNRT1.1基因功能缺失突变体chl1-1和chl1-5在长日照或持续光照的条件下均呈现为晚花表型,抽薹后茎生长速度更慢,开第一朵花的时间更晚[5]。AtNRT1.1点突变株系chl1-9丧失了转运硝酸根的能力,却仍然保留了正常的初级硝酸根反应。突变体chl1-9开花特征与野生型无显著性,表明AtNRT1.1调控的拟南芥晚花表型与硝酸根转运无关,可能与其介导的硝酸根信号转导途径有关[6]。水稻基因组中存在3个AtNRT1.1同源蛋白,依据序列相似性高低分别命名为OsNRT1.1A、OsNRT1.1B和OsNRT1.1C[4]。其中,OsNRT1.1A与AtNRT1.1蛋白序列同源性最高,定位于液泡膜上,主要负责感受细胞内的硝酸根并传递信号;而OsNRT1.1B与AtNRT1.1的生物学功能最为接近,定位于细胞质膜上,主要负责转运硝酸根和感受胞外硝酸根信号[4]。突变体osnrt1.1a呈现出发育迟缓和晚熟的表型,而在水稻和拟南芥中分别超表达OsNRT1.1A均可促进植株早花。特别是在高氮条件下,超表达OsNRT1.1A的植株相较于野生种可提早开花2周以上,从而有效缩短了水稻的成熟时间[7]。因此,硝酸根作为信号分子可能通过不同物种中的NRT1.1s蛋白介导的信号途径调控植物开花(图 1)。

|
| ON:适宜浓度硝酸根;HN:高浓度硝酸根 图 1 拟南芥和水稻硝酸根信号调控植物开花的基因网络示意图 |
转录因子NLPs(Nin-like proteins)蛋白家族成员是硝酸根信号传导途径中的关键调控因子。在拟南芥中,硝酸根可能通过PLC(Phospholipase C)/InsP3(Inositol 1,4,5-trisphosphate)途径诱导胞内钙离子浓度的大幅度升高,从而激活钙依赖激酶CPKs(Calcium-dependent kinases)家族成员CPK10、CPK30和CPK32。CPK10/30/32进而将AtNLP7磷酸化,使其滞留在细胞核内[8]。经磷酸化修饰后的AtNLP7可结合到多个硝酸根转运基因(NRT1.1、NRT1.2、NRT2.1、NRT2.2和NAR2.1等)、同化基因(NIA1、NIA2和GS2等)以及响应基因(HYH、LBD37和LBD38)的启动子上,启动初级硝酸根反应[9]。拟南芥单突变体nlp6、nlp7和双突变体nlp6nlp7均表现出晚花的表型,而且双突变体开花比单突变体推迟时间更长[10]。水稻中,OsNLP3和OsNLP4是AtNLP7两个最为接近的同源蛋白。突变体osnrt1.1a中,OsNLP3和OsNLP4的核定位受到明显抑制;而在氮缺乏的条件下,在水稻原生质体中共表达OsNRT1.1A和OsNLP3或OsNLP4可促进后两者的核定位[7]。因此,不同物种中的NRT1.1s蛋白可能通过下游的NLPs转录调控因子调控植物开花(图 1)。
1.2 硝酸根信号通路与其他开花调控途径互作基因对模式植物拟南芥的研究表明,植物开花调控途径主要包括光周期途径(Photoperiod pathway)、自主途径(Autonomous pathway)、春化途径(Vernalization pathway)和赤霉素途径(Gibberellin pathway)四大类[11]。CO(Constans)编码一个具有锌指结构的转录因子,是光周期途径的核心蛋白[12]。长日照条件下,光敏色素(Phytochrome A,PHYA)、PHYB与隐花色素(Cryptochrome 1,CRY1)、CRY2互作,调节生物钟基因GI(Gigante)的表达,进而诱导CO的积累,促进拟南芥开花。FLC(Floweringlocus C)编码一个MADS-box家族的转录因子,是自主途径和春化途径中关键开花抑制因子[13]。春化作用负调控FLC的转录和FLC蛋白的表达水平,进而诱导拟南芥开花。其中,VRN1(Vernalization 1)、VRN2和VIN3(Vernalization insensitive 3)等通过修饰组蛋白维持FLC在发育后期的低水平表达[14]。自主途径中,FCA(Flowering control locus A)、FY、FVE和FLD(Flowering locus D)等是FLC的抑制因子,通过调节其mRNA水平影响开花[15]。赤霉素(Gibberellin,GA)是调节开花的主要激素之一[16]。成花诱导前,SAM组织可检测到活性GA的积累,而阻断GA合成或者信号转导过程均会影响拟南芥开花过程。下游因子FT(Flowering locus T)和SOC1(Suppressor of constans 1)整合上述多种调控途径,进而作用于花分生组织决定基因AP1(Apetala1)、CAL(Cauliflower)和LFY(Leafy)等,促进植株开花[17]。
拟南芥中硝酸根信号途径调控开花的基因网络相对独立于光周期、自主、春化和赤霉素等经典开花调控途径,但又与其中的关键基因互作[18](图 1)。CRY1是硝酸根途径与光周期途径互作的关键因子。硝酸根通过调控FNR1(Ferredoxin-NADP-+oxidoreductase 1)的表达改变NADPH/NADP+和ATP/AMP的比率,从而影响腺苷酸活化蛋白激酶(Adenosine monophosphate-activated protein kinase,AMPK)的活性[19]。AMPK可调节细胞核内CRY1蛋白丰度和昼夜节律基因以及光周期途径关键基因GI 和CO的转录水平,从而影响植株开花[19]。高浓度硝酸根以GA信号依赖的方式抑制拟南芥开花。高浓度硝酸根环境中,活性GA的合成减弱,GA信号途径的底物蛋白DELLA积累,能够上调其下游转录因子CGA1(Cytokinin-responsive gata factor 1)和GNC(Gata transcription factor 21)的表达[6]。AP2类转录因子SMZ(Schlafmutze)和SNZ(Schnarchzapfen)的表达水平随之提高,SMZ和SNZ蛋白通过与FT启动子结合,抑制FT表达,从而延迟开花[6]。各途径整合因子SOC1是硝酸根调控开花过程的重要元件。低浓度硝酸根环境下,SAM中SOC1的表达水平较低,引起晚花表型。硝酸根信号途径下游转录因子AtNLP7和AtNLP6也在SAM中表达,可能通过结合到SOC1两个上游调控基因SPL3(Squamosa promoter binding protein-like 3)和SPL5的启动子上,影响SOC1表达[10]。
水稻是短日照植物,但是其开花过程的光周期调控机制与拟南芥较为相似。水稻中的Hd3a(Heading date 3a)是与拟南芥中FT功能一致的开花促进因子,其表达在短日照下被诱导,促进植株开花[20]。RFT1(Rice flowering locus T1)与Hd3a同源性最高,是水稻长日照条件下的开花促进因子[21]。Ehd1(Early heading date 1)编码B型反应调控因子,是水稻中特有的开花调控基因。无论在短日照还是长日照条件下,Ehd1均是开花促进因子,分别在Hd3a和RFT1上游诱导二者的表达进而促进开花[22]。上文提到的水稻中感受胞内硝酸根信号的OsNRT1.1A晚花突变体中,Hd3a、RFT1和Ehd1的表达水平均显著下调,而早熟的OsNRT1.1A超表达植株中上述3个基因的表达水平均明显上调[7]。上述研究表明,水稻中硝酸根信号途径与光周期途径协同作用调控开花抽穗(图 1)。
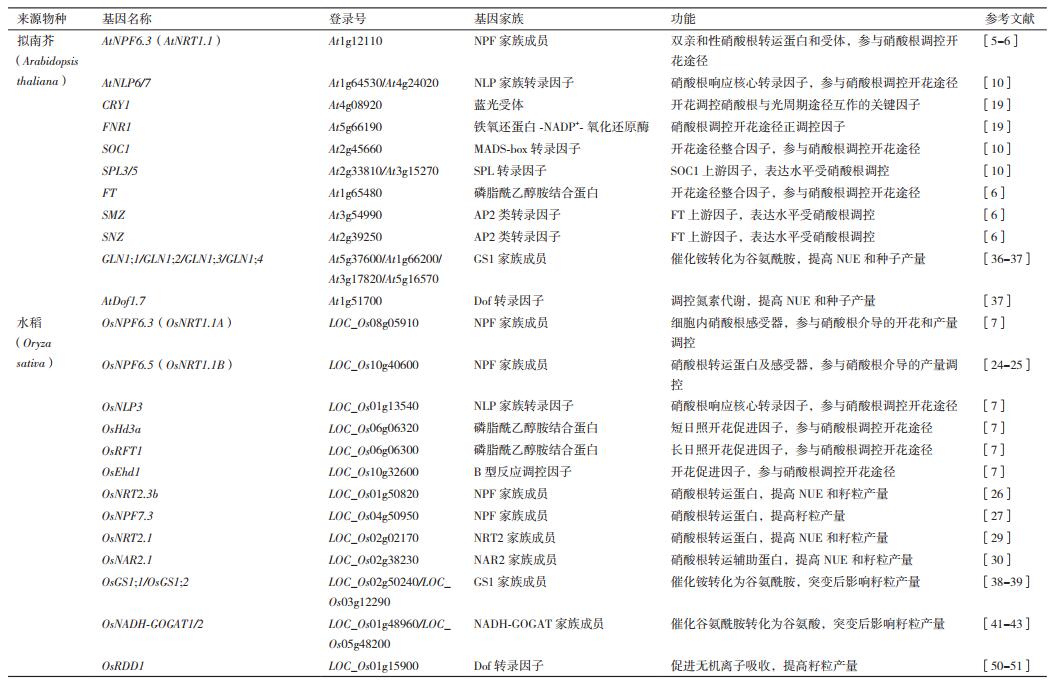
2 硝酸根调控植物产量的分子机制硝酸根的摄取和转运,即根系对硝酸根的摄取、硝酸根在根和茎中的长距离运输以及硝酸根再分配等过程,主要通过各类硝酸根转运蛋白完成。硝酸根的同化则由一系列氮素同化酶共同完成,包括硝酸还原酶NIA(Nitrate reductase)、亚硝酸还原酶NIR(Nitrite reductase)、谷氨酰胺合成酶(Glut-aminesynthase,GS)和谷氨酸合成酶(Glutamate synthase,GOGAT)等[1]。近年来,利用基因工程策略进行的转基因研究表明,调控硝酸根转运、同化和信号转导相关基因的表达可以影响植物的生长和产量(图 2)。

|
| 图 2 植物中硝酸根调控种子产量的相关基因总结 |
拟南芥中共有53个NPF(Nitrate transporter 1 /Peptide transporter)家族成员和7个NRT2家族成员。其中,2个NRT1类转运蛋白NPF6.3(NRT1.1)、NPF4.6(NRT1.2)和4个NRT2类转运蛋白NRT2.1、NRT2.2、NRT2.4和NRT2.5负责从外界吸收硝酸根[23]。3个NRT1类转运蛋白NPF7.3(NRT1.5)、NPF7.2(NRT1.8)和NPF2.9(NRT1.9)以及NPF2.3在硝酸根长距离转运过程中发挥作用[23]。4个NRT1类转运蛋白NPF2.12(NRT1.6)、NPF2.13(NRT1.7)、NPF1.1(NRT1.11)和NPF1.2(NRT1.12)和3个NRT2类转运蛋白NRT2.4、NRT2.5和NRT2.7以及NPF5.5在硝酸根循环再利用过程中发挥重要作用[23]。目前,虽有部分研究证明拟南芥中NPF家族和NRT2家族成员可影响氮素利用效率,但未见其调控结实产量的报道。
水稻中有多于80个NPF(NRT1/PTR)家族成员和4个NRT2家族成员[23]。虽然仅有部分硝酸根转运蛋白的功能得到解析,但仍有多个成员的功能显示与水稻结实产量密切相关。其中,NRT2.1、NRT2.2、NRT2.4、NPF6.3(NRT1.1A)、NPF6.5(NRT1.1B)、NPF8.9(NRT1)和NPF2.4定位于根系,负责从外界吸收硝酸根和传递硝酸根信号[23]。OsNRT1.1A的突变不但导致水稻开花期延长,同时降低了氮素利用效率和水稻产量;而过表达OsNRT1.1A在不同水稻品种及高氮低氮条件下均可显著提高水稻生物量和产量[7]。结合前文可知,OsNRT1.1A兼具促进植株早熟高产的双重效应。在籼稻与粳稻间,OsNRT1.1B蛋白一个氨基酸的区别导致二者氮素利用效率和产量产生显著差异[24]。籼稻具有更高的硝酸盐吸收及转运活性和硝酸根同化能力,在低氮和正常施氮条件下产量均显著高于粳稻。根系微生物组分析进一步表明,OsNRT1.1B在水稻根系微生物构成中发挥关键作用。与粳稻相比,籼稻根系富集的微生物类群更加多样化,微生物功能与氮素代谢更加相关,这可能是导致其氮素利用效率高于粳稻的重要原因之一[25]。NPF2.2、NRT2.3a、NRT2.3b、NPF6.5和NPF2.4参与硝酸根从根部到茎部的转运[23]。OsNRT2.3b主要定位于韧皮部细胞的细胞质膜,在其胞质侧具有一个通过pH感受机制调节硝酸根转运活性的结构域[26]。超表达OsNRT2.3b可提高植物pH平衡缓冲能力和对多种矿质元素的吸收能力。因此在不同氮素条件下,OsNRT 2.3b超表达水稻植株的NUE和产量均大幅提高[26]。OsNPF7.3定位于液泡膜,在硝酸根循环再利用过程中发挥作用。超表达OsNPF7.3不但可以促进水稻的生长,还可以促进生殖生长后期氮素由叶片向籽粒的转运从 而提高作物的产量[27]。OsNAR2.1是NRT2家族成员OsNRT2.1、OsNRT2.2和OsNRT2.3a等的互作因子,调控不同氮素条件下硝酸根的摄取[28]。转基因水稻中,以OsNAR2.1基因启动子分别调控OsNRT2.1和OsNAR2.1的表达均可大幅提高植株的NUE和产量[29-30]。
其他主要农作物中,玉米中有51个NPF和3个NRT2家族成员;大豆中有96个NPF和3个NRT2家族成员;小麦中有7个NPF和5个NRT2家族成员;高粱中有67个NPF和4个NRT2家族成员;谷子中有67个NPF和5个NRT2家族成员[31-32]。对于上述硝酸根转运蛋白的研究多集中在基因家族系统进化分析方面,生物学功能研究尚不深入,调控产量相关机制更不多见。高浓度硝酸根环境下,超表达小麦中高亲和硝酸根转运蛋白TaNRT2.1或TaNRT2.1及其辅助蛋白TaNAR2.1后,转基因拟南芥角果干重和整体生物量均较高[33]。小麦中另外一个NRT2家族成员TaNRT2.5也需要与TaNAR2.1互作,在根系硝酸根吸收和花期硝酸根循环再利用过程中发挥转运功能。超表达TaNRT2.5-3B的小麦植株生物量和籽粒产量均大幅提高,而其RNAi植株产量显著低于对照植株[34]。
2.2 转录因子与植物产量调控植物从外界环境中摄取硝酸根后,首先在胞质中被NIA转化为亚硝酸根(NO2-),随后转运到叶绿体或质体中被NIR1还原为铵(NH4+)。拟南芥中有2个NIA基因(AtNIA1和AtNIA2)和1个NIR基因(AtNIR),而水稻中有3个NIA基因(OsNIA1和OsNIA2等)和两个NIR基因(OsNIR1和OsNIR2)[23]。对NIA和NIR基因突变体及超表达植株的研究表明,上述基因的表达水平影响植株的氮素摄取和利用效率,但是对植株的生长和产量没有显著作用[35]。
铵由GS和GOGAT进一步同化,其中GS将铵转化为谷氨酰胺,GOGAT将其进一步转化为谷氨酸。在高等植物中,存在两条铵同化途径,一条定位于胞质中,受GS1和NADH-GOGAT调控,主要参与根部铵根代谢和叶片衰老进程中氮素的重新利用;另一条定位于质体中,受GS2和Fd-GOGAT调控,主要负责同化质体中硝酸根还原形成的铵和光呼吸产生的铵根[1]。
GS1在拟南芥和水稻中分别有5个成员(GLN1;1、GLN1;2、GLN1;3、GLN1;4和GLN1;5)和3个成员(OsGS1;1、OsGS1;2和OsGS1;3);GS2在拟南芥和水稻中均只有一个成员(AtGS2和OsGS2)[23]。相比GS2,GS1在种子发育调控中起到更为重要的作用。不同氮素条件下,拟南芥gln1;1/gln1;2/gln1;3三突变体叶中氨基酸和铵根的含量更高,而氮素再利用效率以及多个产量指标如种子总产量、单个种子重量和收获指数等均显著低于野生型植株[36]。在烟草中超表达拟南芥的GLN1;4基因,可提高植株在低氮条件下的GS活性、Glu含量以及种子产量[37]。水稻GS1基因突变体也有不同程度的产量降低。OsGS1;1在成熟叶片和韧皮部中表达丰度最高,其突变体成熟期的株高明显低于野生型,而且籽粒灌浆受到抑制导致穗粒体积较小 [38]。OsGS1;2以铵根依赖的方式在根部皮层细胞表达,其突变体分蘖数和穗数与野生型相比明显减少[39]。过表达OsGS1;1或OsGS1;2的水稻植株虽然GS活性升高,但是增产效果并不显著,仅有个别研究表明在特定的生长环境(人工生长箱)中超表达OsGS1;2可提高水稻在高氮条件下的产量[40]。
拟南芥中,Fd-GOGATs分别有2个同源基因GLU1和 GLU2编码,前者在叶片中丰度较高,而后者主要在根中表达;NADH-GOGAT仅有GLT一个基因编码[23]。与拟南芥相反,水稻中Fd-GOGAT有一个基因编码,而NADH-GOGAT有两个基因编码(OsNADH-GOGAT1和OsNADH-GOGAT2)[23]。OsNADH-GOGAT2与OsGS1;1组织定位类似,二者协同作用负责将衰老叶片中的氮素经韧皮部转运到穗中。在大田中,OsNADH-GOGAT2基因的突变导致水稻成熟期每穗小穗数与产量均大幅减少[41]。OsNADH-GOGAT1与OsGS1;2组织定位类似,二者协同作用负责根系中铵根的代谢。OsNADH-GOGAT1基因的突变不但影响水稻苗期根系的发育,同样影响其在大田中的产量。与对照植株相比,OsNADH-GOGAT1突变体每株穗数、每穗小穗数、千粒重等产量指标均小幅下调[42]。在其自身启动子的调控下过表达OsNADH-GOGAT,转基因水稻植株谷物重量提升幅度最高可达到80%[43]。
其他主要农作物中,硝酸根同化相关基因功能研究主要集中在GS1和GS2家族成员上。玉米中ZmGln1-3基因定位于叶肉细胞,是GS1家族中调控产量的主要成员[44]。其突变体籽粒数目明显减少,而超表达植株籽粒数目显著增加[44]。小麦中,TaGS2基因的4个单元型2Ab、2Ba、2Bb和2Da等与氮素利用和多种农艺性状密切相关[45]。导入其中一个优良单元型TaGS2-2Ab转基因小麦,不但氮素利用效率和生物量均明显高于对照植株,而且穗数、每穗粒数和千粒重等籽粒产量指标均有不同程度的提升[46]。高粱中,GS1家族成员之一的SbGln1.2在叶片中表达活性较高[47]。超表达SbGln1.2的转基因植株在冬季温室中生长,产量高于对照植株;而在春季实验中则无显著差异。这说明光照等其他环境因素可能会影响GS1的活性,进而调控植物的生长发育[47]。另外,将大豆中GS1家族成员GS15和Gmglnb1分别在百脉根和紫花苜宿中异源表达,均未发现对产量有显著影响[48-49]。
2.3 转录因子与植物产量调控越来越多的研究表明除NLPs外,许多其它类型的转录因子参与到硝酸根信号通路中连接外界氮素信号和细胞核内响应基因的表达。但是,仅有少数类型的转录因子与植物产量的调控关系得以阐明。
Dof(DNA binding with one finger)蛋白是植物特有的一类转录因子。其N末端保守的单锌指Dof结构域是既与DNA结合又和蛋白相互作用的双重功能域;C末端序列多变,是特异转录调控结构域。拟南芥、水稻和玉米等多个物种中的Dof转录因子家族成员均在植物增产中发挥重要调控作用。水稻中的Dof转录因子OsRDD1(Dof daily fluctuations 1)可通过促进包括氮素在内的各类无机离子的转运和吸收提高产量[50]。反义OsRDD1转基因水稻籽粒大小和千粒重均小于对照植株[51],而超表达OsRDD1水稻的氮素响应能力和每穗粒数、单株粒数及单株总粒重等产量指标均有不同幅度的提高[51]。玉米中Dof转录因子ZmDof1可平衡植株中N/C代谢。利用组织特异性启动子rbcS1调控ZmDof1在小麦中的表达,转基因植株的氮素利用效率、生物量和产量均有显著增加[52]。同时,在拟南芥和水稻中分别超表达ZmDof1均可提高植株在低氮条件下的氮素利用效率和营养生长[53-54]。此外,研究人员还将拟南芥的AtDof1.7基因转入烟草中进行过量表达。低氮条件下,转基因植株中总蛋白、叶绿素、蔗糖和葡萄糖含量以及参与碳氮代谢的多种酶类的活性明显高于野生型植株,种子产量也得到提高[37]。
NAC(NAM,ATAF and CUC)蛋白也是植物特有的一类转录因子,其N端具有一个约由150个氨基酸组成的保守NAC结构域,C端具有一个高度变异的转录激活区。TaNAC2-5A是小麦中一个受硝酸根诱导的NAC调控因子[55]。超表达TaNAC2-5A不但可以增强小麦根系吸收氮素的能力,还可以促进氮素向籽粒的转运进而提高籽粒产量[55]。进一步分析表明,TaNAC2-5A可直接与硝酸根转运蛋白TaNRT2.5-3B、TaNRT2.1-6B、TaNPF7.1-6D和谷氨酰胺合成酶TaGS2-2A等编码基因的启动子结合,并调控相应基因的表达[34, 55]。因此,TaNAC2-5A可直接通过调控硝酸根的吸收和同化促进小麦产 量的提高。
NF-Y是一类结合CCAAT-box的重要转录因子,包括NF-YA、NF-YB和NF-YC三个亚基。氮饥饿或磷饥饿的条件可诱导小麦中多个TaNF-YA基因的表达[56]。过表达其中的一个成员TaNF-YA-B1后,转基因小麦侧根分枝、硝酸根和磷酸根转运蛋白表达水平以及氮素吸收能力显著增强[56]。同时,不论在低氮还是低磷条件下,转基因小麦的穗数和籽粒产量与对照相比均有提高[56]。因此,TaNF-YA-B1可有助于投入较少的肥料达到高产的目标。
研究人员最近还鉴定了一个负调控小麦硝酸根吸收和籽粒产量的转录因子TabZIP60。与正调控转录因子不同,硝酸根可抑制TabZIP60的表达[57]。利用RNAi技术下调TabZIP60的表达后,NADHGOGAT酶活性增强,转基因小麦的侧根分枝、硝酸根吸收和穗数和籽粒产量均不同程度的增加;而超表达植株的表型与之相反[57]。ChIP-qPCR和EMSA实验表明,TabZIP60可结合到TaNADH-GOGAT-3B基因启动子中的ABRE结构域并负调控其表达[57]。进一步探究表明,将TaNADH-GOGAT-3B在超表达TabZIP60-6D的转基因株系中过表达,可恢复其穗数和产量减少的性状[57]。因此,TabZIP60影响小麦NUE和产量主要是通过其对TaNADH-GOGAT表达的负调控实现的。
3 总结与展望综上所述,植物通过感知外界环境硝酸根浓度变化、传递硝酸根信号以及有效地吸收分配硝酸根到不同的组织器官中等多种方式调节开花时间和种子产量。其中每一个步骤都受到外界因子和内在遗传调控网络的复杂调控。目前,已经对模式植物拟南芥和水稻以及部分农作物中促进早熟或高产的硝酸根感受器、转运蛋白、代谢调控基因和信号转导因子等的作用机理进行了阐释(表 1)。但是,在深入挖掘分子机制和应用于生产实践等方面仍然存在多个挑战。
许多氮素转运和代谢蛋白具有不同的表达模式及组织、细胞或亚细胞定位特征。精确动态地调控这些基因的时空表达对合理发挥其生物学功能至关重要。目前,大多数转基因研究中均采用CaMV 35S和Ubiquitin等组成型启动子驱动目的基因表达,进而提高NUE和植物产量。鉴于氮素转运和代谢蛋白的复杂性,在后续研究工作中可选择组织特异性启动子或诱导型启动子调控基因表达,进而更为精准有效地改善NUE。例如,通过低氮诱导的启动子调控硝酸根转运蛋白的表达进而增强植株在低氮环境中的硝酸根吸收能力;通过衰老诱导启动子调控硝酸根再利用蛋白的表达从而促进成熟期硝酸根从叶片向种子中的转运;通过根部或光诱导特异性启动子调控根系或叶片中硝酸根转运和代谢基因的表达等。
增强硝酸根的吸收和转运,在提高产量的同时可能会推迟开花而延长成熟时间。因此,具有早熟高产双重效应的调控因子还不多见。对拟南芥AtNRT1.1和水稻OsNRT1.1A的研究表明,多种作物中的NRT1.1s作为调节氮素利用和促进早熟高产的重要候选基因(图 3)。另外,水稻中OsNRT1.1A和OsNRT1.1B存在较大的功能异化,也提示我们同一作物中NRT1.1s的不同拷贝可在植物环境适应性中发挥重要调控作用。除NRT1.1s外,转录组学、蛋白组学、代谢组学和表型组学等多组学联用将为在今后工作中大力挖掘更多的双重效应调控因子提供有力手段。

|
| Le:Lycopersicon esculentum, 番茄;Os:Oryza sativa, 水稻;Gm:Glycine max, 大豆;Zm:Zea mays, 玉米;Sb:Sorghum bicolor, 高粱;At:Arabidopsis thaliana, 拟南芥;Bn:Brassica napus, 欧洲油菜;Cs:Cucumis sativus, 黄瓜;Gh:Gossypium hirsutum, 陆地棉;Ta:Triticum aestivum, 小麦 图 3 多种植物中NRT1.1s进化树分析和序列比对图 |
硝酸根作为信号分子调控植物生长,植物激素主要生物学功能之一也是调节植物发育。长期以来,人们对硝酸根和植物激素信号通路的解析大多分开进行,从而导致对营养元素和激素互作协同调控植物发育机制的理解比较有限。除了上文提到的开花调控外,根系形态建成和植株分枝性状与硝酸根利用和产量性状密切相关。为了适应土壤中硝酸根非均匀供应条件,植物根系表现出较强的可塑性反应,合理利用养分,维持适当生长状态和生理功能。同时,生长素、乙烯和脱落酸等3类激素在根系形态建成过程中发挥调节作用。分枝不但是植物株型的重要决定因子,也是与作物产量直接相关的生物性状。生长素、细胞分裂素和独脚金内酯等三类激素互作,共同调控植株分枝过程。因此,深入探究硝酸根和激素信号协同调控植物生长的分子机制,并进一步挖掘其中的核心作用元件,将是未来重要研究方向之一。
总之,揭示硝酸根相关重要基因的分子调控机制,并通过基因工程的手段进行遗传改良是改善植物开花及产量性状的可行方法。目前,除继续利用单双子叶模式植物进行基因功能解析外,仍亟需将模式植物的研究成果延伸到其他农作物品种中,寻找同源基因或者探究类似的调控机制,为合理利用氮肥、提高氮素利用效率和培育早熟高产作物新品种发挥更大的应用潜力。
| [1] |
Xu G, Fan X, Miller AJ. Plant nitrogen assimilation and use efficiency[J]. Annu Rev Plant Biol, 2012, 63: 153-182. DOI:10.1146/annurev-arplant-042811-105532 |
| [2] |
Wang YY, Cheng YH, Chen KE, et al. Nitrate transport, signaling, and use efficiency[J]. Annu Rev Plant Biol, 2018, 69: 85-122. DOI:10.1146/annurev-arplant-042817-040056 |
| [3] |
Lin YL, Tsay YF. Influence of differing nitrate and nitrogen availability on flowering control in Arabidopsis[J]. J Exp Bot, 2017, 68: 2603-2609. DOI:10.1093/jxb/erx053 |
| [4] |
Wang W, Hu B, Li A, et al. NRT1. 1s in plants :functions beyond nitrate transporter[J]. J Exp Bot, 2019, 71(15): 4373-4379. |
| [5] |
Guo FQ, Wang R, Chen M, et al. The Arabidopsis dual-affinity nitrate transporter gene AtNRT1. 1(CHL1)is activated and functions in nascent organ development during vegetative and reproductive growth[J]. Plant Cell, 2001, 13: 1761-1777. DOI:10.1105/TPC.010126 |
| [6] |
Gras DE, Vidal EA, Undurraga SF, et al. SMZ/SNZ and gibberellin signaling are required for nitrate-elicited delay of flowering time in Arabidopsis thaliana[J]. J Exp Bot, 2018, 69: 619-631. DOI:10.1093/jxb/erx423 |
| [7] |
Wang W, Hu B, Yuan D, et al. Expression of the nitrate transporter gene OsNRT1. 1A/OsNPF6. 3 confers high yield and early maturation in rice[J]. Plant Cell, 2018, 30: 638-651. DOI:10.1105/tpc.17.00809 |
| [8] |
Liu KH, Niu Y, Konishi M, et al. Discovery of nitrate-CPK-NLP signalling in central nutrient-growth networks[J]. Nature, 2017, 545: 311-316. DOI:10.1038/nature22077 |
| [9] |
Marvhive C, Roudier F, Castaings L, et al. Nuclear retention of the transcription factor NLP7 orchestrates the early response to nitrate in plants[J]. Nat Commun, 2013, 4: 1713. DOI:10.1038/ncomms2650 |
| [10] |
Olas JJ, Van D J, Abel C, et al. Nitrate acts at the Arabidopsis thaliana shoot apical meristem to regulate flowering time[J]. New Phytol, 2019, 223: 814-827. DOI:10.1111/nph.15812 |
| [11] |
Blimel M, Dally N, Jung C. Flowering time regulation in crops-what did we learn from Arabidopsis?[J]. Curr Opin Biotechnol, 2015, 32: 121-129. DOI:10.1016/j.copbio.2014.11.023 |
| [12] |
Suarez-Lopez P, Wheatley K, Robson F, et al. CONSTANS mediates between the circadian clock and the control of flowering in Arabidopsis[J]. Nature, 2001, 410: 1116-1120. DOI:10.1038/35074138 |
| [13] |
Michaels SD, He Y, Scotecci KC, et al. Attenuation of FLOWERING LOCUS C activity as a mechanism for the evolution of summerannual flowering behavior in Arabidopsis[J]. Proc Natl Acad Sci USA, 2003, 100: 10102-10107. DOI:10.1073/pnas.1531467100 |
| [14] |
Sung S, Amasino RM. Vernalization in Arabidopsis thaliana is mediated by the PHD finger protein VIN3[J]. Nature, 2004, 427: 159-164. DOI:10.1038/nature02195 |
| [15] |
Michaels SD, Amasino RM. Loss of FLOWERING LOCUS Cactivity eliminates the late-flowering phenotype of FRIGIDA and autonomous pathway mutations but not responsiveness to vernalization[J]. Plant Cell, 2001, 13: 935-941. DOI:10.1105/tpc.13.4.935 |
| [16] |
Mutasa-Gottgens E, Hedden P. Gibberellin as a factor in floral regulatory networks[J]. J Exp Bot, 2009, 60: 1979-1989. DOI:10.1093/jxb/erp040 |
| [17] |
Srikanth A, Schmid M. Regulation of flowering time :all roads lead to Rome[J]. Cell Mol Life Sci, 2011, 68: 2013-2037. DOI:10.1007/s00018-011-0673-y |
| [18] |
Marin IC, Loef I, Bartetizo L, et al. Nitrate regulates floral induction in Arabidopsis, acting independently of light, gibberellin and autonomous pathways[J]. Planta, 2011, 233: 539-552. DOI:10.1007/s00425-010-1316-5 |
| [19] |
Yuan S, Zhang ZW, Zheng C, et al. Arabidopsis cryptochrome 1 functions in nitrogen regulation of flowering[J]. Proc Natl Acad Sci USA, 2016, 113: 7661-7666. DOI:10.1073/pnas.1602004113 |
| [20] |
Itoh H, Nonoue Y, Yano M, et al. A pair of floral regulators sets critical day length for Hd3a florigen expression in rice[J]. Nat Genet, 2010, 42: 635-638. DOI:10.1038/ng.606 |
| [21] |
Komiya R, Yokoi S, Shimamito K. A gene network forlong-day flowering activates RFT1 encoding a mobile flowering signal in rice[J]. Development, 2009, 136: 3443-3450. DOI:10.1242/dev.040170 |
| [22] |
Doi K, Izawa T, Fuse T, et al. Ehd1, a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-like gene expression independently of Hd1[J]. Genes Dev, 2004, 18: 926-936. DOI:10.1101/gad.1189604 |
| [23] |
Li H, Hu B, Chu C. Nitrogen use efficiency in crops :lessons from Arabidopsis and rice[J]. J Exp Bot, 2017, 68: 2477-2488. DOI:10.1093/jxb/erx101 |
| [24] |
Hu B, Wang W, Ou S, et al. Variation in NRT1. 1B contributes to nitrate-use divergence between rice subspecies[J]. Nat Genet, 2015, 47: 834-838. DOI:10.1038/ng.3337 |
| [25] |
Zhang J, Liu YX, Zhang N, et al. NRT1. 1B is associated with root microbiota composition and nitrogen use in field-grown rice[J]. Nat Biotechnol, 2019, 37: 676-684. DOI:10.1038/s41587-019-0104-4 |
| [26] |
Fan X, Tang Z, Tan Y, et al. Overexpression of a pH-sensitive nitrate transporter in rice increases crop yields[J]. Proc Natl Acad Sci USA, 2016, 113: 7118-7123. DOI:10.1073/pnas.1525184113 |
| [27] |
Fang Z, Bai G, Huang W, et al. The rice peptide transporter OsNPF7. 3 is induced by organic nitrogen, and contributes to nitrogen allocation and grain yield[J]. Front Plant Sci, 2017, 8. |
| [28] |
Yan M, Fan XR, Feng HM, et al. Rice OsNAR2. 1 interacts with OsNRT2. 1, OsNRT2. 2 and OsNRT2. 3a nitrate transporters to provide uptake over high and low concentration ranges[J]. Plant Cell Environ, 2011, 34: 1360-1372. DOI:10.1111/j.1365-3040.2011.02335.x |
| [29] |
Chen JG, Zhang Y, Tan Y, et al. Agronomic nitrogen-use efficiency of rice can be increased by driving OsNAR2. 1 promoter[J]. Plant Biotechnol, 2016, 14: 1705-1715. DOI:10.1111/pbi.12531 |
| [30] |
Chen J, Fan X, Qian K, et al. pOsNAR2. 1 : OsNAR2. 1 expression enhances nitrogen uptake efficiency and grain yield in transgenic rice plants[J]. Plant Biotechnol J, 2017, 15: 1273-1283. DOI:10.1111/pbi.12714 |
| [31] |
Von W W, Neil JJB, Le C H, et al. Evolutionary classification of ammonium, nitrate, and peptide transporters in land plants[J]. BMC Evol Biol, 2014, 14: 11. DOI:10.1186/1471-2148-14-11 |
| [32] |
轩红梅, 王永华, 魏利婷, 等. 小麦幼苗叶片中硝酸盐转运蛋白NRT1和NRT2家族基因对氮饥饿响应的表达分析[J]. 麦类作物学报, 2014, 34: 1019-1028. Xuan HM, Wang YH, Wei LT, et al. Transcription analysis of the genes encoding nitrate transporter NRT1 and NRT2 Families in response to starvation in wheat seedling[J]. Journal of Triticeae Crops, 2014, 34: 1019-1028. |
| [33] |
李文虎, 田汇, 高亚军. 超表达小麦硝态氮转运蛋白基因TaNRT2. 1对拟南芥生长及氮吸收的影响[J]. 植物生理学报, 2018, 54: 651-659. Li WH, Tian H, Gao YJ, et al. Influence of over-expressing the wheat nitrate transporter genes TaNRT2.1 on affected the growth and nitrogen uptake of Arabidopsis thaliana[J]. Plant Physiology Journal, 2018, 54: 651-659. |
| [34] |
Li W, He X, Chen Y, et al. A wheat transcription factor positively sets seed vigour by regulating the grain nitrate signal[J]. New Phytol, 2020, 225: 1667-1680. DOI:10.1111/nph.16234 |
| [35] |
Good AG, Shrawat AK, Muench DG. Can less yield more? Is reducing nutrient input into the environment compatible with maintaining crop production[J]. Trends Plant Sci, 2004, 9: 597-605. DOI:10.1016/j.tplants.2004.10.008 |
| [36] |
Moison M, Marmagne A, Dinant S, et al. Three cytosolic glutamine synthetase isoforms localized in different-order veins act together for N remobilization and seed filling in Arabidopsis[J]. J Exp Bot, 2018, 69: 4379-4393. DOI:10.1093/jxb/ery217 |
| [37] |
Wang Y, Fu B, Pan L, et al. Overexpression of Arabidopsis Dof1, GS1 and GS2 enhanced nitrogen assimilation in transgenic tobacco grown under low-nitrogen conditions[J]. Plant Mol Biol Rep, 2013, 31: 886-900. DOI:10.1007/s11105-013-0561-8 |
| [38] |
Tabuchi M, Sugiyama K, Ishiyama K, et al. Severe reduction in growth rate and grain filling of rice mutants lacking OsGS1;1, a cytosolic glutamine synthetase1;1[J]. Plant J, 2005, 42: 641-651. DOI:10.1111/j.1365-313X.2005.02406.x |
| [39] |
Funayama K, Kojima S, Tabuchi-Kobayashi M, et al. Cytosolic glutamine synthetase1;2 is responsible for the primary assimilation of ammonium in rice roots[J]. Plant Cell Physiol, 2013, 54: 934-943. DOI:10.1093/pcp/pct046 |
| [40] |
Brauer EK, Rochon A, Bi YM, et al. Reappraisal of nitrogen use efficiency in rice overexpressing glutamine synthetase1[J]. Physiol Plant, 2011, 141: 361-372. DOI:10.1111/j.1399-3054.2011.01443.x |
| [41] |
Tamura W, Kojima S, Toyokama A, et al. Disruption of a novel NADH-glutamate synthase2 gene caused marked reduction in spikelet number of rice[J]. Front Plant Sci, 2011, 2: 57. DOI:10.3389/fpls.2011.00057 |
| [42] |
Tamura W, Hidaka Y, Tabuchi M, et al. Reverse genetics approach to characterize a function of NADH-glutamate synthase1 in rice plants[J]. Amino Acids, 2010, 39: 1003-1012. DOI:10.1007/s00726-010-0531-5 |
| [43] |
Yamaya T, Obara M, Nakajima H, et al. Genetic manipulation and quantitative-trait loci mapping for nitrogen recycling in rice[J]. J Exp Bot, 2002, 53: 917-925. DOI:10.1093/jexbot/53.370.917 |
| [44] |
Martin A, Lee J, Kichey T, et al. Two cytosolic glutamine synthetase isoforms of maize are specifically involved in the control of grain production[J]. Plant Cell, 2006, 18: 3252-3274. DOI:10.1105/tpc.106.042689 |
| [45] |
Li XP, Zhao XQ, He X, et al. Haplotype analysis of the genes encoding glutamine synthetase plastic isoforms and their association with nitrogen-use- and yield-related traits in bread wheat[J]. New Phytol, 2011, 189: 449-458. DOI:10.1111/j.1469-8137.2010.03490.x |
| [46] |
Hu M, Zhao X, Liu Q, et al. Transgenic expression of plastidic glutamine synthetase increases nitrogen uptake and yield in wheat[J]. Plant Biotechnol J, 2018, 16: 1858-1867. DOI:10.1111/pbi.12921 |
| [47] |
Urriola J, Rathore KS. Overexpression of a glutamine synthetase gene affects growth and development in sorghum[J]. Transgenic Res, 2015, 24: 397-407. DOI:10.1007/s11248-014-9852-6 |
| [48] |
Vincent R, Fraisier V, Chaillou, S, et al. Overexpression of a soybean gene encoding cytosolic glutamine synthetase in shoots of transgenic Lotus corniculatus L. plants triggers changes in ammonium assimilation and plant development[J]. Planta, 1997, 201: 424-433. DOI:10.1007/s004250050085 |
| [49] |
Ortega JL, Temple SJ, Sengupta-Gopalan C. Constitutive overexpression of cytosolic glutamine synthetase(GS1)gene in transgenic alfalfa demonstrates that GS1 may be regulated at the level of RNA stability and protein turnover[J]. Plant Physiol, 2001, 126: 109-121. DOI:10.1104/pp.126.1.109 |
| [50] |
Iwamotol M, Tagiri A. MicroRNA-targeted transcription factor gene RDD1 promotes nutrient ion uptake and accumulation in rice[J]. Plant J, 2016, 85: 466-477. DOI:10.1111/tpj.13117 |
| [51] |
Iwamoto M, Hi go, K, Takano M. Circadian clock- and phytochromeregulated Dof-like gene, Rdd1, is associated with grain size in rice[J]. Plant Cell Environ, 2009, 32: 592-603. DOI:10.1111/j.1365-3040.2009.01954.x |
| [52] |
Pena PA, Quach T, Sato S, et al. Expression of the maize Dof1 transcription factor in wheat and sorghum[J]. Front Plant Sci, 2017, 8: 434. DOI:10.3389/fpls.2017.00434 |
| [53] |
Yanagisaw AS, Akiyama A, Kisaka H, et al. Metabolic engineering with Dof1 transcription factor in plants :improved nitrogen assimilation and growth under low-nitrogen conditions[J]. Proc Natl Acad Sci USA, 2004, 101: 7833-7838. DOI:10.1073/pnas.0402267101 |
| [54] |
Kurai T, Wakayama M, Abiko T, et al. Introduction of the ZmDof1 gene into rice enhances carbon and nitrogen assimilation under low-nitrogen conditions[J]. Plant Biotechnol J, 2011, 9: 826-837. DOI:10.1111/j.1467-7652.2011.00592.x |
| [55] |
He X, Qu B, Li W, et al. The nitrate-inducible NAC transcription factor TaNAC2-5A controls nitrate response and increases wheat yield[J]. Plant Physiol, 2015, 169: 1991-2005. |
| [56] |
Qu B, He X, Wang J, et al. A wheat CCAAT box-binding transcription factor increases the grain yield of wheat with less fertilizer input[J]. Plant Physiol, 2015, 167: 411-423. DOI:10.1104/pp.114.246959 |
| [57] |
Yang J, Wang M, Li W, et al. Reducing expression of a nitrateresponsive bZIP transcription factor increases grain yield and N use in wheat[J]. Plant Biotechnol J, 2019, 17: 1823-1833. |