生命科学的许多应用研究,如分子诊断和治疗等,都需要基于不同细胞的特性标志物进行,并使用高亲和力和特异性的工具。允许进行这些过程的工具通常称为亲和力工具[1],其中主要是抗体和适配体。
目前,哺乳动物的抗体是最成功的提供广泛分子识别需求的亲和力工具,表征最充分,已经存在了40多年[2]。抗体作为生物体免疫系统自身产生的蛋白质,在当今临床医疗实践中应用甚广,如酶联免疫吸附测定(Enzyme linked immunosorbent assay,ELISA)和Western blotting。但是抗体也有局限性,包括高生产成本和低稳定性。近年来,一些亲和力工具,如工程结合蛋白[3]、适配体[2]和分子印迹聚合物(Molecular imprinted polymer,MIP)[4]都引起了人们的广泛关注。适配体是一类新的具有类似抗体识别功能的核酸分子,它作为新兴的领域,能以高亲和力和特异性识别靶分子,其特异性可与单克隆抗体的同类靶标和特异性相媲美。适配体的出现为生物和医学界提供了一种新的高效快速识别的研究平台,发展迅速,具备良好应用潜力。
细胞特异性核酸适配体是适配体中特殊的一类,其特异性识别的靶分子为细胞。靶标未知的內源状态、细胞活性及细胞表面的复杂性等因素,都对细胞特异性核酸适配体的筛选提出了挑战。该综述针对细胞特异性核酸适配体基于细胞表面标志物、全细胞、组织和体内的筛选,以及细胞适配体的亲和性、特异性、细胞活力、临床组织和体内可行性等几方面的评价进行总结,旨在为未来细胞适配体的筛选提供高效的筛选和评价方式。
1 细胞特异性适配体概述 1.1 适配体简介适配体(Aptamer)最早在1990年被发现和描述[5-6],是长度一般为20-100个核苷酸的单链DNA或RNA,名称“aptamer”来源于拉丁语aptus(合适),和希腊语merus(粒子)[7],中文译名为“核酸适配体”。
适配体能够折叠形成特定且稳定的三级结构,从而以高亲和力和特异性结合靶分子。靶分子类型十分广泛,包括氨基酸、金属离子、蛋白质、病毒、菌和全细胞等[8]。适配体与抗体相似,被称为“化学抗体”。但与传统蛋白质抗体相比,适配体具有独特的优势,包括尺寸小、分子量低、可快速渗透到组织和器官中、特异性强、化学改性较简单、没有批次间的变化、稳定性高、毒性低和免疫原性低等[9-10]。由于适配体的天然状态、构象及优点,使其具备探索临床运用的巨大前景。
1.2 细胞特异性适配体当前,细胞特异性核酸适配体在疾病成像及诊断[11-12]、药物递送[13-15]和免疫治疗[16]等方面得到了很大的发展,所以可用的细胞特异性适配体库的不断扩充尤为重要。
近年来,研究者们已进行大量的努力来筛选适配体,并不断发展和改进筛选方式。适配体一般是通过体外选择寡核苷酸文库中分离出来的,这个过程称为指数富集配体系统进化技术(Systematic evolution of ligands by exponential enrichment,SELEX)。基于SELEX,也有研究者提出了非SELEX(Non-SELEX)的筛选方式,SELEX和非SELEX是通过筛选过程中是否有序列的扩增富集来区分的。Berezovski等[17]首先报道了非SELEX筛选的适配体,他们运用平衡混合物的非平衡毛细管电泳进行分配,仅3个步骤就将DNA文库与靶蛋白的亲和力提高4个数量级以上。非SELEX没有聚合酶链式反应(Polymerase chain reaction,PCR)扩增和链分离富集的重复步骤,一个明显优势就是它的速度和简单性,与典型SELEX需要的数天到数周相比,非SELEX能在1 h内完成,且可以为不能被扩增的小分子库中的筛选提供可能。
在细胞特异性适配体的筛选方法中,SELEX是主要的方式,以全细胞作为靶标的方法最为常用,称为细胞SELEX(Cell-SELEX)。1990年,SELEX由两个实验室独立开发[5-6],它是一个循环的过程,涉及结合、洗脱、扩增等步骤。SELEX过程从1013-1016单链脱氧核糖核酸(Single-stranded deoxyribonucleic acid,ssDNA)或单链核糖核酸(Single-stranded ribonucleic acid,ssRNA)分子的随机库开始,每个序列含有随机区域和侧翼两个已知序列的区域,以便于扩增时的引物结合。筛选DNA适配体时,将随机库与靶分子孵育,除去未结合的序列并分离DNA/靶分子复合物,PCR扩增相应DNA序列,并进行下一轮筛选,多轮筛选后对候选适配体进行测序,并评价其靶亲和力等。筛选RNA适配体的过程与其类似,但需体外转录得到一个RNA随机库,且将结合靶分子的RNA序列进行逆转录并扩增。第一次Cell-SELEX是用于非洲布氏锥虫[18],研究者从RNA适配体库中筛选高亲和力适配体,其结合到血流阶段的非洲锥虫鞭毛袋上。目前Cell-SELEX已经应用于各类肿瘤细胞[19-20]、病毒感染的细胞[21]、正常细胞[22]及寄生虫[23]等特异性适配体的筛选。
常规的SELEX成熟且有效,但是有的富集次数多达20轮,时间从数周到几个月,消耗大量的时间和劳动力,所以用于筛选适配体的方法得到了不断的更新和发展。包括细胞表面表达靶标(Target expressed on cell surface,TECS)-SELX、荧光激活细胞分选(Fluorescence-activated cell sorting,FACS)-SELEX、交叉SELEX、配体引导选择(Ligand-guided selection,LIGS)、3D细胞SELEX、细胞内化SELEX,以及基于组织载玻片的SELEX和体内SELEX等。
2 细胞表面标志物适配体筛选针对细胞的适配体可以使用纯化的蛋白质作为靶标,该种蛋白质往往在某种或某类细胞表面特异性表达。有研究者通过重组人CD4,从含有2'-F-嘧啶的RNA文库中筛选人CD4特异性适配体,将这些适配体与荧光团偶联用于流式细胞术,观察具有不同密度CD4表达能力的细胞的染色情况[24]。Hu等[25]筛选出MUC1特异性适配体MA3,MUC1是在大多数恶性肿瘤细胞上过表达的跨膜糖蛋白,在正常组织中,MUC1的蛋白质核被糖链覆盖,而在肿瘤中,蛋白质核心由于糖基化不足而暴露出来,成为抗癌治疗的靶点[26]。经验证,适配体MA3可特异性地结合到MUC1阳性细胞上,包括A549和MCF-7[25]。此外,SELEX也可以不针对纯化的蛋白质,而针对细胞膜进行,Morris等[27]以人红细胞膜为靶物质筛选适配体,同时获得多个靶标的适配体,且其亲和力与针对纯靶标筛选出来的相当,证明了SELEX在复杂生物体系中的应用。Ababneh等[28]利用人类重组全长CD44蛋白和2’-F-嘧啶修饰的RNA文库,筛选出RNA适配体。细胞表面糖蛋白CD44是用于鉴定癌症干细胞(Cancer stem cell,CSC)最常见表面标记之一。而后使用表达CD44的代表性乳腺癌细胞系评估发现选定的RNA适配体(Apt1)与此类癌细胞发生特异性相互作用。
但是长期以来观察到,筛选自纯化蛋白质的适配体,有的时候可能不能以其内源状态(如细胞中)结合相同的蛋白质[29-30],Elle等[29]通过SELEX分离了针对CD73的含锁核酸(Locked Nucleic Acid,LNA)的DNA适配体,CD73是在实体瘤中经常过度表达的蛋白质。然而,当将适配体与CD73阳性的MDA-MB-231人乳腺癌细胞孵育时发现,没有显示出适配体对细胞的结合。细胞CD73是一种糖基磷脂酰肌醇(Glycosylphosphatidylinosi-tol,GPI)锚定的细胞表面蛋白,而在重组蛋白中适配体显示与CD73的C末端结构域结合,所以适配体识别的表位可能不可用于结合细胞蛋白。
使用活细胞作为靶标筛选适配体的方法则可以克服这一缺陷。在Cell-SELEX中,靶分子具有其自身的天然构象,也不需要对靶标进行纯化或将靶分子进行固定,可以在未知靶标的情况下对细胞的适配体进行筛选,能同时获得几条与靶细胞膜上不同位点结合的适配体,具备广泛的优势[31]。Cell-SELEX为靶向细胞表面蛋白提供了十分有用的策略,特别是对于转录后严重修饰,且不能容易地通过细菌表达系统以其天然修饰和折叠形式获得的蛋白。
3 全细胞适配体SELEX筛选方法 3.1 细胞表面表达靶标SELEX(TECS-SELEX)Cell-SELEX面临着许多挑战,其中一个重要的挑战是细胞在其表面上表达大量的蛋白质和其他化学部分,这些都是适配体的潜在靶标。为了解决这一问题,TECS-SELEX通过使用在其表面上过表达所需靶蛋白的细胞进行筛选,把正向筛选和不表达靶蛋白的细胞的负向筛选相互交替。Ohuchi等[32]在中国仓鼠卵巢细胞(Chinese hamster ovary,CHO)表面上异位表达人转化生长因子-β(TGF-β)Ⅲ型受体(TbRⅢ),在11轮TECS-SELEX后,分离出针对TbRⅢ的RNA适配体,从而无需纯化重组蛋白。Kim等[33]通过逆转录病毒介导的感染,在HepG2细胞中过表达上皮细胞黏附分子(Epithelial cell adhesion molecule,EpCAM),将过表达EpCAM的HepG2细胞作为正筛细胞,普通HepG2细胞作为负筛细胞,筛选出的适配体可用于干细胞和癌症中的研究。
3.2 荧光激活细胞分选SELEX(FACS-SELEX)死细胞的存在是所有类型SELEX的主要缺陷,因为死细胞会对适配体产生非特异性摄取,从而出现假阳性,导致选择过程的失败[34]。运用FACS,通过流式细胞仪根据细胞不同的光散射特性或将细胞用可渗透染料酶促转化成荧光,将活细胞和死细胞分开,能够使得筛选更有效,且周期较短。Raddatz等[34]运用FACS在选择过程中实施了死细胞的分离步骤,筛选CD19+ Burkitt淋巴瘤的适配体,证明了FACS-SELEX方法适用于细胞亚群的有效靶向。Kim等[35]在筛选成熟白色脂肪细胞适配体时,也运用FACS细胞分选富集DNA文库结合的细胞,并在筛选完成后运用其分析适配体对细胞的结合亲和力。
对于适配体筛选过程中死细胞干扰的去除,除了FACS,还有微珠的方法[36],在与DNA文库孵育前除去死细胞,可以减少靶结合序列的损失和富集的适配体被非特异性结合的序列污染。
3.3 交叉SELEX(Cross-over SELEX)在交叉SELEX中,纯化的蛋白质和全细胞都作为靶标,Hicke等[37]首先提出和应用这样的方式,从恶性胶质瘤细胞U251和纯化的tenascin-C蛋白中筛选了tenascin-C蛋白的特异性RNA适配体。与之对应,还存在反向交叉SELEX,即先在蛋白质上进行筛选,再在活细胞上筛选。但交叉SELEX由于涉及其他的筛选方式,所以会相对耗时[25]。
3.4 配体引导选择(LIGS)LIGS利用了SELEX的核心选择步骤,使用预先确定好的天然存在的更强和高度特异性的二价结合剂,与其同源抗原相互作用的抗体(Ab),从部分富集的SELEX文库中竞争特定的适配体,从而获得细胞表面已知靶标的适配体。Zumrut等[38]在表达膜结合的免疫球蛋白M(Membrane-bound immunoglobulin M,mIgM)的Ramos细胞的适配体筛选中,用mIgM的抗体选择性地洗脱特异性mIgM的适配体,将其作为分离步骤鉴定出了3种适配体的候选物。
3.5 细胞内化SELEX有的适配体不仅与细胞表面的靶标结合,还能在与靶标结合之后发生细胞内化。这样的适配体介导的siRNA和药物的递送对于癌症等疾病的治疗具有重要意义。Thiel等[39]将Cell-SELEX与高通量测序(High-throughput sequencing,HTS)和生物信息学相结合,富集了能够选择性内化到血管平滑肌细胞(VSMC)的RNA适配体。
3.6 3D细胞SELEX3D细胞SELEX运用基于磁悬浮技术的磁化噬菌体水凝胶来进行3D细胞培养,从而模拟自然状态下的细胞环境[40],在此基础上与Cell-SELEX相结合的方法。Souza等[41]对PC-3前列腺癌细胞系进行3D培养,形成球形细胞模拟肿瘤微环境,结合Cell-SELEX筛选出了8个PC-3特异性RNA适体。
3.7 消减SELEX(Subtractive SELEX)减法SELEX在每轮选择之前将文库与正常细胞孵育,分化的细胞作为靶标,从而能够区分同源来源和不同亚型等两个密切相关的细胞的适配体。Wang等[42]用分化的PC12细胞作为靶标,每轮选择之前,使用未分化的PC12细胞减去SELEX文库。经过6轮选拔,获得了仅对分化的PC12细胞具有特异性结合能力的高亲和力适配体。
3.8 SWCNTs辅助细胞SELEX(SWCNTs-assisted cell-SELEX)将特异性与非特异性结合的单链DNA(ssDNA)分离是提高筛选效率的重要步骤。为了克服大多数细胞SELEX仅使用洗涤导致分离不完全的问题,运用单壁碳纳米管(Single-walled carbon nanotubes,SWCNT)吸附ssDNA的性质,将其结合到细胞SELEX方法中。分离时,SWCNT吸附未结合或非特异性ssDNA,再对细胞上特异性结合的适配体进行富集。Tan等[43]将两个鼻咽癌(Nasopharyngeal carcinoma,NPC)细胞系CNE2细胞和HONE细胞分别用作靶细胞和阴性细胞,在SWCNTs辅助下,仅6个循环就筛选出了对CNE2细胞显示出高特异性和亲和力的适配体,有效提高了筛选效率。
3.9 高效的SELEX在磁珠(Magnetic bead,MB)上固定靶分子的SELEX方法由Bruno等[44]在1997年提出,磁珠-SELEX(MB-based SELEX)主要针对的靶标是蛋白质,通过磁分离将结合的适配体分离出来。Stoltenburg等[45]在2005年提出了FluMag-SELEX,结合了DNA荧光标记和磁分离技术的优点,每轮选择后通过荧光素标记进行DNA定量。针对不适合固定在固体表面的靶分子,在FluMag-SELEX的基础上发展了Capture-SELEX[46],创建特殊的DNA文库,将其通过对接序列固定在磁珠上。磁珠-SELEX也与微流体应用相结合,开发了微流体-SELEX,Gopinathan等[47]研发了一种能够自动识别胆管癌细胞特异性适配体的集成微流体系统,通过微流控芯片,显著提高了筛选速度,只需6轮即成功筛选出了3种特异性结合胆管癌细胞的适配体。
高通量测序目前也已成为一种相对便宜且用户友好的方法,可以运用其鉴定功能和稀有基序,比较每个寡核苷酸群体中的功能基序并定量其丰度。高通量测序辅助SELEX(HTS-SELEX)[39]也能够使得筛选过程更加高效。单克隆表面展示(Monocl-onal surface display,MSD)SELEX(MSD-SELEX)[48]也是一种发展起来的高效的SELEX技术。
4 基于组织载玻片的SELEX基于组织载玻片的SELEX是把适配体文库和癌组织切片孵育,从载玻片上刮下癌组织和结合序列的复合物,并扩增结合序列,再与正常组织切片的孵育作为负筛步骤。Li等[49]使用乳腺导管癌的石蜡组织切片作为靶标,相同病例或邻近组织作为对照,筛选验证了特异性识别来自不同病理类型的临床组织切片的乳腺癌细胞和具有异质性核糖核蛋白A1(Heterogeneous nuclear ribonucleoprotein A1,hnRNP A1)的细胞内靶标的乳腺癌细胞系的适配体,是一种具备优势的原位筛选策略。
5 体内SELEX体内SELEX与传统SELEX非常相似,但是使用活的生物体进行选择,一般通过尾静脉注射将文库注入特定小鼠体内,再将瘤体或组织取出,分离适配体并扩增,再进入下一轮注射筛选。由于机体内的生理环境复杂多变,所以体内SELEX可以克服体外筛选的一些缺陷,实现活体生物体肿瘤的特异性定位。Wang等[50]在非小细胞肺癌异种移植的小鼠模型中,从聚乙二醇化的RNA文库中筛选出适配体RA16,且该适配体剂量依赖性地抑制小鼠体内NCI-H460肿瘤的增殖。目前,通过体内SELEX筛选适配体已经应用在小鼠模型的多种组织或器官,包括大脑[51]、骨转移的前列腺癌[52]、雄性激素非依赖性前列腺肿瘤[53]及肝内转移的结肠癌[54]等。
6 细胞适配体的评价策略 6.1 适配体的亲和力评价亲和力表征是确定适配体筛选成功与否、评价其可用性的关键步骤。适配体与靶标的结合是一个动态平衡的过程,所以通常用平衡解离常数Kd值来衡量适配体与靶标的结合亲和力,Kd值的定义如式(1)所示,其中c指浓度,Kd值越小,表明二者相互作用越强,即亲和力越高。
 (1)
(1)
细胞特异性适配体最常用的亲和力表征方法是流式细胞术。通过配制一系列不同浓度的带荧光标记的适配体与细胞孵育,将孵育混合物通过流式细胞仪并检测每组的平均荧光强度,流式细胞仪可以将细胞-适配体复合物与游离的适配体或细胞分离,并测定带有荧光标记的复合物的数量。按照式(2)并运用软件(如Origin)即可进行非线性拟合并计算得到Kd值。其中适配体的浓度X为自变量,各组平均荧光强度值与对照组荧光强度值之差Y为因变量,Bmax指饱和荧光强度。
 (2)
(2)
Li等[55]为了确定所选适体的结合亲和力,将LoVo细胞(1 × 10 6)与不同浓度的FITC标记的适体在BB中于4 ℃温育20 min,并通过流式细胞术分析。ssDNA文库用作阴性对照。然后通过Sigma Plot软件(Jandel,San Rafael,CA)获得适配体-细胞相互作用的平衡解离常数(Kd)。Aptaker等[56]将胶质母细胞瘤U87MG细胞与不同浓度的Cy3标记的适配体SA43和SA44分别孵育,PBS洗涤并重悬于PBS中以进行流式细胞术分析,最终非线性回归分析得出,SA43和SA44适配体都以纳摩尔范围的高亲和力结合U87MG靶细胞。
6.2 适配体的特异性评价在评价适配体的特异性时,通常用到流式细胞仪和共聚焦显微镜检测。将荧光修饰的随机文库和适配体与靶细胞及各类非靶细胞孵育,并用流式细胞仪测定细胞表面的平均荧光强度,分析适配体与靶细胞和各类非靶细胞的结合情况,从而评价其特异性。为了使结果更加清晰明了,研究者们通常对流式检测结果进行处理,设置荧光强度的阈值。
Li等[55]为了确定所筛选适配体的细胞选择性,用流式细胞术对各类细胞系进行了适配体结合测定,并设定了流式细胞术分析的荧光强度的阈值,使得与FITC标记的ssDNA文库一起孵育的95%的细胞具有低于其的荧光强度。具有高于设定荧光阈值的细胞的百分比用于评价适配体与细胞的结合能力,并用不同符号加以清晰表示(-,< 10%;+,11%-35%;++,36%-60%;+++,61%-85%;++++,> 85%)。
此外,还可以通过细胞荧光成像评价适配体的特异性。一般把FAM荧光修饰的随机文库和适配体分别与靶细胞孵育,在激光扫描共聚焦显微镜下观察细胞表面的荧光强度。Ababneh等[28]利用流式细胞术和荧光显微镜分析,发现体外筛选的适配体RNA适配体Apt1与CD44阳性的肿瘤干细胞发生特异性相互作用,不同的荧光强度反映了细胞表面CD44的表达水平。
6.3 细胞凋亡评价评价所筛适配体对细胞活力的影响,对于适配体的后续应用是十分关键的。可以用筛选得到的适配体对靶细胞进行处理,测定细胞早期凋亡率和晚期凋亡率[56],同时设置随机文库的对照组。Aptaker等[57]在用适配体处理细胞后,运用显微镜观察细胞形态变化,并分别在并在24 h、48 h和72 h下评估了细胞的增殖活性。判断所筛适配体是否对靶细胞有明显促凋亡作用,能够指导适配体的可用性。当所筛适配体对靶细胞有适度抑制作用时,可能用于肿瘤细胞的抑制[58],而当适配体具有最小细胞毒性时,则可以被应用于药物、siRNA等的靶向递送。
6.4 临床组织和体内可行性评价对于体外筛选的细胞特异性适配体,为了更符合实际医学领域的应用,体内可行性评价也是比较重要的。Huang等[59]为了考察筛选所得适配体能否与实体瘤特异性结合,在裸鼠体内种植移植瘤,而后取出肿瘤做成冰冻切片。核酸适配体apt-A和apt-B与组织切片孵育和清洗后,荧光显微镜下出现明显绿色荧光,而随机文库则没有,证明了适配体与实体瘤的结合,为体内靶向奠定了一定基础。Wu等[60]为了测试适配体XQ-2d是否在体内保持其识别能力,将其用Cy5标记,并通过尾静脉注射到胰腺导管腺癌(Pancreaticductal adenocarcinoma,PDAC)荷瘤小鼠中,小鼠的肿瘤位点比随机文库的对照组显示出更高的荧光信号,且肿瘤中的积累也比心脏、脾、肺中更多,表明了适配体XQ-2d具有体内PDAC靶向能力,为PDAC的诊断和治疗提供了潜在的分子探针。
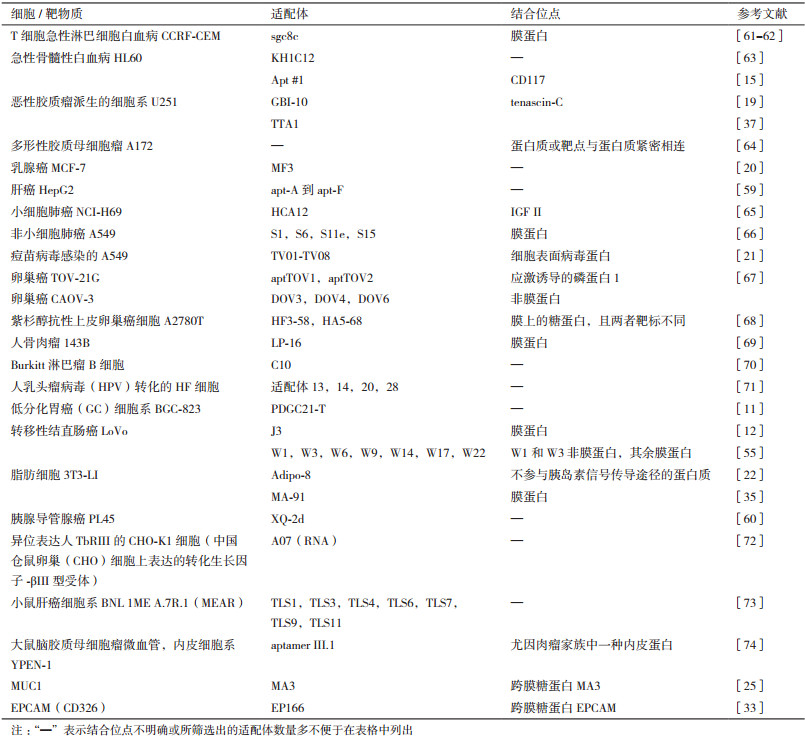
7 总结与展望近年来,核酸适配体的筛选方法得到了不断的发展,其中,细胞特异性适配体的筛选方法也结合细胞本身的特点而不断改进。本文综述了细胞特异性适配体的筛选方式,以及评价适配体的特异性、亲和力、体内可行性等的策略。细胞特异性适配体的筛选方式包括基于细胞标志物的筛选、全细胞筛选和组织及体内筛选等。现列出目前常见的细胞特异性适配体,如表 1所示。
相较于其他靶标,在细胞适配体的筛选过程中,细胞活力的保持及死细胞的去除都十分关键。在整个筛选过程中,要防止吹打和过度酶处理对细胞膜造成的损伤,多种死细胞的去除方法也被结合到细胞适配体的筛选中,包括荧光激活细胞分选和微珠处理等。相较于特定靶标分子,细胞特异性适配体的筛选可以在未知靶标的前提下进行,并且能够一次性获得多条结合不同靶标的适配体,但同时细胞表面的复杂性又会对筛选成功率产生不利影响。
针对现有的筛选方法,筛选所得适配体,及其相应评价策略,从3个方面展开如下讨论。
首先,从细胞适配体的筛选方法角度分析。目前的筛选方法仍有很大的进步空间。虽然筛选细胞适配体的方法有很多,但针对这些筛选方法还没有一个合理且普遍通用的标准,仍需要一些研究来弥补空白。而且,在筛选过程中往往只采用一种细胞作为负筛细胞,这样的实验设置是不严谨的。另外,随着计算机模拟和高通量测序方法在细胞适配体筛选中的应用,核酸适配体的筛选周期被大大缩短。
其次,从筛选得到的细胞适配体的评价角度分析。目前得到的细胞适配体还存在以下不足:针对筛选得到的核酸适配体的评价标准仍不明确;关于适配体在细胞上的结合位点及与细胞的作用力、作用方式的探究是缺乏的;现有研究大多是将筛选得到细胞适配体,与经由胰蛋白酶或蛋白酶K处理的细胞样品结合,从而判定适配体的结合位点是否为膜蛋白,对于非膜蛋白的结合位点鲜有进一步的探索。
再者,从筛选得到的核酸适配体的应用角度分析。人工碱基对的应用、计算机的应用及文库修饰等都迫使适配体的文库设计不断改进,其可能会增加适配体筛选过程的成本,但文库的改善对于筛选具备重要意义。经系列筛选后,适配体的修饰、定点诱变、截短等增强其特性的手段也在不断发展,使筛选得到的适配体在应用性上可以大大加强。
此外,当前细胞特异性适配体的筛选大多集中在癌细胞上。而针对人源正常细胞的适配体筛选还存在很大的空白。希望广大研究者能够进一步努力,继续发展高效省时的适配体筛选方法,逐步丰富和完善适配体库。
| [1] |
Ruigrok VJ, Levisson M, Eppink MH, et al. Alternative affinity tools:More attractive than antibodies?[J]. The Biochemical Journal, 2011, 436(1): 1-13. |
| [2] |
Jayasena SD. Aptamers:An emerging class of molecules that rival antibodies in diagnostics[J]. Clinical Chemistry, 1999, 45(9): 1628-1650. DOI:10.1093/clinchem/45.9.1628 |
| [3] |
Beck A, Wurch T, Bailly C, et al. Strategies and challenges for the next generation of therapeutic antibodies[J]. Nature Reviews Immunology, 2010, 10(5): 345-352. DOI:10.1038/nri2747 |
| [4] |
Ge Y, Turner AP. Too large to fit? Recent developments in macromolecular imprinting[J]. Trends in Biotechnology, 2008, 26(4): 218-224. DOI:10.1016/j.tibtech.2008.01.001 |
| [5] |
Ellington AD, Szostak JW. In vitro selection of rna molecules that bind specific ligands[J]. Nature, 1990, 346(6287): 818-822. DOI:10.1038/346818a0 |
| [6] |
Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment:RNA ligands to bacteriophage T4 DNA polymerase[J]. Science, 1990, 249(4968): 505-510. DOI:10.1126/science.2200121 |
| [7] |
Darmostuk M, Rimpelova S, Gbelcova H, et al. Current approaches in SELEX:An update to aptamer selection technology[J]. Biotechnology Advances, 2015, 33(6): 1141-1161. DOI:10.1016/j.biotechadv.2015.02.008 |
| [8] |
Ye M, Hu J, Peng M, et al. Generating aptamers by Cell-SELEX for applications in molecular medicine[J]. International Journal of Molecular Sciences, 2012, 13(3): 3341-3353. DOI:10.3390/ijms13033341 |
| [9] |
Banerjee J, Nilsen-Hamilton M. Aptamers:Multifunctional molecules for biomedical research[J]. Journal of Molecular Medicine, 2013, 91(12): 1333-1342. DOI:10.1007/s00109-013-1085-2 |
| [10] |
Cai S, Yan J, Xiong H, et al. Investigations on the interface of nucleic acid aptamers and binding targets[J]. Analyst, 2018, 143(22): 5317-5338. DOI:10.1039/C8AN01467A |
| [11] |
Li W, Wang S, Zhou L, et al. An ssDNA aptamer selected by Cell-SELEX for the targeted imaging of poorly differentiated gastric cancer tissue[J]. Talanta, 2019, 199: 634-642. DOI:10.1016/j.talanta.2019.03.016 |
| [12] |
Yuan B, Jiang X, Chen Y, et al. Metastatic cancer cell and tissue-specific fluorescence imaging using a new DNA aptamer developed by Cell-SELEX[J]. Talanta, 2017, 170: 56-62. DOI:10.1016/j.talanta.2017.03.094 |
| [13] |
Li F, Lu J, Liu J, et al. A water-soluble nucleolin aptamer-paclitaxel conjugate for tumor-specific targeting in ovarian cancer[J]. Nature Communications, 2017, 8(1): 1390. DOI:10.1038/s41467-017-01565-6 |
| [14] |
Pusuluri A, Krishnan V, Lensch V, et al. Treating tumors at low drug doses using an aptamer-peptide synergistic drug conjugate[J]. Angewandte Chemie International Edition, 2019, 58(5): 1437-1441. DOI:10.1002/anie.201812650 |
| [15] |
Zhao N, Pei SN, Qi J, et al. Oligonucleotide aptamer-drug conjug-ates for targeted therapy of acute myeloid leukemia[J]. Biomate-rials, 2015, 67: 42-51. DOI:10.1016/j.biomaterials.2015.07.025 |
| [16] |
Yang S, Wen J, Li H, et al. Aptamer-engineered natural killer cells for cell-specific adaptive immunotherapy[J]. Small, 2019, 15(22): e1900903. DOI:10.1002/smll.201900903 |
| [17] |
Berezovski M, Musheev M, Drabovich A, et al. Non-SELEX selection of aptamers[J]. Journal of the American Chemical Society, 2006, 128(5): 1410-1411. DOI:10.1021/ja056943j |
| [18] |
Homann M, Göringer HU. Combinatorial selection of high affinity RNA ligands to live African trypanosomes[J]. Nucleic Acids Research, 1999, 27(9): 2006-2014. DOI:10.1093/nar/27.9.2006 |
| [19] |
Daniels DA, Chen H, Hicke BJ, et al. A tenascin-C aptamer identified by tumor cell SELEX:Systematic evolution of ligands by exponential enrichment[J]. Proceedings of the National Academy of Sciences, 2003, 100(26): 15416-15421. DOI:10.1073/pnas.2136683100 |
| [20] |
Liu M, Yang T, Chen Z, et al. Differentiating breast cancer molecular subtypes using a DNA aptamer selected against MCF-7 cells[J]. Biomaterials Science, 2018, 6(12): 3152-3159. DOI:10.1039/C8BM00787J |
| [21] |
Tang Z, Parekh P, Turner P, et al. Generating aptamers for recognition of virus-infected cells[J]. Clinical Chemistry, 2009, 55(4): 813-822. |
| [22] |
Liu J, Liu H, Sefah K, et al. Selection of aptamers specific for adipose tissue[J]. PLoS One, 2012, 7(5): e37789. DOI:10.1371/journal.pone.0037789 |
| [23] |
Moreno MM, González VM. Advances on aptamers targeting plasmodium and trypanosomatids[J]. Current Medicinal Chemistry, 2011, 18(32): 5003-5010. DOI:10.2174/092986711797535218 |
| [24] |
Davis KA, Abrams B, Lin Y, et al. Staining of cell surface human CD4 with 2'-f-pyrimidine-containing rna aptamers for flow cytometry[J]. Nucleic Acids Research, 1998, 26(17): 3915-3924. DOI:10.1093/nar/26.17.3915 |
| [25] |
Hu Y, Duan J, Zhan Q, et al. Novel MUC1 aptamer selectively delivers cytotoxic agent to cancer cells in vitro[J]. PLoS One, 2012, 7(2): e31970. DOI:10.1371/journal.pone.0031970 |
| [26] |
Burchell JM, Mungul A, Taylor-Papadimitriou J. O-linked glycosylation in the mammary gland:Changes that occur during malignancy[J]. Journal of Mammary Gland Biology and Neoplasia, 2001, 6(3): 355-364. DOI:10.1023/A:1011331809881 |
| [27] |
Morris KN, Jensen KB, Julin CM, et al. High affinity ligands from In vitro selection:Complex targets[J]. Proceedings of the National Academy of Sciences, 1998, 95(6): 2902-2907. DOI:10.1073/pnas.95.6.2902 |
| [28] |
Ababneh N, Alshaer W, Allozi O, et al. In vitro selection of modified RNA aptamers against CD44 cancer stem cell marker[J]. Nucleic Acid Therapeutics, 2013, 23(6): 401-407. |
| [29] |
Elle IC, Karlsen KK, Terp MG, et al. Selection of LNA-containing DNA aptamers against recombinant human CD73[J]. Molecular BioSystems, 2015, 11(5): 1260-1270. DOI:10.1039/C5MB00045A |
| [30] |
Mallikaratchy P. Evolution of complex target SELEX to identify aptamers against mammalian cell-surface antigens[J]. Molecules, 2017, 22(2): 215. DOI:10.3390/molecules22020215 |
| [31] |
Kaur H. Recent developments in cell-SELEX technology for aptamer selection[J]. Biochimica et Biophysica Acta(BBA)-General Subjects, 2018, 1862(10): 2323-2329. DOI:10.1016/j.bbagen.2018.07.029 |
| [32] |
Ohuchi SP, Ohtsu T, Nakamura Y. Selection of RNA aptamers against recombinant transforming growth factor-β type Ⅲ receptor displayed on cell surface[J]. Biochimie, 2006, 88(7): 897-904. DOI:10.1016/j.biochi.2006.02.004 |
| [33] |
Kim JW, Kim EY, Kim SY, et al. Identification of DNA aptamers toward epithelial cell adhesion molecule via cell-SELEX[J]. Molecules and Cells, 2014, 37(10): 742-746. DOI:10.14348/molcells.2014.0208 |
| [34] |
Raddatz MSL, Dolf A, Endl E, et al. Enrichment of cell-targeting and population-specific aptamers by fluorescence-activated cell sorting[J]. Angew Chem Int Edi Engl, 2008, 47(28): 5190-5193. DOI:10.1002/anie.200800216 |
| [35] |
Kim EY, Kim JW, Kim WK, et al. Selection of aptamers for mature white adipocytes by cell SELEX using flow cytometry[J]. PLoS One, 2014, 9(5): e97747. DOI:10.1371/journal.pone.0097747 |
| [36] |
Avci-Adali M, Metzger M, Perle N, et al. Pitfalls of cell-systematic evolution of ligands by exponential enrichment(SELEX):Existing dead cells during in vitro selection anticipate the enrichment of specific aptamers[J]. Oligonucleotides, 2010, 20(6): 317-323. DOI:10.1089/oli.2010.0253 |
| [37] |
Hicke BJ, Marion C, Chang YF, et al. Tenascin-C aptamers are generated using tumor cells and purified protein[J]. Journal of Biological Chemistry, 2001, 276(52): 48644-48654. DOI:10.1074/jbc.M104651200 |
| [38] |
Zumrut HE, Ara MN, Fraile M, et al. Ligand-guided selection of target-specific aptamers:A screening technology for identifying specific aptamers against cell-surface proteins[J]. Nucleic Acid Therapeutics, 2016, 26(3): 190-198. |
| [39] |
Thiel WH, Bair T, Peek AS, et al. Rapid identification of cell-specific, internalizing RNA aptamers with bioinformatics analyses of a cell-based aptamer selection[J]. PLoS One, 2012, 7(9): e43836. DOI:10.1371/journal.pone.0043836 |
| [40] |
Souza GR, Molina JR, Raphael RM, et al. Three-dimensional tissue culture based on magnetic cell levitation[J]. Nature Nanotechnology, 2010, 5(4): 291-296. |
| [41] |
Souza AG, Marangoni K, Fujimura PT, et al. 3D cell-selex:Development of rna aptamers as molecular probes for PC-3 tumor cell line[J]. Exp Cell Res, 2016, 341(2): 147-156. DOI:10.1016/j.yexcr.2016.01.015 |
| [42] |
Wang C, Zhang M, Yang G, et al. Single-stranded DNA aptamers that bind differentiated but not parental cells:Subtractive systematic evolution of ligands by exponential enrichment[J]. Journal of Biotechnology, 2003, 102(1): 15-22. DOI:10.1016/S0168-1656(02)00360-7 |
| [43] |
Tan Y, Guo Q, Xie Q, et al. Single-walled carbon nanotubes(SWCNTs)-assisted cell-systematic evolution of ligands by exponential enrichment(cell-SELEX)for improving screening efficiency[J]. Analytical Chemistry, 2014, 86(19): 9466-9472. DOI:10.1021/ac502166b |
| [44] |
Bruno JG. In vitro selection of DNA to chloroaromatics using magnetic microbead-based affinity separation and fluorescence detection[J]. Biochemical and Biophysical Research Communications, 1997, 234(1): 117-120. DOI:10.1006/bbrc.1997.6517 |
| [45] |
Stoltenburg R, Reinemann C, Strehlitz B. FluMag-SELEX as an advantageous method for DNA aptamer selection[J]. Analytical and Bioanalytical Chemistry, 2005, 383(1): 83-91. DOI:10.1007/s00216-005-3388-9 |
| [46] |
Stoltenburg R, Nikolaus N, Strehlitz B. Capture-selex:Selection of DNA aptamers for aminoglycoside antibiotics[J]. Journal of Analytical Methods in Chemistry, 2012. DOI:10.1155/2012/415697 |
| [47] |
Gopinathan P, Hung LY, Wang CH, et al. Automated selection of aptamers against cholangiocarcinoma cells on an integrated microfluidic platform[J]. Biomicrofluidics, 2017, 11(4): 044101. DOI:10.1063/1.4991005 |
| [48] |
Zhu Z, Song Y, Li C, et al. Monoclonal surface display selex for simple, rapid, efficient, and cost-effective aptamer enrichment and identification[J]. Analytical Chemistry, 2014, 86(12): 5881-5888. |
| [49] |
Li SH, Xu H, Ding HM, et al. Identification of an aptamer targeting hnrnp a1 by tissue slide-based selex[J]. J Pathol, 2009, 218(3): 327-336. |
| [50] |
Wang H, Zhang Y, Yang H, et al. in vivo selex of an inhibitory nsclc-specific rna aptamer from pegylated rna library[J]. Molecular Therapy-Nucleic Acids, 2018, 10: 187-198. DOI:10.1016/j.omtn.2017.12.003 |
| [51] |
Cheng C, Chen YH, Lennox KA, et al. in vivo selex for identification of brain-penetrating aptamers[J]. Molecular Therapy-Nucleic Acids, 2013, 2: e67. DOI:10.1038/mtna.2012.59 |
| [52] |
Chen L, He W, Jiang H, et al. in vivo selex of bone targeting aptamer in prostate cancer bone metastasis model[J]. International Journal of Nanomedicine, 2019, 14: 149-159. |
| [53] |
Civit L, Theodorou I, Frey F, et al. Targeting hormone refractory prostate cancer by in vivo selected DNA libraries in an orthotopic xenograft mouse model[J]. Scientific Reports, 2019, 9(1): 4976. DOI:10.1038/s41598-019-41460-2 |
| [54] |
Mi J, Ray P, Liu J, et al. in vivo selection against human colorectal cancer xenografts identifies an aptamer that targets RNA helicase protein DHX9[J]. Molecular Therapy-Nucleic Acids, 2016, 5(4): e315. |
| [55] |
Li WM, Bing T, Wei JY, et al. Cell-SELEX-based selection of aptamers that recognize distinct targets on metastatic colorectal cancer cells[J]. Biomaterials, 2014, 35(25): 6998-7007. DOI:10.1016/j.biomaterials.2014.04.112 |
| [56] |
黄旭方.基于细胞SELEX技术的MMP14特异性适配体筛选及双模态分子探针的构建与初步功能研究[D].西安: 中国人民解放军空军军医大学, 2018.
|
| [57] |
Aptekar S, Arora M, Lawrence CL, et al. Selective targeting to glioma with nucleic acid aptamers[J]. PLoS One, 2015, 10(8). |
| [58] |
Rosenberg JE, Bambury RM, Van Allen EM, et al. A phase Ⅱ trial of AS1411(a novel nucleolin-targeted DNA aptamer)in metastatic renal cell carcinoma[J]. Investigational New Drugs, 2014, 32(1): 178-187. DOI:10.1007/s10637-013-0045-6 |
| [59] |
Huang R, Chen Z, Liu M, et al. The aptamers generated from HepG2 cells[J]. Science China Chemistry, 2017, 60(6): 786-792. DOI:10.1007/s11426-016-0491-7 |
| [60] |
Wu X, Zhao Z, Bai H, et al. DNA aptamer selected against pancreatic ductal adenocarcinoma for in vivo imaging and clinical tissue recognition[J]. Theranostics, 2015, 5(9): 985-994. DOI:10.7150/thno.11938 |
| [61] |
Shangguan D, Li Y, Tang Z, et al. Aptamers evolved from live cells as effective molecular probes for cancer study[J]. Proceedings of the National Academy of Sciences, 2006, 103(32): 11838-11843. DOI:10.1073/pnas.0602615103 |
| [62] |
Huang YF, Shangguan D, Liu H, et al. Molecular assembly of an aptamer-drug conjugate for targeted drug delivery to tumor cells[J]. Chembiochem, 2009, 10(5): 862-868. DOI:10.1002/cbic.200800805 |
| [63] |
Sefah K, Tang ZW, Shangguan DH, et al. Molecular recognition of acute myeloid leukemia using aptamers[J]. Leukemia Official Journal of the Leukemia Society of America Leukemia Research Fund U K, 2009, 23(2): 235-244. |
| [64] |
Bayrac AT, Sefah K, Parekh P, et al. In vitro selection of DNA aptamers to glioblastoma multiforme[J]. ACS Chemical Neuroscience, 2011, 2(3): 175-181. DOI:10.1021/cn100114k |
| [65] |
Chen H, Medley C, Sefah K, et al. Molecular recognition of small-cell lung cancer cells using aptamers[J]. Chemmedchem, 2008, 3(6): 991-1001. DOI:10.1002/cmdc.200800030 |
| [66] |
Zhao Z, Xu L, Shi X, et al. Recognition of subtype non-small cell lung cancer by DNA aptamers selected from living cells[J]. Analyst, 2009, 134(9): 1808-1814. DOI:10.1039/b904476k |
| [67] |
Van Simaeys D, López-Colón D, Sefah K, et al. Study of the molecular recognition of aptamers selected through ovarian cancer cell-SELEX[J]. PLoS One, 2010, 5(11): e13770. DOI:10.1371/journal.pone.0013770 |
| [68] |
He J, Wang J, Zhang N, et al. In vitro selection of DNA aptamers recognizing drug-resistant ovarian cancer by cell-SELEX[J]. Talanta, 2019, 194: 437-445. DOI:10.1016/j.talanta.2018.10.028 |
| [69] |
Wang L, Li P, Xiao X, et al. Generating lung-metastatic osteosarcoma targeting aptamers for in vivo and clinical tissue imaging[J]. Talanta, 2018, 188: 66-73. DOI:10.1016/j.talanta.2018.05.011 |
| [70] |
Raddatz MS, Dolf A, Endl E, et al. Enrichment of cell-targeting and population-specific aptamers by fluorescence-activated cell sorting[J]. Angewandte Chemie International Edition, 2008, 47(28): 5190-5193. DOI:10.1002/anie.200800216 |
| [71] |
Graham JC, Zarbl H. Use of cell-SELEX to generate DNA aptamers as molecular probes of HPV-associated cervical cancer cells[J]. PLoS One, 2012, 7(4): e36103. DOI:10.1371/journal.pone.0036103 |
| [72] |
Ohuchi SP, Ohtsu T, Nakamura Y. Selection of RNA aptamers against recombinant transforming growth factor-β type Ⅲ receptor displayed on cell surface[J]. Biochimie, 2006, 88(7): 897-904. DOI:10.1016/j.biochi.2006.02.004 |
| [73] |
Shangguan D, Meng L, Cao ZC, et al. Identification of liver cancer-specific aptamers using whole live cells[J]. Analytical Chemistry, 2008, 80(3): 721-728. |
| [74] |
Blank M, Weinschenk T, Priemer M, et al. Systematic evolution of a DNA aptamer binding to rat brain tumor microvessels. selective targeting of endothelial regulatory protein pigpen[J]. Journal of Biological Chemistry, 2001, 276(19): 16464-16468. DOI:10.1074/jbc.M100347200 |