病原菌引起的各种传染病是当今危害人类健康与生命安全最为严重的一类疾病[1-2]。病原菌传统培养检测法是一个金标准方法,但检测周期长、程序复杂[1-3]。同时,由于病原菌种类日益增多,且不少病原菌尚无可行的分离培养方法。而对于临床检验和环境污染应急检测来说,最为关键的要求之一是尽可能简化分析处理过程、尽快获得检测结果和减少所需样品量。传统培养法显然难以这样要求。随着分子生物学、免疫学等现代生物学的飞速发展,分子生物学技术及免疫分析技术被广泛应用于病原菌的检测,如PCR技术、酶联免疫分析技术等。理论上,这些方法可以检测几乎所有病原菌,并且可有效提高病原菌检测的时效性及灵敏度,但是这些分析技术仍然需要较长时间(> 2 h)、前处理过程繁杂、易受杂质干扰、所需的样品量较大及存在假阳/阴性结果等不足之处[1-4]。因此,发展适用于病原菌现场即时高灵敏、高特异性及快速经济的新技术已成为当前的研究热点和前沿[5]。
在融合现代光学、分子生物学和微流控等优势技术的基础上发展起来光学生物传感技术是病原菌现场快速检测领域最为耀眼的明珠[5]。光学生物传感器可大致分为比色生物传感器、基于表面等离子共振的生物传感器和基于激光诱导荧光的生物传感器。其中,基于激光诱导荧光的生物传感器已成为研究最为深入和应用最为广泛的一种光学生物传感器。基于夹心法原理,利用纳米信号增强技术,该类传感器检测细菌和病毒污染的灵敏度可达到10 CFU/mL[5-6]。但要实现光学生物传感器真正的实用化还存在几大挑战。第一个挑战是使用现有的生物传感器要实现病原菌检测的关键步骤需将生物识别分子(如抗体、核酸适体等)固定到传感元件上,以实现病原菌的特异性识别和捕获。但修饰过程会造成生物分子活性的降低,且由于位阻效应,生物分子的固定会影响其对病原菌的捕获,从而影响检测灵敏度[7]。另一方面,生物传感器的优势在于其可重复使用,但当固定抗体或核酸适配体等生物识别分子时,由于再生条件(强酸或强碱)苛刻,使得生物分子的活性快速下降,导致生物传感器再生次数非常有限(< 10次)。第二个挑战是如何简化病原菌检测方式以实现其现场快速分析。现有的病原菌生物传感器一般采用夹心法,这就使得检测过程需要进行多个反应步骤,增加了检测时间。同时,使用试剂种类多,造成检测成本比较高。更为重要的是,为提高检测灵敏度,通常需要使用过滤或免疫磁珠来对病原菌进行富集分离,这进一步延长了检测时间并使得检测过程复杂化。
为此,本研究提出一种新的倏逝波荧光生物传感分析策略来实现病原菌直接快速检测。根据我们前期的研究成果和经验,倏逝波光纤生物传感器是利用光在光纤进行全反射传播时,在光纤表面产生倏逝波,其强度随渗入深度呈指数衰减,有效深度在100 nm以内,当荧光标记的生物分子探针进入倏逝波有效范围内,荧光分子被激发光激发发出荧光,通过检测荧光强度来实现目标物的检测[8-9]。而病原菌(如大肠杆菌、沙门氏菌等)的尺寸约为几百nm到μm级。因此,我们提出基于倏逝波荧光原理及其与病原菌的尺寸效应来实现病原菌的直接快速检测。本研究以E.coli O157:H7为靶标物,结合特异性的核酸适配体来实现其直接快速检测。基本原理是当一定浓度荧光标记核酸适配体加入样品检测池时,倏逝波激发荧光分子发出荧光,利用课题组前期研发的倏逝波全光纤生物传感器即可实现荧光信号的定量检测。而当荧光标记的核酸适配体与E.coli O157:H7混合后加入样品检测池,由于部分核酸适配体结合到E.coli O157:H7表面,其标记荧光分子不能被激发,故导致检测荧光信号降低,利用荧光信号强度与E.coli O157:H7浓度的比例关系即可实现其定量检测。
1 材料与方法 1.1 材料 1.1.1 试剂、缓冲溶液及适配体等浓硫酸、过氧化氢、氢氟酸、氯化钾、磷酸二氢钾、磷酸氢二钠及盐酸等化学试剂均购于北京化学试剂公司,所有试剂均为分析纯或更高。十二烷基硫酸钠(SDS)购自Sigma-aldrich公司(上海,中国)。自配PBS缓冲溶液(pH 7.4,137 mmol/L NaCl+2.7 mmol/L KCl+4.3 mmol/L Na2HPO4+1.4 mmol/L KH2PO4)、Tris-HCl缓冲液、SDS溶液(0.5% SDS,pH 1.9)。
与大肠杆菌特异性结合的核酸适配体序列结构为5'- GCGGGAATAGGATGCGGCTGG AAGGAGAG-GTGTTGGTGGGTGGTG -3',其5'-端标记荧光分子Cy5.5,该核酸适配体由生工生物工程(上海)股份公司合成。E.coli O157:H7、发光细菌、沙门氏菌使用LB培养基培养。不同浓度的菌液由10 mmol/L PBS溶液稀释而成。
1.1.2 倏逝波荧光光纤生物传感器使用的倏逝波荧光光纤生物传感器如图 1所示。其原理是,激发光(635 nm,10 mW)经过单多模光纤耦合器进入光纤传感探头,在光纤传感探头内以全反射方式传播,并在探头表面形成倏逝波。当荧光分子进入倏逝波激发的有效范围内时,倏逝波激发荧光分子发出荧光,部分荧光耦合回光纤传感探头,由单多模光纤耦合器进行收集与传输,经滤光片滤除杂散光后被硅光电探测器进行检测。检测到的荧光信号值实时显示在用户界面上。

|
| 图 1 倏逝波荧光光纤生物传感器原理示意图 |
将5.5 cm长的石英光纤(数值孔径为0.22,南京春晖科技发展有限公司)去除2.5 cm长的涂覆层,将无涂覆层的光纤浸入50% HF溶液中进行腐蚀,光纤芯径腐蚀至225 μm即可。
1.2.2 病原菌的培养E.coli O157:H7的菌落用灼烧后冷却的接种环接种于1 mL LB液体培养基中,将其在置于37 ℃恒温水浴震荡12 h,然后将震荡好的菌液加入到100 mL LB液体培养基中,37 ℃水浴恒温震荡24 h。为保证实验的准确性,每次实验均使用新鲜菌液。
1.2.3 E.coli O157:H7的检测为获得E.coli O157:H7检测标准曲线,将500 nmol/L的E.coli O157:H7核酸适体分别与不同浓度的E.coli O157:H7混合,在25 ℃下预反应15 min,然后输入到样品检测池进行荧光信号检测。每个浓度进行3次平行实验。为绘制标准曲线,对不同浓度E.coli O157:H7所得到荧光信号检测值进行归一化处理,即:
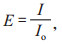 (1)
(1)
式中,I为加入不同浓度的E.coli O157:H7时,传感器检测到的荧光信号值;I0为未加E.coli O157:H7的空白样品荧光信号值。利用Logistics四参数模型进行标准曲线的拟合,即:
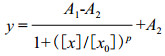 (2)
(2)
式中,[x]为E.coli O157:H7的浓度;y为x对应的荧光信号归一化值;A1为上端渐近线(x=0),常数;A2为下端渐近线(x→∞),常数;[x0]为曲线的拐点,常数;p为拐点处曲线的斜率,常数。根据Sinibaldi等[10]和Chiavaioli等[11],传感器的检测限一般定义为空白样检测的平均信号值加上其3倍标准偏差,依据该定义对该传感器的检测限进行计算。
1.2.4 实际水样的加标检测以校园景观水、自来水、污水处理厂出水为例进行加标分析,考察水体基质对传感分析性能的影响。由于实际样品中所含细菌种类多样,如果不进行灭菌操作,实验样品的各种细菌均可在LB培养基上生长,导致无法使用传统培养法进行比对。因此,首先运用高温灭菌锅对实际水样进行高温灭菌(121 ℃),以便考察实际样品对传感器的影响并于传统培养法进行比对。然后,将扩增培养的菌液使用离心机在10 000 r/min下离心8 min,清洗3次后,加入各种实际水样中,并配成3种不同浓度。各种加标水样与500 nmol/L荧光标记E.coli O157:H7核酸适配体混合,在25 ℃下预反应15 min,然后输入样品检测池进行检测。微生物培养计数法将各种加标水样分别稀释后,取100 μL稀释液在LB固体培养基上涂布,在37 ℃恒温培养24 h后计数。
2 结果 2.1 基于倏逝波荧光原理的E.coli O157:H7传感分析机制与验证基于倏逝波荧光原理的E.coli O157:H7传感分析机制如图 2所示。由菲涅尔公式可知,当光束由折射率n大的介质进入n小的介质时,若入射角θ大于临界角θc,入射光将全部反射回n大的介质中。在全反射的条件下,由于横向电场与横向磁场的Fresnel传递系数不为零,这意味着虽然光能全部被反射,但电磁场却可从两种介质的界面延伸至n小的介质中,即所谓的“倏逝波”,并遵循式(3)呈指数衰减[12]:
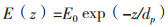 (3)
(3)

|
| 图 2 基于倏逝波荧光原理的E.coli O157:H7传感分析机制示意图 |
式中,z为距光纤探头界面的距离,E(z)为z处倏逝波振幅,E0为界面处电磁场振幅,dp为渗入深度。对于多模光纤探头,渗入深度dp(定义为电磁场为界面处电磁强度1/e的距离),为折射率、入射角和波长的函数:
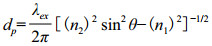 (4)
(4)
式中:λex是激发光的波长;θ是激发光与法线间夹角。本试验中激发光波长为635 nm,光纤探头折射率为1.456,PBS溶液的折射率为1.33,因此,倏逝波的有效深度约为100 nm。
当500 nmol/L荧光标记核酸适配体输入到样品检测池时,从图 3可以看出,倏逝波荧光光纤生物传感器检测到突然上升的荧光信号,这是由于倏逝波激发溶液中游离的核酸适配体标记荧光所致。由于倏逝波的强度有限,荧光漂白不明显,因此传感器检测荧光信号值保持基本不变。当500 nmol/L荧光标记核酸适配体与1×105 CFU/mL大肠杆菌混合反应一段时间后输入到样品检测池时,传感器检测到荧光信号也突然上升,但是荧光信号值明显低于未加入大肠杆菌时的荧光信号值。这说明部分核酸适配体特异性结合到大肠杆菌表面,由于大肠杆菌的尺寸在微米级,倏逝波很难激发结合到大肠杆菌表面的荧光标记核酸适配体,从而导致检测到的荧光信号值下降。为进一步验证该结论,我们将500 nmol/L荧光标记的大肠杆菌核酸适配体分别与1×109 CFU/mL沙门氏菌和发光细菌混合并输入到样品检测池,从图 3可以看出,虽然沙门氏菌和发光细菌的浓度远远高于大肠杆菌的浓度,但是传感器检测到的荧光信号值并没有明显下降。这一方面说明大肠杆菌核酸适配体具有良好的选择性,不会与其他病原菌结合,同时证明本研究提出的基于倏逝波荧光原理和及其与病原菌尺寸效应的检测机制是可行的,可以用于E.coli O157:H7的直接快速检测。

|
|
E.coli O157:H7的浓度1×105 CFU/mL;沙门氏菌和发光细菌的浓度均为1×109 CFU/mL;核酸适配体浓度为500 nmol/L A:传感分析验证的信号响应;B:传感分析验证的荧光信号曲线 图 3 E.coli O157:H7传感分析验证 |
为获得基于倏逝波荧光原理的E.coli O157:H7生物传感分析标准检测曲线,配制不同浓度的E.coli O157:H7溶液,分别与500 nmol/L的荧光标记核酸适配体混合,反应15 min后输入到样品检测池进行荧光信号值检测,得到系列荧光信号检测曲线(图 4)。从图 4可以看出,倏逝波荧光光纤传感器检测到的荧光信号值随E.coli O157:H7浓度的升高而下降。这是由于随着E.coli O157:H7浓度的增加,更多的荧光标记核酸适配体结合E.coli O157:H7表面。因E.coli O157:H7的尺寸在微米级,而倏逝波的渗入深度有限,部分标记在核酸适配体上荧光分子不能被激发,从而导致荧光信号值的下降。各个浓度E.coli O157:H7重复测试3次,荧光信号的标准偏差都少于5%,表明该方法具有良好的稳定性。

|
| E.coli O157:H7的浓度分别为0、1×102、1×103、1×104、1×105、1×106、1×107、1×108、1×109 CFU/mL 图 4 不同浓度E.coli O157:H7的典型荧光信号曲线 |
将不同浓度下的荧光信号值根据式1进行归一化处理,所得归一化值按式2拟合,得到E.coli O157:H7的检测标准曲线(图 5)。通过计算可以得出,E.coli O157:H7检测范围为1.1×103-1.4×107 CFU/mL,检测限[10-11]为610 CFU/mL。以上结果表明该方法具有较好的灵敏度和稳定性。

|
| 图 5 E.coli O157:H7检测标准曲线 |
为验证该方法在实际水样中运用的可行性,考察水体基质对传感分析性能的影响,利用该方法分析了校园景观水、自来水和污水处理厂出水等实际水样的加标回收率并与传统培养法进行了比对。在检测前,先用高温灭菌锅对实际水样进行高温灭菌(121 ℃)。然后,将培养的E.coli O157:H7加入各种实际水样中。各种加标水样与500 nmol/L荧光标记E.coli O157:H7核酸适配体混合,在25 ℃下预反应15 min,然后输入样品检测池进行检测。从表 1可以看出,E.coli O157:H7的回收率在40%-180%之间,相对标准偏差在10%之内。与传统的培养法具有较好的一致性,这些结果表明本研究建立的基于倏逝波荧光原理的E.coli O157:H7生物传感分析方法可以用于实际水样的检测。
E.coli O157:H7是一种肠出血性大肠杆菌,可经食物和饮用水在人群中广泛传播,可导致腹痛、腹泻、急性肾衰,甚至死亡。美国和欧盟曾发生多次E.coli O157:H7的爆发性流行[13]。因此,多国卫生部门已将E.coli O157:H7列为常规检测项目,我国生活饮用水卫生标准中要求不得检出[14-15]。因此,发展高特异性、操作简单、检测迅速及成本低廉的病原菌检测方法具有重要的科学价值和现实意义。
本研究结合倏逝波荧光光纤传感器和核酸适配体的优势,提出了基于倏逝波荧光原理及其与病原菌尺寸效应的E.coli O157:H7直接快速检测方法。该方法可以避免传统生物传感器需要固定生物识别分子造成的诸多不足,如生物识别分子在固定过程中可能导致活性损失等。由于本研究采用的是将特异性核酸适配体与E.coli O157:H7在均相中直接反应,因此,可以有效提高生物识别分子与病原菌的亲和反应效率。这种检测模式无需苛刻的再生条件,仅需在检测完成后,将样品排出样品池即可进行下一个样品的检测。同时,传统生物传感器常采用夹心法方式来实现病原菌的检测,即采用一种生物识别分子(抗体或核酸适配体等)固定在生物传感器表面捕获病原菌,然后用另一种荧光标记生物功能分子结合到病原菌表面作为报告分子[16-19]。本研究提出的方法仅用一种荧光标记核酸适配体即可实现E.coli O157:H7快速识别与定量检测,这可以大大简化病原菌检测程序,减少检测时间和反应试剂的使用,有效降低检测成本。更进一步,本研究提出的检测方法仅需更换荧光标记的生物识别分子即可实现其他不同的病原菌的检测。因此,基于倏逝波荧光原理及其与病原菌尺寸效应的生物传感分析技术具有广泛的适用性和实用性。
4 结论融合倏逝波荧光光纤传感器和特异性核酸适配体的优势,提出一种了基于倏逝波荧光原理及其与病原菌尺寸效应的病原菌新型检测方法。研究表明该方法可实现E.coli O157:H7的直接快速检测,检测限可达610 CFU/mL,线性检测区间为1.1×103-1.4×107 CFU/mL。由于使用特异性核酸适配体与E.coli O157:H7在均相溶液中结合,利用倏逝波荧光原理及尺寸效应来实现其快速检测,无需固定生物识别分子,也无需采用夹心法的检测模式。因此,该方法可有效缩短检测时间(< 30 min),减少使用的反应试剂和检测成本。对实际水样进行了加标回收实验,结果表明加标回收率在40%-180%之间,相对标准偏差在10%之内,水样基质对该方法检测E.coli O157:H7没有明显影响。因此,本研究建立基于倏逝波荧光原理及其与病原菌尺寸效应的生物传感分析方法可以用于实际水样的检测,同时,仅需更换荧光标记的生物识别分子即可实现其他不同的病原菌的检测。
| [1] |
Fournier PE, Drancourt M, Colson P, et al. Modern clinical microbiology:new challenges and solutions[J]. Nature Reviews Microbiology, 2013, 11: 574-585. DOI:10.1038/nrmicro3068 |
| [2] |
Yoo SM, Choi JY, Yun JK, et al. DNA microarray-based identifica-tion of bacterial and fungal pathogens in bloodstream infections[J]. Molecular and Cellular Probes, 2010, 24: 44-52. DOI:10.1016/j.mcp.2009.09.003 |
| [3] |
Joung CK, Kim HN, Lim MC, et al. A nanoporous membrane-based impedimetric immunosensor for label-free detection of pathogenic bacteria in whole milk[J]. Biosensors and Bioelectronics, 2013, 44: 210-215. DOI:10.1016/j.bios.2013.01.024 |
| [4] |
Flekna G, Schneeweiss W, Smulders FJM, et al. Real-time PCR method with statistical analysis to compare the potential of DNA isolation methods to remove PCR inhibitors from samples for diagnostic PCR[J]. Molecular and Cellular Probes, 2007, 21(4): 282-287. DOI:10.1016/j.mcp.2007.02.001 |
| [5] |
Yoo SM, Lee SY. Optical biosensors for the detection of pathogenic microorganisms[J]. Trends in Biotechnology, 2015, 34(1): 7-25. |
| [6] |
Queirós, R B, Gouveia C, Fernandes JRA, et al. Evanescent wave DNA-aptamer biosensor based on long period gratings for the specific recognition of E. coli outer membrane proteins[J]. Biosensors and Bioelectronics, 2014, 62: 227-233. DOI:10.1016/j.bios.2014.06.062 |
| [7] |
Justino CIL, Freitas AC, Pereira R, et al. Recent developments in recognition elements for chemical sensors and biosensors[J]. Trends in Analytical Chemistry, 2015, 68: 2-17. DOI:10.1016/j.trac.2015.03.006 |
| [8] |
Taitt CR, Anderson GP, Ligler FS. Evanescent wave fluorescence biosensors:Advances of the last decade[J]. Biosensors and Bioelectronics, 2015, 76: 103. |
| [9] |
Long F, He M, Shi HC, et al. Development of evanescent wave all-fiber immunosensor for environmental water analysis[J]. Biosensors and Bioelectronics, 2008, 23(7): 952-958. DOI:10.1016/j.bios.2007.09.013 |
| [10] |
Sinibaldi A, Montaño-Machado V, Danz N, et al. Real-time study of the adsorption and grafting process of biomolecules by means of bloch surface wave biosensors[J]. ACS Applied Materials & Interfaces, 2018, 10(39): 33611-33618. |
| [11] |
Chiavaioli F, Zubiate P, Villar ID, et al. Femtomolar detection by nanocoated fiber label-free biosensors[J]. ACS Sensors, 2018, 3: 936-943. DOI:10.1021/acssensors.7b00918 |
| [12] |
Harrick NJ. Internal reflection spectroscopy[M]. New York: Wiley, 1967: 8-25.
|
| [13] |
Huang X, Lu LL, Deng XL, et al. A notice from epidemiological investigation of Shiga-toxin-producing Escherichia coli O104:H4 outbreak in Germany, 2011[J]. Zhonghua Liuxingbingxue Zazhi, 2012, 33(1): 111-114. |
| [14] |
Microbial risk assessment guideline, pathogenic microorganisms with focus on food and water[M]. Washington: United States Department of Agriculture, 2017. EPA.
|
| [15] |
中华人民共和国国务院令(第424号). 病原微生物实验室生物安全管理条例[M]. 北京: 中华人民共和国国务院办公厅, 2004.
|
| [16] |
Shih CM, Chang CL, Hsu MY, et al. Paper-based ELISA to rapidly detect Escherichia coli.[J]. Talanta, 2015, 145: 2-5. DOI:10.1016/j.talanta.2015.07.051 |
| [17] |
Luciani M, Armillotta G, Magliulo M, et al. Production and characterisation of monoclonal antibodies specific for Escherichia coli O157:H7[J]. Veterinaria Italiana, 2006, 42(3): 183-191. |
| [18] |
Donhauser SC, Niessner R, Seidel M. Sensitive quantification of Escherichia coli O157:H7, Salmonella enterica, and Campylobacter jejuni by combining stopped polymerase chain reaction with chemiluminescence flow-through DNA microarray analysis[J]. Analytical Chemistry, 2011, 83(8): 3153-3160. DOI:10.1021/ac2002214 |
| [19] |
Donhauser SC, Niessner R, Seidel M. Quantification of E. coli DNA on a flow-through chemiluminescence microarray readout system after PCR amplification[J]. Analytical Sciences the International Journal of the Japan Society for Analytical Chemistry, 2009, 25(5): 669. DOI:10.2116/analsci.25.669 |





