MicroRNA(miRNA)是一类真核生物中广泛存在的约20-24个核苷酸(Nucleotide,nt)长度的内源非编码单链RNA,成熟的miRNA与Argonaute(AGO)家族蛋白结合形成RNA诱导的沉默复合体(RNA-induced silencing complex,RISC),在转录后水平负调控基因表达。miRNA在生物的细胞增殖分化、生长发育和响应外界环境信号等多种生物过程中发挥重要作用。1993年和2000年,Ambros与Reinhart的研究小组分别先后在秀丽隐杆线虫(Caenorhabditis elegans)中鉴定了最早的动物miRNA,lin-4和let-7[1-2]。2002年Bartel、Carrington与Chen分别带领各自的研究小组发现了植物拟南芥中的miRNA[3-5]。几十年来,科学家已经在动植物中发现了数以万计的miRNA,2018年10月miRBase收录了38 589条miRNA前体信息,这些前体共计产生48 860条不同的成熟miRNA;其中人类基因组中鉴定发现2 654个成熟miRNA,拟南芥中发现428个成熟miRNA[6]。有关miRNA产生的生物学过程、功能分析、逆境胁迫等方面的研究报道逐年攀升,miRNA已然成为科学研究领域炙手可热的“明星分子”。
2002年,研究者们发现植物中第一个miRNA,“miR171”,同时发现植物miRNA与靶基因序列高度互补配对的特性[3-5]。在此之前的1999年,植物中首次发现了小干扰RNA(small interfering RNA,siRNA)。siRNA与靶基因完全互补配对,通过剪切靶基因mRNA调控基因的表达[7]。基于siRNA作用机制的启示,Llave[4]发现miR171也是通过剪切mRNA沉默靶基因。有趣的是,miR171既是植物miRNA作用方式的第一个实例,也是植物中少数与靶基因完全互补配对的miRNA之一。在miRNA被陆续发现和报道的前期,通过定量PCR检测mRNA水平的实验技术非常成熟,对靶基因mRNA水平的检测成为早期主流手段,因此植物miRNA对靶基因mRNA剪切的现象被大量报道。然而,蛋白水平的检测需要借助特异性强且灵敏度高的抗体,植物蛋白抗体开发的不足大大限制了对miRNA靶基因蛋白水平的检测。以上因素综合作用造成了长期以来该领域的观点认为,植物miRNA主要通过剪切靶基因mRNA发挥作用。随后开始有报道发现有的植物miRNA并不影响靶基因mRNA的水平,而是降低靶基因蛋白的水平[8-10],由此提出植物miRNA也存在与动物miRNA类似的抑制蛋白翻译的作用机制。Brodersen等[11]发现了拟南芥中多个miRNA可以介导翻译抑制,并且通过突变体筛选报道了数个影响miRNA翻译抑制的基因,为植物miRNA翻译抑制机制的广泛存在提供了遗传学的证据支持。近些年的研究报道进一步揭示了植物miRNA翻译抑制的普遍性。迄今为止,植物miRNA主要通过剪切mRNA和翻译抑制两种方式对靶基因进行负调控,已经成为该领域最新的共同认知。
自植物miRNA首次被报道以来,近十几年的研究已经对miRNA的生物学发生与降解,及其参与的生物学过程等方面作了清晰的阐述,也有不少文献针对上述内容进行了很好的总结[12-16]。相比在动物中的研究,miRNA在植物中作用方式的分子机制的报道相对匮乏和滞后。对植物miRNA-RISC复合体核心效应因子AGO1的功能解析,结合mRNA降解通路的基因功能研究,阐明了miRNA对靶基因的切割以及随后的mRNA降解机制;相比之下,在翻译抑制方面,尽管发现了一系列影响miRNA翻译抑制的基因,但是这些基因影响这一过程的分子机制仍然不够清晰。本文将回顾和概述植物miRNA作用机制的发现过程与最新的研究进展,系统地总结植物miRNA介导的mRNA剪切和翻译抑制这两种主要作用方式的发生、产生的结果(靶基因mRNA降解、次生小干扰RNA发生)和影响因素等方面的研究进展。本文也将讨论miRNA发生作用的亚细胞场所,以及不同作用机制的联系和区别。综合以上信息,文末将对该领域内未来的研究方向进行展望,同时提出一些亟待解决的问题并进行讨论。
1 miRNA介导的靶基因mRNA剪切动物miRNA与靶基因匹配程度不高(配对仅限于种子序列区域(Seed region)),因此无法直接剪切mRNA[17]。动物miRNA通过RISC复合体招募去腺苷化酶和脱帽蛋白对mRNA进行降解[18]。相比之下,植物miRNA与靶基因的互补配对程度远大于动物miRNA,大部分只存在4个碱基以下的错配,主要通过对靶基因mRNA的剪切发挥沉默效应,作用方式与siRNA类似。迄今为止,既没有生化证据表明在植物中存在与动物中类似的,不依赖剪切的mRNA降解机制[19],在AGO1剪切活性缺陷突变体的转录组数据中也未检测到这一类机制的明确存在[20]。由于实验手段的限制,早期仅能通过Northern Blot和5′cDNA末端快速扩增技术(Rapid amplification of cDNA ends,RACE)鉴定和克隆到少数miRNA剪切的靶基因片段;mRNA被miRNA切割后的3′端片段暴露出5′单磷酸基团,该结构能够被连接酶捕获并添加接头构建得到降解组文库。得益于近些年测序技术的不断发展,对3′切割产物(降解组)进行高通量测序和分析,German等[21]发现大部分拟南芥miRNA都存在对靶基因mRNA进行剪切的作用方式。
植物miRNA主要通过AGO蛋白的核酸内切酶活性发挥作用。AGO蛋白是一类进化上非常保守的蛋白,广泛存在于细菌、古生菌、真菌、植物和动物中[22]。拟南芥AGO蛋白家族有10个成员(AGO1-10),其中AGO1结合绝大多数的miRNA发挥作用[23-24],此外AGO2、AGO4、AGO7和AGO10也可以结合部分miRNA发挥剪切功能[25-30]。成熟的miRNA与AGO蛋白组装成miRNA-RISC复合体,miRNA引导及识别与自身核苷酸互补配对的靶基因mRNA并与之结合;AGO1蛋白保守的PIWI结构域具有类似RNA酶H催化中心的三级结构[31],特异性地在miRNA第10-11位对应的靶基因mRNA位置切断核苷酸之间的磷酸二酯键[32],产生5′端包含帽子结构和3′端带有polyA尾的两段切割产物。
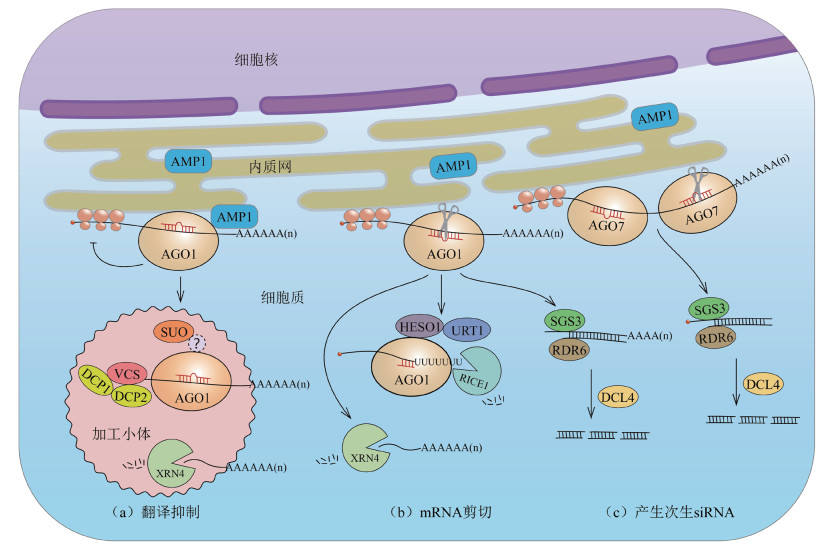
经miRNA剪切后的mRNA 5′和3′端片段进入核酸外切酶降解途径。Northern blot检测结果发现3′切割产物更容易被检测到,意味着5′切割产物更容易被降解,而3′切割产物更稳定[4]。在拟南芥中,催化降解3′切割产物的是一类5′-3′的外切酶EXORIBONUCLEASE 4(XRN4)[33](图 1-b,mRNA剪切)。miRNA 5′切割产物的降解步骤相对复杂,拟南芥的核酸转移酶HEN1 SUPPRESSOR 1(HESO1)以及其同源蛋白RNA URIDYLYLTRANSFERASE 1(URT1)对其3′末端进行尿苷化修饰能够加速5′切割产物的降解[35-36](图 1-b,mRNA剪切)。在莱茵衣藻中,5′切割产物的3′末端被HESO1同源蛋白核酸转移酶MUT68腺苷化[34]。最近的研究发现,拟南芥RICE1(RISC-INTERACTING CLEARING 3′-5′ EXORIBONUCLEASE 1)在尿苷化的5′切割产物随后的降解过程中发挥重要作用[37](图 1-b,mRNA剪切)。RICE1是AGO1蛋白的辅因子,结构上与3′-5′外切酶DnaQ家族类似,RICE1催化活性的缺失导致尿苷化的5′切割产物在突变体内过度积累[37]。此外,在xrn4突变体中也报道了5′切割产物的异常积累,表明XRN4也参与了5′切割产物的降解[35]。早期对细胞质外切体(Exosome)相关基因突变体csl4和rrpl6的研究未发现明显的5′切割产物的积累[35],认为外切体不参与该降解步骤。直到2015年,Branscheid等[38]证实了组成外切体复合体的其他亚基突变体中5′切割产物存在积累的情况,表明该过程需要外切体辅因子的亚基SUPERKILLER2(SKI2),SKI3和SKI8的参与。因此,植物miRNA切割后产生的5′切割产物与果蝇和衣藻中siRNA切割后5′端片段的降解过程类似,均需要外切体的参与,外切体在这个过程中的作用机制是保守的[34, 39]。
2 miRNA介导的翻译抑制 2.1 植物中广泛存在miRNA介导的翻译抑制1993年在C. elegans中报道了第一个鉴定到的miRNA“lin-4”,它的靶基因lin-14的mRNA无明显变化,而蛋白水平下降[1]。基于以上发现,提出miRNA抑制蛋白翻译过程这一作用机理,这是第一次提出“翻译抑制”的概念。由于植物中第一例关于作用机制的报道中miR171是通过剪切靶基因mRNA发挥作用,以及植物蛋白抗体的匮乏,在研究早期,植物miRNA的主要作用方式被认为与siRNA一样通过剪切mRNA实现。植物中被大家所熟知的首例miRNA介导的翻译抑制发生在花序组织中:拟南芥miR172使参与花器官发育调控的靶基因APETALA 2(AP2)的蛋白水平下降,而mRNA水平无明显变化[8-9]。植物中第二个发现的介导翻译抑制的miRNA参与开花时间的调控:miR156/157识别和结合靶基因SQUAMOSA PROMOTER BINDING PROTEIN-LIKE 3(SPL3),通过翻译抑制引起基因沉默,抑制植物早花现象[10]。Brodersen等[11]通过正向遗传学方法筛选影响miRNA作用机制突变体时发现,包括miR171、miR395、miR398和miR834在内的miRNA均可以降低其相应靶基因SCARECROW-LIKE PROTEIN 4(SCL4),ATP SULFURYLASE 1(APS1),COPPER/ZINC SUPEROXIDE DISMUTASE 2(CSD2),COP1-INTERACTING PROTEIN 4(CIP4)的蛋白水平而mRNA水平未出现明显变化,表明翻译抑制是植物中广泛存在的一种机制;该报道不仅发现了影响miRNA剪切靶基因mRNA的突变体,还鉴定到了一系列影响miRNA翻译抑制的突变体(ktn1、vcs、ago1、ago10,后续章节具体阐述),为翻译抑制在植物体内的发生奠定了遗传学基础。2010年,Alonso-Peral等[40]通过比较miR159靶基因MYB DOMAIN PROTEIN 33(MYB33)的转录本和蛋白的相对丰度,发现miR159也能介导翻译抑制。
尽管通过比较靶基因mRNA和靶蛋白稳定状态相对丰度的方法发现了许多植物miRNA介导翻译抑制的例子,然而直到2013年还没有直接的证据证明miRNA-RISC复合体可以抑制靶蛋白的翻译过程。2013年Li等[41]通过放射性同位素标记追踪miRNA靶基因蛋白合成速率的变化,证实miR398和miR165/166抑制其相应靶基因CSD2和PHABULOSA(PHB)的蛋白合成,且新生成的蛋白量降低这一过程依赖ALTERED MERISTEM PROGRAM1(AMP1)。Liu等[42]通过核糖体组学分析发现miRNA靶基因的编码区上附着的核糖体密度降低,miRNA靶基因在核糖体上的翻译效率显著低于非靶基因,而靶基因与非靶基因的稳态mRNA水平并没有明显差异。在影响翻译抑制的DOUBLE-STRANDED RNA-BINDING 2(DRB2)基因的报道中指出,DRB2及其同源基因在苔藓和早期基部被子植物中已经存在,且氨基酸的保守程度非常高,暗示了其功能的保守性,以及在远古的植物中可能已经存在DRB2介导的翻译抑制机制[43]。综上所述,越来越多的证据支持miRNA介导的翻译抑制在植物中的普遍存在。
2.2 影响miRNA翻译抑制的基因动物miRNA主要通过翻译抑制发挥作用,具体机制得到了比较深入的研究。动物miRNA组装成RISC复合体之后,通过miRNA第2-8位的“种子序列”与靶基因mRNA 3′非编码区(Untranslated region,UTR)互补配对。动物的miRNA-RISC复合体包括AGO2蛋白和支架蛋白GW182。GW182是miRNA发挥翻译抑制作用的重要桥梁因子,GW182不仅可以与AGO2蛋白相互作用,也可以与mRNA去稳定化及翻译过程的众多因子相互作用[18]。动物实验研究表明,GW182介导的翻译抑制机制既包括使polyA结合蛋白(Poly A binding protein,PABP)从mRNA上脱落[44],也包含招募翻译阻遏物的参与[18]。然而植物中不存在GW182的同源蛋白[45],暗示了植物翻译抑制机制与动物的差异。对植物AGO1相关蛋白的研究发现了一系列促进RISC复合体组装、miRNA装载的基因,例如HEAT SHOCK PROTEIN 90(HSP90)[46],CYCLOPHILIN-40(CYP40)[47]和TRANSPORTIN1(TRN1)[48]等,遗憾的是,并没有直接线索显示这些与AGO1相关的蛋白与翻译抑制机制相关。
近年来陆续报道了为数不多的影响植物miRNA翻译抑制的基因。2008年,Brodersen等[11]通过正向遗传筛选突变体的方法,发现微管切割酶KATANIN 1(KTN1)基因的突变体中miRNA靶蛋白的水平明显升高而mRNA水平变化不大。KTN1编码微管切割酶中的P60亚基,暗示细胞微管可能参与翻译抑制[11]。Brodersen等[11]还发现加工小体(the processing body,P body)的组分VARICOSE(VCS)影响miRNA介导的翻译抑制。无独有偶,2012年Yang等[49]发现另一个加工小体的组分SUO基因的突变体与AGO1活性降低的突变表型类似,进一步的研究发现该基因的突变导致miRNA翻译抑制功能受到影响。加工小体是细胞质中一类由核糖核蛋白聚集形成的亚细胞结构,目前其已知的组分包含脱帽复合体的成员VCS、DECAPPING1(DCP1)和DECAPPING2(DCP2)以及AGO1[50]、XRN4[51]和SUO[49]。VCS是脱帽复合物中的一种蛋白,其外切酶活性以5′-3′的方向降解mRNA[11]。SUO基因编码的蛋白在C端有两个GW(Glycine and tryptophan)重复序列[49],从蛋白结构猜测SUO有可能是动物GW182在植物中特异存在的发挥类似作用的等价基因,但尚无证据表明SUO直接与AGO1蛋白结合。从AGO1、XRN4与加工小体的相关性可以推测VCS和SUO可能通过在加工小体中对一些需要被翻译抑制调控的基因进行加工或者降解来参与miRNA介导的翻译抑制[52](图 1-a,翻译抑制)。此外,Brodersen等[11]根据AGO家族中各个AGO蛋白与miRNA已知的联系,结合突变体的表型,验证发现AGO1和AGO10也参与植物miRNA介导的翻译抑制。
2013年,Li等[41]报道了一种内质网膜整合蛋白ALTERED MERISTEM PROGRAM 1(AMP1)及其同源蛋白LIKE AMP1(LAMP1)参与植物miRNA介导的翻译抑制,这两个蛋白均可以与AGO1相互作用并且与AGO1共定位在细胞内质网上(图 1-a,翻译抑制)。利用同位素标记检测新合成蛋白量的实验表明,在半衰期内一定时间中,amp1突变体中miRNA对靶基因蛋白生成量的抑制能力降低,表明靶基因的翻译抑制依赖AMP1的作用[41]。相比野生型,miRNA靶基因mRNA在amp1突变体的膜结合多聚核糖体中发生显著富集,以上证据表明AMP1通过抑制mRNA的翻译起始发挥功能[41]。双链RNA结合蛋白DRB是植物中一类保守的蛋白,拟南芥中包含5个家族成员,DRB1-5,其中最被熟知的DRB1(即HYL1)参与miRNA的生物合成。有趣的是,Reis等[43]对野生型和drb2突变体材料同时进行转录组和蛋白组学分析时,发现蛋白组学分析鉴定到的miRNA靶蛋白水平升高而对应的mRNA水平与野生型无异,意味着DRB2参与翻译抑制过程,然而作者并未对DRB2影响翻译抑制的分子机制展开挖掘和论述。
诸多报道发现了一系列仅在蛋白水平影响miRNA靶基因的突变体[11, 41, 49];2009和2013年两篇文献均报道AGO1的剪切活性被抑制时翻译抑制所受影响不显著[19, 53];利用体外组装的RISC复合体检测发现在miRNA-靶基因剪切位点附近(中心配对区)引入错配,依然能发生显著水平的翻译抑制[19]。以上证据表明miRNA介导的剪切和翻译抑制是两种相互独立的机制。目前已知的影响翻译抑制基因的报道并未阐明这一调控方式具体的分子机制。最新的一项以莱茵衣藻为对象的研究发现,VASA INTRONIC GENE 1(VIG1)基因编码的蛋白能够与AGO蛋白相互作用,是RISC复合体的组分之一,也与多聚核糖体组分相关联,影响翻译抑制,并可能调节蛋白质合成的多个步骤[54]。衣藻VIG1在酵母、果蝇和哺乳动物中的同源基因与核糖体小亚基蛋白RACK1(RECEPTOR FOR ACTIVATED PROTEIN C KINASE 1)及影响翻译延伸的蛋白相关,并且影响翻译抑制[55-56]。拟南芥中,RACK1通过与miRNA加工复合体蛋白SERRATE(SE)相互作用影响miRNA前体的加工;此外免疫共沉淀实验和蛋白共定位结果表明RACK1是AGO1复合体的组分。线虫rack1突变体中miRNA表达量升高,然而利用RNA干扰技术敲除线虫的RACK1基因导致miRNA靶基因蛋白水平的升高,表明动物的RACK1参与翻译抑制的过程[57]。在拟南芥中,由于rack1突变体中多数miRNA产生减少,靶蛋白水平的升高不能简单归因于RACK1影响miRNA介导的翻译抑制,这使得RACK1与翻译抑制关系的探究变得富有挑战性[58]。VIG1在拟南芥中有3个同源基因(AT4G16830、AT4G17520、AT5G47210)尚未有这3个基因与miRNA作用机制相关的报道[54]。
2.3 影响mRNA剪切或翻译抑制的因素植物miRNA与动物miRNA的主要差异有两点:第一,与靶基因互补配对程度不同,植物中互补配对程度远高于动物中依赖种子序列的配对;第二,miRNA结合靶基因位置不同,植物miRNA主要结合靶基因编码区,动物主要结合3′ UTR。小鼠miR196除了一个G-U错配,基本与靶基因HOXB8完全互补配对,是动物中为数不多的执行mRNA剪切的miRNA之一[59]。植物中首例解析的miRNA作用机制也是与靶基因完全互补配对并且剪切mRNA[4]。因此早期观点认为近乎完全的互补配对是发生靶基因剪切的分子基础。动物miRNA结合在靶基因的3′ UTR并且翻译抑制是其主要的作用机制[8-10];植物miR156/157是最早发现的翻译抑制的例子之一,它与靶基因SPL3的3′ UTR区结合,暗示结合位点可能也是影响作用机制的重要因素。
早期Brodersen等[11]对结合位点和互补程度各不相同的一系列miRNA靶基因mRNA和蛋白水平分析发现没有明显的规律,提出靶基因剪切或者翻译抑制与mRNA靶位点在基因上的位置(5′ UTR、编码区或3′ UTR)和互补配对程度无关。动物中的研究也发现动物miRNA无论在编码区还是3′ UTR都可以实现翻译抑制[60]。另一方面,植物miRNA与靶基因几乎完全互补配对,而翻译抑制广泛存在,说明互补配对程度并不影响miRNA沉默机制的选择,Brodersen等[11]通过人为引入碱基错配也已经加以证明。衣藻中miRNA报告基因的研究发现miRNA与靶基因mRNA完全互补配对仍然可以引起翻译抑制[61]。利用农杆菌侵染在烟草细胞中瞬时表达miRNA及其靶标的报告基因,研究者们发现模拟动物中以种子序列与靶基因的配对形式,无论将结合位点设计在靶基因的UTR区还是基因编码区,改造后的miRNA都不能发挥剪切或是翻译抑制作用[62]。将纯化的植物AGO蛋白与人工合成的miRNA在体外反应,能够组装成有生物活性的RISC复合体并发挥基因沉默作用[46]。2013年的一项研究基于上述体外系统对翻译抑制的分子机制进行了解析[19]。通过在人工miRNA中引入错配碱基并且检测沉默效应,研究发现植物miRNA与动物相比确实需要更高的碱基配对程度才可以实现翻译抑制;同时与衣藻中的发现类似,当植物miRNA与靶基因的完全互补配对发生在3′ UTR时,也能够实现翻译抑制[19]。综上所述,miRNA的结合位点和与靶基因互补的情况并不是影响作用机制选择的重要因素,真正起决定作用的因素仍有待进一步研究。
2.4 植物miRNA翻译抑制的分子机制初探利用突变体和各种报告系统对miRNA作用方式分子机制的研究没有得到明确的结论,一些体外的生化试验为植物miRNA翻译抑制分子机制的研究提供了一些线索。以荧光素酶作为报告基因,设计人工干扰miRNA使RISC复合体的结合位点位于编码区时发现,miRNA的翻译抑制作用引起全长的靶蛋白水平降低;有趣的是,与miRNA结合位点之前的编码区区域对应的截短蛋白出现明显的积累,然而在报告基因5′ UTR设计的miRNA没有检测到截短蛋白的积累,表明RISC复合体结合在编码区时能够在空间上阻碍靶基因上正在翻译的核糖体延伸[19]。体外实验利用AGO1剪切活性位点突变的RISC复合体(翻译抑制功能正常)与靶基因孵育后进行蔗糖密度梯度组分分析发现,完整的80S核糖体明显减少,说明RISC复合体通过干扰核糖体组装影响翻译起始过程[19]。AMP1是翻译抑制必需的,而amp1突变体中的靶基因在活跃翻译的核糖体上的丰度高于野生型,说明AMP1的存在阻止靶基因mRNA进入翻译机器,表明翻译抑制是通过影响翻译起始实现。此外,利用人工合成miRNA的报告株系检测miRNA沉默效率探索发现,当miRNA与靶基因结合位点位于5′端起始密码子前200碱基对范围时能更加有效的实现翻译抑制,也暗示了miRNA通过抑制翻译起始过程发挥作用[63]。
核糖体印迹测序技术又称ribo-seq(ribosome-sequencing),是近些年新兴的一种翻译组学研究手段。利用RNA酶消化降解没有被核糖体覆盖的mRNA片段后,对被核糖体保护的RNA小片段添加接头并进行测序;可以得到体内转录本上核糖体的分布和密度等信息,从而对翻译过程进行直接研究。利用ribo-seq检测植物体内miRNA对靶基因上核糖体结合情况的影响,能够提供更贴近体内翻译抑制作用机制的线索。当RISC复合体与靶基因的结合导致核糖体的延伸受阻时,在靶基因mRNA结合位点前后可能会出现核糖体的停滞或者堆积,然而在衣藻和拟南芥的ribo-seq数据中,均没有发现剪切位点附近核糖体密度的明显变化[42, 54, 64]。这一发现也更加支持miRNA在植物体内通过影响翻译起始实现翻译抑制的分子机制。
3 miRNA剪切的另一种结果:产生次生siRNA 3.1 phasiRNA与miRNA剪切被miRNA剪切的mRNA除了进入核酸外切酶降解途径以外,还有可能产生phasiRNA。phasiRNA的产生是植物中广泛存在的一种保守机制,即miRNA对一类特殊的靶基因转录本的剪切可以触发产生具有一定相位(Phase)的siRNA,称为phasiRNA(Phased siRNA)。phasiRNA在拟南芥中的数目相对较少[65],近些年伴随着植物基因组数据的不断披露,研究人员发现在玉米、水稻、短柄草等单子叶植物中存在大量的phasiRNA,这些物种的生殖组织中存在的数量尤为丰富[66-68]。通常来说,phasiRNA的生成由22 nt的miRNA触发,与绝大多数编码蛋白的靶基因不同的是,miRNA-RISC复合体切割PHAS转录本,产生的5′和3′片段会被SUPPRESSOR OF GENE SILENCING 3(SGS3)捕获并稳定(图 1-c,产生次生siRNA),阻止了切割产物进入核酸外切酶的降解途径;随后RNA-DEPENDENT RNA POLYMERASE 6(RDR6)将上述剪切片段复制成双链RNA,之后DICER LIKE 4(DCL4)从头开始以21碱基为单位连续剪切双链RNA,形成具有一定相位的,头尾相接的21 nt双链siRNA[69](图 1-c,产生次生siRNA)。成熟的phasiRNA与AGO1组装成沉默复合体,通过碱基互补配对作用于产生phasiRNA的转录本自身或者其同源基因,在转录后水平发挥负调控作用。其中一类特殊的phasiRNA来自一类长链非编码RNA(long noncoding RNA,lncRNA)的转录本“TAS”,因其最终产生的小RNA片段作用于非自身或同源的靶基因而被称为反式作用小干扰RNA(trans acting siRNA,tasiRNA)。
phasiRNA主要来源于长链非编码RNA,但也并非都产生于非编码的转录本。基因组测序及生物信息分析发现部分编码蛋白的基因,包括免疫反应受体NUCLEOTIDE-BINDING LEUCINE-RICH REPEAT(NBS-LRR)和PENTATRICOPEPTIDE REPEAT(PPR)家族以及转录因子MYB家族等,也能够被22 nt的miRNA触发产生phasiRNA,这一类phasiRNA可能在植物与有益微生物的相互作用或者防御外界胁迫中发挥功能[65]。
tasiRNA的产生起始于特定miRNA对TAS转录本的剪切,拟南芥中有4个TAS基因,TAS1-4[69-70]。根据TAS转录本中miRNA结合位点的数目,将tasiRNA的产生机制分为“one-hit model”和“two-hit model”。顾名思义,“one-hit model”中TAS初级转录本只有一个miRNA结合位点,引起切割的miRNA长度为22 nt[70],由AGO1蛋白介导,在5′端进行剪切(图 1-c,产生次生siRNA)。“one-hit model”是主要的tasiRNA/phasiRNA产生模型。而“two-hit model”特异地用于解释TAS3基因转录本的切割,TAS3转录本有两个21 nt长度的miR390的结合位点,参与的AGO蛋白是AGO7而非AGO1,剪切点靠近3′端而非5′端[71](图 1-c,产生次生siRNA)。“two-hit model”在植物中广泛存在,然而在不同物种中的具体分子机制略有差别,在苜蓿和苹果等物种中这些差异涉及参与的miRNA长度,结合的AGO蛋白成员以及剪切发生的位置[72-73]。
3.2 影响phasiRNA产生的因素早期研究发现,“one-hit model”中能触发tasiRNA产生的miRNA长度均为22 nt,而大部分miRNA的长度都是21 nt,不会产生tasiRNA,所以推测miRNA的长度对于tasiRNA的生成至关重要[65]。在这些报道中,一些miRNA以21 nt的标准形式存在时不会触发phasiRNA的产生;当被添加额外一个配对碱基产生22 nt的异构体时,即可触发产生phasiRNA[74-75]。此外在小RNA甲基化基因HEN1(HUA ENHANCER ONE)的突变体中,由于miR171的3′端没有被甲基化保护,在核酸转移酶URT1的催化下miR171的3′端添加了一个尿嘧啶,变成22 nt的miR171也导致phasiRNA的产生,而野生型中HEN1对miRNA 3′端甲基化阻止了尿嘧啶的添加,野生型中21 nt的miR171则不能产生phasiRNA[76]。
除了特定的长度以外,其他影响phasiRNA产生的因素也被陆续报道。2012年,Manavella等[77]发现21 nt的miRNA和22 nt的互补链miRNA*组成的非对称结构也能触发tasiRNA的产生。通过在报告基因不同位置引入miRNA靶位点,Zhang等[78]发现终止密码子紧挨着位于miR173靶位点上游时,tasiRNA产量最高;而终止密码子相距靶位点上游存在一定间隔时,翻译提前终止,tasiRNA生成被抑制,说明翻译过程对于tasiRNA生成也很关键。拟南芥TAS基因上存在一些短的读码框,TAS2的其中一个短读码框包含了miR173的结合位点,体外实验表明这个短的读码框可以被翻译产生短肽;利用烟草报告系统的实验发现,该读码框在miR173识别位点上游一段距离处发生的无义突变,造成tasiRNA的积累降低[79],这一现象与Zhang等[78]的发现一致;然而在拟南芥体内这些读码框对tasiRNA产生的影响并不清楚。miRNA的3′端与靶基因的配对程度也会影响tasiRNA的生成:在miRNA的3′端引入错配碱基会抑制tasiRNA的生成[78]。此外,触发phasiRNA生成的miRNA与细胞内膜结构关联程度的降低,也会使体内phasiRNA的丰度降低或者导致其相位特征的丧失[80]。最近的一项研究报道了与已知tasiRNA生成模型不完全相符的现象:在AGO1剪切活性缺陷的拟南芥幼苗中,体内的tasiRNA位点产生仍然可以产生大量siRNA;这些siRNA的产生同样依赖于RDR6和SGS3,不同的是,它们失去了“相位”特征。该研究表明miRNA-RISC复合体对TAS的剪切决定了tasiRNA产生的“相位”特征,而非tasiRNA产生与否的决定因素[81]。
4 miRNA介导的剪切和翻译抑制发挥活性的亚细胞场所AGO1在miRNA-RISC复合体中发挥核心功能,因此AGO1的亚细胞定位暗示了复合体发挥活性的场所。近几年一系列AGO1在细胞核里的新功能被挖掘和报道[82],然而被调控的靶蛋白的翻译过程在细胞质中完成,因此AGO1在细胞质中的分布更能体现其作用的场所。通过荧光蛋白标记结合激光共聚焦显微镜观察发现,AGO1在细胞质和细胞的核膜周围富集[83]。Li等[41]通过荧光标记发现AGO1在某种细胞质颗粒中积累,这种颗粒状信号与内质网标记重合;AGO1与定位在粗面内质网的膜整合蛋白AMP1的相互作用进一步证实了AGO1在内质网的分布(图 1)。内质网作为一种膜结构,是细胞内蛋白质翻译的重要场所,粗面内质网上附着了mRNA与大量正在执行翻译功能的核糖体。众多核糖体聚集在内质网上被称为膜结合多聚核糖体(Membrane bound poly-ribosome,MBP),多聚核糖体在细胞质和内质网上进行着活跃的蛋白翻译过程。2009年Lanet等[53]在多聚核糖体组分中检测到一些miRNA的分布,随后Li等[41]利用大规模组学测序证实拟南芥中大量miRNA以及其对应的靶基因在MBP的明显富集。AMP1及其同源基因LAMP1参与miRNA介导的翻译抑制,而与mRNA的剪切无关。在amp1 lamp1双突变体中,检测到细胞内总多聚核糖体(包含膜结合多聚核糖体和游离多聚核糖体)中miRNA靶基因mRNA的量与野生型无差别,而MBP中靶基因mRNA的丰度高于野生型,说明AMP1的存在抑制mRNA在粗面内质网的聚集,从而导致翻译抑制,由此推断miRNA介导的翻译抑制可能发生在粗面内质网上。
除了粗面内质网以外,AGO1与细胞中的加工小体紧密相关[84]。加工小体中含有许多与mRNA降解相关的蛋白因子,通过募集靶基因mRNA抑制翻译过程(图 1-a,翻译抑制),可能参与细胞应对环境胁迫的响应[85]。在拟南芥中,早期的研究报道发现两个影响翻译抑制的蛋白,其中VCS是加工小体的组成因子,SUO与加工小体存在共定位[11, 49](图 1-a,翻译抑制)。另一个影响翻译抑制的基因KTN1与细胞微管结构相关[11],可见植物中多种细胞结构都参与了miRNA介导的翻译抑制,然而尚不清楚加工小体与微管如何调控翻译抑制,以及这些细胞活动是如何共同协调发挥作用的。
对miRNA介导mRNA剪切发生的亚细胞场所的报道较少。类异戊二烯可能影响AGO1与膜结构的相关性,在类异戊二烯合成酶基因HYDROXY METHYLGLUTARYL COA REDUCTASE 1(HMG1)突变体中AGO1与膜的相关程度降低,同时miRNA剪切效率也降低[86],结合已知的内质网膜与AGO1的相关性[41],暗示剪切的发生场所也是粗面内质网。进一步针对mRNA降解产物的全基因组测序结果表明,miRNA靶基因在与正在翻译的核糖体结合的状态下被切割[64, 87]。MBP的组分测序分析也发现了许多miRNA剪切后产生的3′切割产物[80]。由此我们可以推测,至少部分miRNA对mRNA的剪切发生在粗面内质网上,同时剪切过程的发生与翻译过程偶联。
phasiRNA产生的第一步也是miRNA的切割,研究发现包括长度为22 nt的miRNA(phasiRNA trigger)在内的所有miRNA都富集在MBP上;ago1-27突变体中检测到22 nt的miRNA与MBP这一膜结构的结合能力下降,伴随phasiRNA的丰度下降[80]。能够生成phasiRNA的TAS虽然是一类长链非编码RNA,但是其序列上存在一些短的开放式阅读框,使TAS转录本与核糖体结合[64],甚至特异地与MBP结合[80]。综合以上发现表明,miRNA对PHAS的剪切步骤也发生在膜结合多聚核糖体上。
5 总结与展望植物miRNA对mRNA进行直接剪切或者抑制其翻译实现对靶基因的转录后负调控。miRNA-RISC复合体利用miRNA与靶基因的互补配对找到靶位点[21],并且发挥AGO1蛋白PIWI结构域的核酸外切酶活性切割靶基因mRNA[31],切割片段随后被5′-3′核酸外切酶XRN4和外切体降解[35]。对于体内的许多miRNA来说,剪切不是唯一的作用机制,类似动物中的翻译抑制也在植物中大量存在[8-10, 11, 41, 43, 49],尽管近十几年的研究揭示了一系列影响miRNA翻译抑制的重要基因[11, 41, 43, 49],但是多数仅限于在相应基因的突变体中发现翻译抑制受到影响的现象,对于这些基因在分子层面调控翻译抑制的机制仍不清楚。基于影响翻译抑制的内质网膜整合蛋白AMP1的相关研究及植物中的ribo-seq数据[41, 54, 64],当前的研究比较支持植物miRNA对翻译抑制的调控是通过抑制翻译起始过程实现的;动物中这一过程的实现主要依靠AGO2的互作蛋白GW182实现[44],然而植物中GW182同源基因的缺失使植物中的作用机制变得更为复杂[45]。植物中影响翻译抑制的蛋白“SUO”含有动物GW182蛋白的GW保守结构域[49],但是其是否与AGO1存在相互作用还有待进一步的验证。尽管有一些研究报道了能够与植物AGO1相关或者存在相互作用的蛋白,但多数与RISC复合体的组装和AGO1自身的降解或者再循环有关[46-48]。核糖体小亚基蛋白RACK1被报道存在于AGO1复合体中,然而一方面RACK1的突变影响了体内大多数miRNA的生成[58],另外也并没有检测到其与AGO1的直接相互作用。综上所述,目前并未找到植物中能够与AGO1互作的,明确与翻译过程相关的因子或者类似GW182的桥梁蛋白,miRNA抑制翻译起始影响蛋白翻译的分子机制有待进一步挖掘。从最新的藻类VIG1的功能研究中可以得到启示[54],植物中存在一些潜在的与动物中已知影响翻译抑制基因的同源基因,其功能机制尚未得到阐述,对这部分基因的深入挖掘将为我们提供新的线索。此外,设计翻译抑制报告株系进行正向遗传学的大量筛选,也将帮助我们尽快地阐明这一机制的分子基础。
植物中miRNA选择翻译抑制还是切割mRNA的决定因素,目前并不清楚。早期研究认为的影响因素(miRNA与靶基因结合的位置和互补配对的程度),目前看来都不能很好地解释两种机制产生的原因。miRNA介导的两种作用机制背后,可能有一些其他原因。miRNA与靶基因在MBP上的富集表明切割和翻译抑制是与翻译过程相偶联的,因此RISC复合体与mRNA的结合在某种程度上是与核糖体的一种竞争。靶基因上的核糖体密度,RISC复合体在细胞内丰度的高低是否会影响机制的选择,都是未来非常值得探索的问题。植物miRNA不仅可以通过胞间连丝实现细胞之间的短距离扩散,还可以通过筛管进行长距离的运输[88]。在miRNA产生的部位原位地和运输到的远程部位中miRNA对靶基因沉默的机制,是否存在差异,仍待进一步的阐明。当然,这两种机制的选择也许由更为复杂的因子综合决定。除了miRNA、靶基因mRNA和miRNA剪切产物在MBP上的富集,翻译抑制中关键的GW182桥梁因子在植物中的缺失,体内ribo-seq数据未显示核糖体在剪切位点附近的异常堆积等发现;在对多个AGO1剪切缺陷突变的转基因株系的研究中发现,个别miRNA靶基因在mRNA剪切活性被阻断以后,其蛋白的翻译抑制情况也受到了影响[20]。综合以上发现,我们可以推测:翻译抑制对某些靶基因来说可能是发生在内质网上的一种mRNA剪切,由于其发生的位点是在活跃翻译的内质网机器上,使我们认为这是一种翻译抑制的机制。
此外,对一些主要执行翻译抑制功能的miRNA来说,在RISC复合体中AGO1的内切酶活性是如何被抑制的也不清楚。动物miRNA-RISC复合体中的AGO2蛋白存在各种翻译后修饰,例如,AGO2不同氨基酸位点的磷酸化可以引起AGO2与miRNA或者靶基因结合能力的变化,以及翻译抑制效率的增强[89]。此外,AGO2的二磷酸腺苷核糖基化(ADP-ribosylation)导致miRNA的抑制效应降低等等[89]。已知植物AGO1蛋白的翻译后修饰在一些病毒与植物的互作中发挥重要作用。病毒中的沉默抑制因子,通常编码能够泛素化底物的F box蛋白,以宿主体内的沉默效应蛋白AGO1为底物进行泛素化。被修饰的AGO1蛋白随之进入自噬体或者“泛素—蛋白酶体”降解途径。此外在非病毒与植物互作背景下,研究者们也发现了F box蛋白编码基因FBW2能够负调控AGO1蛋白水平[90-91],暗示AGO1蛋白泛素化修饰的广泛存在。然而AGO1其他翻译后修饰仍未有报道,这些修饰的存在与否,以及对miRNA作用机制的选择或者RISC复合体的沉默效应的影响,将是未来非常值得关注和讨论的问题。
很多靶基因在植物中同时存在mRNA的剪切和翻译抑制[8, 11, 92]。两种作用方式之间的平衡和分子机制,也需要更多更深入的研究。动物中miRNA诱导的翻译抑制是可逆的[93-95]。研究发现,已经进入加工小体的mRNA分子在人类细胞受到饥饿刺激时可以被重新招募到多聚核糖体上进行翻译[93]。加工小体可以作为mRNA的一个临时储存场所,以保证可逆的翻译抑制的解除,使细胞在响应外界胁迫时做出迅速的反应。植物中也有多个加工小体相关的基因参与了翻译抑制,暗示植物中的翻译抑制可能也是一个可逆的过程。因为剪切后的mRNA进入降解是一种不可逆的生理过程,翻译抑制机制可逆性的存在将丰富miRNA-靶基因调控的层次。此前也有综述中提到,可逆的翻译抑制可能是植物应对环境胁迫的一种临时响应[96],尽管这个假设还没有被相关研究证实。2012年在蒺藜状苜蓿(Medicago truncatula)被根瘤菌侵染的研究中,研究者们发现了miRNA及其靶基因上附着的核糖体存在动态变化[97],该研究在一定程度上支持了这一假说。植物miRNA翻译抑制与植物响应环境胁迫之间的关联,未来仍然需要更多的研究证实。
相信实验技术的不断精进和基因组学的深入探索,必将推动miRNA作用方式分子机制研究的进步。我们期待miRNA及其他小RNA分子参与的表达调控机制的网络逐步明晰,这将有利于我们进一步了解生物体内部的运作机制,促进miRNA技术在抵御逆境胁迫、病毒侵染等领域中的应用。
| [1] |
Lee RC, Feinbaum RL, Ambres V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843-854. DOI:10.1016/0092-8674(93)90529-Y |
| [2] |
Reinhart BJ, Slack FJ, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772): 901-906. DOI:10.1038/35002607 |
| [3] |
Reinhart BJ, Weinstein EG, Rhoades MW, et al. MicroRNAs in plants[J]. Genes & Development, 2002, 16(13): 1616-1626. |
| [4] |
Llave C. Cleavage of scarecrow-like mRNA targets directed by a class of Arabidopsis miRNA[J]. Science, 2002, 297(5589): 2053-2056. DOI:10.1126/science.1076311 |
| [5] |
Park W, Li J, Song R, et al. CARPEL FACTORY, a dicer homolog, and HEN1, a novel protein, act in microRNA metabolism in Arabidopsis thaliana[J]. Current Biology, 2002, 12(17): 1484-1495. DOI:10.1016/S0960-9822(02)01017-5 |
| [6] |
Kozomara A, Birgaoanu M, Griffiths-Jones S. miRBase:from microRNA sequences to function[J]. Nucleic Acids Research, 2019, 47(1): D155-D162. |
| [7] |
Hamilton AJ, Baulcombe DC. A species of small antisense RNA in posttranscriptional gene silencing in plants[J]. Science, 1999, 286(5441): 950-952. DOI:10.1126/science.286.5441.950 |
| [8] |
Aukerman MJ, Sakai H. Regulation of flowering time and floral organ identity by a microRNA and its APETALA2-like target genes[J]. The Plant Cell, 2003, 15(11): 2730-2741. DOI:10.1105/tpc.016238 |
| [9] |
Chen X. A MicroRNA as a translational repressor of APETALA2 in Arabidopsis flower development[J]. Science, 2004, 303(5666): 2022-2025. DOI:10.1126/science.1088060 |
| [10] |
Gandikota M, Birkenbihl RP, Höhmann S, et al. The miRNA156/157 recognition element in the 3′ UTR of the Arabidopsis SBP box gene SPL3 prevents early flowering by translational inhibition in seedlings[J]. The Plant Journal, 2007, 49(4): 683-693. DOI:10.1111/j.1365-313X.2006.02983.x |
| [11] |
Brodersen P, Sakvarelidze-Achard L, Bruun-Rasmussen M, et al. Widespread translational inhibition by plant miRNAs and siRNAs[J]. Science, 2008, 320(5880): 1185-1190. DOI:10.1126/science.1159151 |
| [12] |
熊雪梅, 吴莹, 王洋. 植物体内调控miRNA合成与功能的机制研究进展[J]. 植物研究, 2014, 34(2): 282-288. |
| [13] |
郭韬, 李广林, 魏强, 等. 植物MicroRNA功能的研究进展[J]. 西北植物学报, 2011, 31(11): 2347-2354. |
| [14] |
许硕, 胡正, 姜奇彦, 等. 植物microRNA功能及其在逆境条件下的研究进展[J]. 生物技术通报, 2012(2): 1-7. |
| [15] |
张俊红, 张守攻, 齐力旺. 植物成熟microRNA转录后修饰与降解的研究进展[J]. 植物学报, 2014, 49(4): 483-489. |
| [16] |
Yu Y, Jia T, Chen X. The 'how' and 'where' of plant microRNAs[J]. The New Phytologist, 2017, 216(4): 1002-1017. DOI:10.1111/nph.14834 |
| [17] |
Ipsaro JJ, Joshua-Tor L. From guide to target:molecular insights into eukaryotic RNA-interference machinery[J]. Nature Structural & Molecular Biology, 2015, 22(1): 20-28. |
| [18] |
Iwakawa HO, Tomari Y. The functions of microRNAs:mRNA decay and translational repression[J]. Trends in Cell Biology, 2015, 25(11): 651-665. DOI:10.1016/j.tcb.2015.07.011 |
| [19] |
Iwakawa HO, Tomari Y. Molecular insights into microRNA-mediated translational repression in plants[J]. Molecular Cell, 2013, 52(4): 591-601. DOI:10.1016/j.molcel.2013.10.033 |
| [20] |
Arribas-Hernández L, Kielpinski LJ, Brodersen P. mRNA decay of most Arabidopsis miRNA targets requires slicer activity of AGO1[J]. Plant Physiology, 2016, 171(4): 2620-2632. |
| [21] |
German MA, Pillay M, Jeong DH, et al. Global identification of microRNA-target RNA pairs by parallel analysis of RNA ends[J]. Nature Biotechnology, 2008, 26(8): 941-946. DOI:10.1038/nbt1417 |
| [22] |
Höck J, Meister G. The Argonaute protein family[J]. Genome Biology, 2008, 9(2): 210. |
| [23] |
Baumberger N, Baulcombe DC. Arabidopsis ARGONAUTE1 is an RNA slicer that selectively recruits microRNAs and short interfering RNAs[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(33): 11928-11933. DOI:10.1073/pnas.0505461102 |
| [24] |
Qi Y, Denli AM, Hannon GJ. Biochemical specialization within Arabidopsis RNA silencing pathways[J]. Molecular Cell, 2005, 19(3): 421-428. |
| [25] |
Mi S, Cai T, Hu Y, et al. Sorting of small RNAs into Arabidopsis Argonaute complexes is directed by the 5′ terminal nucleotide[J]. Cell, 2008, 133(1): 116-127. DOI:10.1016/j.cell.2008.02.034 |
| [26] |
Montgomery TA, Howell MD, Cuperus JT, et al. Specificity of ARGONAUTE7-miR390 interaction and dual functionality in TAS3 trans-acting siRNA formation[J]. Cell, 2008, 133(1): 128-141. |
| [27] |
Takeda A, Iwasaki S, Watanabe T, et al. The mechanism selecting the guide strand from small RNA duplexes is different among Argonaute proteins[J]. Plant and Cell Physiology, 2008, 49(4): 493-500. DOI:10.1093/pcp/pcn043 |
| [28] |
Ji L, Liu X, Yan J, et al. ARGONAUTE10 and ARGONAUTE1 regulate the termination of floral stem cells through two microRNAs in Arabidopsis[J]. PLoS Genetics, 2011, 7(3): e1001358. DOI:10.1371/journal.pgen.1001358 |
| [29] |
Maunoury N, Vaucheret H. AGO1 and AGO2 act redundantly in miR408-mediated plantacyanin regulation[J]. PLoS One, 2011, 6(12): e28729. DOI:10.1371/journal.pone.0028729 |
| [30] |
Zhu H, Hu F, Wang R, et al. Arabidopsis Argonaute10 specifically sequesters miR166/165 to regulate shoot apical meristem development[J]. Cell, 2011, 145(2): 242-256. |
| [31] |
Hutvagner G, Simard MJ. Argonaute proteins:key players in RNA silencing[J]. Nature Reviews Molecular Cell Biology, 2008, 9(1): 22-32. |
| [32] |
Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21- and 22-nucleotide RNAs[J]. Genes & Development, 2001, 15(2): 188-200. |
| [33] |
Souret FF, Kastenmayer JP, Green PJ. AtXRN4 degrades mRNA in Arabidopsis and its substrates include selected miRNA targets[J]. Molecular Cell, 2004, 15(2): 173-183. |
| [34] |
Ibrahim F, Rohr J, Jeong WJ, et al. Untemplated oligoadenylation promotes degradation of RISC-cleaved transcripts[J]. Science, 2006, 314(5807): 1893-1893. DOI:10.1126/science.1135268 |
| [35] |
Ren G, Xie M, Zhang S, et al. Methylation protects microRNAs from an AGO1-associated activity that uridylates 5′ RNA fragments generated by AGO1 cleavage[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(17): 6365-6370. DOI:10.1073/pnas.1405083111 |
| [36] |
Wang X, Zhang S, Dou Y, et al. Synergistic and independent actions of multiple terminal nucleotidyl transferases in the 3′ tailing of small RNAs in Arabidopsis[J]. PLoS Genetics, 2015, 11(4): e1005091. DOI:10.1371/journal.pgen.1005091 |
| [37] |
Zhang Z, Hu F, Sung MW, et al. RISC-interacting clearing 3′-5′ exoribonucleases(RICEs)degrade uridylated cleavage fragments to maintain functional RISC in Arabidopsis thaliana[J]. eLife, 2017, 6: e24466. DOI:10.7554/eLife.24466 |
| [38] |
Branscheid A, Marchais A, Schott G, et al. SKI2 mediates degradation of RISC 5′-cleavage fragments and prevents secondary siRNA production from miRNA targets in Arabidopsis[J]. Nucleic Acids Research, 2015, 43(22): 10975-10988. DOI:10.1093/nar/gkv1014 |
| [39] |
Orban TI, Izaurralde E. Decay of mRNAs targeted by RISC requires XRN1, the Ski complex, and the exosome[J]. RNA, 2005, 11(4): 459-469. DOI:10.1261/rna.7231505 |
| [40] |
Alonso-Peral MM, Li J, Li Y, et al. The microRNA159-regulated GAMYB-like genes inhibit growth and promote programmed cell death in Arabidopsis[J]. Plant Physiology, 2010, 154(2): 757-771. |
| [41] |
Li S, Liu L, Zhuang X, et al. MicroRNAs inhibit the translation of target mRNAs on the endoplasmic reticulum in Arabidopsis[J]. Cell, 2013, 153(3): 562-574. DOI:10.1016/j.cell.2013.04.005 |
| [42] |
Liu MJ, Wu SH, Wu JF, et al. Translational landscape of photomorphogenic Arabidopsis[J]. The Plant Cell, 2013, 25(10): 3699-3710. DOI:10.1105/tpc.113.114769 |
| [43] |
Reis RS, Hart-Smith G, Eamens AL, et al. Gene regulation by translational inhibition is determined by Dicer partnering proteins[J]. Nature Plants, 2015, 1: 14027. DOI:10.1038/nplants.2014.27 |
| [44] |
Zekri L, Kuzuoğlu-Öztürk D, Izaurralde E. GW182 proteins cause PABP dissociation from silenced miRNA targets in the absence of deadenylation[J]. The EMBO Journal, 2013, 32(7): 1052-1065. DOI:10.1038/emboj.2013.44 |
| [45] |
Huntzinger E, Izaurralde E. Gene silencing by microRNAs:contributions of translational repression and mRNA decay[J]. Nature Reviews Genetics, 2011, 12(2): 99-110. DOI:10.1038/nrg2936 |
| [46] |
Iki T, Yoshikawa M, Nishikiori M, et al. In vitro assembly of plant RNA-induced silencing complexes facilitated by molecular chaperone HSP90[J]. Molecular Cell, 2010, 39(2): 282-291. DOI:10.1016/j.molcel.2010.05.014 |
| [47] |
Iki T, Yoshikawa M, Meshi T, et al. Cyclophilin 40 facilitates HSP90-mediated RISC assembly in plants[J]. The EMBO Journal, 2012, 31(2): 267-278. DOI:10.1038/emboj.2011.395 |
| [48] |
Cui Y, Fang X, Qi Y. TRANSPORTIN1 promotes the association of microRNA with ARGONAUTE1 in Arabidopsis[J]. The Plant Cell, 2016, 28(10): 2576-2585. DOI:10.1105/tpc.16.00384 |
| [49] |
Yang L, Wu G, Poethig RS. Mutations in the GW-repeat protein SUO reveal a developmental function for microRNA-mediated translational repression in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(1): 315-320. DOI:10.1073/pnas.1114673109 |
| [50] |
Pomeranz M, Lin PC, Finer J, et al. AtTZF gene family localizes to cytoplasmic foci[J]. Plant Signaling & Behavior, 2010, 5(2): 190-192. |
| [51] |
Weber C, Nover L, Fauth M. Plant stress granules and mRNA processing bodies are distinct from heat stress granules[J]. The Plant Journal, 2008, 56(4): 517-530. DOI:10.1111/j.1365-313X.2008.03623.x |
| [52] |
Rogers K, Chen X. Biogenesis, turnover, and mode of action of plant microRNAs[J]. The Plant Cell, 2013, 25(7): 2383-2399. DOI:10.1105/tpc.113.113159 |
| [53] |
Lanet E, Delannoy E, Sormani R, et al. Biochemical evidence for translational repression by Arabidopsis microRNAs[J]. The Plant Cell, 2009, 21(6): 1762-1768. DOI:10.1105/tpc.108.063412 |
| [54] |
Ma X, Ibrahim F, Kim EJ, et al. An ortholog of the Vasa intronic gene is required for small RNA-mediated translation repression in Chlamydomonas reinhardtii[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(1): 761-770. DOI:10.1073/pnas.1908356117 |
| [55] |
Van Dyke N, Pickering BF, Van Dyke MW. Stm1p alters the ribosome association of eukaryotic elongation factor 3 and affects translation elongation[J]. Nucleic Acids Research, 2009, 37(18): 6116-6125. DOI:10.1093/nar/gkp645 |
| [56] |
Bolger GB. The RNA-binding protein SERBP1 interacts selectively with the signaling protein RACK1[J]. Cell Signaling, 2017, 35: 256-263. DOI:10.1016/j.cellsig.2017.03.001 |
| [57] |
Jannot G, Bajan S, Giguère NJ, et al. The ribosomal protein RACK1 is required for microRNA function in both C. elegans and humans[J]. EMBO Reports, 2011, 12(6): 581-586. DOI:10.1038/embor.2011.66 |
| [58] |
Speth C, Willing EM, Rausch S, et al. RACK1 scaffold proteins influence miRNA abundance in Arabidopsis[J]. The Plant Journal, 2013, 76(3): 433-445. DOI:10.1111/tpj.12308 |
| [59] |
Yekta S, Shih IH, Bartel DP. MicroRNA-directed cleavage of HOXB8 mRNA[J]. Science, 2004, 304(5670): 594-596. DOI:10.1126/science.1097434 |
| [60] |
Hausser J, Syed AP, Bilen B, et al. Analysis of CDS-located miRNA target sites suggests that they can effectively inhibit translation[J]. Genome Research, 2013, 23(4): 604-615. DOI:10.1101/gr.139758.112 |
| [61] |
Yamasaki T, Voshall A, Kim EJ, et al. Complementarity to an miRNA seed region is sufficient to induce moderate repression of a target transcript in the unicellular green alga Chlamydomonas reinhardtii[J]. The Plant Journal, 2013, 76(6): 1045-1056. DOI:10.1111/tpj.12354 |
| [62] |
Liu Q, Wang F, Axtell MJ. Analysis of complementarity requirements for plant microRNA targeting using a Nicotiana benthamiana quantitative transient assay[J]. The Plant Cell, 2014, 26(2): 741-753. |
| [63] |
Li JF, Chung HS, Niu Y, et al. Comprehensive protein-based artificial microRNA screens for effective gene silencing in plants[J]. The Plant Cell, 2013, 25(5): 1507-1522. DOI:10.1105/tpc.113.112235 |
| [64] |
Hou CY, Lee WC, Chou HC, et al. Global analysis of truncated RNA ends reveals new insights into ribosome stalling in plants[J]. The Plant Cell, 2016, 28(10): 2398-2416. DOI:10.1105/tpc.16.00295 |
| [65] |
Fei Q, Xia R, Meyers BC. Phased, secondary, small interfering RNAs in posttranscriptional regulatory networks[J]. The Plant Cell, 2013, 25(7): 2400-2415. DOI:10.1105/tpc.113.114652 |
| [66] |
Johnson C, Kasprzewska A, Tennessen K, et al. Clusters and superclusters of phased small RNAs in the developing inflorescence of rice[J]. Genome Research, 2009, 19(8): 1429-1440. DOI:10.1101/gr.089854.108 |
| [67] |
International Brachypodium Initiative. Genome sequencing and analysis of the model grass Brachypodium distachyon[J]. Nature, 2010, 463(7282): 763-768. DOI:10.1038/nature08747 |
| [68] |
Song X, Li P, Zhai J, et al. Roles of DCL4 and DCL3b in rice phased small RNA biogenesis[J]. The Plant Journal, 2012, 69(3): 462-474. DOI:10.1111/j.1365-313X.2011.04805.x |
| [69] |
Yoshikawa M. A pathway for the biogenesis of trans-acting siRNAs in Arabidopsis[J]. Genes & Development, 2005, 19(18): 2164-2175. |
| [70] |
Allen E, Xie Z, Gustafson AM, et al. MicroRNA-directed phasing during trans-acting siRNA biogenesis in plants[J]. Cell, 2005, 121(2): 207-221. |
| [71] |
Axtell M J, Jan C, Rajagopalan R, et al. A two-hit trigger for siRNA biogenesis in plants[J]. Cell, 2006, 127(3): 565-577. DOI:10.1016/j.cell.2006.09.032 |
| [72] |
Zhai J, Jeong DH, De Paoli E, et al. MicroRNAs as master regulators of the plant NB-LRR defense gene family via the production of phased, trans-acting siRNAs[J]. Genes & Development, 2011, 25(23): 2540-2553. |
| [73] |
Xia R, Meyers BC, Liu Z, et al. MicroRNA superfamilies descended from miR390 and their roles in secondary small interfering RNA Biogenesis in Eudicots[J]. The Plant Cell, 2013, 25(5): 1555-1572. DOI:10.1105/tpc.113.110957 |
| [74] |
Chen HM, Chen LT, Patel K, et al. 22-nucleotide RNAs trigger secondary siRNA biogenesis in plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(34): 15269-15274. DOI:10.1073/pnas.1001738107 |
| [75] |
Cuperus JT, Carbonell A, Fahlgren N, et al. Unique functionality of 22-nt miRNAs in triggering RDR6-dependent siRNA biogenesis from target transcripts in Arabidopsis[J]. Nature Structural & Molecular Biology, 2010, 17(8): 997-1003. |
| [76] |
Zhai J, Zhao Y, Simon SA, et al. Plant microRNAs display differential 3′ truncation and tailing modifications that are ARGONAUTE1 dependent and conserved across species[J]. The Plant Cell, 2013, 25(7): 2417-2428. DOI:10.1105/tpc.113.114603 |
| [77] |
Manavella P, Hagmann J, Ott F, et al. Fast-forward genetics identifies plant CPL phosphatases as regulators of miRNA processing factor HYL1[J]. Cell, 2012, 151(4): 859-870. DOI:10.1016/j.cell.2012.09.039 |
| [78] |
Zhang C, Ng WK, Lu J, et al. Roles of target site location and sequence complementarity in trans-acting siRNA formation in Arabidopsis[J]. The Plant Journal, 2012, 69(2): 217-226. DOI:10.1111/j.1365-313X.2011.04783.x |
| [79] |
Yoshikawa M, Iki T, Numa H, et al. A short open reading frame encompassing the microRNA173 target site plays a role in trans-acting small interfering RNA biogenesis[J]. Plant Physiology, 2016, 171(1): 359-368. DOI:10.1104/pp.16.00148 |
| [80] |
Li S, Le B, Ma X, et al. Biogenesis of phased siRNAs on membrane-bound polysomes in Arabidopsis[J]. eLife, 2016, 5: e22750. DOI:10.7554/eLife.22750 |
| [81] |
Arribas-Hernandez L, Marchais A, Poulsen C, et al. The slicer activity of ARGONAUTE1 is required specifically for the phasing, not production, of trans-acting short interfering RNAs in Arabidopsis[J]. The Plant Cell, 2016, 28(7): 1563-1580. |
| [82] |
Bajczyk M, Bhat SS, Szewc L, et al. Novel nuclear functions of Arabidopsis ARGONAUTE1:beyond RNA interference[J]. Plant Physiology, 2019, 179(3): 1030-1039. DOI:10.1104/pp.18.01351 |
| [83] |
Derrien B, Baumberger N, Schepetilnikov M, et al. Degradation of the antiviral component ARGONAUTE1 by the autophagy pathway[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39): 15942-15946. DOI:10.1073/pnas.1209487109 |
| [84] |
Xu J, Chua NH. Processing bodies and plant development[J]. Current Opinion in Plant Biology, 2011, 14(1): 88-93. DOI:10.1016/j.pbi.2010.10.003 |
| [85] |
Teixeira D, Sheth U, Valencia-Sanchez MA, et al. Processing bodies require RNA for assembly and contain nontranslating mRNAs[J]. RNA, 2005, 11(4): 371-382. DOI:10.1261/rna.7258505 |
| [86] |
Brodersen P, Sakvarelidzeachard L, Schaller H, et al. Isoprenoid biosynthesis is required for miRNA function and affects membrane association of ARGONAUTE 1 in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(5): 1778-1783. DOI:10.1073/pnas.1112500109 |
| [87] |
Yu X, Willmann MR, Anderson SJ, et al. Genome-wide mapping of uncapped and cleaved transcripts reveals a role for the nuclear mRNA cap-binding complex in cotranslational RNA decay in Arabidopsis[J]. The Plant Cell, 2016, 28(10): 2385-2397. DOI:10.1105/tpc.16.00456 |
| [88] |
Liu L, Chen X. Intercellular and systemic trafficking of RNAs in plants[J]. Nature Plants, 2018, 4(11): 869-878. DOI:10.1038/s41477-018-0288-5 |
| [89] |
Wilczynska A, Bushell M. The complexity of miRNA-mediated repression[J]. Cell Death and Differentiation, 2015, 22(1): 22-33. DOI:10.1038/cdd.2014.112 |
| [90] |
Earley K, Smith M, Weber R, et al. An endogenous F-box protein regulates ARGONAUTE1 in Arabidopsis thaliana[J]. Silence, 2010, 1(1): 15. DOI:10.1186/1758-907X-1-15 |
| [91] |
Csorba T, Lózsa R, Hutvágner G, et al. Polerovirus protein P0 prevents the assembly of small RNA-containing RISC complexes and leads to degradation of ARGONAUTE1[J]. The Plant Journal, 2010, 62(3): 463-472. DOI:10.1111/j.1365-313X.2010.04163.x |
| [92] |
Beauclair L, Yu A, Bouché N. microRNA-directed cleavage and translational repression of the copper chaperone for superoxide dismutase mRNA in Arabidopsis[J]. The Plant Journal, 2010, 62(3): 454-462. DOI:10.1111/j.1365-313X.2010.04162.x |
| [93] |
Bhattacharyya SN, Habermacher R, Martine U, et al. Stress-induced reversal of microRNA repression and mRNA P-body localization in human cells[J]. Cold Spring Harbor Symposia on Quantitative Biology, 2006, 71: 513-521. DOI:10.1101/sqb.2006.71.038 |
| [94] |
Muddashetty RS, Nalavadi VC, Gross C, et al. Reversible inhibition of PSD-95 mRNA translation by miR-125a, FMRP phosphorylation, and mGluR signaling[J]. Molecular Cell, 2011, 42(5): 673-688. DOI:10.1016/j.molcel.2011.05.006 |
| [95] |
Mazumder A, Bose M, Chakraborty A, et al. A transient reversal of miRNA-mediated repression controls macrophage activation[J]. EMBO Report, 2013, 14(11): 1008-1016. DOI:10.1038/embor.2013.149 |
| [96] |
Voinnet O. Origin, biogenesis, and activity of plant microRNAs[J]. Cell, 2009, 136(4): 669-687. DOI:10.1016/j.cell.2009.01.046 |
| [97] |
Reynoso E, Nesci A, Allegretti P, et al. Kinetic and mechanistic aspects of sensitized photodegradation of β-lactam antibiotics:microbiological implications[J]. Redox Report, 2012, 17(6): 275-283. DOI:10.1179/1351000212Y.0000000032 |