2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences,Beijing 100049
农业生产不断从土壤中带走大量氮素, 农业生产力的维持和提高需要稳定的氮素来源.最初, 人类主要通过种植大豆和水稻来增加土壤氮素, 随后通过开采硝酸盐矿作为肥料和施加有机肥获得更多的作物产量[1].从通过Haber-Bosch法实现氮气工业化生产氨开始, 人类打破了氮素对农业生产的限制, 化学氮肥的生产和应用满足了农业生态系统对活性氮的需求, 为不断增长的人口提供了食物[2-3].然而, 对化学氮肥的过度依赖不利于农业的可持续发展[4].随着化学氮肥生产和施用量的增加, 氮肥利用率越来越低, 气态和溶解态的氮流失导致了水和空气污染的增加, 大气温室气体浓度的升高和生物多样性的丧失等一系列环境问题[2, 5-7].虽然人们正在努力尝试通过各种途径提高农田氮肥利用率(施肥策略, 种植制度, 育种等) [8-12], 但过量施肥条件下的大量氮损失是不可避免的, 适当降低氮肥施用量是最根本的解决办法.
生物固氮是固氮微生物在固氮酶的催化下将空气中的氮气转化成活性氮的自然过程, 是陆地和海洋生态系统中活性氮的主要非人为输入来源[2, 13].在广泛应用氮肥之前, 生物固氮是农业生产中活性氮输入的主要来源[14].随着氮肥施用量的增加, 人们对生物固氮的重视程度逐渐降低, 然而, 更有效地开发和利用自然过程生物固氮作用是逐步降低氮肥施用量的潜在途径[15-16].非共生固氮是非豆科作物栽培过程中的主要生物固氮途径, 在农田氮供应中发挥着广泛的作用[17-18].据估算, 非共生生物固氮提供了作物氮素总量24%的氮素来源[19].接种非共生固氮菌是提高土壤生物固氮能力, 从而降低氮肥施用量的最可行方案之一[20-21].前人研究表明, 从预期发挥功能的环境中分离出的菌株可能更能适应原位生态胁迫[22-23].因此, 为了扩大成功接种固氮菌的可能性, 有必要在各种生态条件下筛选丰富的高效固氮菌株[23].
水稻土在我国分布范围广, 性质差别大, 是微生物菌株分离的重要来源.研究表明紫色土水稻土, 下位砂姜土水稻土和红壤水稻土的固氮效率存在很大差异[24].本研究选取紫色土水稻土, 下位砂姜土水稻土和红壤水稻土3个典型水稻土样品和1物菌种资源, 为进一步稻田生物固氮的微生物调控研究和应用提供菌株材料.
1 材料与方法 1.1 材料 1.1.1 土壤样品供试土壤选取3种不同固氮效率水平的水稻土样品[24] :紫色土水稻土(固氮效率高, 42 d生物固氮量20.1 kg N /hm2, 四川省盐亭县, 中国科学院盐亭紫色土农业生态试验站), 下位砂姜土水稻土(固氮效率中等, 42 d生物固氮量9.4kg N /hm2, 江苏省扬州市, 中国FACE试验站), 红壤水稻土(固氮效率低, 42 d生物固氮量2.2 kg N/hm2, 江西省鹰潭市, 中国科学院鹰潭红壤生态实验站), 另选取一个低肥力水平土壤样品:来自中国科学院鹰潭红壤生态实验站长期定位试验未施肥小区的红壤水稻土(江西省鹰潭市), 共4个水稻土样品.土壤样品于2014年4-5月采自田间耕作层(0-15 cm), 随后进行风干过2 mm筛, 室温保存.2018年4月, 将土壤样品淹水1-2 cm, 28℃黑暗条件下预培养14 d, 以恢复土壤微生物活性, 预培养结束后进行菌株分离.
1.1.2 培养基固氮微生物的分离与培养选用Brown无氮培养基[25-27] :葡萄糖5 g/L, 无水CaCl20.15 g/L, MgSO4·7H2O 0.2 g/L, FeSO4·7H2O 0.04g/L, Na2MoO4 0.005 g/L, K2HPO4 0.8 g/L.pH为6.8-7.0.固体培养基另加琼脂糖10-15 g/L.FeSO4·7H2O单独过滤灭菌, 现配现用.葡萄糖单独过滤灭菌.K2HPO4与金属盐分开灭菌.
1.2 方法 1.2.1 平板稀释法分离固氮微生物取土壤10 g于250 mL无菌三角瓶中, 加入无菌水至100 mL体积, 28℃, 180 r/min振摇30 min, 然后用无菌水进行梯度稀释(10-2, 10-3, 10-4, 10-5, 10-6).吸取100 μL稀释液平板涂布法均匀接种到固体Brown无氮培养基上, 倒置于28℃恒温黑暗培养5-7 d[28].选择菌落数在20-200之间的平板进行计数, 计算无氮培养基上可培养微生物数量密度[18, 29].根据菌落形态选择长势较好, 生长速度较快的不同菌落, 用接种环平板划线法接种于新的固体无氮培养基上单独纯化培养.
1.2.2 富集纯化法分离固氮微生物取土壤10 g于250 mL无菌三角瓶中, 加入液体Brown无氮培养基至100 mL体积, 28℃, 180 r/min振摇富集培养4-5d, 然后1%转接到新的液体无氮培养基中进一步振摇富集培养, 重复转接3-4次后得到在液体无氮培养基中生长较快的以固氮微生物为优势菌群的菌液, 然后进行稀释涂板挑选纯化.
1.2.3 菌株的16S rRNA基因鉴定用细菌基因组DNA提取试剂盒(TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit Ver.3.0)(TaKaRa, Code No.9763)提取菌株基因组DNA.用NanoDrop 1000(Thermo Scientific)进行DNA样品的定量和纯度检测.使用通用引物27F(5'-AGAGTTTGATCMTGGCTCAG-3'), 1492R(5'-GGTTACCTTGTTACGACTT-3')对菌株基因组DNA的16S rRNA基因全长序列进行PCR扩增[25, 30]
PCR产物进行1%琼脂糖凝胶电泳, 目的条带切割后采用SanPrep柱式DNA胶回收试剂盒(SangonBiotech, Order NO. B518131)回收目的DNA片段.使用3730xl DNA Analyzer(Applied Biosystems)进行DNA片段的序列分析, 测序试剂盒使用BigDyeTerminator v3.1(Applied Biosystems).双向测序结果使用SeqMan(DNASTAR Lasergene)进行拼接, 得到接近全长的16S rRNA基因序列.使用EzBioCloud Identify service(https ://www.ezbiocloud.net/identify)[31-33]和NCBI Nucleotide BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)将16S rRNA基因序列结果与GenBank数据库进行比对, 选择有效命名的模式菌株作为参考.
1.2.4 菌株基因组DNA的nifH基因扩增采用引物Pol-F(5'-TGCGAYCCSAARGCBGACTC-3'), Pol-R(5'-ATSGCCATCATYTCRCCGGA-3') [34]对菌株基因组DNA的nifH基因进行PCR扩增, 阴性对照使用ddH2O和大肠杆菌基因组DNA作为模板.
1.2.5 乙炔还原活性测定nifH基因扩增将对数生长中期(OD600=0.4-0.8)的菌液加入125 mL玻璃瓶中, 密封后用注射器将瓶内气体体积的10%空气置换为纯化的高纯乙炔[13, 35-36].继续培养2-24 h后收集瓶内气体, 用带有氢火焰离子化检测器(FID)的气相色谱仪(Shimadzu, GC-2014C)分析乙烯浓度[37-38].采用Bradford法[39]测定菌液的蛋白量来表征其生物量, 计算单位时间单位质量蛋白的乙烯生成速率nmol C2H4 /(mg·min)).以固氮菌属(Azotobacter)模式菌株A. chroococcum ATCC 9043T作为参比菌株.
1.2.6 菌株在无氮培养基中的生长曲线测定以液体Brown无氮培养基中活化培养的对数生长中期菌液(OD600≈0.6)作为种子液, 1%接种于装有100mL液体Brown无氮培养基的250 mL锥形瓶中, 透气封口膜封口, 于28℃, 180 r/min振摇培养, 每2 h取样测定OD600, 绘制菌株在无氮培养基中的生长曲线.
1.2.7 菌株的IAA生成测定基因扩增将菌株种子液(OD600 ≈ 0.6)1%接种于含有500 mg/L L-色氨酸的液体Brown无氮培养基中, 28℃, 180 r/min振摇培养48 h, 测定菌液中的IAA含量.将菌液离心(10 000× g, 10 min), 取上清液加入等体积显色液(10.8 mol/L H2SO4, 4.5 g/L FeCl3), 混匀避光静置30 min, 测定其OD540.用0.5-20 μg/mL IAA标准液测定绘制标准曲线, 根据标准曲线计算菌液IAA含量[40].
1.2.8 菌株的溶磷活性测定将菌株种子液1%接种于以磷酸三钙作为难溶磷酸盐的液体NBRIP培养基[41]中, 28℃, 180 r/min振摇培养7 d.将菌液离心(10 000 × g, 10 min), 取上清液用钼蓝比色法测定菌液中可溶性磷含量的变化[42-43].
1.2.9 菌株的铁载体生成测定将菌株培养在液体Brown无氮培养基(去铁)中, 28℃, 180 r/min振摇培养72 h, 测定菌液中的铁载体.将菌液离心(10 000× g, 10 min), 取上清液加入等体积CAS检测液, 混匀静置1 h.以蒸馏水为空白测定OD630, 未接菌培养基作为参比对照, 铁载体活性单位=[(对照吸光度-样品吸光度)/对照吸光度] [44]
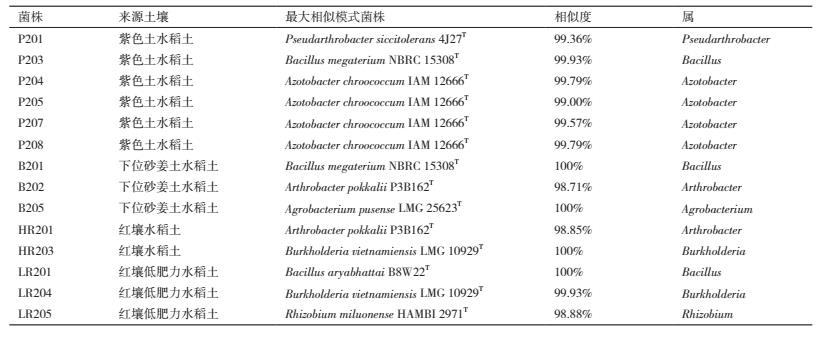
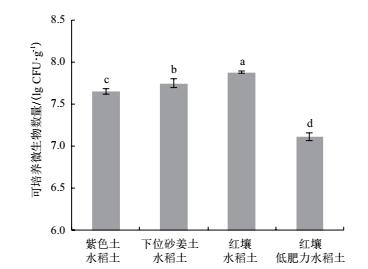
2 结果 2.1 不同土壤样品在无氮培养基上的可培养微生物数量过稀释平板法在Brown无氮培养基上28℃培养7 d测得的可培养微生物数量如图 1所示, 紫色土水稻土在无氮培养基上的可培养微生物数量最多, 其次为下位砂姜土水稻土, 红壤水稻土.无氮培养基上可培养微生物数量的多少与土壤固氮效率的高低规律表现一致.红壤低肥力水稻土样品中在无氮培养基上的可培养微生物数量最少, 约为紫色土水稻土的1/10.
|
| 图中误差线代表标准差, 相同字母表示差异未达显著水平(P < 0.05),下同 图 1 无氮培养基上可培养微生物数量 |
4种土壤样品在Brown无氮培养基中的富集效果不同.在转接富集过程中, 下位砂姜土水稻土, 红壤水稻土, 红壤低肥力水稻土富集液中微生物生长逐渐减缓, 在进行3次转接富集后, 经过5 d的培养依然不可见明显的微生物增殖迹象(图 2).紫色土水稻土富集液在无氮培养基中转接富集效果较好, 1%接种量转接5次后能够持续保持24 h内富集液呈现浑浊状态.对紫色土水稻土富集所得混合菌液进行梯度稀释涂布分离, 平板上的菌落形态较为一致, 通过划线纯化, 混合菌液中仅得到1株菌株(P208).

|
| 图 2 不同土壤样品在无氮培养基中的富集效果 |
采用平板稀释法从4种水稻土样品中分离得到13株能够在固体Brown无氮培养基上较快生长的菌株, 从划线接种到长出稳定的单菌落约需2-5 d, 加上紫色土水稻土富集液中得到的菌株P208, 共得到14株菌株(表 1).通过菌株16S rRNA基因序列与GenBank数据库进行比对, 结合重建的系统发育树信息, 14株新分离菌株分别可归类为:假节杆菌属(Pseudarthrobacter)1株, 芽孢杆菌属(Bacillus)3株, 固氮菌属(Azotobacter)4株, 节杆菌属(Arthrobacter)2株, 农杆菌属(Agrobacterium)1株, 伯克霍尔德氏菌属(Burkholderia)2株, 根瘤菌属(Rhizobium)1株.
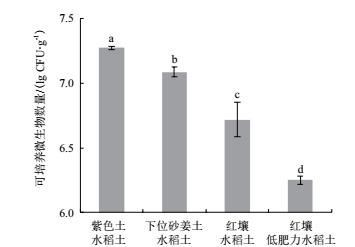
通过检测新分离菌株基因组DNA的nifH基因可以明确其是否具有固氮潜力.结果(图 3)表明, 菌株P204, P205, P207, P208, B205, HR203和LR204共计7个菌株的PCR产物琼脂糖凝胶电泳后在250-500 bp之间均出现明亮条带, 而其余新分离菌株, 阴性对照Escherichia coli与ddH2O无明显条带, 经多次nifH基因PCR扩增检测结果一致, 即在固体无氮培养基上生长的14株菌株中有7株菌株扩增出nifH基因, 占新分离菌株的50%.
|
| M:DL2000 DNA Marker,1:ddH2O,2: Escherichia coli,3:P201,4:P203,5:P204,6 :P205,7 :P207,8 :P208,9 :B201,10 :B202,11 :B205,12 :HR201,13 :HR203,14 :LR201,15 :LR204,16 :LR205 图 3 新分离菌株的nifH基因PCR扩增 |
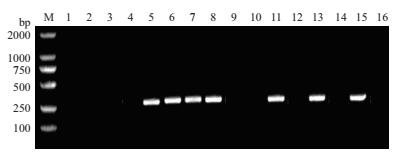
采用乙炔还原法(ARA)测定菌液的乙炔还原活性是定量表征菌株固氮能力的重要方法[37-38, 45-47].在液体Brown无氮培养基中, 菌株P204, P205, P207和P208的乙炔还原活性较高, 对数生长中期菌液在10%乙炔中培养2 h即可检测到适宜计算的乙烯峰, 其他10株菌株在10%乙炔中培养24 h后检测到的乙烯峰较低或未检测到乙烯峰.对数生长中期菌液在10%乙炔中培养2 h的测定结果如图 4所示, 菌株P208的乙炔还原活性最高, 显著高于参比菌株ATCC 9043.
|
| 图 4 菌株的乙炔还原活性 |
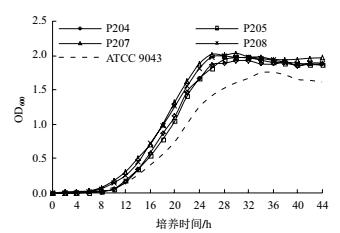
分离纯化得到的14株菌株中, 菌株P204, P205, P207和P208生长较快, 在28℃条件下划线培养2 d后即可长出稳定的单菌落, 1%接种于液体无氮培养基后28℃, 180 r/min培养24 h内可观察到菌液浑浊.其他菌株(除菌株P204, P205, P207和P208外)在28℃条件下划线培养通常需要5 d才可以看到明显菌落, 将这些菌株单菌落接种于液体无氮培养基(3 mL)中, 28℃, 180 r/min培养, 难以较快(5 d内)生长至菌液浑浊状态.
如图 5所示, 菌株P204, P205, P207, P208和参比菌株ATCC 9043在1%接种后于28℃, 180r/min培养, 12 h后能通过OD600检测到明显生长, 菌株P204, P205, P207和P208在26 h后达到稳定生长期, 生长活力较强的时间段为12-24 h.
|
| 图 5 菌株在无氮培养基中的生长曲线 |
结合基因组DNA的nifH基因扩增, 乙炔还原活性和无氮培养基上的生长状况等, 确定菌株P204, P205, P207和P208为本次分离筛选工作得到的优势固氮菌株.
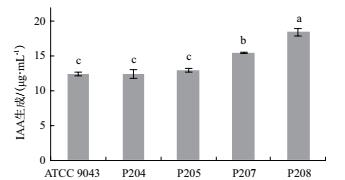
2.7 优势固氮菌株的促生潜力 2.7.1 IAA生成IAA的生成能力是促进植物生长细菌普遍具有的特征之一.如图 6所示, 优势固氮菌株P204, P205, P207, P208与参比菌株ATCC9043均具有IAA生成的能力, 其中菌株P208的IAA生成能力显著高于其他菌株.
|
| 图 6 菌株的IAA生成 |
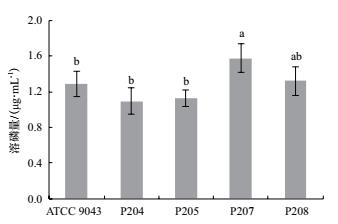
溶磷活性是促进植物生长细菌的重要促生机制之一.如图 7所示, 在含有难溶磷酸盐磷酸三钙的NBRIP培养基中培养7 d后, 优势固氮菌株P204, P205, P207, P208和参比菌株ATCC9043的液体培养液中均检测到可溶性磷含量的增加, 表明其具有一定的溶磷活性.
|
| 图 7 菌株的溶磷活性 |
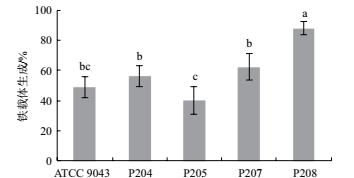
铁载体生成测定值大于50%的菌株为能力较强的菌株.如图 8所示, 优势固氮菌株P204, P205, P207, P208和参比菌株ATCC 9043均具有铁载体生成的能力, 其中P208的铁载体生成能力显著高于其他菌株, 活性单位高达88.21%, 而P205的铁载体合成能力较弱.
|
| 图 8 菌株的铁载体生成 |
从不同生境和样品来源中能够分离出不同适应能力和功能的固氮菌株.根据不同的试验目的, 前人研究中选用尾矿区土壤[48-49], 作物植株[50-51], 林木和作物的根际土壤[52-53]等进行固氮菌株的分离和筛选.本研究中, 为了为稻田生物固氮能力的微生物调控提供菌种资源, 选用稻田土壤作为固氮菌株的分离来源, 得到的菌株大多可归类于固氮菌属(Azotobacter), 芽孢杆菌属(Bacillus), 节杆菌属(Arthrobacter), 伯克霍尔德氏菌属(Burkholderia)等已确定的非共生固氮微生物类群[54].
稀释平板法和富集纯化法是采用选择性培养基分离微生物的主要方法.本研究中, 在较短培养时间(2-3 d)内, 各个稀释度平板上大部分菌落还未明显出现, 但稀释度较低(10-1-10-2)的平板上出现了个别较大的菌落.经过划线纯化, 从这些菌落中能够分离出在固体无氮培养基上快速生长的菌株, 推测这些菌株在土壤中的密度较小, 采用稀释平板法分离时容易在连续稀释过程中遗失.富集纯化法能够将选择性培养基中生长较快的微生物快速富集为优势菌群, 可以避免固氮能力较强但土壤中密度较小的固氮菌株在稀释平板法的稀释过程中遗失, 但得到的菌株类型较少.在微生物分离试验中, 两种方法的结合使用有助于得到相对全面而高效的菌株.不同的培养基和培养条件能分离出不同的微生物菌株[55-56], 本试验中的培养基和培养条件未能在红壤水稻土等3个土壤样品中筛选出固氮能力较强的菌株, 更多培养基和培养条件下的分离筛选工作还有待进一步进行.
琼脂粉是一种较为理想的固体培养基凝固剂, 是包含琼脂糖和琼脂果胶等的混合物.本研究预试验中采用未去杂的生物级琼脂粉作为固体无氮培养基凝固剂, 得到的可培养微生物数量与后期采用琼脂糖作为凝固剂的平板计数相比, 结果显著偏高(图 1, 图 9).因此, 在固氮微生物分离过程中不能直接使用未进行进一步纯化的琼脂粉作为固体无氮培养基的凝固剂, 可在使用前进行水洗去杂(操作繁琐), 或直接采用纯化程度更高的Noble级琼脂(成本较高) [18], 琼脂糖可作为一个较佳的选择.

|
| 图中误差线代表标准差, 相同字母表示差异未达显著水平(P < 0.05), 图 1 无氮培养基上可培养微生物数量 |
|
| 图 9 琼脂粉固化的无氮培养基上可培养微生物数量 |
编码固氮酶铁蛋白亚基的nifH基因在固氮微生物中高度保守, 为明确微生物的固氮潜力提供了依据[57-58].理论上, 经过无氮培养基选择性分离得到的菌株即为以N2为唯一氮源的微生物菌株, 而本研究在无氮培养基上分离得到的14株菌株中, 仅有50%的菌株能够扩增出nifH基因.分析可能与引物和PCR反应程序的选择有关, 引物PolF/PolR能够从大多数代表性固氮微生物基因组中成功扩增出nifH基因[34], 但任何通用引物都无法全面覆盖nifH基因的多样性[59], 这与前人的研究相一致[18].另一方面, 分离得到的菌株也可能包含利用培养基中极微量氮元素和空气中微量氨气作为氮源生存的微生物[18], 这部分菌株在优势固氮菌的筛选过程中逐步被淘汰.本研究中, 所有分离所得菌株同时采用乙炔还原法定量测定了固氮活性, 结合固氮培养基上生长状况等结果筛选优势固氮菌株, 避免了nifH基因引物和PCR反应程序选择对固氮菌株筛选的影响
本研究最终筛选得到优势固氮菌株P204, P205, P207和P208, 4株菌株在乙炔还原活性, IAA生成, 溶磷活性和铁载体生成等方面各有不同, 为同一土壤来源的不同菌株.经过16S rRNA基因相似性和系统发育分析, 可初步鉴定属于固氮菌属(Azotobacter).由于能够产生胞外多糖, 环境适应能力较强[25], 固氮菌属(Azotobacter)具有很高的研究和应用价值.前人研究表明, 除固氮作用外, 固氮菌属(Azotobacter)菌株能够活化土壤中的无机磷, 矿物钾, 提高土壤磷钾的有效性[60-61].陈胜男等[62]研究表明, 接种固氮菌属(Azotobacter)菌株能够提高玉米根际土壤脲酶活性和细菌总代谢活性.Romero-Perdomo等[20]的研究结果显示, 接种固氮菌属(Azotobacter)菌株能够促进棉花植株的生长, 减少氮肥施用量.另外, 该属菌株还可应用于生物表面活性剂, 功能性多糖等生物技术产品的生产[25, 63].本试验中分离筛选得到的固氮菌属(Azotobacter)菌株具有较强的固氮能力和促生潜力, 特别是菌株P208, 乙炔还原活性为模式菌株A. chroococcum ATCC 9043T的2.18倍, IAA生成能力为ATCC 9043T的1.48倍, 铁载体活性单位高达88.21%, 具有很好的开发应用前景.
4 结论共分离纯化得到异养菌株14株, 为土壤微生物研究和应用提供了丰富的试验材料和微生物菌种资源.在新分离纯化得到的菌株中, 菌株P204, P205, P207和P208在无氮培养基上生长速率较快, 乙炔还原活性较高, 且具有IAA生成, 溶磷活性和铁载体生成的促生潜力, 具有很好的应用研究潜力, 可用于进一步功能基因开发和微生物接种应用研究.
| [1] |
Galloway JN, Dentener FJ, Capone DG, et al. Nitrogen cycles :past, present, and future[J]. Biogeochemistry, 2004, 70(2): 153-226. |
| [2] |
Fowler D, Coyle M, Skiba U, et al. The global nitrogen cycle in the twenty-first century[J]. Philosophical Transactions of the Royal Society B-Biological Sciences, 2013, 368(1621): 20130164. |
| [3] |
Smil V. Nitrogen and food production :Proteins for human diets[J]. Ambio, 2002, 31(2): 126-131. |
| [4] |
Tilman D, Cassman KG, Matson PA, et al. Agricultural sustainability and intensive production practices[J]. Nature, 2002, 418(6898): 671-677. |
| [5] |
Erisman JW, Sutton MA, Galloway J, et al. How a century of ammonia synthesis changed the world[J]. Nature Geoscience, 2008, 1(10): 636-639. |
| [6] |
Storkey J, Macdonald AJ, Poulton PR, et al. Grassland biodiversity bounces back from long-term nitrogen addition[J]. Nature, 2015, 528(7582): 401-404. |
| [7] |
Erisman JW, Galloway JN, Seitzinger S, et al. Consequences of human modification of the global nitrogen cycle[J]. Philosophical Transactions of the Royal Society B-Biological Sciences, 2013, 368(1621): 20130116. |
| [8] |
Hitz K, Clark AJ, Van Sanford DA. Identifying nitrogen-use efficient soft red winter wheat lines in high and low nitrogen environments[J]. Field Crops Research, 2017, 200: 1-9. |
| [9] |
Guarda G, Padovan S, Delogu G. Grain yield, nitrogen-use efficiency and baking quality of old and modern Italian bread-wheat cultivars grown at different nitrogen levels[J]. European Journal of Agronomy, 2004, 21(2): 181-192. |
| [10] |
Dai XL, Zhou XH, Jia DY, et al. Managing the seeding rate to improve nitrogen-use efficiency of winter wheat[J]. Field Crops Research, 2013, 154: 100-109. |
| [11] |
Fageria NK, Baligar VC. Enhancing nitrogen use efficiency in crop plants[J]. Advances in Agronomy, 2005, 88: 97-185. |
| [12] |
Dawson JC, Huggins DR, Jones SS. Characterizing nitrogen use efficiency in natural and agricultural ecosystems to improve the performance of cereal crops in low-input and organic agricultural systems[J]. Field Crops Research, 2008, 107(2): 89-101. |
| [13] |
Keuter A, Veldkamp E, Corre MD. Asymbiotic biological nitrogen fixation in a temperate grassland as affected by management practices[J]. Soil Biology and Biochemistry, 2014, 70: 38-46. |
| [14] |
Fowler D, Steadman CE, Stevenson D, et al. Effects of global change during the 21st century on the nitrogen cycle[J]. Atmospheric Chemistry and Physics, 2015, 15(24): 13849-13893. |
| [15] |
Peoples MB, Herridge DF, Ladha JK. Biological nitrogen fixation : An efficient source of nitrogen for sustainable agricultural production?[J]. Plant and Soil, 1995, 174(1-2): 3-28. |
| [16] |
Olivares J, Bedmar EJ, Sanjuan J. Biological nitrogen fixation in the context of global change[J]. Molecular Plant-Microbe Interactions, 2013, 26(5): 486-494. |
| [17] |
Reed SC, Cleveland CC, Townsend AR. Functional ecology of freeliving nitrogen fixation :a contemporary perspective[J]. Annual Review of Ecology, Evolution, and Systematics, 2011, 42: 489-512. |
| [18] |
Dahal B, Nandakafle G, Perkins L, et al. Diversity of free-Living nitrogen fixing Streptomyces in soils of the badlands of South Dakota[J]. Microbiological Research, 2017, 195: 31-39. |
| [19] |
Ladha JK, Tirol-Padre A, Reddy CK, et al. Global nitrogen budgets in cereals :A 50-year assessment for maize, rice, and wheat production systems[J]. Scientific Reports, 2016, 6: 19355. |
| [20] |
Romero-Perdomo F, Abril J, Camelo M, et al. Azotobacter chroococcum as a potentially useful bacterial biofertilizer for cotton (Gossypium hirsutum):Effect in reducing N fertilization[J]. Revista Argentina De Microbiologia, 2017, 49(4): 377-383. |
| [21] |
Jalilian J, Modarres-Sanavya SAM, Saberali SF, et al. Effects of the combination of beneficial microbes and nitrogen on sunflower seed yields and seed quality traits under different irrigation regimes[J]. Field Crops Research, 2012, 127: 26-34. |
| [22] |
van Elsas JD, Duarte GF, Rosado AS, et al. Microbiological and molecular biological methods for monitoring microbial inoculants and their effects in the soil environment[J]. Journal of Microbiological Methods, 1998, 32(2): 133-154. |
| [23] |
Kizilkaya R. Yield response and nitrogen concentrations of spring wheat( Triticum aestivum )inoculated with Azotobacter chroococcum strains[J]. Ecological Engineering, 2008, 33(2): 150-156. |
| [24] |
Wang X, Liu B, Ma J, et al. Soil aluminum oxides determine biological nitrogen fixation and diazotrophic communities across major types of paddy soils in China[J]. Soil Biology and Biochemistry, 2019, 131: 81-89. |
| [25] |
Gauri SS, Mandal SM, Mondal KC, et al. Enhanced production and partial characterization of an extracellular polysaccharide from newly isolated Azotobacter sp. SSB81[J]. Bioresource Technology, 2009, 100(18): 4240-4243. |
| [26] |
Brown ME, Burlingham SK, Jackson RM. Studies on Azotobacter species in soil :1Comparison of media and techniques for counting Azotobacter in soil[J]. Plant and Soil, 1962, 17(3): 309-319. |
| [27] |
Aquilanti L, Favilli F, Clementi F. Comparison of different strategies for isolation and preliminary identification of Azotobacter from soil samples[J]. Soil Biology and Biochemistry, 2004, 36(9): 1475-1483. |
| [28] |
Solanki MK, Wang Z, Wang FY, et al. Intercropping in sugarcane cultivation influenced the soil properties and enhanced the diversity of vital diazotrophic bacteria[J]. Sugar Tech, 2017, 19(2): 136-147. |
| [29] |
Kayasth M, Gera R, Dudeja SS, et al. Studies on salinization in Haryana soils on free-living nitrogen-fixing bacterial populations and their activity[J]. Journal of Basic Microbiology, 2014, 54(3): 170-179. |
| [30] |
Lane DJ. 16S /23S rRNA sequencing[M]. Stackebrandt E, Goodfellow M. Nucleic acid techniques in bacterial systematics.New York: Wiley, 1991: 115-175.
|
| [31] |
Kim OS, Cho YJ, Lee K, et al. Introducing EzTaxon-e :a prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62: 716-721. |
| [32] |
Chun J, Lee JH, Jung Y, et al. EzTaxon :a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57: 2259-2261. |
| [33] |
Yoon SH, Ha SM, Kwon S, et al. Introducing EzBioCloud :a taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J]. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(5): 1613-1617. |
| [34] |
Poly F, Monrozier LJ, Bally R. Improvement in the RFLP procedure for studying the diversity of nifH genes in communities of nitrogen fixers in soil[J]. Research in Microbiology, 2001, 152(1): 95-103. |
| [35] |
Li DJ, Zhang QS, Xiao KC, et al. Divergent responses of biological nitrogen fixation in soil, litter and moss to temperature and moisture in a karst forest, southwest China[J]. Soil Biology and Biochemistry, 2018, 118: 1-7. |
| [36] |
Hyman MR, Arp DJ. Quantification and removal of some contaminating gases from acetylene used to study gas-utilizing enzymes and microorganisms[J]. Applied and Environmental Microbiology, 1987, 53(2): 298-303. |
| [37] |
Hardy RW, Holsten RD, Jackson EK, et al. The acetylene-ethylene assay for N2 fixation :laboratory and field evaluation[J]. Plant Physiology, 1968, 43: 1185-1207. |
| [38] |
Hardy RWF, Burns RC, Holsten RD. Applications of acetyleneethylene assay for measurement of nitrogen fixation[J]. Soil Biology and Biochemistry, 1973, 5(1): 47-81. |
| [39] |
Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of proteindye binding[J]. Analytical Biochemistry, 1976, 72(1): 248-254. |
| [40] |
Glickmann E, Dessaux Y. A critical examination of the specificity of the salkowski reagent for indolic compounds produced by phytopathogenic bacteria[J]. Applied and Environmental Microbiology, 1995, 61(2): 793-796. |
| [41] |
Nautiyal CS. An efficient microbiological growth medium for screening phosphate solubilizing microorganisms[J]. FEMS Microbiology Letters, 1999, 170(1): 265-270. |
| [42] |
Murphy J, Riley JP. A modified single solution method for the determination of phosphate in natural waters[J]. Analytica Chimica Acta, 1962, 27: 31-36. |
| [43] |
Collavino MM, Sansberro PA, Mroginski LA, et al. Comparison of in vitro solubilization activity of diverse phosphate-solubilizing bacteria native to acid soil and their ability to promote Phaseolus vulgaris growth[J]. Biology and Fertility of Soils, 2010, 46(7): 727-738. |
| [44] |
Schwyn B, Neilands JB. Universal chemical assay for the detection and determination of siderophores[J]. Analytical Biochemistry, 1987, 160(1): 47-56. |
| [45] |
Bellenger JP, Xu Y, Zhang X, et al. Possible contribution of alternative nitrogenases to nitrogen fixation by asymbiotic N2-fixing bacteria in soils[J]. Soil Biology and Biochemistry, 2014, 69: 413-420. |
| [46] |
Bellenger JP, Wichard T, Xu Y, et al. Essential metals for nitrogen fixation in a free-living N2-fixing bacterium :chelation, homeostasis and high use efficiency[J]. Environmental Microbiology, 2011, 13(6): 1395-1411. |
| [47] |
Priya H, Prasanna R, Ramakrishnan B, et al. Influence of cyanobacterial inoculation on the culturable microbiome and growth of rice[J]. Microbiological Research, 2015, 171: 78-89. |
| [48] |
李雯, 阎爱华, 黄秋娴, 等. 尾矿区不同植被恢复模式下高效固氮菌的筛选及Biolog鉴定[J]. 生态学报, 2014, 34(9): 2329-2337. |
| [49] |
Navarro-Noya YE, Hernández-Mendoza E, Morales-Jiménez J, et al. Isolation and characterization of nitrogen fixing heterotrophic bacteria from the rhizosphere of pioneer plants growing on mine tailings[J]. Applied Soil Ecology, 2012, 62: 52-60. |
| [50] |
李艳星, 郭平毅, 孙建光. 块根块茎类作物内生固氮菌分离鉴定,系统发育与促生特性[J]. 中国农业科学, 2017, 50(1): 104-122. |
| [51] |
Barraquio WL, Revilla L, Ladha JK. Isolation of endophytic diazotrophic bacteria from wetland rice[J]. Plant and Soil, 1997, 194: 15-24. |
| [52] |
孙建光, 胡海燕, 刘君, 等. 农田环境中固氮菌的促生潜能与分布特点[J]. 中国农业科学, 2012, 45(8): 1532-1544. |
| [53] |
徐秀倩, 吴小芹, 吴天宇, 等. 林木根际细菌JYZ-SD5的促生抗逆性能及种类鉴定[J]. 生物技术通报, 2019, 35(3): 31-38. |
| [54] |
Franche C, Lindstrom K, Elmerich C. Nitrogen-fixing bacteria associated with leguminous and non-leguminous plants[J]. Plant and Soil, 2009, 321: 35-59. |
| [55] |
Li HB, Singh RK, Singh P, et al. Genetic diversity of nitrogen-fixing and plant growth promoting Pseudomonas species isolated from sugarcane rhizosphere[J]. Frontiers in Microbiology, 2017, 8: 1268. |
| [56] |
Kirchhof G, Reis VM, Baldani JI, et al. Occurrence, physiological and molecular analysis of endophytic diazotrophic bacteria in gramineous energy plants[J]. Plant and Soil, 1997, 194(1-2): 45-55. |
| [57] |
Coelho MRR, Marriel IE, Jenkins SN, et al. Molecular detection and quantification of nifH gene sequences in the rhizosphere of sorghum ( Sorghum bicolor )sown with two levels of nitrogen fertilizer[J]. Applied Soil Ecology, 2009, 42(1): 48-53. |
| [58] |
Zhan J, Sun QY. Diversity of free-living nitrogen-fixing microorganisms in the rhizosphere and non-rhizosphere of pioneer plants growing on wastelands of copper mine tailings[J]. Microbiological Research, 2012, 167(3): 157-165. |
| [59] |
Gaby JC, Buckley DH. A comprehensive evaluation of PCR primers to amplify the nifH gene of nitrogenase[J]. PLoS One, 2012, 7(7). |
| [60] |
张亮, 袁玲, 黄建国. 自生固氮菌对土壤钾的活化作用[J]. 土壤学报, 2015, 52(2): 399-405. |
| [61] |
张亮, 杨宇虹, 李倩, 等. 自生固氮菌活化土壤无机磷研究[J]. 生态学报, 2013, 33(7): 2157-2164. |
| [62] |
陈胜男, 谷洁, 付青霞, 等. 接种自生固氮菌对玉米根际土壤酶活性和细菌群落功能多样性的影响[J]. 植物营养与肥料学报, 2012, 18(2): 444-450. |
| [63] |
Thavasi R, Subramanyam Nambaru VR, Jayalakshmi S, et al. Biosurfactant production by Azotobacter chroococcum isolated from the marine environment[J]. Marine Biotechnology, 2009, 11: 551-556. |