基因表达调控是分子生物学研究的重要领域,主要包括转录调控、转录后调控、mRNA翻译、翻译后调控4个方面[1]。随着RNA-seq、RNA技术的成熟,RNA转录后调控机制成为新的研究热点[2]。转录后调控包括RNA包装加工、RNA可变剪接、RNA定位、RNA稳定性、RNA翻译等过程[3],每个环节都需要RNA体内[5]。在微生物中,普城沙雷氏菌G3的RNA结合蛋白Hfq(a host factor for RNA phage)促进YadA(Yersinia adher-ence protient)和InvA(Invasion on as-sociated protein)mRNA与核糖体的结合,进而促进了两种粘连蛋白YadA和InvA的产生,增强菌体的适应性[6]。在植物中,RNA结合蛋白HOS5(High Osmotic Stress Gene Expression 5)结合RS40(Serine/arginine-rich proteins)的前体mRNA,调节其可变剪切,确保水稻对盐胁迫的耐受[7]; 甘氨酸富集的RNA结合蛋白AtRZ-1b结合FLC(Flowering Locus)的mRNA促进FLC第一个内含子高效剪接从而促进拟南芥植物开花[8]。在动物中,人类线粒体RNA结合蛋白C6orf203抑制OXPHOS(Oxidative phosphorylation)mRNA与核糖体结合,进而抑制OXPHOS蛋白的合成[9]。直肠癌细胞RNA结合蛋白HUR(Hu antigen R)通过肠上皮的核仁磷酸蛋白调节Rac1(Ras-related C3botulinum toxin substrate 1)mRNA的核质穿梭[10]。另外,EBP1蛋白通过与AR(Androgen receptor)mRNA 3'-UTR富含UC的motif相互作用,促进ARmRNA的降解,同时EBP1还与AR mRNA 5'编码区的CAG茎环结合,抑制 AR 的翻译,从而抑制前列腺癌细胞的生长[11]。
EBP1是受体酪氨酸激酶ErbB3(Erythroblastic leukemia viral oncogene homolog)的磷酸化底物,是调控动植物生长发育、应激反应重要的DNA和RNA结合蛋白。在动物中,EBP1作为DNA结合蛋白与E2F1(E2F transcription factor1)基因的启动元件相互作用,抑制E2F1基因转录,进而抑制细胞生长[12]。另外,EBP1也可作为RNA结合蛋白,以核糖核蛋白的形式发挥功能,不仅参与不同种rRNA的成熟过程[13],也可以绑定mRNA并调控翻译。例如,人类EBP1蛋白通过富含赖氨酸的基序361SASRKTQKKKKKKAS375与口蹄疫病毒的核糖体进入位点(The internal ribosome entry site,IRES)相互作用,促进48S起始复合物的形成[14]; EBP1也可以通过σ70-like motif(46-54氨基酸)结合5SrRNA,调控其成熟过程[15]。EBP1蛋白在动物和植物中结构和功能高度保守,已有报道EBP1在植物中也可以直接绑定DNA[16]。拟南芥EBP1蛋白直接绑定CML38启动子片段,抑制CML38基因表达[16]。最新质谱结果显示拟南芥EBP1参与核糖体生物生成、蛋白质翻译等过程,可以绑定mRNA、rRNA、snoRNA、tRNA等多种RNA[17]。但已报道的EBP1的靶基因甚少,EBP1绑定RNA的结构域、参与转录后调控具体机制目前仍不清楚。本研究通过RNAEMSA(RNA electrophoretic mobility shift assay)等实验证实EBP1体外结合RNA,并通过生物信息学预测了EBP1结合RNA的结构域,RIP实验找到了3个靶基因RNA(AT1G24792、AT3G25211、AT3G24320)。为了研究EBP1对靶RNA的转录后调控,虫草素处理实验发现,EBP1促进AT1G24792、AT3G24320的mRNA的降解,而对AT3G25211的mRNA稳定性没有影响。通过核糖体提取实验发现EBP1抑制AT1G24792、AT3G24320的mRNA与核糖体结合,而促进AT3G25211的mRNA与核糖体结合。以上实验结果表明,拟南芥EBP1作为一个RNA结合蛋白,参与调控下游靶基因mRNA稳定性与翻译速率,为研究该蛋白的生物学功能提供了新的方向。
1 材料与方法 1.1 材料 1.1.1 主要材料拟南芥野生型Col-0、ebp1-1(SALK_030408)、ebp1-2(SALK_052695)、ebp1-3(CS854731)、35S∷ EBP1-GFP植物材料以及EBP1-pGEX-4T-1 BL21的菌种由中南大学生命科学学院李驰宇提供。将拟南芥种子经表面消毒后,点播在pH为5.8、含0.8%琼脂的1/2 MS固体培养基上,4℃春化3 d后,于22℃,长日照(16 h光照/8 h黑暗)垂直放置培养7 d进行后续实验。
1.1.2 主要试剂GST-Resin(货号:B074B)购于徐州博天生物有限公司; 异丙基-β-D-硫代半乳糖(IPTG)(货号:A100487-0005)、卡那霉素(货号:A506636-0025)以及氨苄青霉素(Amp)(货号:410V033)、还原性谷胱甘肽(货号:A100399-0025)、PMSF(货号:4100745-0005)均购于生工生物工程上海有限公司; Protein GFP Trap(货号:SA070005)购于常州天地人和生物有限公司; 蛋白酶抑制剂(货号:B14001)、磷酸酶抑制剂(货号:B15001)购于Selleck Chemicals; RNA酶抑制剂(货号:N251A)、SYBR Premix Taq kit(货号:SQ121)均购于一诺唯真生物公司,Thermo逆转录试剂盒(货号:K168);FITC修饰探针擎科生物公司合成,其他生化试剂为国产分析纯。
1.2 方法 1.2.1 生物信息学分析EBP1一级结构域预测:将TAIR网站https://www.arabidopsis.org/查找的拟南芥EBP1(AT3G51800)的氨基酸序列导入Pfam网站(http://pfam.xfam.org)预测EBP1蛋白质结构域。EBP1晶体结构预测:将EBP1氨基酸序列输入Phyre2http://www.sbg.bio.ic.ac.uk/phyre2网站进行EBP1晶体三维结构预测分析,通过PyMOL软件进行颜色、结构旋转等修饰。RNA多序列比对分析:利用NCBI网站https://blast.ncbi.nlm.nih.gov/Blast.cgi 的BLAST功能,以“GUCUCUCACUGCGACGGCUU”序列为模版,进行RNA多序列比对,获得包含此RNA序列的潜在靶基因,并下载包含“GUCUCUCACUGCGACGGCUU”长度为60 bp的潜在靶mRNA序列,用Clustal X软件(Invitrogen公司)进行Alignment件,再用Bio-Edit进行颜色、字体、间距的美化。RIP实验引物设计:在TAIR网查找以上潜在靶基因的cDNA,再通过Primer Premier 5软件设计包含潜在结合区域的引物,于擎科生物公司合成,用于后续实验(引物列表见表 1)。
将包含EBP1-pGEX-4T-1或pGEX-4T-1的BL21大肠杆菌蛋白表达菌株,以1:1 000的比例接种于5 mL LB液体培养基中(含100 μg/mL Amp)中,37℃,200 r/min培养12-16 h后,以1:100接种于200 mL含有100 μg/mL Amp的LB液体培养基中,37℃,200 r/min培养,至OD600 =0.8,加入工作浓度为0.5 mmol/L的蛋白表达诱导剂IPTG,28℃,100 r/min诱导6 h。
1.2.3 GST蛋白纯化将诱导好的EBP1-GST菌液分装于50 mL离心管,4℃,5 000 r/min离心10min,去除上清。加入7 mL蛋白裂解缓冲液(50mmol/L Tris,150 mmol/L NaCl,1 mmol/L PMSF,pH 7.5)重悬菌体,冰上超声破碎EBP1-GST菌体(功率200W,超声6 s,冷却9 s,重复破碎约15 min至裂解液澄清)。超声破碎后的悬液于4℃,6 000 r/min离心5 min,将上清转移至10 mL Ep管中。取500 μLGST纯化柱(GST agarose),用1 mL蛋白裂解缓冲液重悬GST纯化柱,4℃,100 × g离心1 min,去除上清,并重复该步骤3次洗去保护液。去除上清后,转移到装有细菌破碎液的10 mL Ep管,4℃旋转孵育8 h。孵育后的液体,4℃,1 000 r/min离心1min,去除上清,并将GST纯化柱转移到2 mL Ep管,加入1 mL蛋白漂洗缓冲液(50 mmol/L Tris,300mmol/L NaCl)重悬,4℃颠倒5 min,以洗去非特异性结合的蛋白,此过程重复3次。离心去上清后加入1 mL蛋白洗脱缓冲液(20 mmol/L Tris pH 7.5,20mmol/L GSH,150 mmol/L NaCl),4℃洗脱3 h。取10 μL纯化的蛋白,进行SDS-PAGE凝胶电泳考马斯亮蓝染色检测。
1.2.4 EBP1-GST与总RNA结合实验8 μL纯化的EBP1-GST蛋白(以GST蛋白作为对照)加到100 μL总蛋白绑定缓冲液(150 mmol/L NaCl,20mmol/L Tris-HCl pH 7.4,0.5 mmol/L EDTA,蛋白酶抑制剂)中,然后在总蛋白绑定缓冲液中分别加入50 μL的GST纯化柱(已用总蛋白绑定缓冲液洗3次),4℃结合3 h后,用总蛋白绑定缓冲液洗3次,每次5 min,洗掉杂蛋白,备用。用TRIzol降解(质量合格的RNA有3条带,从大到小分别是28S、18S、5.8S)。将结合有EBP1-GST、GST蛋白的GST纯化柱以及没有孵育蛋白的GST纯化柱中加入20 μL的RNA和100 μL总蛋白绑定缓冲液(包含8 U/mL RNA酶抑制剂)室温孵育30 min,其余RNA存放于-80℃冰箱,用1 mL总蛋白绑定缓冲液将GST纯化柱洗3次,洗去未结合的RNA,利用TRIzol法提取样品中的RNA,取2 μL -80℃储存的RNA,与其他实验样品一起用琼脂糖凝胶电泳检测RNA含量,同时测定RNA浓度及OD260值[18]。
1.2.5 RIP实验称取生长7 d的EBP1-GFP植物幼苗3 g,置于50 mL离心管中,用清水洗3次,加入35 mL 0.5%甲醛,立即真空固定5 min(压力为9MPa),释放真空,继续固定8 min,然后加入1.5 mL2 mol/L甘氨酸,抽真空1 min,释放真空,再抽真空5min(终止甲醛交联反应),释放真空,清水洗5次,用吸水纸去除水分,液氮速冻。将幼苗在液氮中充分研磨,加入500 μL总蛋白提取液(20 mmol/L TrisHCl pH 8.0,5 mmol/L EDTA,5 mmol/L EGTA,0.05%SDS,10 mmol/L DTT,1 mmol/L PMSF,1%蛋白酶抑制剂,8 U/mL RNA酶抑制剂),冰上静置1 h。4℃,12 000 r/min离心10 min,收集上清到新的1.5 mL Ep管,取100 μL植物抽提液作为Input,其余样品分成两份,一份作为对照组,一份作为实验组。取两份40 μL GFP-Tarp,用RIP结合缓冲液(20 mmol/LTris-HCl pH 8,150 mmol/L NaCl,2 mmol/L EDTA,0.1%SDS,1% TritionX-100)洗3次,去上清,加入300μL RIP结合缓冲液和1 mL Chip稀释缓冲液(16.7mmol/L Tris-HCl pH 8,167 mmol/L NaCl,1.2 mmol/LEDTA,1.1% TritionX-100,8 U/mL RNA酶抑制剂,1%蛋白酶抑制剂),再分别加入实验组和对照组的植物抽提液,4℃结合过夜。将结合有蛋白的GFP-Tarp用RIP结合缓冲液洗3次以洗去杂蛋白,去上清后加入50 μL RIP洗脱缓冲液(100 mmol/L Tris-HCl pH8.0,100 mmol/L EDTA,0.1% SDS,1 U/mL RNA酶抑制剂),65℃振荡10 min,离心后将上清转移至新的Ep管,再向GFP-Tarp中加入50 μL RIP洗脱缓冲液,65℃振荡10 min,将两次上清合并,在Input以及上清加入1 μL 14 mg/mL蛋白酶K,56℃放置1 h,释放结合的RNA,之后用TRIzol法提取RNA,逆转录获得cDNA,作为进一步qRT-PCR的模板,以上试剂均用DEPC水配制。
1.2.6 RNA-EMSA实验将100 ng纯化的EBP1-GST蛋白(GST空载蛋白作为对照)和100 pg FITC荧光修饰的RNA探针(不带FITC修饰的RNA竞争探针作为对照)加入RNA-EMSA结合缓冲液(10mmol/L Tris,5%甘油,1 mmol/L MgCl2,50 mmol/LKCl,0.2 mg/mL BSA,0.5 mmol/L DTT,0.5 mg/mLpolyglutamate pH 7.5)中,室温放置20 min。用6.5%非变性聚丙烯酰胺凝胶(6.5%凝胶储液,10%甘油,1%过硫酸铵,TEMED,0.5×TBE)先在冰上预电泳0.5 h,冲洗胶孔,上样,电泳1 h后,用KODAK4000M Image Station显影[19]。
1.2.7 虫草素(cordycepin)处理实验取1/2 MS固体培养基上生长7 d的拟南芥幼苗,浸没于CRD处理缓冲液(1 mmol/L哌嗪-1,4-二乙磺酸,1 mmol/L柠檬酸钠pH 6.25,1 mmol/L KCl,15 mmol/L蔗糖,150 μg/mL虫草素)中,抽真空3 min后,继续放置于22℃光照处理15 min、30 min、45 min后,提取RNA并逆转录成cDNA,用于后续qRT-PCR检测[20]。
1.2.8 核糖体提取实验取生长7 d的拟南芥幼苗1g,用纸吸干,液氮充分研磨后,每个样加入500 μL提取液(0.2 mol/L Tris-HCl pH 7.5,50 mmol/L KCl,25 mmol/L MgCl2,50 μg /mL放线菌酮,50 μg/mL虫草素,400 U/mL RNA酶抑制剂,10 μL蛋白酶抑制剂Cocktail,1 mmol/L PMSF)冰上抽提30 min,同时在预冷的50 mL离心管铺制10%-60%梯度的蔗糖,将植物提取液4℃,12 000 r/min离心10 min。取上清轻轻滴加在已铺好的蔗糖上层,同时将没有植物样品各梯度蔗糖作为对照。4℃,32 000 r/min离心4 h。离心后取不同梯度组分,测定各个组分OD260吸光值。提取45%-60%蔗糖梯度组分的RNA,逆转录得到的cDNA用于后续qRT-PCR检测[21]。
1.2.9 RNA提取与qRT-PCR用TRIzol法提取拟南芥总RNA后,通过逆转录试剂盒逆转录成cDNA,用Bio-rad定量PCR仪(C1000 TouchTM Thermal Cycler)进行qRT-PCR实验。
2 结果 2.1 EBP1蛋白结合RNA的结构域分析拟南芥EBP1一级结构域预测结果发现,在48-56氨基酸区域存在结合RNA的结构域(图 1-A)。同时我们在Phyre2网站预测了其晶体结构,发现人类与拟南芥EBP1于蛋白表面均含有两个封闭的环状结构(Loop)。拟南芥EBP1蛋白48-56氨基酸区域就位于Loop1环结构中,并且此序列与人类EBP1报道的结合RNA的σ70-like motif(46-54氨基酸)序列类似(图 1-B),这暗示EBP1在结构上具有结合RNA的潜能。
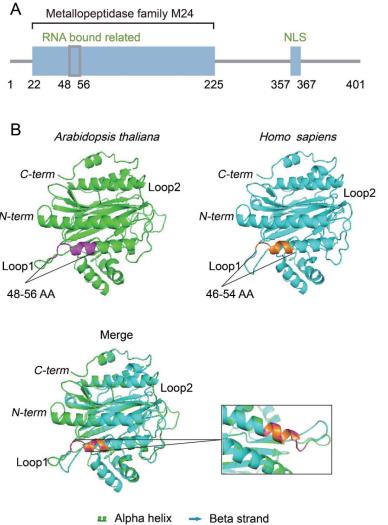
|
| A :拟南芥中EBP1蛋白一级结构域预测。1-401氨基酸序列是EBP1氨基酸总长,22-225氨基酸序列为金属肽合酶M24家族结构域(Metallopeptidasefamily M24)。48-56氨基酸为EBP1与RNA的结合区域。NLS :nuclearlocalization signal。B:EBP1蛋白在拟南芥(Arabidopsis thaliana)和人类(Homo sapiens)中结构保守; 呈现的图为EBP1的带状晶体结构,Loop为闭合环状区域; N-term :N末端; C-term :C末端; Alpha helix : α螺旋; Beta strand :β折叠。粉色线条为拟南芥EBP1 48-56氨基酸区域所处位置,橙色线条为人类EBP1 46-54氨基酸区域 图 1 EBP1蛋白结构域分析 |
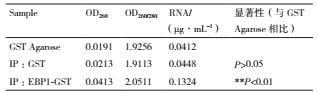
为了进一步验证EBP1蛋白是否可以直接结合RNA,我们首先进行了总RNA结合实验(图 2),实验结果显示:GST蛋白结合的泳道(第三泳道)与对照组GST纯化柱结合的泳道(第二泳道)RNA条带亮度基本一致,EBP1-GST蛋白结合的RNA条带(第四泳道)与第二泳道相比亮度明显增强(图 2-A)。定量分析显示,对照组GST纯化柱、GST蛋白和EBP1-GST蛋白结合的RNA浓度分别是0.041 2μg/μL、0.044 8 μg/μL、0.132 4 μg/μL(表 2)。以上实验数据表明EBP1-GST蛋白结合RNA的浓度明显高于对照组(P <0.05),而GST蛋白与对照组基本一致(P>0.05),即EBP1-GST可以结合部分RNA,GST蛋白不能结合RNA。
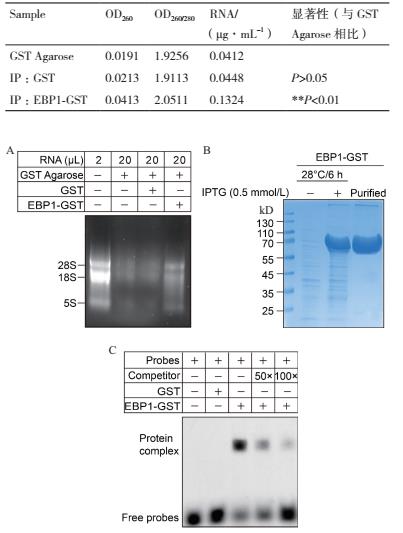
|
| A :EBP1蛋白结合RNA。第一泳道为从Col-0植物提取的总RNA,第二泳道为GST纯化柱结合的RNA(阴性对照),第三泳道为GST蛋白结合的RNA,第四泳道为EBP1-GST蛋白结合的RNA; B :EBP1-GST蛋白纯化图,诱导条件是28℃诱导6 h; C :EBP1结合特定的RNA序列。Probes是FITC修饰的“GUCUCUCACUGCGACGGCUU”RNA片段。Competitor是没有修饰的“GUCUCUCACUGCGACGGCUU”序列。实验进行3次生物学重复,结论一致 图 2 EBP1蛋白与RNA直接相互作用 |
进一步表达并纯化了EBP1-GST蛋白(图 2-B),然后合成FITC修饰的探针“GUCUCUCACUGCGACGGCUU”(Probes)以及不修饰的探针“GUCUCUCACUGCGACGGCUU”(Competitor),通过与EBP1-GST、GST蛋白孵育,同时用50倍和100倍的不修饰的特异性探针(Competitor)进行RNAEMSA实验。实验结果表明:对照组GST纯化柱(第一泳道)和GST(第二泳道)蛋白与探针孵育的泳道没有出现迁移的电泳条带,EBP1-GST与探针孵育泳道(第三泳道),显示出明显迁移的电泳条带,且条带的强度随着竞争性探针(Competitor)浓度的升高而减弱(第四、五泳道)。表明GST蛋白不能结合RNA,而EBP1蛋白可以直接绑定“GUCUCUCACUGCGACGGCUU”这一RNA序列(图 2-C)。
2.3 在拟南芥体内EBP1绑定RNA为了进一步验证拟南芥EBP1在体内结合RNA,首先通过NCBI网站Blast找到12个基因含有“GUCUCUCACUGCGACGGCUU”序列(图 3-A):AT1G24792、AT3G24320、AT1G25211、AT1G07150、AT1G11810、AT5G55950、AT3G13830、AT3G14400、AT3G17265、AT3G21120、AT3G63200、AT3G24700,利用实验室已有的EBP1-GFP转基因过表达植株,结合RIP技术,对这12个潜在EBP1靶mRNA进行了RIP-qRT-PCR验证。结果显示: AT1G24792被对照组GFP-TraP免疫沉淀的RNA相对含量是2.623 40,被实验组EBP1-GFP免疫沉淀的RNA相对含量5.094 07; AT3G24320被免疫沉淀的RNA在对照组和实验组的相对含量分别是2.936 38、13.318 90。AT1G25211被免疫沉淀的RNA在对照组和实验组的相对含量分别是3.462 92、9.613 54,这3个基因的mRNA被EBP1-GFP免疫沉淀后相比对照组有显著性差异(AT1G24792,P <0.05;AT3G24320,P <0.001; AT1G25211,P <0.01),其他9个基因被EBP1-GFP免疫沉淀的RNA相比对照没有显著性差异(P>0.05)(图 3-B)。表明EBP1结合AT1G24792、AT3G24320、AT1G25211的RNA,并可能参与其转录后调控过程。
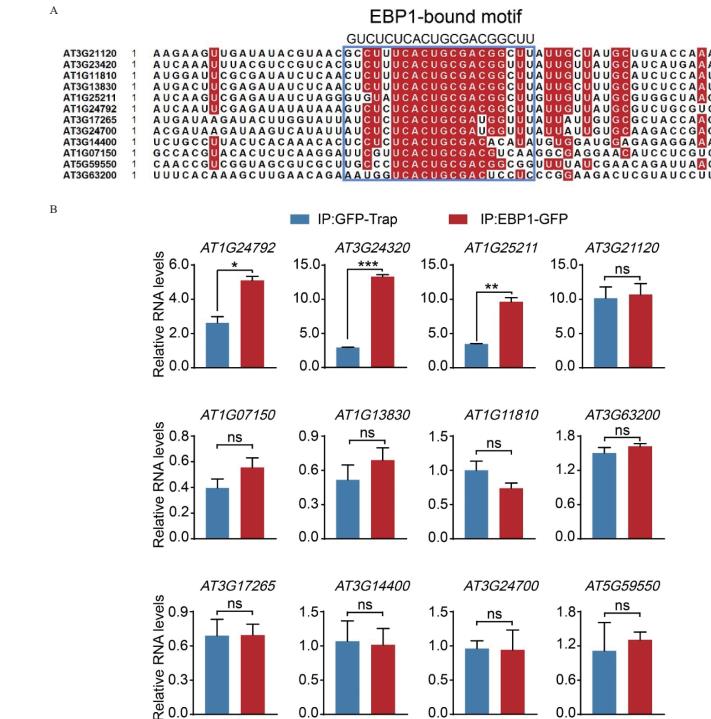
|
| A :EBP1潜在绑定的RNA片段序列比对。蓝色方框所示为与“GUCUCUCACUGCGACGGCUU”对应的RNA序列; B :RIP-qRT-PCR筛选EBP1的靶mRNA基因。EBP1-GFP蛋白用GFP-Trap免疫沉淀获得,以Col-0的蛋白结合GFP-Trap作为阴性对照。图B实验进行3次独立生物学重复,并获得了相似的实验结果。显著性差异分析采用t检验,*P<0.05,**P<0.01,***P<0.001,ns表示无显著性差异 图 3 EBP1在体内与特定基因的mRNA相互作用 |
为了研究EBP1对靶RNA转录后事件的调控,首先我们分析了EBP1对基因AT1G24792、AT3G24320、AT1G25211在RNA稳定性方面的影响。利用虫草素这种转录抑制剂,结合qRT-PCR技术检测靶RNA的降解速度,进而分析RNA的稳定性。于是对Col-0、ebp1-1、ebp1-2、EBP1-GFP 植株用虫草素处理后,进行qRT-PCR检测。ATHSPRO2 基因是一个已被报道mRNA不稳定的基因,虫草素处理后ATHSPRO2 mRNA会迅速降解,证明虫草素处理有效(图 4-A)。Col-0、ebp1-1、ebp1-2、EBP1-GFP 植株在虫草素处理0 min时这3个靶基因的mRNA含量均为1。相对于0 min mRNA含量,EBP1突变后AT1G24792 mRNA的含量在虫草素处理30 min、45 min分别为0.574 7、0.218,而对照组Col-0中AT1G24792 mRNA分别是0.33、0.03,过表达EBP1-GFP AT1G24792 mRNA含量在30 min、45min分别为0.301 58、0.046 03(图 4-B); EBP1突变后AT3G24320 mRNA在处理15 min、30 min后含量分别为0.222、0.191 1,对照组Col-0中AT3G24320mRNA在虫草素处理15 min、30 min时的相对含量是0.642 8、0.33,过表达EBP1-GFP AT3G24320mRNA含量在15 min、30 min分别为0.202 5、0.13(图 4-C); EBP1突变后虫草素处理15 min、45 min后AT1G25211 mRNA相对含量分别是0.807 3、0.218 4,Col-0被虫草素处理15 min、45 min后AT1G25211mRNA相对含量分别是0.802 0、0.263 9(图 4-D)。以上数据表明:EBP1促进AT1G24792、AT3G24320mRNA的降解,而对AT1G25211mRNA的稳定性没有影响。
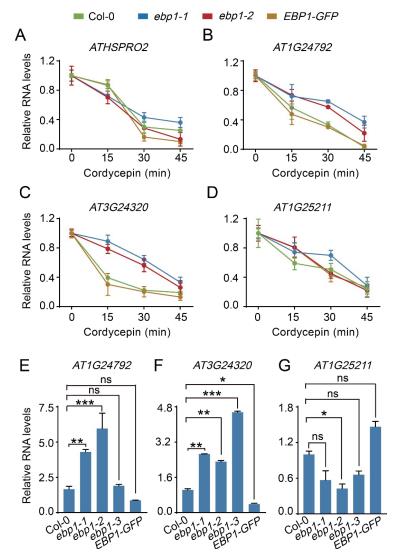
|
| A-D :qRT-PCR实验检测150 μg/mL Cordycepin处理后不同时间梯度样本中特定靶mRNA的含量。ATHSPRO2基因为阳性对照。内参基因使用EIF4A1。图中数值表示+ /-标准差。E-G :在Col-0、ebp1突变体和EBP1-GFP植株中的特定靶mRNA含量。Actin用作内参基因,数据所示为+ /-标准差平均值。实验进行了3次独立生物学重复,并获得了相似的实验结果。统计方法为one-way ANOVA,*P<0.05,**P<0.01,***P<0.001,ns表示无显著性差异 图 4 EBP1对AT1G24792、AT3G24320、AT1G25211mRNA稳定性的影响 |
进一步我们通过qRT-PCR手段检测Col-0、ebp1-1 、ebp1-2 、ebp1-3、EBP1-GFP植物中靶mRNA的含量(图 4-E-G)。数据显示AT1G24792 mRNA在Col-0、ebp1-1 、ebp1-2 、ebp1-3、EBP1-GFP的相对含量为1.660 57、4.297 34、5.955 5、1.904 86、0.873 17,即AT1G24792 mRNA在ebp1-1 、ebp1-2 中明显增多(P <0.05),而在过表达中mRNA没有显著改变(P>0.05)(图 4-E)。AT3G24320的mRNA相对含量在Col-0、ebp1-1 、ebp1-2 、ebp1-3、EBP1-GFP分别为1.034 72、2.661 91、2.310 78、4.561 03、0.392 15,即EBP1突变后AT3G24320 mRNA的含量增加(P <0.05),EBP1过表达AT3G24320 mRNA的含量减少(P <0.05)(图 4-F)。AT1G25211的mRNA相对含量在Col-0、ebp1-1 、ebp1-2 、ebp1-3、EBP1-GFP分别为1、0.567 20、0.426 91、0.659 82、1.466 62,表明EBP1对AT1G25211的mRNA含量没有显著影响(P>0.05)(图 4-G)。
综合RNA稳定性和植物中靶mRNA表达量检测的实验结果,我们发现EBP1突变后AT1G24792、AT3G24320 的mRNA稳定性增强,从而使这些基因的mRNA含量增加,即EBP1可以调控靶RNA的稳定性。
2.5 EBP1调节靶mRNA的翻译速率为了探究在拟南芥中EBP1是否对核糖体的成熟、mRNA的翻译速率起调节作用,本实验通过蔗糖密度梯度离心分离核糖体来检测EBP1对3个靶基因mRNA翻译速率的影响。数据显示: AT1G24792在ebp1-1的多聚核糖体组分(8-11)中mRNA的相对含量分别是1.53、0.95、1.71、1.02,在Col-0植物的多聚核糖体组分(8-11)中相对含量是0.94、0.43、1、0.91(图 5-B); AT3G24320在ebp1-1的多聚核糖体组分(8-11)中mRNA的相对含量分别是2.31、4.69、4.39、2.62,在Col-0植物的多聚核糖体组分(8-11)中相对含量是0.95、2.34、2.65、1.38(图 5-C); AT1G25211在ebp1-1的多聚核糖体组分(8-11)中mRNA的相对含量分别是0.76、0.75、1.15、1.05,在Col-0植物的多聚核糖体组分(8-11)中mRNA的相对含量是1.28、1.91、1.51、1.39(图 5-D); 以上数据表明EBP1突变与对照Col-0比可以促进多聚核糖体组分中AT1G24792、AT3G24320 mRNA增多(P <0.05),而多聚核糖体结合AT1G25211的mRNA明显减少(P <0.05),即EBP1抑制AT1G24792、AT3G24320的mRNA与核糖体的结合,促进AT1G25211 mRNA与核糖体的结合。因此植物中EBP1也可以调控mRNA的翻译过程。
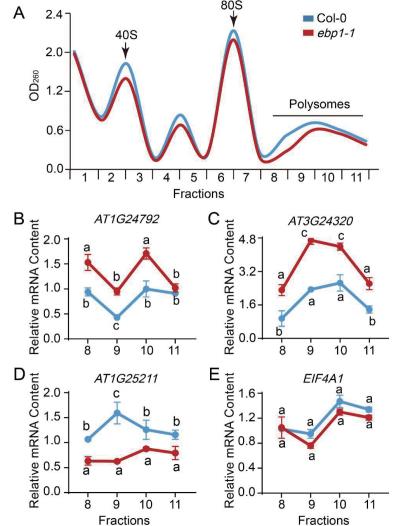
|
| A:各核糖体组OD260吸光度值曲线。40S、80S和多聚核糖体组分已标示。B-E:qRT-PCR检测多聚核糖体组分中 AT1G24792、AT3G24320、AT1G25211 和 EIF4A1 mRNA的含量。Actin用作内参基因,显著性差异分析采用one-wayANOVA(单因素方差分析)方法。a和b之间 P <0.05,a和c之间 P <0.01 图 5 EBP1调节靶mRNA的翻译效率 |
RBPs通过与RNA相互作用在转录后调控过程中发挥重要作用,从而调节细胞功能。植物拥有复杂的RBPs系统,参与调节植物生长的多个过程,包括调控开花时间[22]、激素反应[23]、生物钟[24]、生物[25]及非生物胁迫[26]等过程。研究RBPs的生物学功能并解析其调控的分子机制将有助于挖掘新的功能基因,为农作物改良提供新的种质资源。
RNA与蛋白质的相互作用通常是通过一个特异的RNA识别基序实现的,Foley等[27]利用蛋白质测序技术对拟南芥有根毛和无根毛细胞核中的RBPs进行测序,结果发现“GUCUCUCACUGCGACGGCUU”可能是EBP1潜在的靶RNA序列。本研究发现拟南芥EBP1蛋白可以直接绑定“GUCUCUCACUGCGACGGCUU”RNA序列,并且找到了EBP1的3个靶基因RNA(AT1G24792、AT3G24320、AT1G-25211),进一步研究发现EBP1可以调控AT1G24792和AT3G24320的mRNA稳定性,对AT1G25211的mRNA稳定性没有明显影响。在此过程中EBP1如何实现对靶RNA的特异性识别与结合,是否还有更多的靶RNA没有被发现,今后可综合RIP-seq等技术,筛选更多与EBP1相互作用的RNA。另外,RNA结合蛋白通过识别位于mRNA 3'UTR富含AU的区域途径或者识别含有提前终止密码子的mRNA的NMD(Nonsense-mediated mRNA decay)等途径来影响靶RNA的稳定性[28]。然而EBP1是如何调控AT1G24792和AT3G24320的mRNA稳定性的目前还不清楚,还需要进一步实验研究。
EBP1在动植物中是受体酪氨酸激酶ErbB3磷酸化调控的核质穿梭蛋白[29-30]。哺乳动物中,ErbB3是HRG/NRG(Heregulin/neuregulin)小肽的受体[31]。在乳腺癌细胞中,当HRG配体小肽结合ErbB3受体时,ErbB3与EBP1相互作用,使EBP1磷酸化并促使其在细胞核积累,发挥抑制细胞生长,促进细胞分化的功能[32]。植物中EBP1作为核质穿梭的转录因子,其功能已有研究。CrRLK1L(Catbarantbus roseus RLK1-like)成员FERONIA感知RALF1(RapidAlkalinization Factor 1)小肽信号后,FERONIA与EBP1相互作用,使EBP1磷酸化并在细胞核积累,EBP1入核后抑制 CML38的转录[16]。EBP1磷酸化状态的改变是否影响其对转录后事件的调控仍不清楚。EBP1对RNA稳定性、mRNA翻译的调控过程是否受到上游磷酸化蛋白调控也需要进一步研究。报道显示拟南EBP1与核糖体的结合分布于细胞质、细胞核及核仁之间,且EBP1可以结合不同种类的RNA[17],EBP1蛋白在调控植物器官大小、逆境胁迫等多个方面发挥功能[16],那么,EBP1在不同的位置结合核糖体所行使的功能有何差异,其是否是通过改变蛋白构象或结合特定的基序来识别不同的RNA,从而实现不同的功能?所以,结合遗传学多层次探究EBP1在RNA水平调控的生物学意义,是今后需要关注和研究的重点。
4 讨论本研究通过运用生物信息学分析结合分子生物学手段,发现拟南芥EBP1作为一个RNA结合蛋白,在RNA转录后水平调控过程中发挥功能。EBP1可以直接绑定“GUCUCUCACUGCGACGGCUU”RNA基序; 生物信息学分析结合RIP-qRT-PCR技术,发现了EBP1的3个靶基因RNA( AT1G24792、AT3G24320、AT1G25211); 进一步通过虫草素处理发现拟南芥EBP1促进 AT1G24792、AT3G24320mRNA的降解,而对 AT1G25211 mRNA的稳定性没有影响。核糖体提取实验发现,EBP1抑制 AT1G24792、AT3G24320与核糖体的结合,而促进 AT1G25211与核糖体结合。因此,EBP1在拟南芥中可以与RNA相互作用,并具有调控靶RNA的稳定性和翻译速率的生物学功能。
| [1] |
Lipshitz HD, Claycomb JM, Smibert CA. Post-transcriptional regulation of gene expression[J]. Methods, 2017, 126: 1-2. |
| [2] |
陈伟, 许馨, 孙绍光. RNA结合蛋白与RNA相互作用鉴定技术[J]. 中国生物化学与分子生物学报, 2017, 8(2): 6-10. |
| [3] |
Singh G, Pratt G, Yeo GW, et al. The clothes make the mRNA :past and present trends in mRNP fashion[J]. Annual Review of Biochemistry, 2015, 84(1): 325-354. |
| [4] |
张在宝, 李婉杰, 李九丽, 等. 植物RNA结合蛋白研究进展[J]. 中国农业科学, 2018, 51(21): 6-18. |
| [5] |
Glisovic T, Bachorik JL, Yong J, et al. RNA-binding proteins and post-transcriptional gene regulation[J]. FEBS Letters, 2008, 582(14): 1977-1986. |
| [6] |
郭英飞, 袁玖云, 王玉飞, 等. 细菌RNA结合蛋白Hfq研究进展[J]. 解放军预防医学杂志, 2015, 33(5): 584-586. |
| [7] |
Chen T, Cui P, Chen H, et al. A KH-domain RNA-binding protein interacts with FIERY2/CTD phosphatase-like 1 and splicing factors and is important for pre-mRNA splicing in Arabidopsis [J]. PLoS Genetics, 2013, 9(10). |
| [8] |
Zhe DZ, Lin XY, Jin M. RNA binding proteins RZ-1B and RZ-1C play critical roles in regulating pre-mRNA splicing and gene expression during development in Arabidopsis [J]. Plant Cell, 2016, 28(1): 55-73. |
| [9] |
Shreekara G, Sarah FP, Adam MD, et al. C6orf203 is an RNAbinding protein involved in mitochondrial protein synthesis[J]. Nucleic Acids Research, 2019, 47(17): 17. |
| [10] |
Liu L, Xiao L, Chung HK, et al. RNA-binding protein HuRregulates Rac1 nucleocytoplasmic shuttling through nucleophosmin in the intestinal epithelium[J]. Cellular and Molecular Gastroenterology and Hepatology, 2019, 8(3): 475-486. |
| [11] |
Zhou H, Mazan Mamczarz K, Martindale JL, et al. Posttranscriptional regulation of androgen receptor mRNA by an ErbB3 binding protein 1 in prostate cancer[J]. Nucleic Acids Research, 2010, 38(11): 3619-3631. |
| [12] |
Zhang Y, Hamburger AW. Heregulin regulates the ability of the ErbB3-binding protein Ebp1 to bind E2F promoter elements and repress E2F-mediated transcription[J]. Journal of Biological Chemistry, 2004, 279(25): 26126-26133. |
| [13] |
Zhou H, Zhang Y, Hamburger AW. EBP1 inhibits translation of androgen receptor mRNA in castration resistant prostate cancer cells[J]. Anticancer Research, 2011, 31(10): 3129-3135. |
| [14] |
Squatrito M, Mancino M, Sala L, et al. Ebp1 is a dsRNAbinding protein associated with ribosomes that modulates eIF2α phosphorylation[J]. Biochemical & #38; Biophysical Research Communications, 2006, 344(3): 859-868. |
| [15] |
Monie TP, Perrin AJ, Birtley JR, et al. Structural insights into the transcriptional and translational roles of Ebp1[J]. EMBO Journal, 2007, 26(17): 3936-3944. |
| [16] |
Li C, Liu X, Qiang X, et al. EBP1 nuclear accumulation negatively feeds back on FERONIA-mediated RALF1 signaling[J]. PLoS Biology, 2018, 16(10). |
| [17] |
Lokdarshi A, Papdi C, Pettko-Szandtner A, et al. ErbB3 binding protein 1 regulates translation and counteracts retinoblastoma related to maintain the root meristem[J]. Plant Physiology, 2020, 182(2): 919-932. |
| [18] |
Squatrito M, Mancino M, Donzelli M, et al. EBP1 is a nucleolar growth-regulating protein that is part of pre-ribosomal ribonucleoprotein complexes[J]. Oncogene, 2004, 23(25): 4454-4465. |
| [19] |
Liu H, Yu X, Li K, et al. Photoexcited CRY2 interacts with CIB1 to regulate transcription and floral initiation in Arabidopsis [J]. Science, 2008, 322(5907): 1535-1539. |
| [20] |
Seeley KA, Dennis H. Red light-independent instability of oat phytochrome mRNA in vivo [J]. Plant Cell, 1992, 4(1): 29-38. |
| [21] |
Li W, Ma M, Feng Y, et al. EIN2-directed translational regulation of ethylene signaling in Arabidopsis [J]. Cell, 2015, 163(3): 670-683. |
| [22] |
Liu F, Quesada V, Crevillén P, et al. The Arabidopsi s RNA-binding protein FCA requires a Lysine-specific demethylase 1 homolog to downregulate FLC[J]. Molecular Cell, 2007, 28(3): 398-407. |
| [23] |
Schmal C, Reimann P, Staiger D. A circadian clock-regulated toggle switch explains AtGRP7 and AtGRP8 oscillations in Arabidopsis thaliana[J]. PLoS Computational Biology, 2013, 9(3). |
| [24] |
Lyons R, Iwase A, G?nsewig T, et al. The RNA-binding protein FPA regulates flg22-triggered defense responses and transcription factor activity by alternative polyadenylation[J]. Scientific Reports, 2013, 3: 2866. |
| [25] |
Kim MH, Sasaki K, Imai R. Cold shock domain protein 3 regulates freezing tolerance in Arabidopsis thaliana[J]. Journal of Biological Chemistry, 2009, 284(35): 23454-23460. |
| [26] |
Yamada H, Mori H, Momoi H, et al. A fission yeast gene encoding a protein that preferentially associates with curved DNA[J]. Yeast, 1994, 10(7): 883-894. |
| [27] |
Foley SW, Gosai SJ, Wang D, et al. A global view of RNA-protein interactions identifies post-transcriptional regulators of root hair cell fate[J]. Developmental Cell, 2017, 41(2): 204-220. |
| [28] |
高媛. RNA结合蛋白调节mRNA稳定性的作用机制[J]. 微生物学免疫学进展, 2012, 40(2): 79-83. |
| [29] |
Sliwkowski MX, Schaefer G, Akita RW, et al. Coexpression of ErbB2 and ErbB3 proteins reconstitutes a high affinity receptor for heregulin[J]. Journal of Biological Chemistry, 1994, 269(20): 14661-14665. |
| [30] |
Carraway KL, Sliwkowski MX, Akita R, et al. The ErbB3 gene product is a receptor for heregulin[J]. Journal of Biological Chemistry, 1994, 269(19): 14303-14306. |
| [31] |
Yoo J, Wang X, Rishi A, et al. Interaction of the PA2G4 (EBP1)protein with ErbB3 and regulation of this binding by heregulin[J]. British Journal of Cancer, 2000, 82(3): 683. |
| [32] |
Lessor TJ, Yoo JY, Xia X, et al. Ectopic expression of the ErbB3 binding protein ebp1 inhibits growth and induces differentiation of human breast cancer cell lines[J]. Journal of Cellular Physiology, 2000, 183(3): 321-329. |