2. 河北农业大学生命科技学院,保定 071000
2. College of Life Science, Hebei Agricultural University, Baoding 071000
山羊是人类驯化较早的动物, 为人们提供肉、绒、毛、皮、奶等多种畜产品, 在畜牧生产中占据重要地位。我国山羊品种资源丰富, 有着悠久的驯化和养殖历史, 就河北省及周边地区就存在多个优良地方山羊品种, 如武安山羊、承德无角山羊、太行山羊、燕山绒山羊、辽宁绒山羊、内蒙古绒山羊和济宁青山羊等。这些地方品种, 无论是在促进当地经济发展还是在促进我国出口创汇中, 均占有重要地位。随着国外羊品种的大量引进及肉用绵羊的大量养殖, 这些优秀的地方山羊品种的养殖规模和存栏数量不断减少, 作为生物多样性的重要组成部分, 亟需加强保护和利用。
有许多研究表明, 分子标记可用于基因多样性分析。到目前为止, 有各种分子标记研究遗传多样性, 例如限制性片段长度多态性(Restriction fragment length polymorphi, RFLP)、随机扩增多态性DNA(Random amplified polymorphic DNA, RAPD)、扩增片段长度多态性(Rmplified fragment length polymorphism, AFLP)、简单重复序列(Simple sequence repeat, SSR)和单核苷酸多态性(Single nucleotide polymorphisms, SNP)等[1-5]。SSR又称为微卫星标记, 由于其共显性遗传、高程度的等位基因多样化、相对多态性丰富和广泛的基因组分布, 可以有效地促进遗传关系的建立, 是分析生物遗传多样性和群体遗传结构的常用方法[6]。
为充分保护和利用地方山羊遗传资源的多样性, 本研究选取15对微卫星位点分析河北省及周边地区7个地方山羊群体的遗传多样性和群体结构, 旨在为地方山羊品种的种质资源保护和利用提供理论依据。
1 材料与方法 1.1 材料采集30只燕山绒山羊(青龙利红绒山羊技术服务中心)、23只济宁青山羊(滦平虎什哈养殖场)、30只承德无角山羊(宽城立东养殖有限公司)、30只太行山羊(阜平县银洞山肉羊养殖专业合作社)、36只武安山羊(武安市马家庄太行饲养场)、31只辽宁绒山羊(辽宁绒山羊科技示范场)的血液样品, 以及33只内蒙古绒山羊(阿拉善型)(内蒙古亿维白绒山羊有限责任公司)的耳组织, 213份样品均为随机取样, 且均为没有亲缘关系的母羊。利用血液基因组DNA提取试剂盒(DP348)天根生化科技有限公司(北京)提取基因组DNA(图 1)。
|
| 1-24:部分样品结果;M:DL2000 图 1 DNA琼脂糖凝胶电泳图 |
参照联合国粮农组织(FAO)及国际遗传学会(ISAG)的推荐并查阅相关文献, 选用15对荧光标记微卫星引物(表 1), 引物由通用生物系统有限公司合成(安徽)。
PCR扩增的反应体系为:ddH2O 14.8 µL, dNTP 0.4 µL, Buffer 2 µL, F 0.3 µL(20 µmol/L), R 0.3 µL(20 µmol/L), DNA模板2 µL, Taq 0.2 µL, 共20 µL。PCR反应条件为:94℃预变性5 min; 94℃变性30 s, 54℃退火35 s, 72℃延伸40 s, 共35个循环; 最终72℃延伸3 min(图 2)。
|
| 1-24为部分PCR产物结果;M:DL2000。 图 2 PCR产物琼脂糖凝胶电泳图 |
将甲酰胺与分子量内标按100:1的体积比混匀后, 取15 µL加入上样板中, 再加入1 µL稀释10倍的PCR产物, 然后使用3730XL测序仪进行毛细管电泳(图 3)。
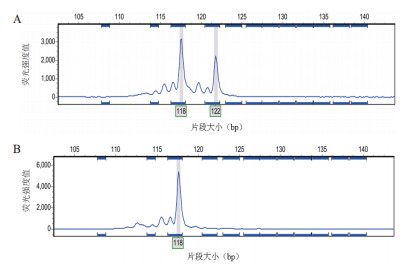
|
| A和B为部分结果图其余未列出,2个峰代表位点的基因型为118/122 bp,为杂合子;1个峰,代表位点的基因型为118/118 bp,为纯合子 图 3 毛细管电泳检测部分结果 |
利用Genemarker中的Fragment(Plant)片段分析软件对测序仪得到的原始数据进行分析, 将各泳道内分子量内标的位置与各样品峰值的位置做比较分析, 得到片段大小。把15个微卫星位点的等位基因片段大小进行编号, 由大到小依次为1-21。利用Popgene32计算等位基因频率、有效等位基因数(Effective numbers of alleles, Ne)、期望杂合度(Expected heterozygosity, He)、观测杂合度(Observed heterozygosity, Ho)、F统计量(Fit和Fst)、基因流(Gene flow, Nm)、Shannon指数(I)等指标。利用PIC-Calc软件计算多态信息含量(Polymorphism information content, PIC)。利用Dispan软件统计各群体Nei’s遗传距离, 并聚类分析。利用MEGA4.0软件构建群体UPGMA系统发育树。
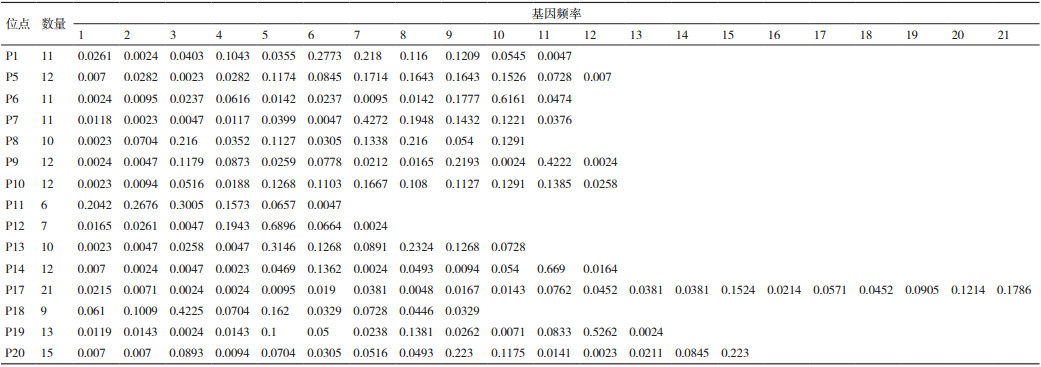
2 结果 2.1 7个地方山羊品种群体内遗传变异分析7个地方山羊品种15个微卫星位点的等位基因频率分析结果见表 2。由表 2可知, 15个位点共检测到了172个等位基因。其中, BM6526位点的等位基因最多, 为21个, 片段大小为152-191 bp; INRA063位点的等位基因最少, 为6个, 片段大小为169-179 bp。在所检测到的等位基因中ILSTS005位点181 bp片段基因频率最高为0.689 6, 其次为BM1225位点221 bp片段, 基因频率为0.669;而OarFCB48标记的163 bp片段、OarFCB20标记的112 bp片段、CSRD247标记的248 bp片段、ILSTS087标记的164 bp片段、TGLA53标记的158 bp片段、BM1225标记的252 bp片段和BMS574标记的122 bp片段的基因频率最低, 均为0.002 3。
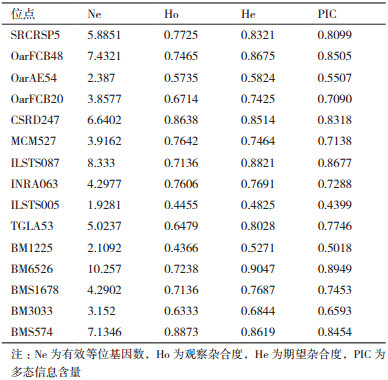
7个地方山羊品种15个微卫星位点的多态性分析结果见表 3。由表 3可知, 所有微卫星位点的有效等位基因数(Ne)范围为1.928 1(ILSTS005)-10.257(BM6526), 观察杂合度(Ho)范围为0.436 6(BM1225)- 0.887 3(BMS574), 期望杂合度(He)范围为0.482 5(ILST005)-0.904 7(BM6526), 平均多态信息含量(PIC)范围为0.439 9(ILSTS005)-0.894 9(BM6526), 均属于高度多态。
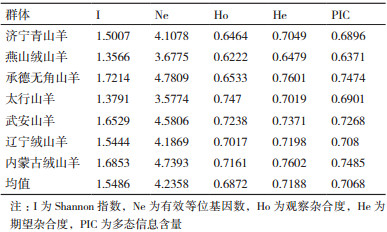
7个地方山羊品种15个微卫星位点的遗传变异分析结果见表 4。由表 4可知, 7个地方山羊群体15个微卫星位点Shannon指数(I)均大于1, 有效等位基因数在3-5之间, 等位基因数较多, 有较高的观察杂合度和较高的期望杂合度, 平均多态信息含量范围为0.637 1-0.748 5, 为高度多态。
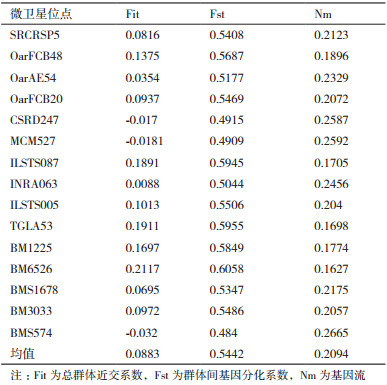
各微卫星位点F-统计量分析见表 5。由表 5可知, 7个山羊群体的平均群体间基因分化系数(Fst)为0.544 2, 平均基因流(Nm)为0.209 4, 总群体近交系数(Fit)为0.088 3。
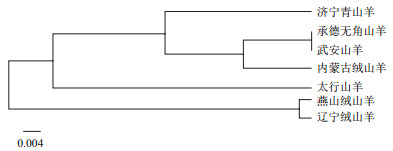
由表 6可知, 7个地方山羊品种的相似系数范围为0.764 8-0.927 4, 遗传距离范围为0.075 4-0268 2。武安山羊和承德无角山羊相似系数最大(0.927 4), 遗传距离最近(0.075 4); 太行山羊和燕山绒山羊相似系数最小(0.764 8), 遗传距离最远(0.268 2)。同时, 依据遗传相似系数对7个地方山羊群体进行聚类分析, 结果见图 4, 7个山羊群体被聚为2类, 济宁青山羊、承德无角山羊、武安山羊、内蒙古绒山羊和太行山羊聚为一类, 燕山绒山羊和辽宁绒山羊聚为一类。
|
| 图 4 7个地方山羊群体聚类系统发育树 |
等位基因频率是衡量动物群体遗传结构、估计遗传变异的重要指标[7], 群体中频率最高、片段最大的等位基因是该物种最保守的基因, 而其余等位基因则是在进化过程中由该基因突变产生的[8]。王可等[9]利用12个微卫星标记研究济宁青山羊群体遗传多样性, 发现SRCRSP8位点的230 bp和232 bp片段的基因频率为0.281 3和0.322 9, 远大于236 bp和240 bp的基因频率0.010 4, 认为前两个片段可能为原始基因, 后两个片段可能是突变基因。本试验也发现了基因频率相差较大的片段, 如ILSTS005位点181 bp片段基因频率为0.689 6, 173 bp片段基因频率为0.002 4, 推测181 bp片段可能为原始基因, 173 bp片段可能为突变基因。有效等位基因数可反映群体遗传变异, 其数值越接近等位基因数, 等位基因在群体中分布越均匀[10]。本试验发现BM6526位点等位基因数最多(21个)片段大小为152-191 bp, 说明BM6526位点变异程度大, 对研究山羊选育更有价值。武志娟等[11]利用14个微卫星标记分析5个黑山羊群体, 发现其中有11个位点的等位基因数和有效等位基因数差异大, 认为这种不平衡可能是由于地理隔离和人工选择导致的。Lin等[12]研究亚洲10个山羊群体的遗传多样性和遗传结构发现, 群体离驯养中心越远, 其遗传多样性渐渐下降。王可等[13]研究济宁青山羊4个群体同样发现等位基因数要高于Ne。Hines等[14]认为等位基因分布不均可能与近交有关。本试验中15个微卫星位点等位基因数与Ne相差较多, 片段大小不同, 频率分布不均匀, 说明7个地方山羊群体有丰富的遗传多样性。
基因杂合度与品种的变异程度具有正相关关系, 即基因杂合度越大, 品种的变异程度越大[15]。若一个群体的平均杂合度大于0.5, 表明该群体的遗传多样性丰富[16]。郭丹等[17]利用7个微卫星位点分析辽宁绒山羊遗传多样性, 发现群体的平均杂合度为0.638, 说明辽宁绒山羊品种内的遗传变异处于较高水平, 选择潜力大。赵艳红[18]分析阿拉善型内蒙古绒山羊的遗传多样性, 发现群体的平均杂合度为0.613 6, 同样说明内蒙古绒山羊遗传多样性丰富。本研究中, 所检测的群体内平均杂合度比较高, 说明群体的遗传变异较大, 适应环境的能力较强。并且, 不同微卫星在同一山羊群体, 同一微卫星在不同山羊群体中变异程度也有很大差别。BM6526位点的He最高为0.904 7, ILSTS005位点相对其它微卫星位点较低为0.482 5;燕山绒山羊最低为0.647 9, 内蒙古绒山羊的He最高为0.760 2。7个群体中仅太行山羊Ho > He, 其他6个群体均是Ho < He, 说明除太行山羊外, 其他6个群体均存在不同程度的近交, 承德无角山羊近交程度高于其他群体。王可等[9]研究济宁青山羊遗传多样性发现, 各标记的观测杂合度在0.428 6-0.904 8之间, 期望杂合度在0.598 1-0.839 7之间, 平均期望杂合度为0.757 4, 属于高度杂合, 遗传变异大。I是研究物种多样性的指标之一, 与群体的遗传变异程度有正相关关系, 即I值越大, 群体的遗传变异程度越高[19]。本试验中, 7个地方山羊群体I值均大于1, 表明试验群体遗传变异程度较高, 受近交的影响较小。多态信息含量(PIC)反映了微卫星标记的变异程度和估计群体内的遗传变异程度, PIC < 0.25时为低度多态, 0.25 < PIC < 0.5时为中度多态, PIC > 0.5时为高度多态[20], PIC > 0.7时认为是最优的遗传标记[21]。本试验的15个微卫星位点仅有ILSTS005为中度多态, 其余均为高度多态, 有11个微卫星位点大于0.7, 说明试验地方山羊群体具有丰富的遗传基础, 有很大的保种潜力。
3.2 7个地方山羊品种的遗传距离及聚类分析Fst反映了群体间的遗传分化程度, Fst在0-0.05时, 各群体间遗传分化很小, 在0.05-0.15时, 为中等程度的遗传分化, 在0.15-0.25时, 为高度遗传分化; Nm数值小于1时, 群体可能向遗传分化的方向发展, Nm数值大于1时, 群体能有效地抑制由遗传漂变引起的遗传分化[22]。Radhika等[23]利用10个微卫星位点分析印度本地山羊和杂交山羊品种的遗传多样性和群体结构发现, 二者均具有高的遗传多样性, 并且Fst值为0.02, 说明群体间遗传分化很小。本试验发现14个微卫星位点在7个山羊群体的Fst值均在0.484以上, 说明群体间的遗传分化程度很大。Nm值均在0.162 7以上, 说明7个群体间的基因交流较少。Fit反应了总群体的近交程度, 表示整个群体中携带的一对等位基因是同源的概率[24]。本试验Fit值为0.088 3, 表明群体间存在8.83%的遗传变异来自远交, 占的比例相对较小。
遗传距离表示不同种群或品种间的基因差异程度, 通常利用等位基因频率计算[25], 是研究物种遗传多样性的基础, 可反映群体的系统进化, 群体分化时间越短, 遗传距离就越小[26]。赵艳红等[27]分析中国6个山羊群体遗传多样性发现辽宁绒山羊和内蒙古绒山羊(阿拉善型)遗传距离为0.134, 与本试验的结果(0.1589)相似。杨清芳[28]利用15个微卫星研究5个山羊品种遗传多样性发现, 辽宁绒山羊与承德无角山羊的遗传距离为0.2270, 与本试验研究结果(0.2341)相似。刘恩民[29]利用SNP芯片分析中国地方山羊品种的遗传结构发现内蒙古绒山羊和太行山羊聚在一大类, 与本试验结果相一致。Alatiyat等[30]利用17个微卫星位点UPGMA聚类分析3种沙特山羊发现, Jabli、Bishi和Tohami聚为一大类。本研究结果显示, 燕山绒山羊与辽宁绒山羊聚为一类, 说明这两个群体遗传距离较近, 燕山绒山羊与太行山羊遗传距离为0.268 2, 相对于其他群体最远。
4 结论燕山绒山羊、辽宁绒山羊、承德无角山羊、济宁青山羊、太行山羊、武安山羊和内蒙古绒山羊7个地方山羊群体遗传多样性丰富, 群体间遗传分化程度大, 基因交流少, 受近交程度影响小, 具有较高的利用价值和潜力。
| [1] |
白俊艳, 曹恒, 王旭, 等. 绵羊GH基因的PvuⅡ位点的多态性与其生长性状的关联分析[J]. 浙江农业学报, 2019, 31(9): 1416-1422. |
| [2] |
高茜.安徽三个湖羊场种群遗传结构的RAPD分析[D].合肥: 安徽农业大学, 2018.
|
| [3] |
徐尚荣. 杜泊羊、藏羊和小尾寒羊AFLP多态性分析[J]. 青海大学学报:自然科学版, 2015, 33(3): 58-63. |
| [4] |
Tefiel H, Ata N, Chahbar M, et al. Genetic characterization of four Algerian goat breeds assessed by microsatellite markers[J]. Small Ruminant Reserve, 2018, 160: 65-71. |
| [5] |
刘家鑫, 魏霞, 邓天宇, 等. 绵羊全基因组ROH检测及候选基因鉴定[J]. 畜牧兽医学报, 2019, 50(8): 1554-1566. |
| [6] |
Carneiro V, Luciane S, Lima D, et al. Microsatellite markers:what they mean and why they are so useful[J]. Genetics and Molecular Biology, 2016, 39(3): 312-328. |
| [7] |
郎侠, 吕潇潇. 兰州大尾羊微卫星DNA多态性研究[J]. 中国畜牧杂志, 2011, 47(1): 14-17. |
| [8] |
樊月圆, 余淑青, 曹振辉, 等. 云南红骨圭山山羊微卫星DNA位点遗传多样性分析[J]. 云南农业大学学报:自然科学版, 2013, 28(3): 329-335. |
| [9] |
王可, 刘昭华, 崔绪奎, 等. 济宁青山羊微卫星标记多态性分析[J]. 山东农业科学, 2017, 49(2): 142-146. |
| [10] |
雒林通, 杨博辉, 史兆国, 等. 甘肃高山细毛羊优质毛品系微卫星标记与经济性状的相关性分析[J]. 江苏农业学报, 2010, 26(2): 331-338. |
| [11] |
武志娟, 陈明华, 万洁, 等. 5个黑山羊品种(群体)微卫星标记的遗传多样性研究[J]. 中国畜牧兽医, 2013, 40(6): 146-152. |
| [12] |
Lin BZ, Kato T, Kaneda M, et al. Genetic diversity and structure in Asian native goat analyzed by newly developed SNP markers[J]. Animal Science Journal, 2013, 84(8): 579-584. |
| [13] |
王可, 刘昭华, 曹洪防, 等. 山东省济宁青山羊群体遗传多样性分析[J]. 山东农业科学, 2019, 51(5): 129-134. |
| [14] |
Hines HC, Zikakis JP, Haenlein GF, et al. Linkage relationships among loci of polymorphisms in blood and milk of cattle[J]. Journal of Dairy Science, 1981, 64(1): 71-76. |
| [15] |
Bai JY, Jia XP, Yang YB, et al. Polymorphism analysis of henan fat-tailed sheep using microsatellite markers.[J]. Journal of Animal & Plant Sciences, 2014, 24(3): 965-968. |
| [16] |
张瑞, 郎侠, 吴建平, 等. 岷县黑裘皮羊微卫星遗传多样性分析[J]. 中国畜牧兽医, 2019, 46(8): 2360-2370. |
| [17] |
郭丹, 韩迪, 王春艳, 等. 辽宁绒山羊7个微卫星位点的遗传多样性分析[J]. 中国畜牧兽医, 2010, 37(12): 99-103. |
| [18] |
赵艳红.利用微卫星标记分析部分山羊品种的遗传多样性[D].呼和浩特: 内蒙古农业大学, 2000.
|
| [19] |
Magurran AE. Biological diversity[J]. Current Biology, 2005, 15(4): 116-118. |
| [20] |
Helentjaris T, Slocum M, Wright S, et al. Construction of genetic linkage maps in maize and tomato using restriction fragment length polymorphisms[J]. Theoretical and Applied Genetics, 1986, 72(6): 761-769. |
| [21] |
Purvis WI, Franklin IR. Major genes and QTL influencing wool production and quality:a review[J]. Genetics Selection Evolution, 2005, 37(S1): 97-107. |
| [22] |
Powell R. Evolution and the genetics of populations:variability within and among natural populations by sewall wright[J]. American Scientist, 1979, 67(1): 108. |
| [23] |
Radhika G, Raghavan KC, Aravindakshan TV, et al. Genetic diversity and population structure analysis of native and crossbred goat genetic groups of Kerala, India[J]. Small Ruminant Research, 2015, 131(2015): 50-57. |
| [24] |
Weir BS, Cockerham CC. Estimation F-statistics for the analysis of population structure[J]. Evolution, 1984, 38: 1358-1370. |
| [25] |
Nei M. Genetic distance[J]. Brenners Encyclopedia of Genetics, 2013, 11(138): 248-250. |
| [26] |
赵东伟, 李春苗, 黎寿丰, 等. 13个地方鸡品种6个微卫星座位遗传多样性分析[J]. 扬州大学学报:农业与生命科学版, 2017, 38(4): 39-45. |
| [27] |
赵艳红, 何晓红, 关伟军, 等. 中国6个山羊群体微卫星标记的遗传多样性分析[J]. 畜牧兽医学报, 2007(1): 20-24. |
| [28] |
杨清芳.五个山羊品种遗传多样性的微卫星分析[D].保定: 河北农业大学, 2011.
|
| [29] |
刘恩民.中国地方山羊品种群体遗传结构和选择信号分析[D].兰州: 兰州大学, 2018.
|
| [30] |
Alatiyat RM, Alobre MM, Aljumaah RS, et al. Microsatellite based genetic diversity and population structure of three Saudi goat breeds[J]. Small Ruminant Research, 2015, 130: 90-94. |