2. 山东第一医科大学(山东省医学科学院)山东省医药生物技术研究中心, 济南 250062
2. Shandong Medicinal Biotechnology Center, Shandong First Medical University & Shandong Academy of Medical Sciences, Jinan 250062
克拉维酸(Clavulanic acid, CA)是一种氧青霉烷类广谱β-内酰胺酶抑制剂, 为治疗β-内酰胺类抗生素耐药菌感染的临床药物。CA由棒状链霉菌(Streptomyces clavuligerus)发酵产生, 发酵条件优化是提高CA产量的有效手段之一[1-3]。
克拉维酸的生物合成途径已经基本阐明。同位素喂养实验证实3-磷酸甘油醛(G3P)和精氨酸是克拉维酸生物合成的前体物质[3-4], 然后经过六步催化反应合成关键中间产物-克拉维胺酸[5], 克拉维胺酸是CA和5S克拉维烷合成代谢的分支点, 在此后的反应中部分克拉维胺酸在克拉维酸合成晚期基因如cad、cyp450和oppA1等作用下合成克拉维酸[6-7], 另一部分则通过5S分支途径形成克拉维烷-2-羧酸、丙氨酰克拉维烷和2-羟甲基克拉维烷等多种5S克拉维烷产物[8-10]。多株S. clavuligerus的基因组已经完成测序[11-14], 研究发现3个基因簇与克拉维酸生物合成有关, 分别为克拉维酸基因簇、克拉维烷基因簇和旁系同源基因簇。克拉维酸基因簇包含克拉维酸生物合成途径多数合成酶的基因, 比如ceaS2、bls2、pah2、cas2、oat2、gcas、car、cyp和fd等, 位于头霉素C合成基因簇的下游; 克拉维烷基因簇包含克拉维酸合成酶基因cas1和5S分支途径的合成基因; 旁系同源基因簇位于内生质粒pSCL4, 包含多个同源基因比如ceas1、bls1、pah1和oat1[14]。此外, CA合成受到复杂的调控作用, ClaR和CcaR是CA合成的途径特异性调控因子[15-16], 而双组份信号转导系统CagRS可以同时调控CA和CA前体物质的合成基因[17], 此外, 研究还显示γ-丁内酯信号系统[18]、上游调控因子BldG[19]、δ-因子Orf21[20]、氨基酸匮乏[21]都影响CA的合成。
S. clavuligerus F613-1是CA的工业生产菌株, 来源于标准菌株ATCC27064的长期育种筛选。工业生产经验表明发酵过程中提高搅拌转速能够提高F613-1菌株的克拉维酸产量和缩短发酵周期, 推测可能与高转速能提高发酵液溶氧水平和传质效果有关, 但并不清楚其具体的分子机制。因此, 本研究拟通过转录组测序比较分析在高转速发酵和低转速发酵条件下F613-1菌株转录组差异, 分析CA合成相关基因簇、前体物质G3P和精氨酸代谢相关基因的变化情况, 以期从转录水平阐明高转速发酵条件影响CA合成的分子机制, 为进一步通过基因工程方法提高CA产量提供依据。
1 材料与方法 1.1 材料 1.1.1 菌株棒状链霉菌S. clavuligerus F613-1。
1.1.2 培养条件F613-1孢子的收集采用BSCA固体培养基(麦芽提取物1.5%, 胰蛋白胨0.3%, 葡萄糖0.4%, 琼脂粉2%, pH7.5), 25℃培养10 d收集孢子。CA液体发酵培养:按106个/mL的接种量将孢子接种于SCZ培养基(大豆超细粉2%, 玉米淀粉1.2%, 酵母自溶物0.5%, 磷酸氢二钾0.08%, 甘油酸三油酯1.1%(V/V), pH 7.1)中, 25℃、250 r/min培养48 h得到种子液, 然后按5%接种量转接至SCF发酵培养基(大豆超细粉2.7%, 大豆蛋白提取物2.2%, 麦芽糊精3.0%, 氯化钾0.15%, 六水氯化镁0.1%, 磷酸氢二钾0.2%, 二水氯化钙0.04%, 六水三氯化铁0.008%, 氯化锌0.001%, 氯化钠0.018%, MOPS 4.2%, 甘油酸三油酯1.6%(V/V), pH 7.1), 25℃培养, 转速分别设为200 r/min和300 r/min。
1.2 方法 1.2.1 发酵液总糖含量和还原糖含量的测定液体发酵过程中分别在发酵0、24、48、72、96、120和144 h取1 mL发酵液测定发酵液中总糖和还原糖浓度, 检测方法参照前期研究进行[22]。
1.2.2 胞内精氨酸含量的测定分别在发酵24、48、72、96、120和144 h取1 mL发酵液, 离心收集菌体, 测量菌体湿重, 然后按照精氨酸(Arg)检测试剂盒(上海纪宁, 96T)测定胞内游离精氨酸的含量。
1.2.3 发酵液CA含量的HPLC检测取1.2.1中发酵液, 离心取上清液后用0.22 μm滤膜过滤, 参照前期研究采用HPLC测定CA含量[22]。
1.2.4 转录组测序与分析收集1.2.1中发酵72 h的发酵液, 采用TRIzol(Ambion)抽提法提取链霉菌F613-1总RNA, 质检合格后去除核糖体RNA并进行片段化处理, 逆转录合成单链cDNA, 再合成双链cDNA并纯化, 接着末端修复并加接头引物后进行PCR扩增并纯化, 采用illuminaHiseq2500测序平台和PE150测序方式进行转录组测序(锐博生物)。原始测序数据首先进行数据质量分析, 然后用参考基因S. clavuligerus ATCC27064(NZ_CM001015.1-NZ_CM001019.1)进行比对。转录组数据采用tbtools软件进行火山图制作, 采用Prism8进行数量差异分析。通过差异倍数(|log2FoldChange| > 1)和显著水平(q-value < 0.001)两个水平判断样本间差异表达的基因。
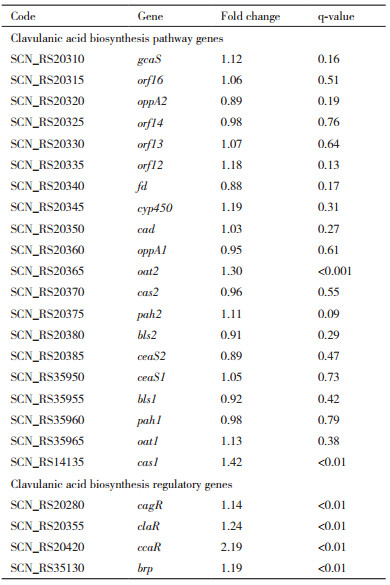
2 结果 2.1 不同转速条件下CA产量的比较每隔24 h收集不同转速条件下的发酵液, 分别进行CA含量、总糖含量和还原糖含量的测定。结果显示在高转速(300 r/min)条件下CA产量显著高于低转速(200 r/min)条件, 其中在48、72和96 h时CA产量的提高趋势最显著, 分别达到0.67、2.15和3.08 g/L, 比低转速条件提高了约2.23倍、2.44倍和1.32倍, 而在发酵后期(120 h和144 h)CA产量的提高幅度降低, 分别约为1.19倍和1.16倍(图 1-A)。高转速条件下F613-1对总糖的消耗速度也显著高于低转速条件(图 1-B), 说明高转速条件下F613-1对糖的消耗更大, 代谢水平更加旺盛。发酵过程中还原糖总体趋势为先升高后降低, 而高转速发酵液在48 h和72 h时还原糖含量的升高幅度显著高于低转速发酵液, 随后还原糖含量快速下降(图 1-B), 与高转速发酵液中CA合成速度的趋势一致, 表明高转速条件下高水平还原糖可能与CA合成速度增加有关, 而且高转速条件能够缩短发酵周期。研究显示糖代谢中间产物3-磷酸甘油醛是克拉维酸合成的前体物质[1,3,23], 因此, 高转速条件下旺盛的糖代谢水平可能是其高产CA的原因之一。
|
| A:发酵过程中CA产量变化; B:发酵过程中总糖和还原糖变化趋势 图 1 不同转速条件下克拉维酸产量比较分析 |
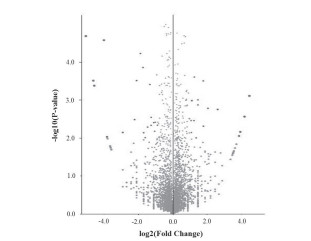
本研究收集了高转速和低转速发酵条件下第72小时(CA合成高峰期)的菌丝体进行转录组测序。转录组测序数据经过质量筛选, 高转速样品平均获得16 721 348个有效mRNA信息, 基因覆盖率为97.04%, 低转速样品平均获得16 154 651个有效mRNA信息, 基因覆盖率为96.84%。通过tbtools软件进行基因转录水平差异分析并绘制样本间基因差异火山图(图 2), 结果显示高转速样品和低转速样品的转录水平之间存在统计意义上的显著差异。与低转速样品相比较, 高转速样品共有789个基因的转录水平发现显著变化, 其中419个基因转录水平显著下降, 370个基因转录水平显著升高, 说明发酵转速在整体上显著影响F613-1菌株的基因转录水平。
|
| 纵坐标代表显著水平, 即-log10(P-value); 横坐标代表(log ratios), 表示log2(Fold Change) 图 2 不同样本间基因差异分析火山图 |
S. clavuligerus F613-1中与克拉维酸合成相关的基因簇共有3个:克拉维酸基因簇、克拉维烷基因簇和旁系同源基因簇。对转录组数据进行差异分析, 结果显示与低转速发酵条件下的转录组数据相比较, 在高转速条件下F613-1中CA合成相关3个基因簇中的大部分基因表达量无明显变化(表 1)。调控CA合成的调节基因claR, cagR和brp的表达量增加了1-1.5倍, 而CA的途径特异性正调控基因ccaR的表达量增加了2.19倍。
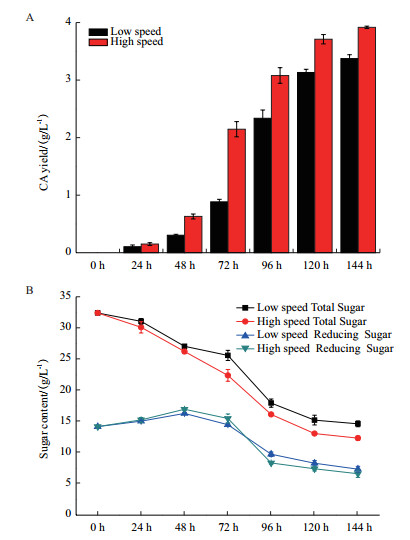
精氨酸和3-磷酸甘油醛(G3P)是CA生物合成途径的2个起始物质。差异分析显示, 与低转速发酵条件下的转录组数据相比较, 在高转速条件下F613-1中精氨酸生物合成基因簇中所有基因(argH, argG, argR, argD, argB, argJ, argC)表达量整体上调大约1-2倍(表 2)。本研究进一步分析了F613-1菌株在发酵不同时间段胞内精氨酸的含量, 数据显示在高转速条件下F613-1胞内的精氨酸含量明显增加(图 3), 与转录组数据一致, 说明F613-1在高转速条件下可以积累更多的精氨酸, 进而促进CA的合成。
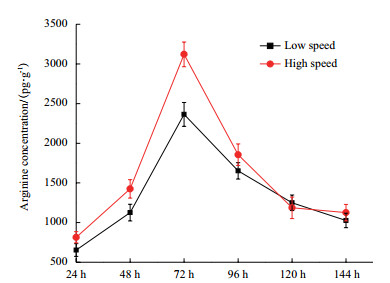
|
| 图 3 不同转速条件下F613-1菌株胞内精氨酸含量比较分析 |
G3P既是CA生物合成途径的直接前体物质, 同时也是糖酵解途径和磷酸戊糖途径的重要中间产物。差异分析显示, 与低转速发酵条件下的转录组数据相比较, 在高转速条件下F613-1中甘油转化途径中3-磷酸甘油醛脱氢酶编码基因(SCN_RS26820)表达量上调3.55倍(图 4), 加速了甘油代谢, 有利于G3P的合成。糖酵解(Glycolysis)途径中多个酶的编码基因表达量下调(图 4-A), 从转录水平看, 尽管通过糖酵解途径产生的G3P和乙酰辅酶A(Acetyl-CoA)下降, 但通过甘油酸三油酯(Glycerol trioleate)代谢途径产生的G3P和Acetyl-CoA却增加。另外, 三羧酸循环(TCA)途径中多个基因的表达量也不同程度的增加(图 4-C), 有利于增强TCA过程。精氨酸是CA生物合成途径的另一个前体物质, 从转录水平看, 在高转速条件下F613-1中精氨酸生物合成基因簇中所有基因(argB, argC, argD, argJ, argG, argH)表达量整体上调大约1-2倍(图 4-B), 有利于增强精氨酸的合成。根据转录组数据和总糖含量测定的实验结果, 本研究绘制了低转速和高转速两种发酵条件下F613-1菌株的代谢通路变化(图 4)。结果显示CA的合成能力与能量代谢正相关, 与低转速发酵条件相比, F613-1菌株在高转速发酵条件下甘油转化产生G3P过程、TCA过程和精氨酸合成途径都有所提高, 因此初级代谢途径的改变可能是CA合成能力增强的原因之一。
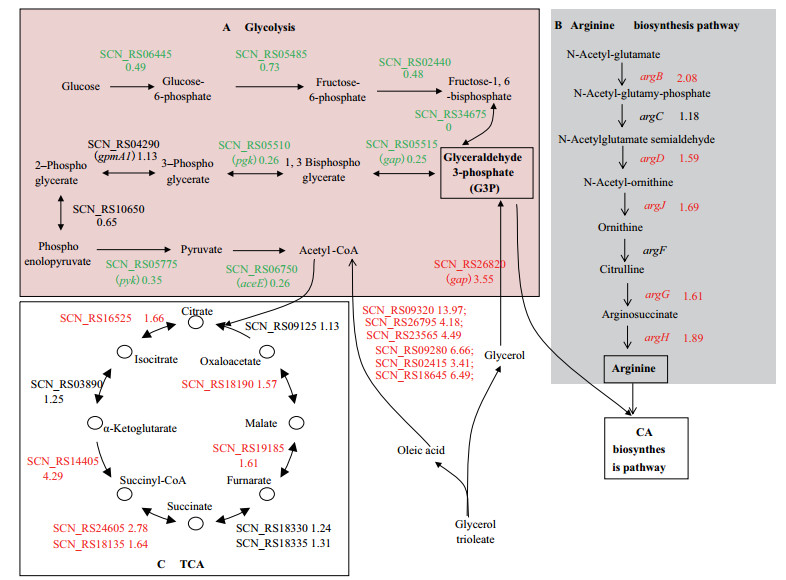
|
| 红色代表显著上调基因, 绿色代表显著下调基因, 黑色为无明显差异基因 图 4 F613-1菌株在不同转速条件下代谢途径变化情况 |
S. clavuligerus F613-1是发酵生产克拉维酸的高产菌株, 由长期的诱变育种和筛选获得。与标准菌株ATCC27064的全基因组比较分析显示:相比ATCC27064菌株含有pSCL1、pSCL2、pSCL3和pSCL4等4个质粒, F613-1只有一个0.7 Mb的质粒, 进化分析其可能来源于ATCC27064菌株中pSCL2和pSCL4质粒的部分序列, 因此F613-1菌株相比ATCC27064菌株具有较小的基因组; 比较基因组分析发现F613-1和ATCC27064基因组具有较高的一致性, 但F613-1基因组存在一个RGP(Region of genomic plasticity)区域; 此外, CA基因簇、克拉维烷基因簇和旁系同源基因簇的序列比对分析显示两个菌株之间仅在基因间隔区存在7个SNP位点。F613-1和ATCC27064的转录组比较分析显示[22]:F613-1中CA基因簇和合成基因的转录水平整体显著高于ATCC27064菌株, 全霉素(另一个次级代谢产物)合成基因的转录水平则大幅下调, 与此同时, 精氨酸(CA合成的前体物质)的合成基因显著上调, 提示F613-1菌株高产CA的原因可能与CA基因簇的转录水平上调有关, 同时菌株代谢流向的变化也可能促进了CA合成。
发酵条件优化是提高CA产量的有效手段之一[1-3]。本研究进一步分析了CA液体发酵过程中搅拌转速对S. clavuligerus合成CA能力的影响。F613-1菌株在高转速发酵条件下克拉维酸产量显著提高, 可能与高转速能提高发酵液溶氧水平和传质效果有关; 转录组测序比较分析发现, 在高转速发酵条件下, CA合成基因簇中基因表达量变化虽不显著, 但CA合成途径的调控基因ccaR表达量显著增加, CcaR可能通过间接调控初级代谢进而影响了CA的合成; 此外, 转录组分析显示F613-1菌株在高转速发酵条件下, 整个精氨酸合成基因簇中基因上调表达, 因此本研究中F613-1精氨酸合成基因的上调可能会促进CA的合成。G3P是CA合成的另一个直接前体物质, 总糖含量测定和转录组数据显示, F613-1菌株在高转速发酵条件下甘油代谢转化为G3P过程、TCA过程明显提高, 这可能是F613-1在高转速条件下CA产量提高的一个主要原因。
4 结论发酵转速显著影响F613-1合成CA的速度, 高转速条件下CA合成速度加快并且CA产量提高。转录组测序显示高转速条件下基因转录水平发生显著变化, 涉及CA合成途径相关基因及其途径特异性正调控基因、精氨酸生物合成基因簇、G3P代谢途径相关基因、TCA循环途径相关基因。高转速发酵条件促进精氨酸和G3P的合成可能对CA产量提高起到重要作用。
| [1] |
Ser HL, Law JW, Chaiyakunapruk N, et al. Fermentation conditions that affect Clavulanic Acid production in Streptomyces clavuligerus:A systematic review[J]. Frontiers in Microbiology, 2016, 7: 522. |
| [2] |
Viana Marques DA, Machado SEF, Ebinuma VC, et al. Production of beta-Lactamase inhibitors by Streptomyces species[J]. Antibiotics(Basel), 2018, 7(3): pii:E61. |
| [3] |
Rodrigues K, Souza AT, Badino AC, et al. Screening of medium constituents for clavulanic acid production by Streptomyces clavuligerus[J]. Brazilian Journal of Microbiology, 2018, 49(4): 832-839. DOI:10.1016/j.bjm.2018.01.006 |
| [4] |
Townsend CA, Ho MF. Biosynthesis of clavulanic acid:origin of the C3 Unit[J]. Journal of the American Chemical Society, 1985, 107: 1066-1068. DOI:10.1021/ja00290a057 |
| [5] |
Jensen SE. Biosynthesis of clavam metabolites[J]. Journal of Industrial Microbiology & Biotechnology, 2012, 39(10): 1407-1419. |
| [6] |
Alvarez-Alvarez R, Rodriguez-Garcia A, Martinez-Burgo Y, et al. Transcriptional studies on a Streptomyces clavuligerus oppA2 deletion mutant:N-Acetylglycyl-Clavaminic acid is an intermediate of clavulanic acid biosynthesis[J]. Applied and Environmental Microbiology, 2018, 84(22): pii:e01701-18. |
| [7] |
Kwong T, Zelyas NJ, Cai H, et al. 5S clavam biosynthesis is controlled by an atypical two-component regulatory system in Streptomyces clavuligerus[J]. Antimicrobial Agents and Chemotherapy, 2012, 56(9): 4845-4855. DOI:10.1128/AAC.01090-12 |
| [8] |
Abusara NF, Piercey BM, Moore MA, et al. Comparative genomics and metabolomics analyses of clavulanic acid-producing Streptomyces species provides insight into specialized metabolism[J]. Frontiers in Microbiology, 2019, 10: 2550. DOI:10.3389/fmicb.2019.02550 |
| [9] |
Hamed RB, Gomez-Castellanos JR, Henry L, et al. The enzymes of beta-lactam biosynthesis[J]. Natural Product Reports, 2013, 30(1): 21-107. |
| [10] |
Ozcengiz G, Demain AL. Recent advances in the biosynthesis of penicillins, cephalosporins and clavams and its regulation[J]. Biotechnology Advances, 2013, 31(2): 287-311. DOI:10.1016/j.biotechadv.2012.12.001 |
| [11] |
Cao G, Zhong C, Zong G, et al. Complete genome sequence of Streptomyces clavuligerus F613-1, an industrial producer of clavulanic acid[J]. Genome Announcements, 2016, 4(5): e01020-16. |
| [12] |
Alvarez-Alvarez R, Martinez-Burgo Y, Rodriguez-Garcia A, et al. Discovering the potential of S. clavuligerus for bioactive compound production:cross-talk between the chromosome and the pSCL4 megaplasmid[J]. BMC Genomics, 2017, 18(1): 907. DOI:10.1186/s12864-017-4289-y |
| [13] |
Srivastava SK, King KS, Abusara NF, et al. In vivo functional analysis of a class A beta-lactamase-related protein essential for clavulanic acid biosynthesis in Streptomyces clavuligerus[J]. PLoS One, 2019, 14(4): e0215960. DOI:10.1371/journal.pone.0215960 |
| [14] |
Medema MH, Trefzer A, Kovalchuk A, et al. The sequence of a 1. 8-mb bacterial linear plasmid reveals a rich evolutionary reservoir of secondary metabolic pathways[J]. Genome Biology and Evolution, 2010, 2: 212-24. DOI:10.1093/gbe/evq013 |
| [15] |
Martinez-Burgo Y, Alvarez-Alvarez R, Rodriguez-Garcia A, et al. The pathway-specific regulator ClaR of Streptomyces clavuligerus has a global effect on the expression of genes for secondary metabolism and differentiation[J]. Appl Environ Microbiol, 2015, 81(19): 6637-6648. DOI:10.1128/AEM.00916-15 |
| [16] |
Alvarez-Alvarez R, Rodriguez-Garcia A, Santamarta I, et al. Transcriptomic analysis of Streptomyces clavuligerus DeltaccaR::tsr:effects of the cephamycin C-clavulanic acid cluster regulator CcaR on global regulation[J]. Microbial Biotechnology, 2014, 7(3): 221-31. |
| [17] |
Fu J, Qin R, Zong G, et al. The CagRS Two-component system regulates clavulanic acid metabolism via multiple pathways in Streptomyces clavuligerus F613-1[J]. Frontiers in Microbiology, 2019, 10: 244. DOI:10.3389/fmicb.2019.00244 |
| [18] |
Santamarta I, Perez-Redondo R, Lorenzana LM, et al. Different proteins bind to the butyrolactone receptor protein ARE sequence located upstream of the regulatory ccaR gene of Streptomyces clavuligerus[J]. Molecular Microbiology, 2005, 56(3): 824-835. |
| [19] |
Bignell DR, Tahlan K, Colvin KR, et al. Expression of ccaR, encoding the positive activator of cephamycin C and clavulanic acid production in Streptomyces clavuligerus, is dependent on bldG[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(4): 1529-1541. DOI:10.1128/AAC.49.4.1529-1541.2005 |
| [20] |
Jnawali HN, Liou K, Sohng JK. Role of sigma-factor(orf21)in clavulanic acid production in Streptomyces clavuligerus NRRL3585[J]. Microbiological Research, 2011, 166(5): 369-79. DOI:10.1016/j.micres.2010.07.005 |
| [21] |
Gomez-Escribano JP, Martin JF, Hesketh A, et al. Streptomyces clavuligerus relA-null mutants overproduce clavulanic acid and cephamycin C:negative regulation of secondary metabolism by(p)ppGpp[J]. Microbiology, 2008, 154(Pt 3): 744-755. |
| [22] |
覃荣活, 付加芳, 宗工理, 等. 转录组分析棒状链霉菌工业生产菌株高产克拉维酸的分子基础[J]. 生物技术通报, 2017, 33(9): 235-243. |
| [23] |
Khaleeli N, Li RF, Townsend CA. Origin of the β-Lactam carbons in clavulanic acid from an unusual thiamine pyrophosphate-mediated reaction[J]. Journal of the American Chemical Society, 1999, 121: 9223-9224. DOI:10.1021/ja9923134 |