代谢工程改造是构建工业化细胞工厂的一种重要的合成生物学手段。微生物则是目前代谢工程改造的主要研究对象之一。其合成的丰富的代谢产物涵盖了食品、医疗、燃料、高分子材料等应用领域。目前, 基于代谢工程改造构建的微生物细胞工厂已被广泛应用到萜类化合物[1]、聚酮类化合物[2]、生物塑料制品[3]等高值产品的生产中。除此之外, 工程化改造的微生物在活体功能材料、水土修复等众多领域均具有较好的应用潜力。因此, 细胞工厂的工程化改造无论在高值产物合成还是在功能化微生物的改造中, 都有着重要的工业化应用价值。然而, 由于微生物自身复杂的代谢途径和调控关系, 给细胞工厂的理性改造带来很多挑战与不确定性。因此开发高效的代谢工程改造方法, 不仅可以有效地提升细胞工厂构建的效率, 而且还可以进一步强化工程化细胞的工作效能。
随着分子生物学、合成生物学[4-5]、基因组学和蛋白组学[6]等领域的快速发展, 新的代谢工程改造技术也不断被开发、丰富, 涵盖了代谢通路构建、表达, 复杂且精细的通路调谐、调控, 以及代谢网络重构分析等基于系统与合成生物学的新技术[7]。而静态调控改造策略是目前研究最多、应用最广的细胞改造策略。近年来, 随着前沿生物技术的创新推动和工业生物产业的不断发展, 动态调控策略在代谢工程领域的作用和优势逐渐凸显出来[8-9]。因此, 本文将重点综述关于动态调控的原理、特征, 以及在代谢工程领域的应用范例, 同时总结了动态调控系统设计与构建的关键步骤及其在未来细胞工厂改造中的潜在应用方式。
1 代谢工程中的调控方式 1.1 静态调控静态调控是指通过设计和改造遗传编码组件对目标代谢通路进行静态优化的代谢工程改造策略[10]。这种改造方式的最大特征是定向、不可逆性, 改造后的细胞在生长过程中不具备感应指定信号而做出特定调整的能力。目前, 静态调控主要涵盖了基因插入和敲除[11-12], 单/多基因的表达水平的调谐如启动子工程以调控转录水平[13]、核糖体结合位点(Ribosome binding site, RBS)改造以实现翻译水平调节[14], 引入蛋白支架[15-16], 代谢流平衡分析[17]等方面(图 1-A)。

|
| A:主要包括启动子改造、核糖体结合位点(RBS)改造、基因敲除、代谢网络模型指导的代谢流分析等;B:P34HB生物聚酯在嗜盐单胞菌中的合成及静态优化 图 1 静态调控概览 |
其中, 敲除影响菌株目标代谢物积累量的基因是构建工程化菌株的重要手段之一, 也是典型的静态调控方式[18]。例如, Alper和Stephanopoulos等[19]报道了一种基于全局化学计量学分析的单基因和多基因敲除的合理设计方法。该方法以大肠杆菌为出发菌, 以番茄红素为目标产物, 经组合构建了64个基因敲除突变株, 筛选分析后番茄红素的产量得到极大的提高。此外, 通过构建底盘菌的代谢网络模型来进行需敲除基因的预测, 从而实现目标代谢产物的高效合成[20], 是近年来基于系统与合成生物学开发的一种更高效的方法。而且, 随着计算工具的进一步发展, 代谢网模型可用于预测基因敲除引起的全局基因表达水平的变化[21], 进而可以系统地评估基因敲除对产物合成和细胞生长的影响。该方法甚至还能用于计算上调、下调及微调目的基因表达水平后的代谢水平变化[22], 从而预测菌株改造的变异性, 提高菌株改造的效率[23]。相关研究工作如提高琥珀酸、丁二醇等在大肠杆菌中的产量[24]及乙醇在酵母中产量[25]等是该研究领域的典型范例。
除基因敲除外, 启动子工程和RBS改造也是静态调控中较常用的方法。该方法可有效地调节代谢通路相关基因表达过程中的转录和翻译水平, 从而控制通路中相关代谢物的合成或消耗速率。如通过基因簇表达量的调控, 在谷氨酸棒杆菌(Cornyebacterium glutamicum)中实现赖氨酸的高产[26]。然而为了应对更复杂的代谢通路表达量调谐, 许多研究在不同的生物底盘中开发了多种启动子和RBS库, 从而实现多基因表达调谐的高通量建库筛选[13-14]。此外, Salis等[27]利用翻译初始化的热力学模型, 通过减少具有抑制性的RNA二级结构的引入, 从而实现RBS与核糖体结合能的预测和设计, 从而理性设计和调控基因表达过程中的翻译水平。
静态调控因其易操作、周期短、效果显著、设计简单等特点, 在合成生物学领域被广泛应用 以基于嗜盐单胞菌合成聚(3-羟基丁酸-4羟基丁酸)P(3HB4HB)为例, 首先实现4-羟基丁酸辅酶A(4HB-CoA)合成通路的构建及验证, 然后对相关的旁路基因进行敲除, 最后对4HB-CoA合成路径的代谢通量进行调谐, 从而提高P34HB的产量(图 1-B) [11-12]。然而, 在细胞生长过程中, 由于细胞代谢物或目标代谢产物的组成和浓度会随着培养基营养物质的组成、生长环境(主要是溶解氧浓度)以及细胞生长速率的变化而发生改变, 所以静态调控所获得的突变株无法感应细胞生长过程中的代谢变化从而进行自身的细胞活动调节。因此, 单一地减少或增加关键蛋白的表达量可能会对细胞生长产生不可逆的副作用, 尽管突变菌株相对于出发菌株具有更高水平的产物积累能力, 但受到细胞生长率抑制、生长鲁棒性差、代谢紊乱等因素影响, 使得目标产物的合成未达到最优化水平, 且在后期放大生产过程中也可能面临众多挑战[28-29]。
1.2 动态调控由于目标产物的产率和产量往往受到细胞代谢失衡的限制难以被进一步提升。因此, 若只采用静态调控, 相关基因表达水平过低, 会导致产物合成的代谢流不足, 而过高的表达量则不仅会导致较多冗余的mRNA、蛋白质或中间产物积累, 而且还可能对细胞生长有一定的抑制作用, 如细胞生长迟缓或产生适应性不良反应, 从而降低产量[10]。为了适时地平衡产物合成所需的基因表达与全局代谢水平之间的关系, 动态调控基因通路的引入能够实时响应代谢信号, 并及时进行反馈调节, 以便适应宿主内部代谢或环境的变化[30-32], 实现稳定合成所需产物, 提高发酵培养的稳定性和可放大性。
通常, 动态调控是通过设计与构建基因通路单元, 使重组细胞可以动态地感应并处理特定信号, 从而根据信号的改变进行适应性调控, 达到产物合成或目标系统的最优化状态[33]。该系统中的特定信号可以是代谢通路中的中间产物、细胞系统的代谢物或蛋白等因子, 也可以是外源引入合成的信号分子等。相比于静态调控, 动态调控是一个以信号响应为基础的动态调控过程, 因此可以在线感知细胞状态并进行实时代谢调控[34]。该策略已经在较多研究中得以应用并实现目标产物的大幅度增产, 如番茄红素、脂肪酸、氨基酸、肌醇等高附加值生物合成制品[35-36]。
2 动态调控系统根据构建原理或响应机制不同, 动态调控系统可以分为代谢物依赖型动态调控和非代谢物依赖型动态调控; 而根据其调控方式不同, 又可以分为单向动态调控和双向动态调控。其中, 代谢物依赖型动态调控是以目标通路的代谢产物以及因引入目标通路而引起变化的内源代谢物为响应信号的调控方式。而非代谢物依赖型动态调控是以外界环境因素或外源引入的与细胞系统正交的调控因子作为响应信号的调控方式(图 2)。单向动态调控是基于响应信号对目标基因表达只进行单一上调或下调调节[37], 而双向动态调控则可以实现上调和下调同时调节。表 1中列出了近年来动态调控用于代谢工程改造的典型应用案例, 包括了产物产量优化、调控网络构建、嵌段大分子合成等。
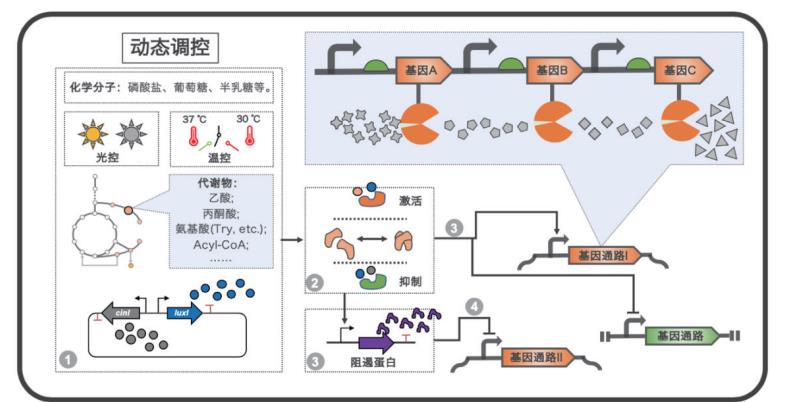
|
| 第一层为输入信号,包括化学分子、光控、声控、代谢物及基因通路合成分子等;第二层为信号响应蛋白;第三层可以是直接受调控的基因通路,或者级联控制阻遏蛋白表达,以实现双向调控或更复杂的调控网络 图 2 动态调控系统概览 |
代谢物依赖型动态调控通常是通过构建生物传感器感应代谢物的浓度, 从而该传感器输出信号可直接或间接调控目标基因的表达[44-45]。如番茄红素的高效合成就是一个典型的研究案例。因为大肠杆菌生长过程中葡萄糖的过量供给会导致乙酸的积累, 不仅抑制细胞生长, 而且削弱了番茄红素合成通路的代谢流通量, 从而降低番茄红素在大肠杆菌中的产率。研究发现, 磷酸烯醇丙酮酸合成酶(Pps)是控制细胞内甘油醛-3-磷酸酯和丙酮酸之间平衡的关键, 而异戊烯基焦磷酸异构酶(Idi)是番茄红素合成通路中的限速步骤。而在糖酵解通路中, 乙酰磷酸是从丙酮酸生成乙酸的中间产物, 因此构建以乙酰磷酸分子为响应信号的生物传感器是实现番茄红素合成通路动态调控的关键。为此, 研究者构建响应乙酰磷酸浓度的动态调控开关, 在胞内乙酰磷酸出现异常累积的情况下, 上调磷酸烯醇丙酮酸合成酶(Pps)和异戊烯基焦磷酸异构酶(Idi)的表达水平, 从而将丙酮酸逆向转化成番茄红素的上游合成前体磷酸烯醇丙酮酸, 同时由于异戊烯基焦磷酸异构酶(Idi)的过表达, 番茄红素合成的关键酶表达量增加, 增强了番茄红素的代谢流通量, 从而起到减少乙酸积累和增强番茄红素合成的目的。通过对比发现, 基于动态调控所获得的番茄红素滴定产量比简单的基因过表达方式所合成的产量提高18倍, 同时产率也得到较显著的提升[46]。
因此, 此类动态调控系统的关键有两点:一是找到与目标代谢通路关联且影响显著的代谢物; 二是构建以该代谢物作为响应信号的生物传感器[47]。有趣的是, 细胞系统内通常天然存在各种各样的动态调控系统, 用于调节自身的细胞活动。因此, 我们可以通过分子生物学和合成生物学手段对这种类型的生物传感器进行挖掘和二次开发, 变成实际代谢工程改造中所适用的动态调控元件[48]。如研究发现脂肪酸合成通路中的关键中间产物脂肪酰辅酶A能与配体响应型转录调控因子FadR蛋白结合, 解除FadR蛋白对转录起始位点的抑制作用[49]。因此, 有研究者通过启动子工程改造获得具有更严谨转录调控性能的响应开关, 建立基于脂肪酰辅酶A响应的动态调控系统, 从而实现生物柴油合成通路中各基因模块的动态调控, 平衡目标通路代谢物在胞内的有效流通, 减少宿主细胞的代谢压力并提高生物柴油合成的稳定性和滴定产量[50-51]。
这种基于天然调控因子改造而来的动态调控系统, 可以有效地拓宽动态调控的应用范围, 如拓展至其他代谢途径的代谢物, 可应用于多种宿主进行调控等。经研究表明, 大肠杆菌内至少有36种此类配体响应型转录因子可供开发使用[50]。另外, 来自其他生物体的配体响应型转录因子也非常多。例如, 氨基酸响应的转录因子[52-53], 葡萄糖[54]、乙酸[55-56]、磷酸根应答型转录调控蛋白等[57]。基于这些转录因子组成的动态调控系统可以用于宿主或异源宿主的代谢工程改造中, 以实现目标代谢物合成关联基因的动态调控, 从而提高产物产量和重组菌株的鲁棒性。
2.2 非代谢物依赖型动态调控非代谢物依赖型动态调控是通过感应外界环境因素或与宿主细胞系统正交的信号分子而实现基因表达的动态调控。与代谢物依赖型动态调控相比, 该调控方式的要点在于不依赖目标代谢通路, 通用性强, 但需重点评估外源信号引入对宿主代谢的影响。目前, 该类型动态调控大致包括群体感应[58]、光控[41]、非编码RNA[39, 59]、温控[60]等(图 2)。
群体感应是细胞群体中, 由于特定信号分子(如内酯类)随着细胞生长而不断积累, 在一定生长密度下积累的信号分子可以诱导或关闭特定基因表达, 是细胞生长过程中响应细胞密度的典型调控方式之一, 目前已被广泛用于代谢调控领域[61-62]。利用群体感应系统对导向内源代谢路径的代谢通量进行可控限制, 以增加碳源进入目标产物合成路径的代谢通量, 进而动态地控制大肠杆菌中糖酵解过程和产物合成途径的代谢流, 以平衡细胞生长和产物合成的协同, 达到较优的产物滴定度。该方法已用于合成莽草酸、葡萄糖二酸、肌醇、甜没药烯等[40, 63-64]。
近年来, 光遗传工具的开发也取得了较大的进展, 并逐步解决动态调控中响应速度慢、便携性差、动态表达能力低等难题。光感应蛋白可以通过响应不同波长的光源形成单聚体或多聚体结构, 从而改变蛋白和特定核酸序列的结合能, 实现基因表达的可控调控。最近, 研究报道在酵母中通过构建光控动态双向调控系统, 大幅提升异丁醇和2-甲基-1-丁醇的产量, 其小试发酵罐产量分别高达8.49 g/L和2.38 g/L[41]。目前, 光控系统还被用于控制蛋白发生快速、可逆的相分离, 从而提高代谢通路中相关酶的催化效率以及实现更精细的代谢调控, 以脱氧紫色杆菌素的合成为模型的验证实验表明, 最终产量提高6倍, 产品特异性提高18倍, 效果显著[65]。
此外, 基于温度响应的动态调控系统也可被用于细胞代谢工程改造的相关研究。例如温敏阻遏蛋白(CI857)是被研究和应用最广泛的。它在30oC时以单聚体的形式存在, 从而结合并抑制lamda启动子的转录活性; 在37℃时, 该蛋白形成二聚体从启动子区释放出来, 转录活性得以恢复。研究通过将重组细胞的培养温度从37℃转换到30℃, 使得细胞在37℃表达保持细胞的正常生长所需的异柠檬酸脱氢酶, 生长期结束后调整温度至30℃, 抑制异柠檬酸脱氢酶的表达, 以减少细胞代谢中α-酮戊二酸合成对目标产物合成的分流, 因此细胞也将从生长期转移到生产阶段实现产量提升[60]。
目前, 光控和温控调控系统得益于其本身输入信号的可移除性, 在代谢工程中有着更广泛的潜在应用价值。但由于需要光敏和温敏蛋白的表达, 其表达水平对宿主全局代谢以及目标代谢通路可能存在的影响需要谨慎评估, 尽量减少系统噪音。其次, 由于光控设备引入以及光在放大过程中的非均一性分布, 也限制了光控在更大培养体系中的应用。而基于群体感应系统构建的动态调控线路, 对于细胞密度的敏感性控制难度大, 在放大培养中容易失活, 但易于实现自调控。相对而言, 基于代谢物响应的动态调控系统与宿主有较好的相容性, 也便于实现自调控, 但响应信号分子的筛选及其对应生物传感器的改造难度大。
2.3 动态双向调控前文提到的单向动态调控系统是目前研究最多、应用最广的调控方式之一。而相比于单向动态调控, 双向动态调控可以同时实现不同基因或代谢路径的激活和抑制表达[66]。近年来, 因为双向动态调控可实现更复杂的控制逻辑, 为代谢调控提供更多元化的选择, 在代谢工程改造中有较好的灵活性, 关于双向动态调控的研究报道也越来越多[67-69]。
目前, 动态双向调控主要应用于解决细胞前期生长和后期产物合成的矛盾, 实现目标合成通路及与其竞争的生长依赖型代谢通路之间的分段式调控, 实现细胞生长和产物积累的最大化[70-72]。如研究结合光控动态表达系统和非门基因电路设计的动态双向调控在酵母中实现异丁醇的高产[41]。此外, Dinh等[62]通过组合基于同一种内酯分子(OC6)响应的抑制型和激活型两种群体感应系统, 构建了感应细胞密度的双向动态自调控系统, 分别在重组大肠杆菌中实现柚皮素和水杨酸的高效合成。
除此之外, 动态双向调控目前还可以被用于合成人工定制化的生物大分子(图 3-B), 比如嵌段型蛋白复合体[43]、高分子生物聚酯材料等。
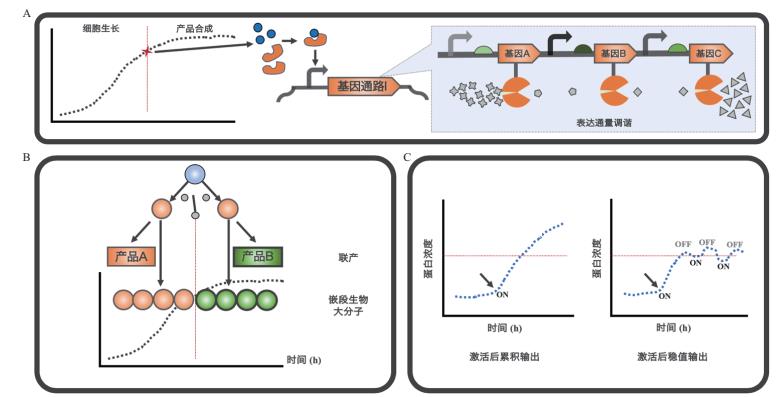
|
| A:静态调控与动态调控组合使用;B:动态调控用于多产品联产和嵌段型生物大分子的体内合成;C:动态调控用于诱导后稳定输出,减少基因表达过载 图 3 动态调控的应用展望 |
动态调控系统一般主要由3部分组成, 包括输入信号, 信号处理模块(生物传感器), 信号输出(或执行机构) [73-75]。其中, 这3个部分元件的选择、改造以及组合方式, 决定着动态调控系统的功能与性能。输入信号可以为直接添加的化学分子(如阿拉伯糖、半乳糖、乙酸等)、外界物理条件(如光、温度等)或者通过基因通路设计合成的生物分子(如群体感应分子、RNA、脂肪酸、氨基酸等), 如图 2所示。而生物传感器的设计是整个动态调控系统的关键[76]。目前主要的构建方法包括响应蛋白突变改造[77-78]、响应蛋白复合体的组合设计[79-80]、响应蛋白与核酸结合区域的设计[49]等, 这类型方法均可以改变生物传感器的“输入-响应”曲线, 从而改变生物传感器对输入信号的响应范围和灵敏度、输出值的动态范围、响应阈值等参数(图 4-A), 以匹配实际的应用需求。

|
| A:传感器的“输入-响应”特征曲线;B:生物传感器在细胞不同细胞密度下(不同生长期)的响应评价 图 4 动态调控的应用展望 |
动态调控系统在控制上一般需要满足两个要求, 分别是可定时控制[81]和可控值输出。定时控制可以很好地平衡细胞生长和产物合成之间的关系, 可控值输出可以为目的产物合成提供适配的胞内蛋白丰度的同时, 尽可能实现细胞系统能效转换的最大化。
随着合成生物学的发展, 动态调控在精细化生物合成中的作用将日趋重要, 优势显像.因此, 动态调控单元的组合使用, 理论上将实现更为复杂多样的控制逻辑.但是, 目前受限于对细胞系统的深入认知, 多层级(>3层)信号传递的调控系统在实际应用中容易出现信号衰减甚至丢失而导致控制系统失活的现象[82]。因此, 在动态调控系统的设计过程中, 充分考量其简单易用性, 能很好地保障其性能发挥的稳定性和生物工程应用的可放大性。此外, 目前低层级信号级联(≤ 3层)动态调控系统的开发与应用仍具有重要的意义, 因其不仅可以为未来复杂的动态调控系统奠定丰富的元件库和研究基础, 而且还能启发研究者更好地理解生命系统错综复杂的代谢调控网络。
3.2 基于报告基因的系统表征由于调控系统一般涉及复杂的信号传递, 因此很难确定信号的识别和反馈控制。为了解决这个问题, 很多研究通常引入荧光报告基因编码的荧光蛋白或基因簇(如绿色、红色、黄色荧光蛋白等), 通过检测荧光蛋白的表达量, 实现响应性能表征。一般来讲, 基因元件的基础表征对于元件的利用是极其重要的。元件本身的特性直接影响其在实际使用中的表现。例如, 当传感器在无输入状态下的背景输出较大时, 不适用于对低表达敏感的基因的控制等[12]。
对于动态调控系统, 除了传感器本身的“输入-响应”关系表征外, 还需要考虑传感器在细胞不同生长期的响应表现[83], 以及响应速度等特征(图 4-B)。因为动态调控在实际代谢工程的应用中容易受细胞培养浓度的影响, 往往在高细胞密度条件下失去功能。而传感器对输入的响应速度对信号级联式控制也至关重要[82]。最后, 系统地评估传感器与宿主之间的相容性, 有利于改善因引入外源系统带来的系统噪音影响[83]。目前, 可以通过转录组学、蛋白组学等系统生物学方法对全局代谢系统进行差异分析, 以评估影响的显著性[83]。同时, 基于细胞生长表型, 如生长的趋势、营养物代谢速率等参数, 来进行综合性生物学评价[12]。结合以上评估方法, 可以有效地提高动态调控系统在不同培养规模下的可用性和稳定性。
4 展望动态调控的关键点在于输入信号响应, 既包括响应特定代谢物的生物传感器构建, 也包括生物传感器在更高细胞浓度下的有效响应, 这两个技术难点目前仍是动态调控的核心挑战。尤其对于细胞生长进入平台期后的基因表达调控。因为在大多数胞内外产物积累末期, 实现细胞形态变化[84]或诱导裂解细胞释放产品[85]便于下游分离提纯, 可以很大程度上解决细胞人工破壁的成本。除此之外, 信号的快速输入与移除, 也将影响动态调控的效率。如温控的均一性好, 但升温降温响应存在滞后; 而光控的输入快, 但是均一性差, 且放大过程中装置的设计或改造难度大。综上所述, 动态调控在构建可工业化应用的细胞工厂中仍存在很多挑战, 并需要进一步研究探索。但动态调控仍然是代谢工程领域的一个高效、创新的重要技术方向。
最近, 根据RNA调控[86]和“蛋白-蛋白”调控[87]等研究表明, 动态调控未来有实现转录后或翻译后调控的可能, 从而减少目前基于中心法则的长行程基因表达带来的噪音和迟滞引发的不稳定性, 同时也可以实现更快速、精准的输入和输出响应, 强化代谢调控的特异性。
另外, 动态调控与其他代谢工程改造方法的创新性组合使用也有利于细胞工厂在产物合成中产量、转化率和产率(TYP :Titer, Yield, Productivity)的进一步提升[88]。如结合静态和动态调控的各自优点, 实现基因表达在时间和表达水平上的最优化组合。静态调控可以通过基因敲除、启动子或RBS改造等方法, 将目标合成路径的代谢流进行调谐, 有效地导引底物向产物的催化转化, 减少副产物或中间产物的积累; 而动态调控可以控制激活合成通路和抑制竞争旁路的时机。从而当依据静态调控水平去激活目标基因表达时, 既保证了细胞在对数期的正常生长, 又实现细胞工厂高效的产物积累(图 3-A)。以大肠杆菌合成肌醇和葡萄糖二酸为例, 研究者已通过构建感应细胞密度的生物传感器, 调控葡萄糖-6-磷酸(G6P)的下游代谢分流。即在低细胞密度的条件下, 葡萄糖-6-磷酸进入糖酵解和磷酸戊糖途径供给细胞生长, 在高细胞密度下, 葡萄糖-6-磷酸则作为肌醇合成的底物, 从而提高肌醇和葡萄糖二酸的产量[40]。因此, 被激活的肌醇或葡萄糖二酸的合成通路可以先通过基因表达调谐来确定合适的表达强度, 然后再通过细胞密度传感器来激活表达, 以减少过量表达导致细胞出现过载的代谢负荷(图 3-A) [89]。
此外, 从光控诱导合成异丁醇的生物发酵过程可得, 动态调控系统与基因振荡器联用控制目标基因表达也可能带来更好的产品积累量.研究基于光控动态表达系统对酵母中乙醇和异丁醇的合成通路的关键酶进行定时的激活和抑制控制[41].有趣的是, 在小试发酵实验中, 在特定时间激活异丁醇合成通路表达后, 间歇式的光控诱导所合成的产物浓度比持续性的诱导方式要高.因此, 在激活未经调谐的代谢通路时, 采用间歇式的转录调控方式可以减少细胞内目的蛋白的过度积累, 从而维持细胞正常的代谢水平和提高产物合成效率(图 3-C)。
除代谢工程改造以外, 动态调控系统还可以用于研究微生物群落[90-93]、细胞靶向药物释放[94]、细胞表型变化[95]、相分离控制[65]等领域。尤其是多个动态调控单元级联的复杂调控网络构建[42], 既可用于构建仿细胞代谢调控的网络动态[96], 也有利于研究者深入探索和解析复杂的细胞代谢调控网络机制。随着更多生物传感器的开发, 以及工程化理性设计的发展, 结合日渐丰富先进的生物技术, 如高通量自动化平台、DNA合成及测序等, 细胞工厂的工程化改造将实现快周期、智能化、高效率的革命性突破。
| [1] |
Martin Vincent JJ, Pitera Douglas J, Withers Sydnor T, et al. Engineering a mevalonate pathway in Escherichia coli for productionof terpenoids[J]. Nature Biotechnology, 2003, 21(7): 796-802. DOI:10.1038/nbt833 |
| [2] |
Peirú S, Menzella Hugo G, Rodríguez E, et al. Production of thepotent antibacterial polyketide erythromycin C in Escherichiacoli[J]. Applied and Environmental Microbiology, 2005, 71(5): 2539-2547. DOI:10.1128/AEM.71.5.2539-2547.2005 |
| [3] |
Aldor IS, Keasling JD. Process design for microbial plasticfactories :metabolic engineering of polyhydroxyalkanoates[J]. Current Opinion in Biotechnology, 2003, 14(5): 475-483. DOI:10.1016/j.copbio.2003.09.002 |
| [4] |
Xia PF, Ling H, Foo JL, et al. Synthetic genetic circuits forprogrammable biological functionalities[J]. BiotechnologyAdvances, 2019, 37(6): 107393. |
| [5] |
Nielsen J, Keasling JD. Engineering cellular metabolism[J]. Celi, 2016, 164(6): 1185-1197. DOI:10.1016/j.cell.2016.02.004 |
| [6] |
Lempp M, Farke N, Kuntz M, et al. Systematic identification ofmetabolites controlling gene expression in E. coli[J]. NatureCommunications, 2019, 10(1): 1-9. |
| [7] |
Kent R, Dixon N. Contemporary tools for regulating gene expressionin bacteria[J]. Trends in Biotechnology, 2020, 38(3): 316-333. DOI:10.1016/j.tibtech.2019.09.007 |
| [8] |
Xu P. Production of chemicals using dynamic control of metabolicfluxes[J]. Current Opinion in Biotechnology, 2018, 53: 12-19. DOI:10.1016/j.copbio.2017.10.009 |
| [9] |
Chen X, Liu L. Gene circuits for dynamically regulatingmetabolism[J]. Trends Biotechnol, 2018, 36(8): 751-754. DOI:10.1016/j.tibtech.2017.12.007 |
| [10] |
Holtz WJ, Keasling JD. Engineering static and dynamic control ofsynthetic pathways[J]. Cell, 2010, 140(1): 19-23. |
| [11] |
Ye J, Hu D, Che X, et al. Engineering of Halomonas bluephagenesisup for low cost production of poly(3-hydroxybutyrate-co-4-hydroxybutyrate)from glucose[J]. Metabolic Engineering, 2018, 47: 143-152. DOI:10.1016/j.ymben.2018.03.013 |
| [12] |
Ye J, Hu D, Yin J, et al. Stimulus response-based fine-tuning ofpolyhydroxyalkanoate pathway in Halomonas[J]. MetabolicEngineering, 2020, 57: 85-95. |
| [13] |
Shen R, Yin J, Ye JW, et al. Promoter engineering for enhanced P(3HB-co-4HB)production by Halomonas bluephagenesis[J]. ACS Synthetic Biology, 2018, 7(8): 1897-1906. DOI:10.1021/acssynbio.8b00102 |
| [14] |
Li T, Ye J, Shen R, et al. Semirational approach for ultrahighpoly(3-hydroxybutyrate)accumulation in Escherichia coli bycombining one-step library construction and high-throughputscreening[J]. ACS Synth Biol, 2016, 5(11): 1308-1317. DOI:10.1021/acssynbio.6b00083 |
| [15] |
Dueber JE, Wu GC, Malmirchegini GR, et al. Synthetic proteinscaffolds provide modular control over metabolic flux[J]. NatureBiotechnology, 2009, 27(8): 753. |
| [16] |
Baek JM, Mazumdar S, Lee SW, et al. Butyrate productionin engineered Escherichia coli with synthetic scaffolds[J]. Biotechnology and Bioengineering, 2013, 110(10): 2790-2794. DOI:10.1002/bit.24925 |
| [17] |
Zhou H, Vonk B, Roubos JA, et al. Algorithmic co-optimization ofgenetic constructs and growth conditions :application to 6-ACA, apotential nylon-6 precursor[J]. Nucleic Acids Research, 2015, 43(21): 10560-10570. |
| [18] |
Zhao L, Lu Y, Yang J, et al. Expression regulation of multiple keygenes to improve l-threonine in Escherichia coli[J]. MicrobialCell Factories, 2020, 19(1): 1-23. DOI:10.1186/s12934-019-1269-8 |
| [19] |
Alper H, Miyaoku K, Stephanopoulos G. Construction of lycopeneoverproducing E.coli strains by combining systematic andcombinatorial gene knockout targets[J]. Nature Biotechnology, 2005, 23(5): 612-616. DOI:10.1038/nbt1083 |
| [20] |
Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1, 4-butanediol[J]. Nature Chemical Biology, 2011, 7(7): 445. DOI:10.1038/nchembio.580 |
| [21] |
Burgard AP, Pharkya P, Maranas CD. Optknock :a bilevelprogramming framework for identifying gene knockout strategiesfor microbial strain optimization[J]. Biotechnology andBioengineering, 2003, 84(6): 647-657. DOI:10.1002/bit.10803 |
| [22] |
Pharkya P, Maranas CD. An optimization framework for identifyingreaction activation/inhibition or elimination candidates foroverproduction in microbial systems[J]. Metabolic Engineering, 2006, 8(1): 1-13. |
| [23] |
Ranganathan S, Suthers PF, Maranas CD. OptForce :anoptimization procedure for identifying all genetic manipulationsleading to targeted overproductions[J]. PLoS ComputationalBiology, 2010, 6(4). |
| [24] |
Lee SJ, Lee DY, Kim TY, et al. Metabolic engineering of Escherichia coli for enhanced production of succinic acid, based ongenome comparison and in silico gene knockout simulation[J]. Applied Environmental Microbiology, 2005, 71(12): 7880-7887. DOI:10.1128/AEM.71.12.7880-7887.2005 |
| [25] |
Bro C, Regenberg B, Förster J, et al. In silico aided metabolicengineering of Saccharomyces cerevisiae for improved bioethanolproduction[J]. Metabolic Engineering, 2006, 8(2): 102-111. DOI:10.1016/j.ymben.2005.09.007 |
| [26] |
Becker J, Zelder O, Häfner S, et al. From zero to hero-design-basedsystems metabolic engineering of Corynebacterium glutamicum forl-lysine production[J]. Metab Eng, 2011, 13(2): 159-168. DOI:10.1016/j.ymben.2011.01.003 |
| [27] |
Salis HM, Mirsky EA, Voigt CA. Automated design of syntheticribosome binding sites to control protein expression[J]. Nature Biotechnology, 2009, 27: 946-950. |
| [28] |
Rugbjerg P, Sommer MOA. Overcoming genetic heterogeneity in industrial fermentations[J]. Nat Biotechnol, 2019, 37(8): 869-876. DOI:10.1038/s41587-019-0171-6 |
| [29] |
Schmitz AC, Hartline CJ, Zhang F. Engineering microbial metabolite dynamics and heterogeneity[J]. Biotechnology Journal, 2017, 12(10): 1700422. DOI:10.1002/biot.201700422 |
| [30] |
Shen X, Wang J, Li C, et al. Dynamic gene expression engineering as a tool in pathway engineering[J]. Current Opinion in Biotechnology, 2019, 59: 122-129. DOI:10.1016/j.copbio.2019.03.019 |
| [31] |
Lalwani MA, Zhao EM, Avalos JL. Current and future modalities of dynamic control in metabolic engineering[J]. Current Opinion in Biotechnology, 2018, 52: 56-65. DOI:10.1016/j.copbio.2018.02.007 |
| [32] |
Immethun CM, DeLorenzo DM, Focht CM, et al. Physical, chemical, and metabolic state sensors expand the synthetic biologytoolbox for Synechocystis sp. PCC 6803[J]. Biotechnology and Bioengineering, 2017, 114(7): 1561-1569. DOI:10.1002/bit.26275 |
| [33] |
Cress BF, Trantas EA, Ververidis F, et al. Sensitive cells :enabling tools for static and dynamic control of microbial metabolicpathways[J]. Curr Opin Biotechnol, 2015, 36: 205-214. DOI:10.1016/j.copbio.2015.09.007 |
| [34] |
Venayak N, Anesiadis N, Cluett WR, et al. Engineering metabolism through dynamic control[J]. Current Opinion in Biotechnology, 2015, 34: 142-152. DOI:10.1016/j.copbio.2014.12.022 |
| [35] |
Zhou L, Ren J, Li Z, et al. Characterization and engineering of a Clostridium glycine riboswitch and its use to control a novel metabolic pathway for 5-aminolevulinic acid production in Escherichia coli[J]. ACS Synthetic Biology, 2019, 8(10): 2327-2335. DOI:10.1021/acssynbio.9b00137 |
| [36] |
Gao C, Xu P, Ye C, et al. Genetic circuit-assisted smart microbial engineering[J]. Trends Microbiol, 2019, 27(12): 1011-1024. DOI:10.1016/j.tim.2019.07.005 |
| [37] |
Brockman IM, Prather KLJ. Dynamic knockdown of E.coli central metabolism for redirecting fluxes of primary metabolites[J]. Metabolic Engineering, 2015, 28: 104-113. DOI:10.1016/j.ymben.2014.12.005 |
| [38] |
Dahl RH, Zhang F, Alonso-Gutierrez J, et al. Engineering dynamic pathway regulation using stress-response promoters[J]. Nature Biotechnology, 2013, 31(11): 1039. DOI:10.1038/nbt.2689 |
| [39] |
Yang Y, Lin Y, Wang J, et al. Sensor-regulator and RNAi based bifunctional dynamic control network for engineered microbial synthesis[J]. Nature Communications, 2018, 9(1): 3043. DOI:10.1038/s41467-018-05466-0 |
| [40] |
Gupta A, Reizman I MB, Reisch CR, et al. Dynamic regulation of metabolic flux in engineered bacteria using a pathway-independent quorum-sensing circuit[J]. Nat Biotechnol, 2017, 35(3): 273. DOI:10.1038/nbt.3796 |
| [41] |
Zhao EM, Zhang Y, Mehl J, et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production[J]. Nature, 2018, 555(7698): 683-687. DOI:10.1038/nature26141 |
| [42] |
Moser F, Borujeni AE, Ghodasara AN, et al. Dynamic control of endogenous metabolism with combinatorial logic circuits[J]. Molecular Systems Biology, 2018, 14(11): e8605. |
| [43] |
Chen AY, Deng Z, Billings AN, et al. Synthesis and patterning of tunable multiscale materials with engineered cells[J]. Nature Materials, 2014, 13(5): 515-523. DOI:10.1038/nmat3912 |
| [44] |
Wang J, Zhang R, Zhang Y, et al. Developing a pyruvate-driven metabolic scenario for growth-coupled microbial production[J]. Metabolic Engineering, 2019, 55: 191-200. DOI:10.1016/j.ymben.2019.07.011 |
| [45] |
Qian S, Cirino PC. Using metabolite-responsive gene regulators to improve microbial biosynthesis[J]. Current Opinion in Chemical Engineering, 2016, 14: 93-102. DOI:10.1016/j.coche.2016.08.020 |
| [46] |
Farmer WR, Liao JC. Improving lycopene production in Escherichia coli by engineering metabolic control[J]. Nature Biotechnology, 2000, 18(5): 533-537. DOI:10.1038/75398 |
| [47] |
Solomon KV, Sanders TM, Prather KLJ. A dynamic metabolite valve for the control of central carbon metabolism[J]. Metabolic Engineering, 2012, 14(6): 661-671. DOI:10.1016/j.ymben.2012.08.006 |
| [48] |
Johnson AO, Gonzalez-Villanueva M, Wong L, et al. Design and application of genetically-encoded malonyl-CoA biosensors for metabolic engineering of microbial cell factories[J]. Metabolic Engineering, 2017, 44: 253-264. DOI:10.1016/j.ymben.2017.10.011 |
| [49] |
DiRusso CC, Heimert TL, Metzger AK. Characterization of FadR, a global transcriptional regulator of fatty acid metabolism in Escherichia coli[J]. Journal of Biological Chemistry, 1992, 267(12): 8685-8691. |
| [50] |
Zhang F, Carothers JM, Keasling JD. Design of a dynamic sensorregulator system for production of chemicals and fuels derived from fatty acids[J]. Nature Biotechnology, 2012, 30(4): 354. DOI:10.1038/nbt.2149 |
| [51] |
Morgan SA, Nadler DC, Yokoo R, et al. Biofuel metabolic engineering with biosensors[J]. Current Opinion in Chemical Biology, 2016, 35: 150-158. DOI:10.1016/j.cbpa.2016.09.020 |
| [52] |
Mustafi N, Grünberger A, Kohlheyer D, et al. The development and application of a single-cell biosensor for the detection of l-methionine and branched-chain amino acids[J]. Metabolic Engineering, 2012, 14(4): 449-457. DOI:10.1016/j.ymben.2012.02.002 |
| [53] |
Schendzielorz G, Dippong M, Grünberger A, et al. Taking control over control :Use of product sensing in single cells to remove flux control at key enzymes in biosynthesis pathways[J]. ACS Synthetic Biology, 2014, 3(1): 21-29. |
| [54] |
Maury J, Kannan S, Jensen NB, et al. Glucose-dependent promotersfor dynamic regulation of metabolic pathways[J]. Frontiers in Bioengineering and Biotechnology, 2018, 6: 63. DOI:10.3389/fbioe.2018.00063 |
| [55] |
Farmer WR, Liao JC. Acetate-inducible protein overexpressionfrom the glnAp2 promoter of Escherichia coli[J]. Biotechnologyand Bioengineering, 2001, 75(5): 504-509. DOI:10.1002/bit.10049 |
| [56] |
Bulter T, Lee SG, Wong WWC, et al. Design of artificial cell-cellcommunication using gene and metabolic networks[J]. PNAS, 2004, 101(8): 2299-2304. DOI:10.1073/pnas.0306484101 |
| [57] |
Teo WS, Chang MW. Development and characterization of ANDgate dynamic controllers with a modular synthetic GAL1 corepromoter in Saccharomyces cerevisiae[J]. Biotechnology and Bioengineering, 2014, 111(1): 144-151. DOI:10.1002/bit.25001 |
| [58] |
Dinh CV, Chen X, Prather KLJ. Development of a quorum-sensing based circuit for control of co-culture population composition in a naringenin production system[J]. ACS Synthetic Biology, 2020. |
| [59] |
Krishnamurthy M, Hennelly SP, Dale T, et al. Tunable riboregulator switches for post-transcriptional control of gene expression[J]. ACS Synthetic Biology, 2015, 4(12): 1326-1334. DOI:10.1021/acssynbio.5b00041 |
| [60] |
Harder BJ, Bettenbrock K, Klamt S. Temperature-dependent dynamic control of the TCA cycle increases volumetric productivity of itaconic acid production by Escherichia coli[J]. Biotechnology and Bioengineering, 2018, 115(1): 156-164. DOI:10.1002/bit.26446 |
| [61] |
Kim EM, Woo HM, Tian T, et al. Autonomous control of metabolic state by a quorum sensing(QS) -mediated regulator for bisabolene production in engineered E[J]. Metabolic Engineering, 2017, 44: 325-336. DOI:10.1016/j.ymben.2017.11.004 |
| [62] |
Dinh CV, Prather KLJ. Development of an autonomous and bifunctional quorum-sensing circuit for metabolic flux control in engineered Escherichia coli[J]. PNAS, 2019, 116(51): 25562-25568. DOI:10.1073/pnas.1911144116 |
| [63] |
Doong SJ, Gupta A, Prather KLJ. Layered dynamic regulation for improving metabolic pathway productivity in Escherichia coli[J]. PNAS, 2018, 115(12): 2964-2969. DOI:10.1073/pnas.1716920115 |
| [64] |
Soma Y, Hanai T. Self-induced metabolic state switching by a tunable cell density sensor for microbial isopropanol production[J]. Metabolic Engineering, 2015, 30: 7-15. DOI:10.1016/j.ymben.2015.04.005 |
| [65] |
Zhao EM, Suek N, Wilson MZ, et al. Light-based control of metabolic flux through assembly of synthetic organelles[J]. Nature Chemical Biology, 2019, 15(6): 589-597. DOI:10.1038/s41589-019-0284-8 |
| [66] |
Brockman IM, Prather KLJ. Dynamic metabolic engineering :new strategies for developing responsive cell factories[J]. Biotechnology Journal, 2015, 10(9): 1360-1369. DOI:10.1002/biot.201400422 |
| [67] |
Deng J, Chen C, Gu Y, et al. Creating an in vivo bifunctional gene expression circuit through an aptamer-based regulatory mechanism for dynamic metabolic engineering in Bacillus subtilis[J]. Metabolic Engineering, 2019, 55: 179-190. DOI:10.1016/j.ymben.2019.07.008 |
| [68] |
Cui S, Lv X, Wu Y, et al. Engineering a bifunctional Phr60-Rap60-Spo0A quorum-sensing molecular switch for dynamic fine-tuning of menaquinone-7 synthesis in Bacillus subtilis[J]. ACS Synthetic Biology, 2019, 8(8): 1826-1837. DOI:10.1021/acssynbio.9b00140 |
| [69] |
Wu Y, Chen T, Liu Y, et al. Design of a programmable biosensorCRISPRi genetic circuits for dynamic and autonomous dual-control of metabolic flux in Bacillus subtilis[J]. Nucleic Acids Research, 2020, 48(2): 996-1009. DOI:10.1093/nar/gkz1123 |
| [70] |
Lv Y, Qian S, Du G, et al. Coupling feedback genetic circuits with growth phenotype for dynamic population control and intelligent bioproduction[J]. Metab Eng, 2019, 54: 109-116. DOI:10.1016/j.ymben.2019.03.009 |
| [71] |
Gu F, Jiang W, Mu Y, et al. Quorum sensing-based dual-function switch and its application in solving two key metabolic engineering problems[J]. ACS Synthetic Biology, 2020, 9(2): 209-217. DOI:10.1021/acssynbio.9b00290 |
| [72] |
Burg JM, Cooper CB, Ye Z, et al. Large-scale bioprocess competitiveness :the potential of dynamic metabolic control in two-stage fermentations[J]. Current Opinion in Chemical Engineering, 2016, 14: 121-136. DOI:10.1016/j.coche.2016.09.008 |
| [73] |
Liu D, Xiao Y, Evans B S, et al. Negative feedback regulation of fatty acid production based on a malonyl-CoA sensoractuator[J]. ACS Synthetic Biology, 2015, 4(2): 132-140. DOI:10.1021/sb400158w |
| [74] |
Zargar A, Quan DN, Bentley WE. Enhancing intercellular coordination :Rewiring quorum sensing networks for increased protein expression through autonomous induction[J]. ACS Synthetic Biology, 2016, 5(9): 923-928. DOI:10.1021/acssynbio.5b00261 |
| [75] |
Shi S, Ang EL, Zhao H. In vivo biosensors :mechanisms, development, and applications[J]. Journal of Industrial Microbiology & Biotechnology, 2018, 45(7): 491-516. |
| [76] |
Zhang F, Keasling J. Biosensors and their applications in microbial metabolic engineering[J]. Trends Microbiol, 2011, 19(7): 323-329. DOI:10.1016/j.tim.2011.05.003 |
| [77] |
Cao J, Yao Y, Fan K, et al. Harnessing a previously unidentified capability of bacterial allosteric transcription factors for sensing diverse small molecules in vitro[J]. Science Advances, 2018, 4(11): eaau4602. |
| [78] |
Venayak N, von Kamp A, Klamt S, et al. MoVE identifies metabolic valves to switch between phenotypic states[J]. Nature Communications, 2018, 9(1): 1-9. DOI:10.1038/s41467-017-02088-w |
| [79] |
Younger AKD, Su PY, Shepard AJ, et al. Development of novel metabolite-responsive transcription factors via transposon-mediated protein fusion[J]. Protein Eng Des Sel, 2018, 31(2): 55-63. |
| [80] |
Younger AKD, Dalvie NC, Rottinghaus AG, et al. Engineering modular biosensors to confer metabolite-responsive regulation of transcription[J]. ACS Synth Biol, 2017, 6(2): 311-325. DOI:10.1021/acssynbio.6b00184 |
| [81] |
Ceroni F, Blount BA, Ellis T. Sensing the right time to be productive[J]. Cell Systems, 2016, 3(2): 116-117. |
| [82] |
Pinto D, Vecchione S, Wu H, et al. Engineering orthogonal synthetic timer circuits based on extracytoplasmic function σ factors[J]. Nucleic Acids Res, 2018, 46(14): 7450-7464. DOI:10.1093/nar/gky614 |
| [83] |
Meyer AJ, Segall-Shapiro TH, Glassey E, et al. Escherichia coli "Marionette" strains with 12 highly optimized small-molecule sensors[J]. Nature Chemical Biology, 2019, 15(2): 196-204. DOI:10.1038/s41589-018-0168-3 |
| [84] |
Tan D, Wu Q, Chen JC, et al. Engineering Halomonas TD01 for the low-cost production of polyhydroxyalkanoates[J]. Metabolic Engineering, 2014, 26: 34-47. DOI:10.1016/j.ymben.2014.09.001 |
| [85] |
Dai Z, Lee A J, Roberts S, et al. Versatile biomanufacturing through stimulus-responsive cell-material feedback[J]. Nature Chemical Biology, 2019, 15(10): 1017-1024. DOI:10.1038/s41589-019-0357-8 |
| [86] |
Chappell J, Westbrook A, Verosloff M, et al. Computational design of small transcription activating RNAs for versatile and dynamic gene regulation[J]. Nature Communications, 2017, 8(1): 1-12. DOI:10.1038/s41467-016-0009-6 |
| [87] |
Baumschlager A, Aoki SK, Khammash M. Dynamic blue lightinducible T7 RNA polymerases(Opto-T7RNAPs)for precise spatiotemporal gene expression control[J]. ACS Synthetic Biology, 2017, 6(11): 2157-2167. DOI:10.1021/acssynbio.7b00169 |
| [88] |
Tan SZ, Prather KLJ. Dynamic pathway regulation :recent advances and methods of construction[J]. Current Opinion in Chemical Biology, 2017, 41: 28-35. DOI:10.1016/j.cbpa.2017.10.004 |
| [89] |
Borkowski O, Bricio C, Murgiano M, et al. Cell-free prediction of protein expression costs for growing cells[J]. Nature Communications, 2018, 9(1): 1-11. DOI:10.1038/s41467-017-02088-w |
| [90] |
Stephens K, Pozo M, Tsao CY, et al. Bacterial co-culture with cell signaling translator and growth controller modules for autonomously regulated culture composition[J]. Nature Communications, 2019, 10(1): 1-11. DOI:10.1038/s41467-018-07882-8 |
| [91] |
Honjo H, Iwasaki K, Soma Y, et al. Synthetic microbial consortium with specific roles designated by genetic circuits for cooperative chemical production[J]. Metab Eng, 2019, 55: 268-275. DOI:10.1016/j.ymben.2019.08.007 |
| [92] |
He X, Chen Y, Liang Q, et al. Autoinduced AND gate controls metabolic pathway dynamically in response to microbial communities and cell physiological state[J]. ACS Synthetic Biology, 2017, 6(3): 463-470. DOI:10.1021/acssynbio.6b00177 |
| [93] |
Pai A, Tanouchi Y, Collins CH, et al. Engineering multicellular systems by cell-cell communication[J]. Current Opinion in Biotechnology, 2009, 20(4): 461-470. DOI:10.1016/j.copbio.2009.08.006 |
| [94] |
Sedlmayer F, Hell D, Müller M, et al. Designer cells programming quorum-sensing interference with microbes[J]. Nature Communications, 2018, 9(1): 1-13. DOI:10.1038/s41467-017-02088-w |
| [95] |
Kobayashi H, Kaern M, Araki M, et al. Programmable cells :interfacing natural and engineered gene networks[J]. PNAS, 2004, 101(22): 8414-8419. DOI:10.1073/pnas.0402940101 |
| [96] |
Saltepe B, Kehribar ES, Su Yirmibeşoğlu SS, et al. Cellular biosensors with engineered genetic circuits[J]. ACS sensors, 2018, 3(1): 13-26. |





