近年来,随着生物技术及相关专业的发展,针对转基因作物的研究和应用愈发广泛,截至2018年底,全球转基因作物种植面积已达1.917亿hm2[1];与此同时,各国家和地区对于转基因作物的安全管理也日趋严格,开始实施标识制度和系统化管理[2]。
RNA干扰(RNAi,RNA interference)作为一种新兴的热门转基因生物技术,常指一些短链的双链RNA(dsRNA)可以通过促使特异性mRNA降解来高效地阻断生物体内特异性基因的表达[3]。该技术在基因功能研究领域发挥了重要作用,并被认为是极具应用潜力的育种新方法,已在转基因番茄、马铃薯等作物上实现了深入研究[4-6]。但由于RNAi技术存在沉默机制的特异性可能在传代、生长环境等过程中产生变化、沉默在物种间可能产生的漂移、偶然性触发等因素,RNAi转基因作物的安全评价便显得尤为重要,但目前国内外尚未见对RNAi转基因植物有权威的评价及管理方法,这将对我国转基因安全监管十分不利。因此,寻找到RNAi转基因植物的高效检测方法,对其进行精确定量的意义重大。
目前,针对转基因产物定量检测主要依靠PCR技术[7]。实时荧光定量PCR技术(Real-time PCR)和普通PCR法相比灵敏度更高,并且能对基因拷贝数进行定量。但由于它是依靠Ct值和标准曲线进行相对定量的,因此对于低拷贝数和来源复杂样品的检测有很大的局限性[8]。随着PCR技术的不断发展,第三代数字PCR(digital PCR,dPCR)以其灵敏度高、绝对定量精确、检测线性范围宽、特异性好等优势脱颖而出。dPCR采用分散手段,将完整的PCR体系分散至成千上万个独立体系中,使得每个独立PCR体系中至多包含一个模板(实际样品中的模板数符合泊松分布),并对这些体系是否发生了PCR反应进行检测从而达到精确的绝对定量[9]。由于上述多种优势,dPCR技术在近年来飞速发展,已经多次被应用于转基因成分的定量检测实验研究中[10]。
我国作为农业大国,在过去数十年中一直致力于开发用于作物抗昆虫侵袭的方法。以前采用的传统手段如化学杀虫剂等,虽效果较理想,但对非目标生物如蚯蚓等也会造成伤害;并且由于其难以代谢,长期使用会导致田野贫瘠;更严重的是会积累于食物链中,对更高端物种将会诱导疾病的产生。RNAi能以序列特异性方式下调基因的表达,在害虫防治方面的应用已较为成熟。本研究中选用的转基因玉米DBN11061,是一种利用RNAi技术以降低或关闭昆虫体内靶序列表达来削弱昆虫存活、繁殖或是侵袭寄主能力的玉米品系。本研究中将以该转基因玉米品系的特异性dsRNA为实验目标,建立并优化其逆转录数字PCR(RT-dPCR)定量检测方法,为RNAi转基因作物的定量检测和基于RNAi技术的转基因作物安全评价做出贡献。
1 材料与方法 1.1 材料RNAi转基因玉米DBN11061(北京大北农生物技术有限公司);植物RNA提取试剂盒(ExtrationKits,ET Kits)(PureLinkTM RNA Mini Kit,赛默飞世尔科技(中国)有限公司;柱式植物总RNA抽提纯化试剂盒,生工生物工程(上海)股份有限公司;MiniBEST Plant RNA Extraction Kit,宝生物工程(大连)有限公司);反转录试剂盒(Reverse Transcription Kits,RT Kits)(ProtoScriptcDNA第一链合成试剂盒,NEB(北京)有限公司;PrimeScriptTM II 1st Strand cDNA Synthesis Kit,宝生物工程(大连)有限公司;Maxima H Minus First Strand cDNA Synthesis Kit,赛默飞世尔科技(中国)有限公司);引物、探针合成(赛默飞世尔科技(中国)有限公司);数字PCR系统(QuantStudioTM 3D,赛默飞世尔科技(中国)有限公司);核酸定量仪(Nanodrop2000,赛默飞世尔科技(中国)有限公司)。
1.2 方法 1.2.1 引物探针的设计针对转基因玉米DBN11061中引发特异序列基因沉默的dsRNA序列设计dPCR正反向引物及探针。
1.2.2 转基因玉米总RNA的抽提方法优化在提取过程中,均先使用液氮将叶片磨成粉末,再分别使用不同方法对转基因玉米叶片进行总RNA提取,得到的RNA存放于-80℃。使用同一反转录方法对RNA进行反转录后得到cDNA,并按照QuantStudioTM 3D数字PCR仪默认程序进行数字PCR并比较结果,实验均重复3次。
1.2.3 RNA反转录为cDNA的方法优化使用1.2.2中最优提取方法得到的转基因玉米叶片RNA按照不同方法进行反转录,得到的cDNA按照QuantStudioTM 3D默认程序进行数字PCR并比较结果,实验均重复3次。
1.2.4 数字PCR反应条件的优化不同的模板及引物探针所需的最佳反应条件均不相同,其中退火温度为最关键因素。我们选择的dPCR反应条件为:(1)酶激活和预变性阶段:96℃,10 min;(2)循环扩增阶段:60、58、56、54和52℃(不同退火温度)/2 min,98℃/30 s,共40个循环。升温速率为0.8℃/s,降温速率为1.2℃/s,分别根据不同退火温度进行数字PCR并比较结果。
1.2.5 数字PCR反应体系的优化数字PCR方法的准确性和反应体系中的引物、探针浓度密切相关,针对转基因玉米DBN11061中dsRNA序列PCR反应体系中的引物、探针浓度进行优化。dPCR反应体系(总体积为20 μL)中,分别选取探针终浓度为:10 nmol/L、25 nmol/L、50 nmol/L、75 nmol/L、100 nmol/L;外源基因的正反向引物终浓度为50 nmol/L、100 nmol/L、200 nmol/L、400 nmol/L、1 000 nmol/L;Master mix体积为10 μL,H2O体积为8 μL,依次根据1.5中最优反应条件进行数字PCR并比较结果。
1.2.6 实验方法的定量检测低限的测定将cDNA样品依次稀释1、10、20、50、100倍,根据1.2.4及1.2.5的反应体系及条件进行dPCR,每个扩增实验重复3次,并计算测得平行数据的RSD值,以相对标准偏差(Relative standard deviation,RSD)≤25%[11]作为有效定量数据的判断依据,判断本文建立的dPCR体系定量检测低限的浓度值。
2 结果 2.1 引物探针的设计针对转基因玉米品系DBN11061的dsRNA序列进行引物探针设计,序列如表 1。
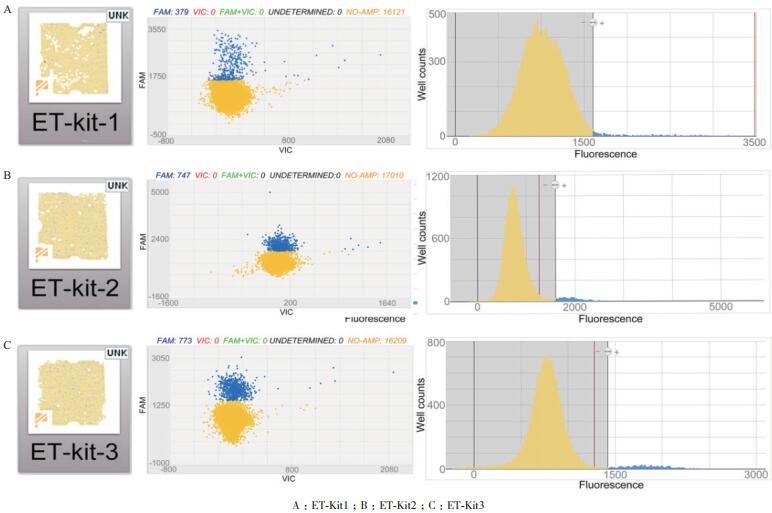
不同的植物总RNA提取方法的原理基本类似,提取流程主要包括:(1)加入液氮充分研磨,以彻底破碎植物细胞的细胞壁;(2)使核蛋白复合体中的蛋白质变性,致使蛋白质与核酸分离;(3)抑制RNA酶活性,以防止释放出的RNA被污染;(4)分离RNA与DNA、蛋白质、酚类物质、多糖等;(5)溶解并得到总RNA,检测质量与浓度。本实验主要选取3种转基因植物总RNA提取试剂盒,在称取相同质量的转基因玉米叶片提取得到RNA后,使用同种反转录方法得到cDNA,再运用同种反应体系与条件进行数字PCR,通过比较数字PCR的结果筛选出相对最优的提取方法。实验结果如图 1。
|
| 图 1 不同反应提取方法得到的dPCR芯片图 |
取等体积RNA进行逆转录后,将得到的cDNA按1.2.4、1.2.5的PCR体系(引物终浓度为200 nmol/L,探针终浓度为50 nmol/L)及反应条件(退火温度为52℃)进行数字PCR扩增,并比较各提取方法得出的结果。
综合3种提取方法得到RNA的dPCR结果,我们发现,ET-Kit3提取得的RNA经过反转录后得到的cDNA在进行dPCR实验中,信号区分最显著、阈值线易划分。因此后续相关转基因玉米RNA提取实验均采用ET Kit 3。
2.3 转基因植物RNA的反转录方法优化反转录反应过程为:(1)65℃反应不同时间,冰上冷却,以使RNA变性,打开其二级结构;(2)加入随机引物、dNTP、buffer、逆转录酶在较低温度(不同提取方法温度不同)反应,以使引物与模板RNA退火结合;(3)在逆转录酶工作温度(不同提取方法温度不同)下反应;(4)变性:使合成的cDNA和RNA区分开;(5)在较高温度(不同提取方法温度不同)反应,以使逆转录酶失活。在上述实验过程中,RNA逆转录酶的活性和效率至关重要。因此我们选择3种不同的条件进行dPCR,筛选最优的反转录方法。
结果(表 2)发现,RT Kit B的逆转录效率远高于另两种方法,因此后续实验所涉及的反转录步骤均使用RT Kit B。
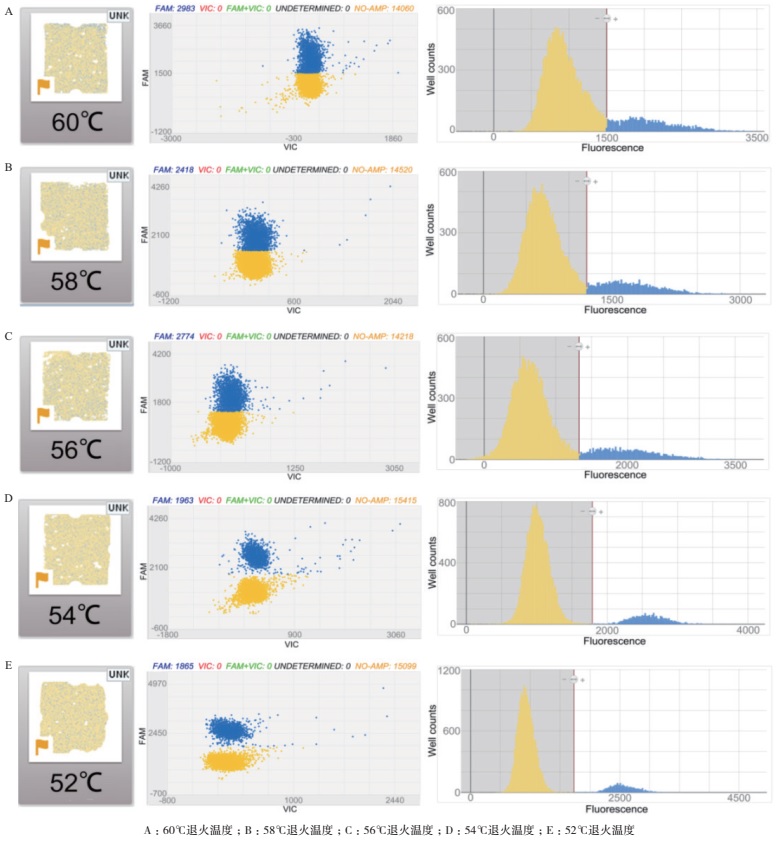
在PCR反应中,退火温度是最重要的参数。在反应过程中,退火温度需要足够低,以确保引物和目的序列有效退火;并且还需要足够高,以减少非特异性结合。在dPCR反应过程中,合理的退火温度约为60℃。本文中分别选取退火温度为60、58、56、54和52℃并按照1.2.5中的PCR体系(引物终浓度为200 nmol/L,探针终浓度为50 nmol/L)依次独立进行实验,以优化得到最佳退火温度。
根据实验结果的芯片图(图 2)我们可以发现,当退火温度为60-56℃时,信号尚区分不明显,阈值线不能明确划分;当降至52℃时,信号区分最明显,阈值线显著。因此dPCR反应条件确定为:酶激活和预变性阶段:96℃,10 min;循环扩增阶段:52℃/2 min,98℃/30 s,共40个循环。升温速率为0.8℃/s,降温速率为1.2℃/s。
|
| 图 2 不同反应条件得到的dPCR芯片图 |
数字PCR方法的准确性和反应体系中的引物、探针浓度密切相关,直接关系到阳性信号和阴性信号的区分,以及后续dPCR结果的准确度,为此,我们对引物、探针浓度进行了优化,依次进行dPCR。
2.4.2.1 探针浓度的优化按照1.2.5中体系,分别选取探针终浓度为:10、25、50、75、100 nmol/L依次进行dPCR,结果如图 3,当探针终浓度为50 nmol/L时,信号区分最佳,因此确定该浓度为探针浓度。

|
| 图 3 不同探针浓度得到的dPCR芯片图 |
按照1.2.5中体系,采用外源基因的正反向引物终浓度为50、100、200、400、1 000 nmol/L,依次进行dPCR,结果如图 4。当引物终浓度为200 nmol/L时信号区分最佳,因此确定200 nmol/L为引物终浓度。

|
| 图 4 不同引物浓度得到的dPCR芯片图 |
为了得到通过上述实验所建立方法的定量检测低限,我们将使用ETKit 3试剂盒提取得到的RNA(600 ng/μL)使用RT Kit B反转录后得到的cDNA依次稀释1、10、20、50、100倍,并依照2.4中得到的最佳反应体系及条件进行数字PCR,结果如表 3。
当cDNA模板稀释至50倍时,检测浓度为21.7 copies/μL,RSD为14.6%,小于转基因成分定量检测平行数据RSD为25%的接受阈值[11],可以用于转基因的定量检测。而当检测空白样品时,平均值为1.8 copies/μL,标准差为0.23 copies/μL,RSD为12.4%,根据定量检测中LOD的计算方法,判定本文建立的dPCR体系绝对定量检测低限约为2.5 copies/μL。
3 讨论在我国,由于害虫侵袭造成危害的区域已逐渐从西北等地蔓延到东北、华北等玉米主要生产区域。因此,研究出能控制昆虫虫害并对植物损害较小的方法一直是相关领域研究者的目标。本研究中提及的RNAi技术通过下调靶序列来控制昆虫的侵袭,例如破坏昆虫生长必须的生物过程[12]。当特定的靶序列表达被下调或抑制时,会减少至少30%的虫害威胁。此外,这些靶序列可以仅在昆虫的生长特定阶段表达。本实验以DBN11061品系中dsRNA序列为目标,设计引物探针进行dPCR定量检测,从玉米的总RNA提取方法、RNA反转录方法、dPCR退火温度、所用引物探针浓度等方面均进行条件优化,从而建立了一套完整的转基因玉米RT-dPCR定量检测方法。
在得到转基因品系玉米的cDNA过程中发现,用ET-Kit 3提取试剂盒所得RNA的浓度及纯度优于其他两种提取方法;在反转录过程中,针对不同的总RNA,不同的反转录酶活性也大相径庭。在本实验中RT-Kit B反转录活性显然优于其他两种反转录酶。因此后续实验检测所用的cDNA均由ET-Kit 3提取玉米中的总RNA后,使用RT-Kit B进行反转录。
在数字PCR的体系及反应条件优化过程中我们将反应条件确定为:酶激活和预变性阶段:96℃,10 min;循环扩增阶段:52℃/2 min,98℃/30 s,共40个循环。升温速率为0.8℃/s,降温速率为1.2℃/s;反应体系为总体积为20 μL中,探针终浓度为50 nmol/L,外源基因的正反向引物终浓度为200 nmol/L,Master mix体积为10 μL,H2O体积为8 μL,模板为2 μL。
依照本文建立的数字PCR检测方法,绝对定量检测低限约为2.5 copies/μL,RSD为12.4%;低浓度实际样品达21.7 copies/μL时,相对偏差为2.7%,RSD为14.6%,优于欧盟的转基因定量标识限[13]。
与传统PCR相比,本实验所采用的dPCR更高效、更稳定,且由于不需要制作标准曲线,也避免了昂贵的标准物质的使用和PCR扩增效率差异对实验结果的影响。将本实验构建并优化的方法应用于转基因RNAi品系玉米dsRNA检测研究,将为我国转基因作物的定量检测工作提供技术支撑和方法依靠。
4 结论本研究针对基于RNAi技术的转基因玉米品系,通过对植物提取方法、逆转录方法、数字PCR条件及体系的摸索,建立了一套逆转录芯片式数字PCR定量检测该品系玉米双链RNA的方法,该方法的绝对定量检测低限约为2.5 copies/μL,RSD为12.4%。
| [1] |
国际农业生物技术应用服务组织. 2018年全球生物技术/转基因作物商业化发展态势[J]. 中国生物工程杂志, 2019, 39(8): 1-6. |
| [2] |
徐琳杰, 刘培磊, 熊鹂, 等. 国际上主要国家和地区农业转基因产品的标识制度[J]. 生物安全学报, 2014, 23(3): 301-304. |
| [3] |
Andrew F. RNA-triggered gene silencing[J]. Trends in Genetics, 1999, 15(9): 358-363. DOI:10.1016/S0168-9525(99)01818-1 |
| [4] |
Schwind N, Zwiebel M, Itaya A, et al. RNAi-mediated resistance to potato spindle tuber viroid in transgenic tomato expressing a viroid hairpin RNA construct[J]. Molecular Plant Pathology, 2010, 10(4): 459-469. |
| [5] |
Yang Y, Sherwood T, Patte C, et al. Use of tomato yellow leaf curl virus(TYLCV)rep gene sequences to engineer TYLCV resistance in tomata[J]. Phytopathology, 2004, 94(5): 490-496. DOI:10.1094/PHYTO.2004.94.5.490 |
| [6] |
Tenlado F, Dlaz-Rufz JR. Double-stranded RNA-mediated interference with plant virus infection[J]. J Virol, 2001, 75(24): 12288-12297. DOI:10.1128/JVI.75.24.12288-12297.2001 |
| [7] |
李夏荧, 高鸿飞, 刘鹏程, 等. 转基因作物快速检测技术的研究进展[J]. 江苏农业科学, 2018, 46(3): 5-9. |
| [8] |
王荣谈, 刘冬儿, 厉建萌, 等. 实时荧光定量PCR技术及其在植物转基因产品检测中的应用[J]. 植物生理学通讯, 2009, 45(6): 591-597. |
| [9] |
Quan PL, Sauzade M, Brouzes E. dPCR:A technology review[J]. Sensors(Basel), 2018, 18(4): 1271. |
| [10] |
Tigst D, David D. Critical assessment of digital PCR for the detec-tion and quantification of genetically modified organisms[J]. Anal BioanalChem, 2018, 410(17): 4039-4050. DOI:10.1007/s00216-018-1010-1 |
| [11] |
European Network of GMO Laboratories. Definition of minimum performance requirements for analytical methods of GMO testing[J]. ResearchGate, 2015, 5: 1-24. |
| [12] |
左洪亮, 李海怡, 高璐, 等. RNAi作用机制及其在害虫防治领域的应用前景[J]. 公共植保与绿色防护, 2010, 379-384. |
| [13] |
The European Parliament and The Council of The European Union. Regulation(EC)NO 1829/2003 of the European Parliament and of the Council of 22 September 2003 on genetically modified food and feed[J]. Official Journal of the European Communities, 2003, L268: 1-23. |