肝癌是全球恶性肿瘤患者死亡的主要原因之一,在我国2017年肿瘤死亡排名中高居第二位,而肝细胞癌(Hepatocellular carcinoma,HCC)在原发性肝癌中占比高达70%-90%[1-3]。近年来,HCC的治疗措施有了很大的改善,包括手术切除、射频消融术、肝移植、栓塞化疗术、靶向药物治疗等,但是HCC患者的5年生存率仍然偏低[4-5]。另一方面,处于同一肿瘤分期的HCC患者可能有不同的预后表现,故仅基于临床指标来预测HCC患者预后可能不尽人意[6]。因此,引入分子水平信息对患者进行更加精准的风险分型十分必要,将促进精准医疗的开展。
目前,利用分子标志物进行HCC风险分型预测的模型的大部分研究利用基因差异表达分析筛选出的mRNA或miRNA建立风险打分模型进行HCC患者风险预测,每次通过中位数或软件设置单数据集打分分界点将HCC患者分成高低风险组来验证模型的准确性,但用这些模型进行分型难以保证不同数据集的打分分界点的统一性[7-12]。Liu等[13]整合多组学数据将HCC分成5种类型,部分类型却呈现类似的总体生存期表现。有些模型虽然稳健,但需要较多的分子信息来进行模型构建,如Chaudhary等[2]用深度学习的方法整合mRNA、miRNA及甲基化等多组学数据,Fa等[14]基于多条通路或通路子集将HCC分成两类生存风险有差异的组别。因此,如何利用较少的分子信息来建立预后模型,既能保证较高的预测稳健性与准确性,又具备操作简单、诊断成本低廉的特点,是本研究的主要目标。
本研究首先针对HCC预后相关的多条通路,通过主成分分析筛选出41个核心基因。然后通过k均值聚类、支持向量机等机器学习的方法在TCGA数据集上建立HCC患者分组预后模型,并在TCGA数据集内部及3个外部数据集上进行稳健性、有效性验证。最后通过HCC患者组间基因差异表达分析和富集分析,筛选出与HCC疾病进展有关的基因与通路,以期为个性化医疗提供理论依据,同时进一步验证模型分类的有效性。
1 材料与方法 1.1 材料肝细胞癌患者基因表达和临床数据来自4个数据集TCGA、LIRI-JP、GSE14520及GSE54236。其中,TCGA数据集通过TCGA-Assembler 2.0.6软件从癌症和肿瘤基因图谱(The Cancer Genome Atlas,TCGA)数据库下载(https://tcga-data.nci.nih.gov/tcga/)[15];LIRI-JP数据集来自国际肿瘤基因组协作组(International Caner Genome Consortium,ICGC)数据库(https://dcc.icgc.org/projects/LIRI-JP)[16];GSE14520和GSE54236两个数据集来自NCBI的GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)[17-18]。去除生存时间不正确或缺失及非HCC患者数据,最终得到不同样本量的HCC患者数据集如表 1所示。其中,数据集GSE54236缺乏生存时间删失(Censoring)信息。
根据文献中提到的与肝细胞癌生存风险显著相关的13个信号通路或通路子集[14],我们分别对TCGA、LIRI-JP及GSE14520三个HCC数据集相应信号通路中的基因表达数据进行主成分分析(Principle component analysis,PCA),分别截取主成分载荷(Loading)大于0.3及总方差的累积解释比例(Cumulative proportion)大于0.8的主成分后取交集,得到通路中的关键基因。PCA分析使用R语言内置的prcomp函数。基于上一步得到的与生存相关的关键基因,首先对整个TCGA数据集做无监督K均值聚类(K-means clustering,k=2),将HCC患者分为两组。用log-rank检验(R包survival[19])比较两组间生存风险差异,将高风险组记为S1(Subgroup 1),低风险组记为S2(Subgroup 2)。然后使用支持向量机(Support vector machine,SVM)进行有监督建模。模型构建与验证采用R语言内置的kmeans函数和R包e1071中svm函数(https://cran.r-project.org/web/packages/e1071/index.html),其中SVM模型的最优参数使用交叉验证的方法选择。
为了验证该模型的稳定性,我们用整个TCGA数据集进行交叉验证,即将数据集随机分成5个大小一致的组,任意选择3组做训练集,其余2组做测试集,共得到10个数据组合进行验证。为了评价模型的预测能力,我们将整个TCGA数据集作为训练集,验证模型在LIRI-JP、GSE14520及GSE54236数据集中的预测能力。
对组别间生存风险差异评价使用以下3种衡量指标:一致性指数(Concordance index,C-index,R包survcomp[20])、log-rank检验的P值(Log-rank P value of Cox-PH model)及Brier分数(Brier score,R包survcomp)。一致性指数由Harrell提出[21],该值范围在0到1之间,越大表示模型预测结果与实际生存风险排序越一致。总生存期(Overall survival,OS)的KM(Kaplan-Meier)曲线绘制使用R包survminer中的ggsurvplot函数(https://cran.r-project.org/web/packages/survminer/index.html),并用log-rank检验比较组间生存差异。Brier分数衡量预测结果与真实结果间的差异,该值越小表示模型预测能力越好。
在建模之前,对训练集及测试集进行了两步归一化处理。首先,对所有数据集分别进行中位数标准化,即利用该数据集所有肿瘤样本对应基因表达值的中位数(Median)和绝对中位差(Median absolute deviation,MAD)对该基因表达值进行标准化。其次,利用TCGA训练集的各基因归一化后的均值及方差对测试集数据进一步标准化。需要指出的,无论是测序数据还是芯片数据,我们均用各数据内最小值(近似仪器检测下限)来填补缺失值,未log2处理的数据进行对数化。针对芯片数据多个探针对应一个基因的情况,我们取其平均值。
1.2.2 基于单因素Cox回归的基因筛选及HCC患者预后分组模型的建立为了与上述基于信号通路中关键基因的模型做对比,利用单因素Cox回归进行基因筛选。取TCGA、LIRI-JP及GSE14520 3个HCC数据集的共有基因,然后在TCGA数据集上,利用单因素Cox回归筛选上述基因中与生存风险最显著相关的子集(Log-rank P值最小)。为保证可比性,筛选的基因数目、HCC患者预后分组模型的建立方法及稳定性、预测能力的检验与1.2.1相同。
1.2.3 HCC疾病进展相关的差异基因筛选应用1.2.1中构建的风险预测模型可分别将3个主要数据集TCGA、LIRI-JP及GSE14520中的HCC患者分成两个生存风险存在显著差异的组别。我们对两组间的肿瘤样本(HCC高风险与低风险组相比)进行差异表达基因(Differentially expressed genes,DEGs)筛选,其中测序数据、芯片数据分别使用R包DESeq2和limma进行分析[22-23]。差异检验使用BH(Benjamini-Hochberg)方法控制假阳性率[24]。对3个数据集中满足|log2FoldChange| > 1、校正后P值< 0.05的差异表达基因取交集进行后续分析。
1.2.4 差异表达基因的功能富集及注释使用R包clusterProfiler对上一步得到的与HCC疾病进展有关的差异基因进行基因本体(Gene ontology,GO)富集分析和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)信号通路分析,探索与HCC疾病进展有关的生物学过程[25]。筛选条件均为BH校正后的P值< 0.01,q值< 0.05。
1.2.5 差异表达基因的蛋白质互相作用网络及子网络构建使用STRING 11.0数据库(https://string-db.org)分析与HCC疾病进展有关的差异表达基因的蛋白质相互作用网络(Protein-protein interaction network,PPI),并用Cytoscape软件进行可视化处理[26-27]。其中蛋白质节点的度(Degree)用插件CentiScaPe计算,图示节点大小与度成正比,并用插件MCODE(Molecular complex detection)分析子网络[28-29]。对度排名前20的蛋白质,与KEGG通路过程(P值< 0.05)取交集,得到hub基因(Hub genes)。使用插件ClueGo+CluePedia分析hub基因参与的重要KEGG通路过程[30-31]。
1.2.6 hub基因的预后相关性分析将TCGA数据集中355例HCC患者的总体生存时间与hub基因表达特征进行统计检验,探索这些hub基因对肝细胞癌的预后价值。根据每个hub基因表达水平的中位数将HCC患者分为高表达组(表达值> 中位数)和低表达组(表达值≤中位数),通过KM曲线图、log-rank检验来研究两组之间的总生存期差异。
2 结果 2.1 基于信号通路的基因筛选结果及HCC患者预后分组模型的表现根据文献中提到的与HCC生存风险显著相关的13条信号通路或通路子集,我们分别对TCGA、LIRI-JP及GSE14520三个HCC数据集中相应信号通路中的基因表达数据进行主成分分析,取交集后得到41个核心基因:CDK1、DUT、EGF、ENO1、FOXM1、G6PD、GMDS、GNAS、GNG4、GPI、GPX7、HEXA、IRAK1、ITPR1、PPP2R5A、MAPK1、MAPK9、MAPK13、MAP2K2、RAC1、RAD51、RAF1、RENBP、RRM2、TGFA、TK1、TKT、TYMS、UGDH、UCK2、IKBKG、SQSTM1、WASF1、GNPDA1、GNB5、RALBP1、PLCB1、CYB5R1、HKDC1、NPL和UAP1L1。
根据信号通路筛选的41个基因及无监督聚类得到的标签对TCGA基因表达数据建立HCC风险预测模型,其中稳健性及准确性分析结果如表 2所示。TCGA数据集交叉验证结果表明,模型的预测准确率在0.71附近,HCC患者组间总体生存期的log-rank检验P值平均为0.03,且Brier分数较低。在其他3个数据集上,该模型区分的HCC患者组间总体生存期均存在显著差异(log-rank检验P值< 0.05),预测准确率均高于0.7(一致性指数 > 0.7)。4个数据集的KM曲线(图 1)可进一步直观表明,该模型可将HCC患者分成生存风险存在显著差异的两组(S1高风险组和S2低风险组)。其中,TCGA数据集log-rank检验P值为0.000 18,LIRI-JP数据集log-rank检验P值为0.000 38,GSE14520数据集log-rank检验P值为0.002 1,GSE54236数据集log-rank检验P值为0.012。
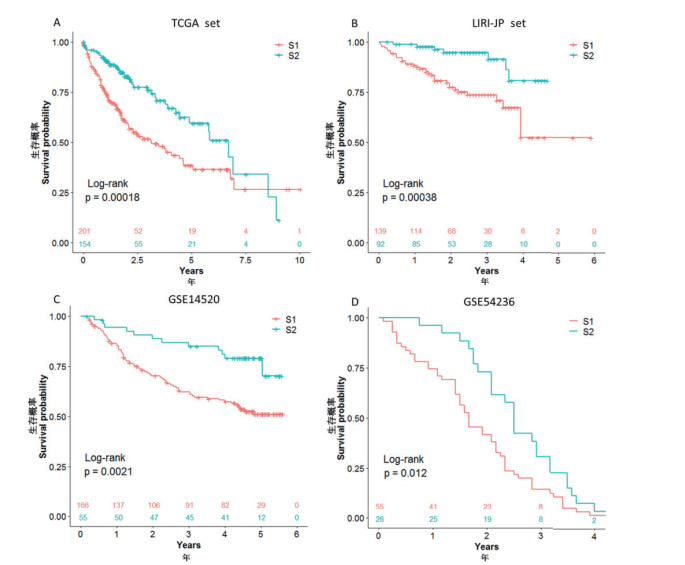
|
| 图 1 四个数据集的两组间总体生存期差异 |
针对TCGA、LIRI-JP及GSE14520三个HCC数据集的共有基因,利用单因素Cox回归得到TCGA数据集中与生存风险最显著相关的41个基因:CDCA8、SFPQ、KIF20A、G6PD、PSRC1、UTP11、TTC26、VRK2、KIF2C、CENPA、NEIL3、CCNJL、UCK2、TTK、ZNF643、NCAPG、YBX1、NDC80、PDE6A、TXNRD1、TPX2、SEPHS1、HDAC2、DYNC1LI1、DBF4、HJURP、DLGAP5、CHORDC1、C19orf26、CEP85、MCM10、CPSF6、EZH2、CBX2、GLMN、NCAPD2、GNL2、ZNF239、NUP205、KIF18A及TRIP13。
根据单因素Cox回归筛选的41个基因及无监督聚类得到的标签对TCGA基因表达数据再次建立HCC风险预测模型,其中稳健性及准确性分析结果如表 2所示。TCGA数据集交叉验证结果表明,该模型的预测准确率也在0.71附近,HCC患者组间总体生存期的log-rank检验P值平均为0.02,且Brier分数较低。在其他3个数据集上,该模型区分的HCC患者组间总体生存期均存在显著差异(log-rank检验P值< 0.05),预测准确率均高于0.65(一致性指数 > 0.65)。
2.3 HCC疾病进展相关的基因差异表达分析与富集分析的结果基于信号通路中41个核心基因的模型可将HCC患者分成高风险组S1和低风险组S2,S1组患者的总体生存时间显著低于S2组。通过对TCGA、LIRI-JP及GSE14520三个HCC数据集各自S1与S2组之间进行基因差异表达分析,最终筛选出61个上调基因及122个下调基因。3个数据集所得的差异表达基因数目具体如图 2-A所示。
对上述得到的HCC疾病进展相关的差异表达基因进行GO富集分析(前10个显著结果见图 2-B),发现上调基因主要富集在染色体分离、细胞核分裂、细胞器分裂、细胞核有丝分裂等生物学过程,下调基因主要富集在小分子分解代谢过程、类固醇代谢过程、羧酸生物合成过程等生物学代谢过程。对差异表达基因进一步进行KEGG通路富集分析(图 2-C),发现上调基因主要涉及细胞周期、卵母细胞减数分裂、p53信号通路、DNA复制等信号通路,下调基因主要涉及视黄醇代谢、细胞色素P450蛋白负责的药物代谢等代谢通路。
2.4 差异表达基因的蛋白质互作网络及hub基因筛选利用STRING 11.0数据库与Cytoscape 3.7.2软件,构建如图 3所示的162个节点、1 172条边的差异表达基因的蛋白质互作网络。通过插件MCODE分析后发现两个主要的子网络模块1(35个节点,557条边,Score=34.353)和模块2(28个节点,174条边,Score=12.889),图示环状。将编码度排名前20的结点蛋白质的差异基因与KEGG通路富集过程取交集,得到12个hub基因:CCNB1、CDK1、RRM2、BUB1B、CCNB2、TTK、CDC20、MCM4、RFC4、PTTG1、MCM2和MAD2L1。这12个基因均属于模块1的相互作用网络,包含66条边的互作关系(图 4-A),经过KEGG富集分析发现主要集中在细胞周期、卵母细胞减数分裂等信号通路(图 4-B)。
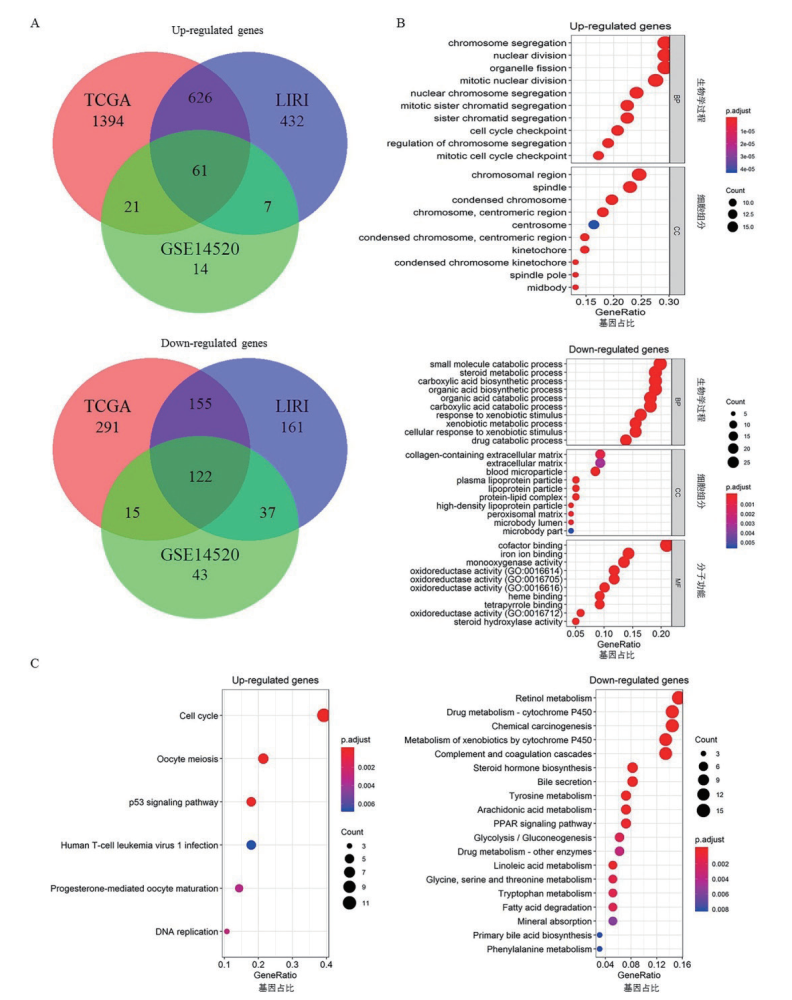
|
| A:3个数据集的上调基因及下调基因;B:差异表达基因的GO富集分析;C:差异表达基因的KEGG通路富集分析 图 2 差异表达基因(S1和S2组相比)的筛选及功能注释 |
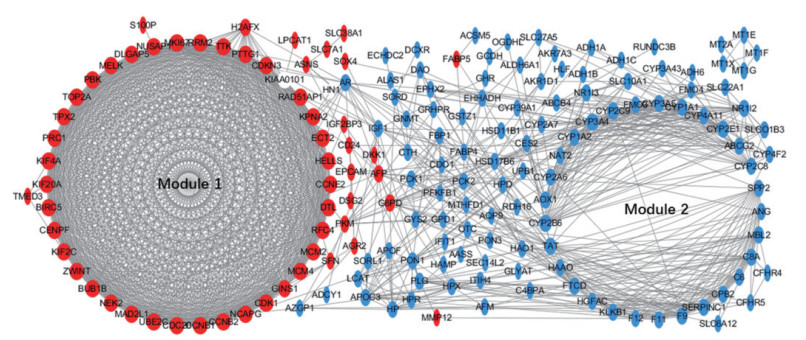
|
| 红色表示上调基因,蓝色表示下调基因,白色表示非差异表达基因 图 3 差异表达基因的蛋白质相互作用网络分析(组S1和S2相比) |

|
| A:hub基因的蛋白质相互作用网络分析;B:hub基因的KEGG通路分析 图 4 hub基因的分析研究 |
根据TCGA数据集中的355例HCC患者的生存数据,对12个hub基因分别进行预后差异分析。如图 5所示,除了基因MCM4,每个基因高表达组的生存风险率均显著高于低表达组(Log-rank P值< 0.05)。
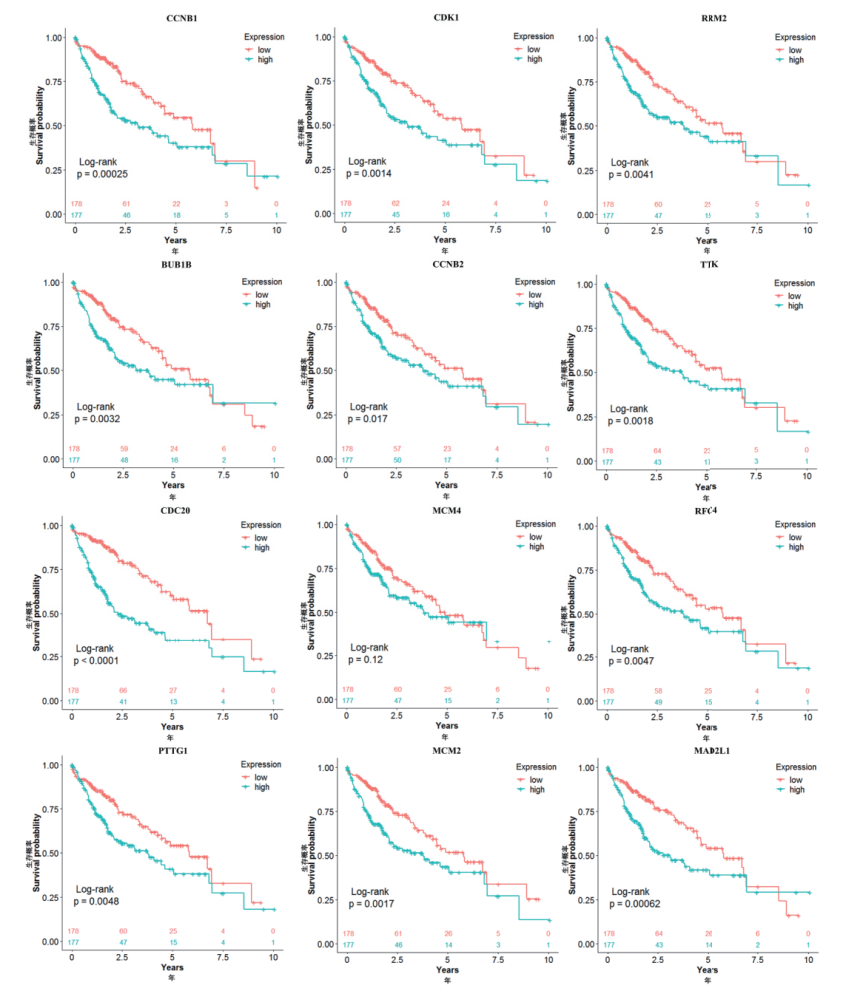
|
| 图 5 12个hub基因在肝细胞癌中的预后价值(总体生存期) |
肝细胞癌是全球常见的恶性肿瘤之一,呈现出发病机制复杂、风险因素多样的特点[4]。因此,融入分子水平信息对患者进行准确分类十分关键,能够指导不同分型患者的个性化治疗方案设计。
本研究利用信号通路中41个核心基因的表达数据,通过k均值聚类、支持向量机等方法在TCGA数据集上建立了HCC分类模型。该模型能够将HCC患者分成生存风险存在显著差异的两组,交叉验证结果表明模型具有较好的稳健性,并且3个外部数据集的测试结果证明该模型能够对HCC患者进行准确分类。基于信号通路中41个核心基因的模型与基于单因素Cox回归筛选41个基因的模型在TCGA数据集上交叉验证和LIRI-JP数据集上准确性验证的结果差别不大,甚至从某种程度上来说(log-rank检验P值),基于单因素Cox回归的模型表现更好。但是从GSE14520和GSE54236两个数据集上的表现来看,基于信号通路核心基因的模型预测准确性(一致性指数 > 0.7)远好于基于单因素Cox回归的模型(一致性指数 > 0.65)。综上所述,这两个模型均能对患者的生存风险进行有效区分,但是基于信号通路中核心基因的模型在多个数据集上的整体表现更好,更稳定并准确预测HCC患者的生存风险。本研究的工作也是围绕信号通路中核心基因的模型而展开。与基于深度学习方法进行HCC预后预测的文献结果相比,模型在3个相似的数据集上有不逊色的表现,而且需要的基因数更少,无需甲基化、miRNA的数据,能够进一步降低患者检测成本[2]。
对分类后生存风险差异显著的两组HCC患者的表达数据进行生物信息学分析,发现高风险组的上调基因主要富集在染色体分离、细胞核分裂、细胞器分裂、细胞核有丝分裂等生物学过程(GO分析)及细胞周期、卵母细胞减数分裂、p53信号通路、DNA复制等信号通路(KEGG分析)。其中染色体分离、细胞核分裂、细胞器分裂、细胞核有丝分裂、卵母细胞减数分裂及DNA复制等都是发生在细胞周期中的生物学过程,p53信号通路最终也诱导细胞周期的调控,而细胞周期调控和癌症进展关系紧密,细胞周期失调在促进肝细胞癌发生中起核心作用[32-33]。
本研究共发现12个hub基因,除基因MCM4外,每个基因高表达组的生存风险率均显著高于低表达组,而它们在S1组中的相对高表达或许部分解释了S1组与S2组HCC患者间的生存风险差异。这些基因CCNB1[4, 34-37]、CCNB2[35-37]、CDK1[34-37]、RRM2[35-37]、BUB1B[34, 36-37]、TTK[35, 37]、CDC20[4, 34, 36-37]、MCM2[36]、MCM4[36]、RFC4[36]、PTTG1[35-36]和MAD2L1[4, 34, 36-37]均在HCC癌组织中表达显著上调,该结果进一步表明分类模型的有效性,并说明这些基因可能在HCC的发生、进展中起关键性作用,可以作为疾病预警的重要标志物或者潜在治疗靶点。要说明的是,MCM2和MCM4基因在HCC预后中的作用与有些研究不一致[38-39],可能与数据集的构成有关,需要进一步验证。
4 结论通过在TCGA数据集上对信号通路中41个核心基因建模,可以对HCC患者的预后风险进行准确分类。交叉验证表明该模型具备较高的稳健性,且在另外3个数据集上得到准确的预测结果。将生存风险差异显著的两组进行生物信息学分析,发现HCC高风险组上调基因富集在细胞周期信号通路中,并发现了11个hub基因的上调与HCC风险显著相关,生物信息学分析也进一步验证模型的有效性。
| [1] |
Zhou M, Wang H, Zeng X, et al. Mortality, morbidity, and risk factors in China and its provinces, 1990-2017:a systematic analysis for the Global Burden of Disease Study 2017[J]. The Lancet, 2019, 394(10204): 1145-1158. DOI:10.1016/S0140-6736(19)30427-1 |
| [2] |
Chaudhary K, Poirion OB, Lu L, et al. Deep learning-based multi-omics integration robustly predicts survival in liver cancer[J]. Clinical Cancer Research, 2018, 24(6): 1248-1259. DOI:10.1158/1078-0432.CCR-17-0853 |
| [3] |
Zhu RX, Seto WK, Lai CL, et al. Epidemiology of hepatocellular carcinoma in the Asia-Pacific region[J]. Gut and Liver, 2016, 10(3): 332-339. |
| [4] |
Fan G, Tu Y, Chen C, et al. DNA methylation biomarkers for hepatocellular carcinoma[J]. Cancer Cell International, 2018, 18(1): 140. |
| [5] |
Montironi C, Montal R, Llovet JM. New drugs effective in the systemic treatment of hepatocellular carcinoma[J]. Clinical Liver Disease, 2019, 14(2): 56-61. DOI:10.1002/cld.796 |
| [6] |
Bruix J, Gores GJ, Mazzaferro V. Hepatocellular carcinoma:clinical frontiers and perspectives[J]. Gut, 2014, 63(5): 844-855. DOI:10.1136/gutjnl-2013-306627 |
| [7] |
Wang X, Gao J, Zhou B, et al. Identification of prognostic markers for hepatocellular carcinoma based on miRNA expression profiles[J]. Life Sciences, 2019, 232: 116596. DOI:10.1016/j.lfs.2019.116596 |
| [8] |
Wang Y, Ruan Z, Yu S, et al. A four-methylated mRNA signature-based risk score system predicts survival in patients with hepatocellular carcinoma[J]. Aging, 2019, 11(1): 160-173. |
| [9] |
Long J, Zhang L, Wan X, et al. A four-gene-based prognostic model predicts overall survival in patients with hepatocellular carcinoma[J]. J Cell Mol Med, 2018, 22(12): 5928-5938. DOI:10.1111/jcmm.13863 |
| [10] |
Zheng Y, Liu Y, Zhao S, et al. Large-scale analysis reveals a novel risk score to predict overall survival in hepatocellular carcinoma[J]. Cancer Manage Res, 2018, 10: 6079-6096. DOI:10.2147/CMAR.S181396 |
| [11] |
Liu GM, Zeng HD, Zhang CY, et al. Identification of a six-gene signature predicting overall survival for hepatocellular carcinoma[J]. Cancer Cell International, 2019, 19(1): 138. |
| [12] |
Wang Z, Teng D, Li Y, et al. A six-gene-based prognostic signature for hepatocellular carcinoma overall survival prediction[J]. Life Sciences, 2018, 203(24): 83-91. |
| [13] |
Liu G, Dong C, Liu L. Integrated multiple "-omics" data reveal subtypes of hepatocellular carcinoma[J]. PLoS One, 2016, 11(11): e0165457. DOI:10.1371/journal.pone.0165457 |
| [14] |
Fa B, Luo C, Tang Z, et al. Pathway-based biomarker identification with crosstalk analysis for robust prognosis prediction in hepatocellular carcinoma[J]. EBioMedicine, 2019, 44: 250-260. DOI:10.1016/j.ebiom.2019.05.010 |
| [15] |
Wei L, Jin Z, Yang S, et al. TCGA-assembler 2:software pipeline for retrieval and processing of TCGA/CPTAC data[J]. Bioinformatics, 2018, 34(9): 1615-1617. DOI:10.1093/bioinformatics/btx812 |
| [16] |
Fujimoto A, Furuta M, et al. Whole-genome mutational landscape and characterization of noncoding and structural mutations in liver cancer[J]. Nature Genetics, 2016, 48(5): 500-509. DOI:10.1038/ng.3547 |
| [17] |
Roessler S, Jia HL, Budhu A, et al. A unique metastasis gene signature enables prediction of tumor relapse in early-stage hepatocellular carcinoma patients[J]. Cancer Research, 2010, 70(24): 10202-10212. DOI:10.1158/0008-5472.CAN-10-2607 |
| [18] |
Villa E, Critelli R, et al. Neoangiogenesis-related genes are hallmarks of fast-growing hepatocellular carcinomas and worst survival. Results from a prospective study[J]. Gut, 2016, 65(5): 861-869. DOI:10.1136/gutjnl-2014-308483 |
| [19] |
Therneau TM. A package for survival analysis in S. version 2. 38[J]. 2015.
|
| [20] |
Schröder MS, Culhane AC, et al. survcomp:An R/Bioconductor package for performance assessment and comparison of survival models[J]. Bioinformatics, 2011, 27(22): 3206-3208. DOI:10.1093/bioinformatics/btr511 |
| [21] |
Harrell FE, Lee KL, Mark DB. Multivariable prognostic models:issues in developing models, evaluating assumptions and adequacy, and measuring and reducing errors[J]. Statistics in Medicine, 1996, 15(4): 361-387. |
| [22] |
Love MI, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 |
| [23] |
Ritchie ME, Phipson B, Wu D, et al. limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Research, 2015, 43(7): e47. DOI:10.1093/nar/gkv007 |
| [24] |
Benjamini Y, Hochberg Y. Controlling the false discovery rate:a practical and powerful approach to multiple testing[J]. JRS Soc:Series B Methodol, 1995, 57(1): 289-300. |
| [25] |
Yu G, Wang LG, Han Y, et al. ClusterProfiler:an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 |
| [26] |
Szklarczyk D, Gable AL, Lyon D, et al. STRING v11:protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Research, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 |
| [27] |
Su G, Morris JH, Demchak B, et al. Biological network exploration with Cytoscape 3[J]. Curr Protoc Bioinformatics, 2014, 47: 8. 13. 1-8. 24. DOI:10.1002/0471250953.bi0813s47 |
| [28] |
Scardoni G, Tosadori G, Faizan M, et al. Biological network analysis with CentiScaPe:Centralities and experimental dataset integration[J]. F1000Research, 2015, 3: 139. DOI:10.12688/f1000research.4477.2 |
| [29] |
Bader GD, Hogue CWV. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003, 4(1): 2. |
| [30] |
Bindea G, Mlecnik B, et al. ClueGO:a Cytoscape plug-in to deci-pher functionally grouped gene ontology and pathway annotation networks[J]. Bioinformatics, 2009, 25(8): 1091-1093. DOI:10.1093/bioinformatics/btp101 |
| [31] |
Bindea G, Galon J, Mlecnik B. CluePedia Cytoscape plugin:pathway insights using integrated experimental and in silico data[J]. Bioinformatics, 2013, 29(5): 661-663. DOI:10.1093/bioinformatics/btt019 |
| [32] |
Bisteau X, Caldez M, Kaldis P. The Complex relationship between liver cancer and the cell cycle:a story of multiple regulations[J]. Cancers, 2014, 6(1): 79-111. DOI:10.3390/cancers6010079 |
| [33] |
陈珂, 丁艳平, 王建林, 等. p53参与代谢调控的研究进展[J]. 生物技术通报, 2016, 32(11): 52-58. |
| [34] |
Zhuang L, Yang Z, Meng Z. Upregulation of BUB1B, CCNB1, CDC7, CDC20, and MCM3 in tumor tissues predicted worse overall survival and disease-free survival in hepatocellular carcinoma patients[J]. Biomed Res Int, 2018, 7897346. |
| [35] |
Li N, Li L, Chen Y. The Identification of core gene expression signature in hepatocellular carcinoma[J]. Oxidative Medicine and Cellular Longevity, 2018, 3478305. |
| [36] |
Yang WX, Pan YY, You CG. CDK1, CCNB1, CDC20, BUB1, MAD2L1, MCM3, BUB1B, MCM2, and RFC4 may be potential therapeutic targets for hepatocellular carcinoma using integrated bioinformatic analysis[J]. Biomed Res Int, 2019, 1245072. |
| [37] |
Liu ZK, Zhang RY, Yong YL, et al. Identification of crucial genes based on expression profiles of hepatocellular carcinomas by bioinformatics analysis[J]. Peer J, 2019, 7(8): e7436. |
| [38] |
Liu Z, Li J, Chen J, et al. MCM family in HCC:MCM6 indicates adverse tumor features and poor outcomes and promotes S/G2 cell cycle progression[J]. BMC Cancer, 2018, 18(1): 200. DOI:10.1186/s12885-018-4056-8 |
| [39] |
Liu J, Li W, Zhang J, et al. Identification of key genes and long non-coding RNA associated ceRNA networks in hepatocellular carcinoma[J]. Peer J, 2019, e8021. |






