2. 湖北工业大学发酵工程省部共建教育部重点实验室,武汉 430068
2. Key Laboratory of Fermentation Engineering, Ministry of Education, Hubei University of Technology, Wuhan 430068
β-甘露聚糖酶(β-1, 4-mannanase,EC 3.2.1.78)是降解甘露聚糖的关键酶,属于内切水解酶,能降解含β-1, 4-甘露糖苷键的甘露寡糖和甘露多糖[1-2]。β-甘露聚糖酶在饲料、食品、医药、石油开采等方面具有广泛应用前景[3-4]。然而目前甘露聚糖酶的工业生产成本较高,使其在实际生产应用中受到一定制约。利用基因工程的方法提高甘露聚糖酶的产量在其工业化生产中具有十分重要的意义,而共表达分子伴侣是提高外源蛋白表达的有效策略之一[5-6]。
当蛋白高水平表达时,未折叠的蛋白质容易在内质网中错误折叠和累积,内质网主要通过UPR调控这一现象,UPR激活调控因子HAC1的表达,可促进UPR相关蛋白质及分子伴侣的高水平表达,介导蛋白质的正确折叠分泌,及错误折叠蛋白在内质网中的相关降解(ERAD)[7-8]。大量研究表明分子伴侣对外源蛋白的分泌表达有着重要意义,其主要作用是辅助蛋白质的正确折叠,减少由于蛋白质错误折叠而导致的降解,从而提高蛋白质的分泌表达[7, 9]。ERO1在内质网中氧化还原态的蛋白二硫键异构酶PDI成氧化态,PDI的氧化活性帮助蛋白形成正确的折叠结构而分泌到胞外,对内质网腔内蛋白质二硫键的形成具有促进作用,且ERO1、PDI两种分子伴侣都负责内质网氧化蛋白的折叠[10-11]。分子伴侣如PDI1,也属于蛋白二硫化物异构酶,是内质网腔中多功能分子伴侣[12];CPR5亲环蛋白,参与蛋白质翻译后修饰,维持细胞内金属离子的内稳定[13]。BiP / Kar2p,重链结合蛋白,防止蛋白质分子内和分子间的相互作用而导致的错误折叠或聚集,并与错误折叠或未折叠的蛋白质结合,并阻止它们离开内质网[14]。Huang等[15]通过将HAC1在分泌α-淀粉酶的毕赤酵母重组菌胞内共表达,使重组菌的α-淀粉酶在摇瓶水平上的酶活提高约2倍。Zhang等[16]将内质网中的KAR2(BiP)与PDI两种分子伴侣组合,并同时在分泌G-CSF蛋白的毕赤酵母重组菌胞内共表达,结果重组菌在摇瓶水平分泌的G-CSF蛋白总量增加6.5倍。Zhang等[17]通过在分泌表达β-葡萄糖苷酶的毕赤酵母重组菌中胞内共表达PDI,共表达PDI的重组菌在3 L发酵罐中诱导发酵时,其发酵上清液的酶活提高46%,同时发酵上清液中β-葡萄糖苷酶的比活力提高54%。
本研究将HAC1及5种内质网折叠相关分子伴侣分别在分泌表达ManA的重组菌中胞内共表达,并进一步将HAC1、ERO1、PDI进行两基因或三基因组合后再进行共表达,以研究HAC1及不同分子伴侣对ManA在毕赤酵母GS115中分泌表达的影响。
1 材料与方法 1.1 材料 1.1.1 菌种与质粒分泌表达ManA的重组菌GS115/pPIC9K-ManA由实验室构建并保藏(ManA基因编号为Accession No. KJ806637[18])。表达载体pPICZA、巴斯德毕赤酵母GS115、大肠杆菌Top10均由实验室保藏。
1.1.2 主要试剂与仪器限制性内切酶、Prime STAR Max DNA聚合酶、T4 DNA连接酶等工具酶、DNA Marker、TaKaRa Bradford Protein Assay Kit均购自宝生物工程(大连)有限公司;Prestained protein ladder购自赛默飞公司。质粒小量提取试剂盒、DNA凝胶回收试剂盒、PCR清洁回收试剂盒等购自上海AxyPrep公司;SDS-PAGE凝胶快速配置试剂盒购于碧云天公司;底物角豆胶购于Sigma公司;Tryptone和Yeast Extract购自于Oxoid公司;生物素、无氨基酵母氮源YNB、山梨醇和琼脂购自Biosharp公司;博来霉素Zeocin购自Invitrogen公司;其他试剂均为国产或进口分析纯。
1.1.3 培养基LBL(1 L):10 g蛋白胨,5 g NaCl,5 g酵母提取物(使用时按需要加入zeocin,根据Invitrogen使用说明书)
YPD(1 L):10 g酵母提取物,20 g蛋白胨,20 g葡萄糖(使用时按需要加入zeocin,根据Invitrogen使用说明书)
BMGY(1 L):10 g酵母提取物,20 g蛋白胨,13.4 gYNB,1 mL 500×生物素(4×10-4 g/L),100 mL 1 mol/L pH 6.0磷酸钾缓冲溶液,1%甘油(根据毕赤酵母表达手册配置)
BMMY(1 L):10 g酵母提取物,20 g蛋白胨,13.4 gYNB,1 mL 500×生物素(4×10-4 g/L),100 mL 1 mol/L pH 6.0磷酸钾缓冲溶液,甲醇根据培养所需添加(根据毕赤酵母表达手册配置)。
1.2 方法 1.2.1 HAC1与5种分子伴侣胞内表达重组质粒及对应重组菌的构建 1.2.1.1 六种重组质粒的构建HAC1与5种分子伴侣基因序列NCBI数据库编号分别是CPR5(XM_002489874.1,669 bp);HAC1(XM_002489994.1,996 bp);PDI1(XP_002489806.1,1 110 bp);PDI(AJ302014.1,1 554 bp);ERO1(XM_002489600.1,1 584 bp);BiP(XM_002490982.1,2 037 bp)。以GS115基因组为模板,分别扩增6种基因,各PCR扩增引物如表 1,下划线为酶切位点。将PCR所得的产物与载体pPICZA分别双酶切、连接并转化大肠杆菌Top10,挑取阳性转化子培养后提取质粒送至武汉擎科生物技术有限公司进行测序验证。
采用Sac I分别线性化上述构建好的重组质粒,电转化至已制备成感受态的宿主菌GS115/pPIC9K-ManA中,涂布于YPDZ博来霉素抗性筛选平板上,筛选获得阳性转化子,并分别命名为GS115/ManA-CPR5、GS115/ManA-HAC1、GS115/ManA-PDI1、GS115/ManA-PDI、GS115/ManA-ERO1、GS115/ManA-BiP。
1.2.2 六种重组菌的诱导表达及相关分析 1.2.2.1 六种重组菌的摇瓶发酵将各重组菌接种于10 mL BMGY培养基中,30℃、250 r/min培养24 h,离心收集适量菌体,重悬于25 mL BMMY培养基中,控制起始OD600值约为1.0,在30℃、250 r/min条件下培养至192 h,每隔24 h取样0.25 mL并补加1%甲醇,检测各样品的OD600值(通过测量600 nm处的OD值来确定酵母细胞生长状况)和酶活力,以GS115/pPIC9K-ManA作为对照。所有实验数据均重复3次,并通过GraphPad Prism 5软件对数据进行显著性分析。
甘露聚糖酶酶活力采用DNS法[19]测定,将收取的样品离心并收集上清,以pH6.0的Na2HPO4-柠檬酸缓冲液配置的0.5%角豆胶为底物,酶活力单位定义为在其最适温度75℃和最适pH 6.0条件下,1 min内水解甘露聚糖底物产生1 μmol甘露糖所需要的甘露聚糖酶的量。
1.2.2.2 六种重组菌胞内滞留酶活检测摇瓶发酵至168 h时取等量菌体,10 000 r/min离心,弃上清,用等体积预冷的pH 6.0的Na2HPO4-柠檬酸缓冲液重悬,重复洗涤2次。再用等量的缓冲液重悬,向每管中加入0.73 g酸性玻璃珠,振荡器上处理30 min,离心去除细胞碎片,收集上清,测定胞内甘露聚糖酶酶活力。所有实验数据均重复3次。
1.2.2.3 六种重组菌发酵上清液蛋白浓度的测定及SDS-PAGE电泳摇瓶发酵至168 h时,取等体积的发酵上清液进行SDS-PAGE电泳分析,并根据TaKaRa Bradford Protein Assay Kit使用说明检测发酵上清液总蛋白含量。所有实验数据均重复3次。
1.2.3 HAC1、ERO1、PDI组合重组质粒及对应重组菌的构建 1.2.3.1 四种组合重组质粒的构建胞内共表达HA-C1、ERO1和PDI的重组菌发酵上清液中的ManA酶活力分别有不同程度的提升,因此选择HAC1、ERO1和PDI构建4种组合质粒pPICZA/HAC1-ERO1、pPICZA/HAC1-PDI、pPICZA/ERO1-PDI、pPICZA/HAC1-ERO1-PDI。首先采用重叠延伸PCR将HAC1、ERO1和PDI基因中的Bgl II和BamH I位点在不影响翻译后蛋白序列的条件下定点突变,去除各基因中的Bgl II和BamH I位点,重叠延伸PCR引物见表 2。然后根据同尾酶法构建各两基因组合重组质粒,以pPICZA/HAC1-ERO1为例,流程图如图 1所示。在已构建好的pPICZA/HAC1-ERO1的基础上进一步构建pPICZA/HAC1-ERO1-PDI,方法同上。
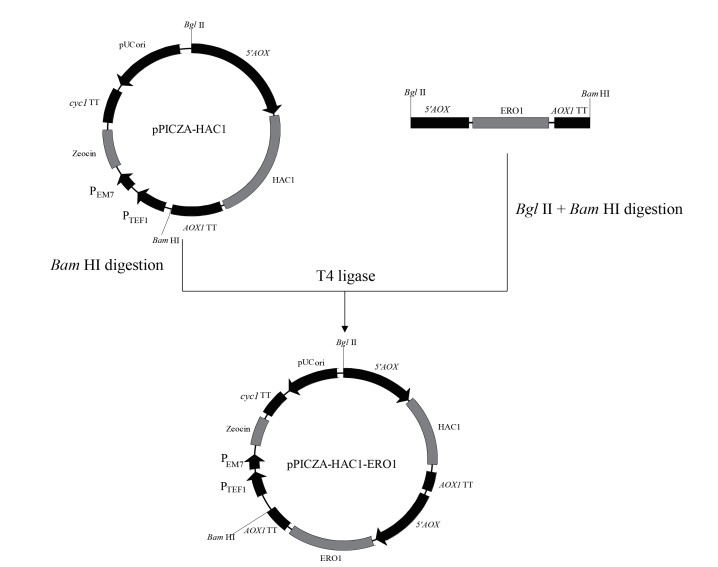
|
| 图 1 pPICZA/HAC1-ERO1质粒构建示意图 |
将4种组合重组质粒分别电转化至已制备成感受态的宿主菌GS115/pPIC9K-ManA中,筛选阳性转化子,并分别命名为GS115/ManA-HAC1-ERO1、GS115/ManA-HAC1-PDI、GS115/ManA-ERO1-PDI、GS115/ManA-HAC1-ERO1-PDI。
1.2.4 四种重组菌的诱导表达及相关分析各重组菌摇瓶发酵及相关分析实验同1.2.2。
2 结果 2.1 HAC1与5种分子伴侣重组质粒及对应重组菌的构建以提取的GS115基因组为模板,PCR扩增获得6种基因,CPR5(669 bp),HAC1(996 bp),PDI1(1 110 bp),PDI(1 554 bp),ERO1(1 584 bp),BiP(2 037 bp),扩增结果如图 2,大小与预期一致。将目的基因分别与载体pPICZA连接获得6种重组质粒,测序结果表明6种重组质粒均构建成功。然后将各重组质粒电转化至GS115/pPIC9K-ManA中,筛选获得阳性转化子。
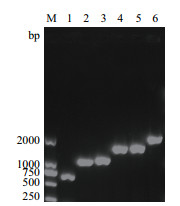
|
| M:DL2000 DNA Maker;Lane 1:CPR5;Lane 2:HAC1;Lane 3:PDI1;Lane 4:PDI;Lane 5:ERO1;Lane 6:BiP 图 2 六种基因的PCR产物电泳图 |
各重组菌发酵生长曲线及酶活曲线如图 3所示,从图 3-A可以看出各重组菌的生长趋势一致,表明HAC1及5种分子伴侣的胞内共表达不会影响重组菌的正常生长。从图 3-B中发现各重组菌摇瓶发酵至168 h时,上清的酶活力达到最高,其中GS115/ManA-HAC1、GS115/ManA-ERO1、GS115/ManA-PDI的酶活水平相比GS115/ManA分别提高26%、15%、20%,分别达到1 014 U/mL、925 U/mL、965 U/mL。
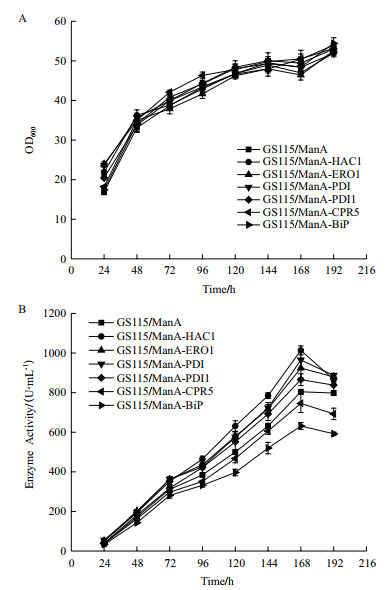
|
| 图 3 六种重组菌生长曲线(A)及酶活力曲线(B) |
摇瓶发酵至168 h时,检测各重组菌上清的酶活及胞内滞留酶活,并计算各重组菌的总酶活见图 4。重组菌GS115/ManA-HAC1、GS115/ManA-ERO1、GS115/ManA-PDI分泌至上清液的酶活分别提高26%、15%、20%,而GS115/ManA-HAC1胞内滞留的酶活力下降34%,且GS115/ManA-ERO1和GS115/ManA-PDI胞内滞留酶活力也略有下降,同时3种重组菌发酵产生的总酶活分别提高了22%、13%、18%;重组菌GS115/ManA-PDI1和GS115/ManA-CPR5发酵上清液的酶活及发酵产生的总酶活力相对GS115/ManA无明显变化;而重组菌GS115/ManA-BiP发酵上清液的酶活下降了22%,胞内滞留酶活反而上升了42%,总酶活力下降17%。

|
| *表明存在显著性差异,*= P < 0.05,**= P < 0.01,*** = P < 0.001,下同 图 4 六种重组菌酶活力分析(168 h) |
摇瓶发酵至168 h时,对各重组菌上清液进行SDS-PAGE电泳(图 5),测定各重组菌上清液总蛋白含量并计算出比活力(表 3),结果发现胞内共表达HAC1、ERO1、PDI基因的重组菌,发酵上清液中的比活力分别提高了65%、55%、44%,说明上清液中外源蛋白ManA的纯度相对提高。

|
| M:Prestained protein ladder; Lane 1:GS115/ManA; Lane 2:GS115/ManA-HAC1;Lane 3:GS115/ManA-ERO1;Lane 4:GS115/ManA-PDI; Lane 5:GS115/ManA-PDI1;Lane 6:GS115/ManA-CPR5;7:GS115/ManA-BiP 图 5 六种重组菌发酵上清液的SDS-PAGE分析(168 h) |
将HAC1 ERO1和PDI进行两基因或三基因组合,按照图 1流程构建4种组合重组质粒,使用Bgl II与BamH I进行双酶切鉴定结果如图 6,双酶切鉴定大小与预期一致。将各组合重组质粒电转化至GS115/pPIC9K-ManA中,筛选各重组菌阳性转化子进行后续实验。
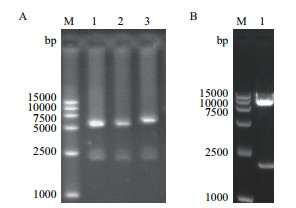
|
| (A) M:DL15000 DNA maker; Lane 1:pPICZA/HAC1-ERO1;Lane 2:pPICZA/HAC1-PDI; Lane 3:pPICZA/ERO1-PDI; (B) M:DL15000 DNA maker; Lane 1:pPICZA/HAC1-ERO1-PDI 图 6 四种重组质粒双酶切鉴定 |
各重组菌生长曲线与酶活曲线见图 7。图 7-A显示各重组菌生长趋势无明显差异,表明各重组菌均生长正常。从图 7-B可以看出,各重组菌摇瓶发酵至168 h时,GS115/ManA-HAC1-PDI上清酶活比GS115/ManA提高10%,达到884 U/mL,而其它重组菌上清酶活力与GS115/ManA相比均无明显变化。

|
| 图 7 四种重组菌生长曲线(A)及酶活曲线(B) |
摇瓶发酵至168 h时,测定各重组菌上清酶活及胞内滞留酶活,并计算各重组菌的总酶活力见图 8。从图 8可以看出,GS115/ManA-HAC1-PDI发酵上清液酶活提高10%,且发酵产生的总酶活力提高10%,而其它3种重组菌的上清酶活、胞内滞留酶活及总酶活都无明显变化。基于各重组菌的酶活力比较,GS115/ManA-HAC1-PDI对ManA的分泌表达有一定促进作用,但相较于GS115/ManA-HAC1、GS115/ManA-ERO1和GS115/ManA-PDI却并没有进一步的提升。

|
| 1:GS115/ManA; 2:GS115/ManA-HAC1-ERO1;3:GS115/ManA-HAC1-PDI; 4:GS115/ManA-ERO1-PDI; 5:GS115/ManA-HAC1-ERO1-PDI 图 8 四种重组菌酶活力分析(168 h) |
摇瓶发酵至168 h时,检测各重组菌发酵上清液的总蛋白含量并计算比活力,结果见表 4,同时对各上清液进行SDS-PAGE电泳分析见图 9。对比GS115/ManA,GS115/ManA-HAC1-PDI比活力提高39%,GS115/ManA-HAC1-ERO1-PDI比活力提高27%,而其它3种重组菌发酵上清液的比活力无明显变化。但GS115/ManA-HAC1-PDI发酵上清液的ManA比活力相比GS115/ManA-HAC1、GS115/ManA-ERO1、GS115/ManA-PDI却没有进一步提高。
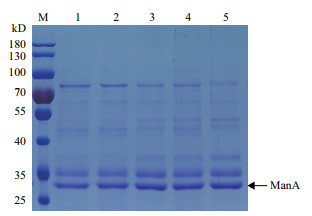
|
| M:Prestained protein ladder; Lane 1:GS115/ManA; Lane 2:GS115/ManA-HAC1-ERO1;Lane 3:GS115/ManA-HAC1-PDI; Lane 4:GS115/ManA-ERO1-PDI; Lane 5:GS115/ManA-HAC1-ERO1-PDI 图 9 四种重组菌发酵上清液的SDS-PAGE分析(168 h) |
外源蛋白的大量表达可能会使ER折叠和分泌外源蛋白质的能力造成负担,进而错误折叠或未折叠的蛋白在ER中大量积聚,导致未折叠蛋白应答(UPR)途径的激活,该途径通过诱导参与蛋白折叠的分子伴侣和ER相关降解(ERAD)途径的基因来减轻ER的压力[7-8]。分泌表达ManA的重组菌胞内共表达HAC1、ERO1和PDI时,各重组菌发酵上清液的酶活水平分别提高26%、15%和20%,且胞内滞留酶活分别下降,表明了HAC1及分子伴侣ERO1和PDI的共表达对甘露聚糖酶ManA的正确折叠及分泌表达具有一定的促进效果,说明其中HAC1的表达,可能是其介导的UPR途径,辅助外源蛋白ManA的正确折叠及分泌,减少由于ManA错误折叠而导致的ER相关降解途径(ERAD)的降解,从而提高ManA的分泌表达[7-8]。而分子伴侣如ERO1,PDI的共表达,主要是直接或间接通过其蛋白的分子伴侣活性介导未折叠的蛋白质正确折叠,及错误折叠蛋白的再折叠来辅助表达ManA[5]。在分泌表达ManA的重组菌胞内共表达PDI1、CPR5和BiP时,各重组菌发酵液的酶活力没有明显提高,表明并非所有的内质网分子伴侣都能辅助ManA的正确折叠及分泌表达。对于HAC1与分子伴侣组合并在分泌表达ManA的重组菌,其分泌表达ManA的酶活力没有进一步提升,研究发现外源蛋白的正确折叠与不同的分子伴侣,或者不同的分子伴侣组合之间没有特别对应的规律。已报道的研究中,不同的分子伴侣的组合协同表达外源蛋白,有时比单独一种分子伴侣与外源蛋白共表达的效果要更明显[16, 20],但也有不同分子伴侣的组合与外源蛋白共表达时,效果反而不明显的现象[21]。分子伴侣相互之间的影响仍有待进一步研究,针对分子伴侣对酵母表达系统进行优化,应该根据目的蛋白的分子结构特征而区别对待,构建相对最适合的表达工程菌[22]。
在胞内共表达HAC1、ERO1和PDI后,各重组菌ManA发酵上清液的比活力分别提高了65%、55%和44%,表明ManA发酵上清液的质量也相应得到提高,对工业上生产甘露聚糖酶的过程中简化生产流程以及降低生产成本有着一定的实际意义,但HAC1及分子伴侣对毕赤酵母分泌表达上清中ManA的比活力的变化还需要进一步纯化论证。在提高甘露聚糖酶表达量的相关研究中,其它研究策略如改变启动子[23]、信号肽[24]等,虽然对酶活的提升都有一定作用,但同时也可能会导致胞内滞留酶活的积累,增加内质网蛋白折叠的压力,若同时共表达HAC1及分子伴侣ERO1、PDI等基因,能协同分泌表达甘露聚糖酶,从而构建高效分泌表达甘露聚糖酶的工程菌。
蛋白质分泌的限制可能是由低折叠率造成,原因可能是内质网相关蛋白折叠分子伴侣的表达水平不足[25]。已报道的研究中发现,HAC1或者分子伴侣基因以多拷贝的形式在毕赤酵母重组菌胞内共表达可以进一步提高其重组菌外源蛋白的分泌产量,如Huang等[15]在分泌表达不同拷贝α-淀粉酶基因的毕赤酵母重组菌胞内共表达HAC1,并随着HAC1拷贝数在一定范围的增加,分泌表达不同拷贝α-淀粉酶基因的重组菌在摇瓶发酵水平分泌表达α-淀粉酶酶活都不断提高,其中分泌表达12拷贝α-淀粉酶基因,并在胞内共表达6拷贝HAC1的重组菌酶活达到最大值,相比分泌表达12拷贝α-淀粉酶基因,并在胞内共表达1拷贝HAC1的重组菌提高了约1倍。故本研究中的HAC1及分子伴侣ERO1、PDI还可以进一步研究其多拷贝基因在分泌表达不同拷贝ManA的毕赤酵母重组菌胞内共表达,从而构建高效分泌表达ManA的工程菌。
4 结论本研究在工程菌GS115/pPIC9K-ManA的基础上,分别同时共表达HAC1、ERO1、PDI,各重组菌在摇瓶发酵水平,发酵上清液的ManA酶活力都有所提高,其中整合HAC1基因的重组菌最高提高了26%。进一步将HAC1、ERO1、PDI进行两基因或三基因组合后再进行共表达,但其重组菌发酵上清液酶活力并没有进一步提升。
| [1] |
Le NJ, Anderson L, Stoll D, et al. The structure and characterization of a modular endo-β-1, 4-mannanase from Cellulomonas fimi[J]. Biochemistry, 2005, 44(38): 12700-12708. |
| [2] |
Van ZWH, Rose SH, Trollope K, et al. Fungal β-mannanases:mannan hydrolysis, heterologous production and biotechnological applications[J]. Process Biochemistry, 2010, 45(8): 1203-1213. |
| [3] |
张建新, 宋宜乐, 冯军厂, 等. 微生物β-甘露聚糖酶的研究进展[J]. 中国酿造, 2019, 38(4): 7-10. |
| [4] |
Srivastava PK, Kapoor M. Production, properties, and applications of endo-β-mannanases[J]. Biotechnology Advances, 2017, 35(1): 1-19. |
| [5] |
Puxbaum V, Mattanovich D, Gasser B. Quo vadis? The challenges of recombinant protein folding and secretion in Pichia pastoris[J]. Applied Microbiology and Biotechnology, 2015, 99(7): 2925-2938. |
| [6] |
Damasceno LM, Huang CJ, Batt CA. Protein secretion in Pichia pastoris and advances in protein production[J]. Applied Microbiology and Biotechnology, 2012, 93(1): 31-39. |
| [7] |
Hampton RY. ER stress response:getting the UPR hand on misfolded proteins[J]. Current Biology, 2000, 10(14): 518-521. |
| [8] |
Hwang J, Qi L. Quality control in the endoplasmic reticulum:Crosstalk between ERAD and UPR pathways[J]. Trends in Biochemical Sciences, 2018, 43(8): 593-605. |
| [9] |
Ping Y, Qi Z, Chen K, et al. Improving the secretory production of the heterologous protein in Pichia pastoris by focusing on protein folding[J]. Appl Biochem Biotechnol, 2015, 175(1): 535-548. |
| [10] |
高庆华, 董聪, 王玥, 等. 共表达分子伴侣PDI和Ero1对葡萄糖氧化酶在毕赤酵母中表达的影响[J]. 生物技术通报, 2018, 34(7): 174-179. |
| [11] |
Onda Y, Kobori Y. Differential activity of rice protein disulfide isomerase family members for disulfide bond formation and reduction[J]. FEBS Open Bio, 2016, 4(1): 730-734. |
| [12] |
林小琼.基于iTRAQ技术的高效表达木聚糖酶重组毕赤酵母细胞的蛋白组学研究[D].广州: 华南理工大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10561-1014153353.htm
|
| [13] |
Arevalo-Rodriguez M, Wu X, Hanes SD, et al. Prolyl isomerases in yeast[J]. Frontiers in Bioscience, 2004, 9(1-3): 2420-2446. |
| [14] |
Liu YY, Woo JH, Neville DM. Overexpression of an anti-CD3 immunotoxin increases expression and secretion of molecular chaperone BiP/Kar2p by Pichia pastoris[J]. Appl Environ Microbiol, 2005, 71(9): 5332-5340. |
| [15] |
Huang M, Gao Y, Zhou X, et al. Regulating unfolded protein response activator HAC1p for production of thermostable raw-starch hydrolyzing α-amylase in Pichia pastoris[J]. Bioprocess and Biosystems Engineering, 2017, 40(3): 341-350. |
| [16] |
Zhang W, Zhao H, Xue C, et al. Enhanced secretion of heterologous proteins in Pichia pastoris following overexpression of Saccharomyces cerevisiae chaperone proteins[J]. Biotechnology Progress, 2006, 22(4): 1090-1095. |
| [17] |
Zhang J, Wu D, Chen J, et al. Enhancing functional expression of β-glucosidase in Pichia pastoris by co-expressing protein disulfide isomerase[J]. Biotechnology and Bioprocess Engineering, 2011, 16(6): 1196-1200. |
| [18] |
张巍, 王亚伟, 陈丰, 等. 一株全基因合成耐热甘露聚糖酶的表达及酶学性质分析[J]. 中国生物工程杂志, 2014, 34(8): 41-46. |
| [19] |
Yu YH, Sun MY, Wang JW, et al. Purification and properties of Bacillus subtilis SA22 Endo-1, 4-β-D mannarnase[J]. Chinese Journal of Biotechnology, 2003, 19(3): 327-331. |
| [20] |
Li X, Liu Z, Wang G, et al. Overexpression of Candida rugosa lipase Lip1 via combined strategies in Pichia pastoris[J]. Enzyme and Microbial Technology, 2016, 82: 115-124. |
| [21] |
Damasceno LM, Anderson KA, Ritter G, et al. Cooverexpression of chaperones for enhanced secretion of a single-chain antibody fragment in Pichia pastoris[J]. Applied Microbiology and Biotechnology, 2007, 74(2): 381-389. |
| [22] |
杜济良.分子伴侣与信号肽对分泌蛋白在巴斯德毕赤酵母中表达水平的影响研究[D].北京: 中国人民解放军军事医学科学院, 2011. http://cdmd.cnki.com.cn/Article/CDMD-90106-1012325886.htm
|
| [23] |
熊海容, 汪斌, 杨青, 等. 不同启动子调控的两种半纤维素酶分泌表达的比较[J]. 中南民族大学学报:自然科学版, 2019, 38(1): 39-44. |
| [24] |
杨青, 汪斌, 王亚伟, 等. 介导两种半纤维素酶分泌表达的信号肽比较[J]. 中国生物工程杂志, 2017, 37(8): 15-22. |
| [25] |
Ahmad M, Hirz M, Pichler H, et al. Protein expression in Pichia pastoris:recent achievements and perspectives for heterologous protein production[J]. Applied Microbiology & Biotechnology, 2014, 98(12): 5301-5317. |








