2. 中国科学院天津工业生物技术研究所, 天津 300308;
3. 中国科学院大学, 北京 100049
2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308;
3. University of Chinese Academy of Sciences, Beijing 100049
核黄素又名维生素B2, 是一种异咯嗪衍生物[1]。这种黄色晶体最早从乳清当中分离, 因其分子结构中含有核糖醇基而被命名为核黄素[1-2]。核黄素属于B族维生素家族, 其在哺乳动物生长发育过程中起着重要的作用[3]。因而核黄素在饲料行业、食品医药行业中都着有广泛的应用[4]。微生物发酵法是现在最主要的核黄素生产方法[5]。高产菌株的获取从初期的诱变筛选和简单代谢工程改造[6-9], 发展到结合系统生物学与基因组、转录组、代谢组学等方法进行全面解析[10-11]。历经近70年的发展, 工业菌株的产量与转化率均不断提高, 逐步替代了传统的化学合成法和化学半合成法, 成为了微生物发酵法替代化学合成法生产复杂有机分子的典型范例[12]。
虽然理性改造的技术手段不断丰富, 但是以诱变-筛选为首的非理性改造在突破代谢瓶颈与寻找新的突变位点等方面仍具有不可替代的作用[13-17]。此外, 随着流式细胞仪[18-19]、液滴微流控[20-21]、自动化育种筛选等高通量设备的应用, 筛选的效率也大幅提升。和其他大多数代谢产物不同, 核黄素作为一种天然的黄绿色荧光物质, 具有和绿色荧光蛋白类似的发射光[22-23], 这种得天独厚的条件为以荧光筛选高产菌株提供了方便。然而利用高通量筛选技术进行核黄素高产菌株筛选的工作直到最近才有所报道。Chen等[24]利用液滴微流控对一株产核黄素的乳酸菌的随机突变库进行筛选, 菌株的核黄素产量从1.51 mg/L提升到2.81 mg/L。Wagner等[25]在对解脂耶氏酵母进行诱变-筛选研究中则发现使用流式细胞筛选得到的高产菌其胞内核黄素产量较高, 而使用液滴微流控筛选到的菌株其胞外核黄素产量较高。然而两篇文献中所用出发菌株的核黄素产量都比较低, 对于产量较高的工业菌株尚未建立合适的高通量筛选方法。因此本文评价了流式细胞分选, 液滴微流控分选, 96孔板筛选等方法在核黄素工业菌株筛选中的适用性, 建立了利用96孔板静置培养-荧光检测筛选核黄素工业菌株的方法, 并应用该方法提高了工业菌株核黄素的产量。
1 材料与方法 1.1 材料 1.1.1 菌株R1, P1均为实验室保藏的核黄素生产菌株; S1为实验室保藏的核黄素生产工业菌株; 1#、2#和3#为本研究所得菌株。
1.1.2 培养基LB培养基:蛋白胨10 g/L、酵母抽提物5 g/L、氯化钠10 g/L;
YP发酵培养基:玉米浆干粉(0.5%)、蔗糖(1%)、硫酸镁(0.5%)、酵母抽提物(0.5%)、磷酸氢二钾(0.3%)、磷酸二氢钾(0.2%), 灭菌前用NaOH调节pH至7.2。
1.2 方法 1.2.1 核黄素不同浓度标品荧光强度的测定配置10 mg/L、20 mg/L、30 mg/L、40 mg/L、50 mg/L、60 mg/L、70 mg/L、80 mg/L的核黄素标准品水溶液, 分别取100 μL于Costar黑色底透酶标板中, 测定激发光473 nm, 发射光520 nm下的荧光值, 绘制核黄素不同浓度标品与荧光强度的关系曲线。
1.2.2 流式细胞分析挑取活化36 h的R1、P1新鲜的单菌落, 分别接种至LB固体试管斜面培养基, 37℃静置培养36 h。分别用接种环刮取斜面上的所有菌苔于1.5 mL发酵液中, 混匀, 取500 μL菌液接种至含有100 mL发酵培养基的500 mL三角瓶中, 37℃、200 r/min振荡培养。分别将发酵液13 000 r/min, 离心1 min, 弃上清, 用1 mL 1×PBS稀释重悬, 使重悬后细胞的OD600=1, 然后取50 μL重悬后菌液到990 μL 1×PBS中, 混匀后去进行流式分析, 检测R1、P1的细胞内荧光强度。
1.2.3 液滴微流控分选挑取活化36 h的R1、P1新鲜单菌落, 分别接种于含有40 mL LB培养液的250 mL三角瓶中, 37℃、200 r/min振荡培养12 h。分别将种子液4 000 r/min离心8 min, 弃上清。然后用40 mL发酵培养基重悬菌体沉淀, 得到种子悬液。将种子悬液接种至发酵培养基中, 使接种后发酵液的OD600=0.1。然后用液滴微流控进行液滴包埋。包埋后液滴置于37℃静置培养。筛选液滴微流控激发波长473 nm, 发射波长520 nm下, 荧光信号最高的1%个液滴, 每管收集2 000个液滴左右。将收集到的液滴涂布LB固体培养基平板, 37℃倒置培养36 h。
1.2.4 96深孔板静置和振荡培养挑取突变库单菌落接种于含有500 μL LB液体培养基的96孔板中, 37℃、800 r/min、80%湿度、培养18 h。转接27 μL种子液于含有473 μL发酵培养基的96深孔板中, 同一来源的种子液分别接入静置和振荡培养的96孔板中。静置培养的孔板放置于37℃培养箱中, 振荡培养的孔板置于孔板摇床中, 37℃、800 r/min、80%湿度培养。
1.2.5 发酵液中核黄素浓度的分光光度检测和荧光检测核黄素浓度的分光光度检测法如下:取适量体积的发酵液, 用0.01 mol/L NaOH碱溶20 min后, 10 000 r/min, 离心10 min, 取上清测定444 nm下的吸光度。根据公式:产量(mg/L)=(稀释倍数×吸光度)/0.032 1计算核黄素产量。
核黄素浓度的荧光检测法如下:取100 μL发酵液, 测定其在激发光473 nm, 发射光520 nm下的荧光强度。
1.2.6 菌种保藏方法取样后在孔板中加入终浓度20%的甘油, 混匀。将孔板冻存于-20℃, 以备进行下一轮筛选。
1.2.7 96孔板静置培养复筛取3 μL孔板初筛得到的高荧光菌株冻存液, 点于LB固体平板上, 37℃培养24 h后, 挑取单菌落于含有500 μL LB液体培养基的96孔板中, 37℃、800 r/min、80%湿度、培养18 h。转接27 μL种子液于含有473 μL发酵培养基的96深孔板中。放置于37℃培养箱静置培养24 h后, 进行荧光检测。
1.2.8 摇瓶发酵验证取3 μL经96孔板静置复筛验证后, 荧光最高的4株菌及S1的初筛冻存液, 点于LB固体平板上, 37℃静置培养36 h。分别用接种环刮取斜面上的所有菌苔于1.5 mL发酵液中, 混匀, 取500 μL菌液接种至含有100 mL发酵培养基的500 mL三角瓶中, 37℃、200 r/min振荡培养, 用分光光度法测定12 h、24 h和36 h的核黄素产量。
2 结果 2.1 流式细胞分选与液滴微流控分选在核黄素高产菌株选育的应用使用1.2.5中荧光检测法, 测试液滴微流控和流式细胞仪检测核黄素的可行性, 在0-80 mg/L的浓度范围内, 核黄素水溶液的荧光强度与核黄素浓度呈正相关(图 1-A), 表明通过荧光信号可以对核黄素浓度进行定量。同时测定参数满足流式细胞仪和液滴微流控细胞分选仪的检测要求, 故可用于核黄素含量的检测。
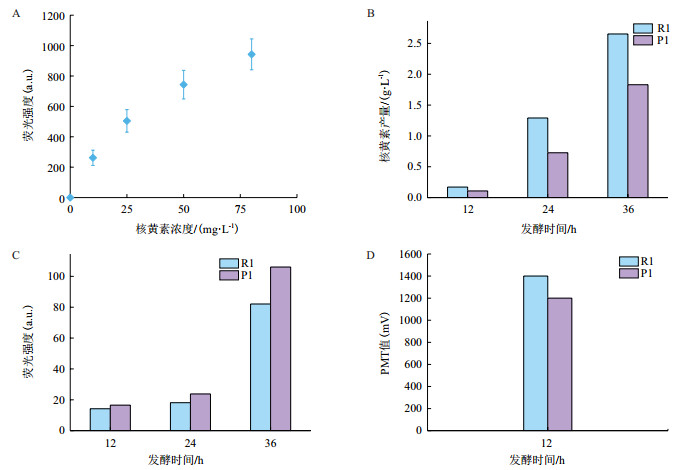
|
| A:核黄素浓度与荧光强度的关系; B:摇瓶发酵核黄素产量结果; C:R1、P1流式细胞荧光强度; D:R1、P1液滴微流控PMT信号值 图 1 荧光与核黄素浓度的关系及流式细胞分析与液滴微流控分析结果 |
对来源于同一工业菌株的R1与P1进行了摇瓶发酵, 使用分光光度法测定核黄素产量。发酵36 h后, P1的产量明显低于R1(图 1-B)。为评价流式细胞仪和液滴微流控对不同核黄素产量菌株的区分能力, 收集R1与P1的发酵液细胞, 对其进行流式分析发现, 36 h时, R1和P1的平均荧光强度分别为82.08 a.u.和106.01 a.u.(图 1-C), 表明流式细胞仪无法衡量菌株产量的高低。此外, 将12 h发酵液的上清包埋到微液滴中进行检测, R1上清PMT值为1 400 mV, P1上清PMT值为1 200 mV(图 1-D)。故液滴微流控技术可对胞外核黄素产量的高低进行区分, 与实际测得产量结果呈正相关。
为探究液滴微流控筛选高产菌株的可能性, 按1.2.3所述方法将R1和P1使用液滴微流控系统进行包埋。在培养18 h、24 h和36 h时使用荧光显微镜对液滴进行观察。如图 2中所示, 在上述3个时间点, R1与P1的液滴荧光均有差异, 其中液滴培养24 h时差异最为显著。使用液滴微流控分选仪收集了每个时间点荧光值前1%的2 000个液滴, 将其涂布在LB平板上, R1与P1在不同培养时间点均只长出1-2个单菌落, 极低的细胞成活率阻碍了液滴微流控对高产菌株的筛选。同时进一步优化种子液培养时间、液滴分选电压以及发酵培养基的糖浓度均未能提高细胞的成活率。因此, 液滴微流控虽然能区分出不同产量的核黄素工程菌株, 但不适用于目标菌株的分选。后续研究中我们建立了通量相对较低的96孔板筛选方法, 用于高产菌株的筛选。

|
| 图 2 R1和P1液滴核黄素荧光信号图 |
为了建立最优的96孔板筛选方法, 将荧光检测/分光光度检测, 振荡培养/静置培养两组条件进行了组合分析。组合后的筛选操作流程如图 3所示。
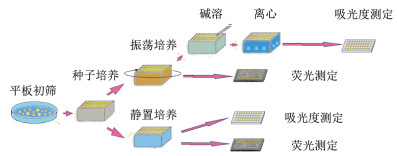
|
| 图 3 96孔板筛选示意图 |
96孔板振荡培养条件下, 18 h-36 h时分光光度法测定的R1的核黄素产量均高于P1(图 4-A); 24 h后的荧光强度不能正确反应产量高低趋势(图 4-B), 荧光检测不能对菌株产量进行有效区分。96孔板静置培养条件下, 18 h-36 h时, 分光光度法测定的R1和P1的核黄素产量差异不显著(图 4-C); 18 h、24 h、36 h时R1的平均荧光强度均高于P1(图 4-D), 荧光检测法则可明显区分菌株产量高低。因此, 96孔板振荡培养-分光光度法和96孔板静置培养-荧光法均可实现核黄素不同产量菌株的区分。如图 3所示, 静置-荧光法检测流程更加简便, 故是一种更优的筛选方法。
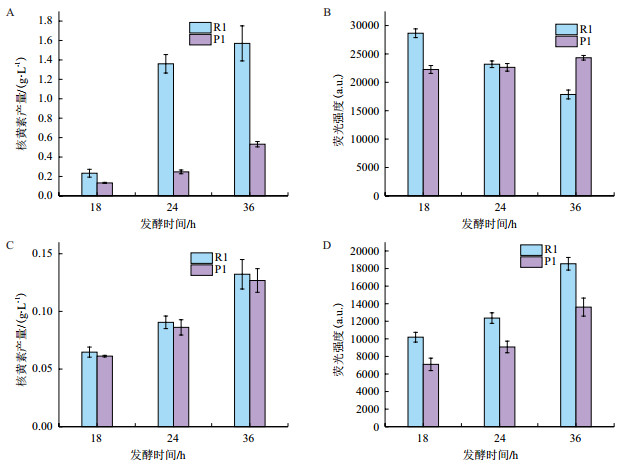
|
| A:96孔板振荡培养条件下分光光度法测核黄素产量; B:96孔板振荡培养条件下荧光法测核黄素浓度; C:96孔板静置培养条件下分光光度法测核黄素产量; D:96孔板静置培养条件下荧光法测核黄素浓度 图 4 96孔板振荡和静置培养 |
使用96孔板静置培养-荧光检测法对工业菌株S1突变体库进行筛选。首先对S1的突变体库在LB平板上进行初筛。在之前的实验中发现, 单菌落过小的个体发酵后产量明显低于平均水平。因此, 依据菌落直径大于0.5 mm的原则, 在约3 500个菌落中挑选了约1 300个菌落进行初筛, 初筛结果如图 5-A所示。将初筛选得到的菌株按荧光强度排序, 选取荧光强度前12%的菌株进行复筛。复筛结果与初筛的趋势相似, 从复筛得到的菌株中选取荧光相对较高的3株菌(1#、2#、3#)进行摇瓶发酵, 发现1#、2#、3#相对出发菌S1核黄素产量均有提高, 36 h时产量分别为2.3 g/L、2.4 g/L、2.53 g/L(图 5-B), 较出发菌分别提高了4.5%、9%和15%, 说明96孔板静置培养-荧光检测法可以用于高产核黄素工业枯草芽孢杆菌的筛选。
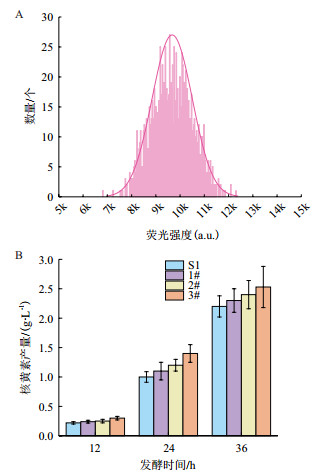
|
| A:96孔板静置培养-荧光检测初筛结果; B:摇瓶发酵S1, 1#, 2#, 3#核黄素产量结果 图 5 96孔板静置初筛、复筛及摇瓶验证 |
核黄素作为经典的生物发酵产品, 已有许多关于菌株非理性改造与理性改造的研究[26-27], 然而与筛选方法相关的研究却比较少, 这可能是由于直接通过观察单菌落的颜色即可粗略评价产量高低[28]。近年来随着流式细胞分选, 液滴微流控分选等方法的发展, 国外已经有一些关于核黄素菌株高通量筛选方法的研究[24-25]。然而这些研究的出发菌株的核黄素产量并不高, 因而有必要开发针对高产菌株的筛选方法。在本研究中, 我们首先评价了超高通量的流式细胞筛选和液滴微流控筛选两种方法在核黄素高产工业菌株筛选中的应用。我们发现, 正如文献中推测的, 核黄素由于具有胞外分泌的特性, 因而其高产菌株的胞内核黄素含量并不一定高[25]。这就造成了使用流式细胞检测不能正确评价总产量的高低。而液滴微流控相当于一个微反应器, 细胞生产的核黄素全部封闭在液滴中。因而可以将液滴的荧光高低等同于核黄素产量的高低。遗憾的是, 在该研究中, 我们并没有使用液滴微流控成功分选到足够数量的细胞。由于一些未知原因, 经过分选的细胞成活率非常低。我们推测这可能与分选时的脉冲电压, 培养基的组分以及核黄素生产菌本身的自溶现象有关。对于液滴微流控分选成活率低的问题, 我们测试了实验室保藏的其他核黄素生产菌株, 其中一些菌株的成活率可以满足分选的需要。虽然这些菌株的核黄素产量低于S1、R1等高产菌株, 但可以利用其作为底盘菌株筛选对产量有提升作用的核黄素代谢途径酶的突变体, 利用代谢工程可以将突变体整合到高产菌株中以提高核黄素产量。研究并解决相关问题, 将成为高通量筛选核黄素高产工业菌株方法的突破, 将更有推广应用的价值。而流式细胞分选的研究也并非没有实际意义。由于现在核黄素外排的分子机制还不清楚, 因此检测胞内核黄素的变化可以了解其外排的情况, 筛选胞内核黄素含量变化的菌株, 也可以帮助寻找相关转运蛋白。
本文通过对孔板培养方式与核黄素检测方法的比较, 建立了孔板静置培养-荧光检测筛选方法。发酵培养基中含有对检测造成干扰的不溶颗粒、细胞以及色素物质, 并且部分核黄素会以颗粒形式存在于发酵液中, 因此使用分光光度法对发酵液进行检测前需用稀碱溶解, 然后离心取上清进行检测。不但流程繁琐, 而且需要孔板离心机, 操作相对复杂。使用荧光进行检测时, 由于核黄素的荧光具有特异性, 培养基中的其他成分不会对其进行干扰, 因此可以直接进行测定, 步骤简单、快速。并且静置培养可以作为液滴培养的预试验方法, 为后期液滴微流控方法的建立提供参考。从S1出发, 通过该方法进行一轮的筛选, 突变体中产量最高的菌株产量达到2.53 g/L。实验室中保藏的菌株R1, 为从S1出发, 通过大量平板筛选, 摇瓶验证, 在3年中筛选得到的产量最高的菌株。使用S1作为出发菌株进行孔板筛选, 同时对比了平板筛选和孔板筛选的效率。然而与流式细胞筛选和液滴微流控筛选相比较, 其通量有差距, 但在现有的流式分选与液滴微流控分选不适用于目标菌株的条件下, 可通过多轮的筛选实现产量的进一步提高。
4 结论本研究考察了不同的筛选技术, 如流式细胞分选、液滴微流控、96孔板静置及振荡培养, 在核黄素菌株筛选方面的效果。流式细胞仪不能实现高低产菌的区分; 液滴微流控可以对高低产菌株进行区分, 但现有方法不适用于菌株分选。因此我们建立了96孔板筛选方法, 静置培养-荧光检测法可以实现高产菌株与低产菌株的区分, 且该方法简便、快速、具有较好的稳定性和准确性。利用静置培养-荧光检测法从工业菌突变体库中筛选到了核黄素产量提高的菌株, 证明了该方法具有实际应用意义。
| [1] |
Karrer P, Pfaehler K, Benz F, et al. Synthesen von Flavinen Ⅲ[J]. Helvetica Chimica Acta, 18(1): 69-79. DOI:10.1002/hlca.19350180111 |
| [2] |
Kis K, Bacher A. Substrate Channeling in the Lumazine Synthase/Riboflavin Synthase Complex of Bacillus subtilis[J]. Journal of Biological Chemistry, 1995, 270(28): 16788-16795. DOI:10.1074/jbc.270.28.16788 |
| [3] |
Paracchini V, Petrillo M, Reiting R, et al. Molecular characte rizati on of an unauthorized genetically modified, Bacillus subtilis, production strain identified in a vitamin B 2, feed additive[J]. Food Chemistry, 2017, 230: 681-689. DOI:10.1016/j.foodchem.2017.03.042 |
| [4] |
Schwechheimer SK, Park EY, José LR, et al. Biotechnology of riboflavin[J]. Applied Microbiology and Biotechnology, 2016, 100(5): 2107-2119. DOI:10.1007/s00253-015-7256-z |
| [5] |
Althöefer H, Revuelta JL, Seulberger H, Zelder O. Genetic method for producing riboflavin: WO, WO1999061623A2[P]. 1999-02-12.
|
| [6] |
Lehmann M, Degen S, Hohmann HP, et al. Biosynthesis of riboflavin:Screening for an improved GTP cyclohydrolase Ⅱ mutant[J]. FEBS Journal, 2009, 276(15): 4119-4129. DOI:10.1111/j.1742-4658.2009.07118.x |
| [7] |
Duan YX, Chen T, Chen X, et al. Overexpression of glucose-6-phosphate dehydrogenase enhances riboflavin production in Bacillus subtilis[J]. Applied Microbiology and Biotechnology, 2010, 85(6): 1907-1914. DOI:10.1007/s00253-009-2247-6 |
| [8] |
Shi T, Wang YC, Wang ZW. Deregulation of purine pathway in Bacillus subtilis and its use in riboflavin biosynthesis[J]. Microbial Cell Factories, 2014, 13: 101. |
| [9] |
Wang Z, Chen T, Ma X, et al. Enhancement of riboflavin production with Bacillus subtilis by expression and site-directed mutagenesis of zwf and gnd gene from Corynebacterium glutamicum[J]. Bioresource Technology, 2011, 102(4): 3934-3940. DOI:10.1016/j.biortech.2010.11.120 |
| [10] |
Guanglu W, Ting S, Tao C, et al. Integrated whole-genome and transcriptome sequence analysis reveals the genetic characteristics of a riboflavin-overproducing, Bacillus subtilis[J]. Metabolic Engineering, 2018, 48: 138-149. DOI:10.1016/j.ymben.2018.05.022 |
| [11] |
Hu J, Lei P, Mohsin A, et al. Mixomics analysis of Bacillus subtilis:effect of oxygen availability on riboflavin production[J]. Microbial Cell Factories, 2017, 16(1): 150. DOI:10.1186/s12934-017-0764-z |
| [12] |
Revuelta JL, Ledesma-Amaro R, Lozano-Martinez P, et al. Biopro-duction of riboflavin:a bright yellow history[J]. Journal of Industrial Microbiology & Biotechnology, 2017, 44(4-5): 659-665. |
| [13] |
Mahr R, Cornelia Gätgens, Jochem Gätgens, et al. Biosensor-driven adaptive laboratory evolution of L-valine production in Corynebacterium glutamicum[J]. Metabolic Engineering, 2015, 32: 184-194. DOI:10.1016/j.ymben.2015.09.017 |
| [14] |
Gong J, Zheng H, Wu Z, et al. Genome shuffling:Progress and applications for phenotype improvement[J]. Biotechnology Advances, 2009, 27(6): 996-1005. DOI:10.1016/j.biotechadv.2009.05.016 |
| [15] |
Parekh S, Vinci VA, Strobel RJ. Improvement of microbial strains and fermentation processes[J]. Applied Microbiology and Biotechnology, 2000, 54(3): 287-301. |
| [16] |
Portnoy VA, Bezdan D, Zengler K. Adaptive laboratory evolution—harnessing the power of biology for metabolic engineering[J]. Curr Opin Biotechnol, 2011, 22(4): 590-594. DOI:10.1016/j.copbio.2011.03.007 |
| [17] |
Dragosits M, Mattanovich D. Adaptive laboratory evolution - principles and applications for biotechnology[J]. Microbial Cell Factories, 2013, 12(1): 64. DOI:10.1186/1475-2859-12-64 |
| [18] |
Barahona E, Jiménez-Vicente, Emilio, Rubio LM. Hydrogen overproducing nitrogenases obtained by random mutagenesis and high-throughput screening[J]. Scientific Reports, 2016, 6(1): 38291. DOI:10.1038/srep38291 |
| [19] |
Vlaardingerbroek I, Beerens B, Shahi S, et al. Fluorescence Assisted Selection of Transformants(FAST):Using flow cytometry to select fungal transformants[J]. Fungal Genetics and Biology, 2015, 76: 104-109. DOI:10.1016/j.fgb.2015.02.003 |
| [20] |
Sjostrom SL, Bai Y, Huang M, et al. High-throughput screening for industrial enzyme production hosts by droplet microfluidics[J]. Lab on a Chip, 2014, 14(4): 806-813. |
| [21] |
Lagus TP, Edd JF. A review of the theory, methods and recent applications of high-throughput single-cell droplet microfluidics[J]. Journal of Physics D:Applied Physics, 2013, 46(11). DOI:10.1088/0022-3727/46/11/114005 |
| [22] |
José L. Revuelta, Ledesma-Amaro R, Alberto Jiménez. Industrial Production of Vitamin B2 by Microbial Fermentation[M]// Industrial Biotechnology of Vitamins, Biopigments, and Antioxidants. Wiley-VCH Verlag GmbH & Co. KGaA, 2016.
|
| [23] |
Abbas CA, Sibirny AA. Genetic control of biosynthesis and transport of riboflavin and flavin nucleotides and construction of robust biotechnological producers[J]. Microbiology & Molecular Biology Reviews Mmbr, 2011, 75(2): 321. |
| [24] |
Chen J, Vestergaard M, Jensen TG. Finding the Needle in the Haystack—the Use of Microfluidic Droplet Technology to Identify Vitamin-Secreting Lactic Acid Bacteria[J]. mBio, 2017, 8(3): e00526-17. |
| [25] |
Wagner JM, Liu L, Yuan SF, et al. A comparative analysis of single cell and droplet-based FACS for improving production phenotypes:Riboflavin overproduction in Yarrowia lipolytica[J]. Metabolic Engineering, 2018, 47: 346-356. DOI:10.1016/j.ymben.2018.04.015 |
| [26] |
Pedrolli DB, Kühm C, Sévin DC, et al. A dual control mechanism synchronizes riboflavin and sulphur metabolism in\r, Bacillus subtilis[J]. Proceedings of the National Academy of Sciences, 2015, 112(45): 14054-14059. DOI:10.1073/pnas.1515024112 |
| [27] |
Silva R, Aguiar TQ, Domingues, Lucília. Blockage of the pyrimidine biosynthetic pathway affects riboflavin production in Ashbya gossypii[J]. Journal of Biotechnology, 2015, 193: 37-40. DOI:10.1016/j.jbiotec.2014.11.009 |
| [28] |
Liu D, Huang C, Guo J, et al. Development and characterization of a CRISPR/Cas9n-based multiplex genome editing system for Bacillus subtilis[J]. Biotechnol Biofuels, 2019, 12: 197-213. DOI:10.1186/s13068-019-1537-1 |




