自20世纪60年代首次实现发酵法生产谷氨酸以来,微生物发酵技术凭借其成本低廉、工艺简单和易于分离等特点,迅速扩大生产规模,增加多种氨基酸生产工艺,将氨基酸产品产值升至逾百亿美金,涉及饲料添加剂、保健品和化妆品等多种行业[1]。目前,除蛋氨酸等个别氨基酸仍依赖于化学合成及酶催化以外,其余均可采用微生物发酵技术生产,不少厂商已开始通过转向合成氨基酸衍生物等高附加值产品以缓解趋于饱和的氨基酸市场[2]。
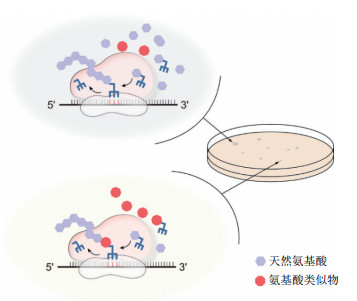
高产稳定的微生物菌株无疑是支撑大规模发酵生产氨基酸的基本保障。然而,野生菌株通过精细的代谢调控机制以避免产生过多的氨基酸。最初研究人员通过筛选营养缺陷型菌株,成功获得了可以积累鸟氨酸和赖氨酸的多种缺陷型菌株[3-4]。然而,营养缺陷型菌株并没有真正意义上解除反馈机制,其应用范围也止步于此。1950年有研究发现具有氨基酸类似物抗性的菌株同时具有向胞外分泌氨基酸的特性[5-6],并于1970年首次利用氨基酸类似物筛选出高产赖氨酸的菌株[7]。该方法的本质是对胞内翻译过程的竞争性抑制(图 1)。这些类似物的结构、分子量和电荷特性与组成蛋白质的氨基酸十分相近,可以与对应的氨基酸竞争数量有限的tRNA,却无法参与肽链的延伸[8]。它们所结合的tRNA比例越高,对蛋白质正常翻译的阻碍就越大,最终导致菌体生长减缓甚至死亡[9-10]。
|
| 图 1 氨基酸类似物筛选氨基酸高产菌株作用机理 |
因此,通过简单正筛实验,从突变菌中挑取能够在高浓度氨基酸类似物条件下生长最快的菌落,即有可能获得高产氨基酸的突变菌株。氨基酸类似物的筛选工作几乎覆盖了20种天然氨基酸(表 1),也涉及不同种属的菌株,极大地推进了微生物大规模生产氨基酸的工业化进程。然而,高浓度的氨基酸类似物会干扰细胞代谢甚至影响细胞结构[11-12],部分突变会赋予细胞隔离、外排甚至降解氨基酸类似物的能力[10, 13],从而摆脱筛选压力。
目前,使用氨基酸类似物筛选氨基酸高产菌株的方法已经贡献出许多优良的生产菌株,但是对菌株产量提升的幅度越来越小。一方面是因为长期使用的类似物种类有限,导致从筛选菌株中获得新突变的难度变大;另一方面是由于早期的生产菌株多是通过对随机突变库进行多轮筛选得到,在提高氨基酸发酵产量的同时在菌株基因组中引入了不确定的突变,可能导致进一步提高氨基酸积累的困难。近年来,氨基酸生物传感器相关研究不断产生突破。利用富含稀有密码子的基因筛选氨基酸高产菌株的策略也被提出,提供了面向更加广谱的微生物菌种的高效、准确和简便的筛选方法,利用该方法筛选获得的菌株可以为微生物生产和积累氨基酸的研究提供更丰富的基因信息。
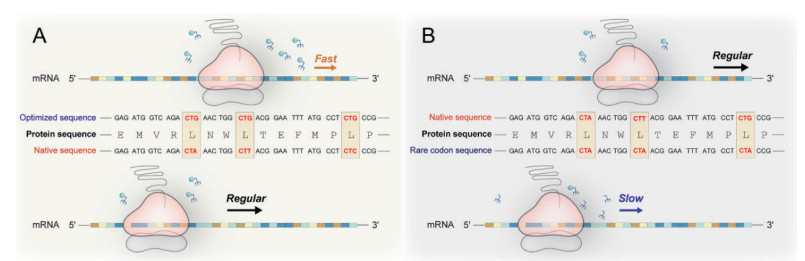
1 利用稀有密码子筛选氨基酸高产菌株在肽链合成过程中,密码子可被对应的氨酰tRNA识别并翻译成氨基酸。然而,不同密码子对应的tRNA的相对丰度并不相同,翻译高频密码子的tRNA在胞内的含量最为丰富,而翻译稀有密码子的tRNA含量较低[26]。在同类tRNA竞争游离氨基酸的过程中,含量丰富的tRNA有更大的概率竞争到氨基酸并完成高频密码子的翻译。因此,将异源蛋白编码基因中的密码子替换为宿主偏好的高频密码子可以显著提高目标蛋白质的表达量,由此催生了“密码子优化”策略[27]。在氨基酸饥饿的情况下,稀有tRNA在与其他tRNA的竞争中难以捕获到足够的氨基酸,常处于“空载”状态,难以完成稀有密码子的翻译。因此在氨基酸饥饿条件下,富含稀有密码子的异源蛋白编码基因的翻译速率普遍缓慢,甚至无法进行。只有当胞内氨基酸浓度大幅提升并使大部分tRNA维持“满载”时,稀有tRNA才有机会捕获到剩余的氨基酸[28],富含稀有密码子的基因才能正常表达。这些能够在胞内累积氨基酸的菌株即是所谓的氨基酸高产菌株(图 2)[29]。
与氨基酸类似物竞争性地抑制翻译过程相似,利用富含稀有密码子的筛选标记可以将翻译过程与胞内氨基酸浓度建立关联,并在氨基酸饥饿的条件将差异进一步的放大,最终通过生长和表型差异筛选氨基酸高产菌株。根据所用筛选标记的不同,该方法可以分为选择系统和筛选系统。前者使用抗生素抗性基因,表现为菌体的生长水平差异,后者使用颜色蛋白、荧光等表型基因,表现为菌体特征差异。在两种筛选系统中,质粒拷贝数和标记基因的启动子强度会影响菌体的生长。改变稀有密码子的频率,以及更换复制起始位点或启动子可调节系统的选择强度。实验表明使用弱启动子和拷贝数较低的复制起始位点更有助于提高系统的选择或筛选能力。当使用诱导型启动子时,可设定不同的诱导剂量逐级改变启动子强度,从而确定最佳的诱导水平。
突变不仅能够赋予菌株高产某种氨基酸的特性,还有可能造成对抗生素的抗性,导致以抗生素抗性基因为筛选标记的选择系统中出现假阳性的结果。以上假阳性现象可通过两种途径消除:其一是向选择系统质粒中插入gfp等颜色指示基因,存活于抗生素胁迫的菌株可通过荧光或颜色表型进行复筛,以确保选择系统功效的发挥;还可构建含有两种抗生素抗性基因的选择系统以消除假阳性菌株。除富含稀有密码子的抗性基因外,该质粒系统上还插入了针对另外一种抗生素的野生型抗性基因。在诱变中获得了一种抗生素抗性的菌株几乎不可能同时获得针对另一种抗生素的抗性。因此,任何存活于两种抗生素胁迫条件下的菌株都应含有筛选质粒并具备更强的氨基酸合成能力。除常见的抗性基因外,sacB[30]、tolC[31]和ccdAB[32]等毒素-解毒系统也可用于选择及筛选系统的构建。当采用这类基因时,可将解毒基因上的密码子替换为其稀有形式。此时,只有能够高产对应氨基酸的菌株才可以维持解毒基因的表达,从而产生足够量的解毒蛋白使菌株得以生存。
以上高、低丰度tRNA对胞内游离氨基酸的竞争机制为氨基酸生物传感器的设计提供了新思路。将基因中的密码子替换为其稀有形式即可提高翻译过程对氨基酸含量要求的“门槛值”,此时,蛋白的表达速率可反映胞内氨基酸的浓度。通过将蛋白质的表达水平与菌体生长或颜色表型相偶联,即可从菌株库中快速鉴别出可能的氨基酸高产菌株。该策略对细胞结构及胞内其他生化过程无负面影响,理论上可以克服氨基酸类似物筛选法的缺陷。
2 稀有密码子筛选策略的应用拓展密码子偏好性的发现及其延伸出的稀有密码子的概念和密码子优化的技术方案,该方案解决了基因工程中异源蛋白表达困难的问题[27]。但是不断有研究发现,稀有密码子的作用不仅仅是限制mRNA的翻译速率,它们在稳定mRNA二级结构、调控肽链延伸速率和辅助蛋白折叠等方面也有非常重要的作用。替换某些必需基因中的稀有密码子(如精氨酸稀有密码子AGR)可能会影响序列结构和翻译起始,从而造成细胞死亡[33-35]。利用稀有密码子造成的翻译胁迫筛选氨基酸高产菌株方法,本质上是在饥饿条件下多种tRNA竞争氨基酸的过程,证明了增加稀有密码子数量这一“密码子负优化”方式可以显著减缓甚至终止mRNA的翻译。在某些基因(如dCas9)中增加稀有密码子的数量,限制其在生长前期的表达量,在需要的时候通过补加氨基酸来调控特定基因的翻译。这种类似诱导表达的模式具有调节精确,无泄露表达,不影响细胞正常生长等特点,且价格远低于常用诱导剂。
部分氨基酸的密码子数量较少,细胞对这部分同义密码子的使用频率没有明显的差异,限制了该方法在此类氨基酸高产菌株筛选中的应用。通过引入异源的tRNA和氨酰tRNA合成酶(Aminoacyl tRNA Synthetase,aaRS)正交对能够针对性的解决以上问题。通常经过筛选得到的tRNA和aaRS正交对相互识别,且独立于宿主翻译系统中的其它正交对,可以成功将非天然氨基酸插入到肽链中,实现蛋白质功能的改造[36]。目前使用非天然氨基酸改造蛋白质的研究已经较为成熟。利用终止密码子等三联体密码子将非天然氨基酸插入肽链的策略可能干扰基因组其它基因的正常翻译。一方面有研究通过人工修改基因组序列,构建仅有63种甚至61种密码子的大肠杆菌[37-38];另一方面使用翻译四联体密码子的tRNA和aaRS正交对,既能够解决菌株中没有稀有密码子的问题,又可以避免干扰胞内正常tRNA的工作[39]。因此,通过改造tRNA反密码子环或者引入翻译四联体密码子的tRNA及其aaRS正交对,可以人为地在胞内引入“稀有”密码子及tRNA,从而实现对其余氨基酸高产菌株的筛选。这一策略不仅可以为常用的生产菌株筛选出更多新的突变位点,更重要的是可以凭借密码子的普适性,将筛选范围拓展至具有多密码子的氨基酸及所有微生物物种,为氨基酸高产菌株的筛选提供了高效解决方案。
3 基于转录与翻译调控胞内氨基酸合成微生物细胞通过不同层级的转录调控来实现基因功能的选择性发挥,进而将资源引导至不同的生命活动形式,而翻译是转录的终点,是实施调控的关键环节,它允许细胞在mRNA合成终止后对蛋白质合成进行再次干预,这对于延长发酵时间、增加底物利用率和目标代谢物产量有重要意义。目前通过转录和翻译调控的强化,以分配合理化、功能多样化及途径最优化等理念,成功构建了用于生产生物燃料、糖醇和芳香族化合物等物质的高产菌株[40]。基于转录和翻译水平的工程改造可针对翻译速率与翻译的持续性两部分展开,直接影响了途径酶的生成速率与总量[41-42]。
3.1 翻译速率许多目标合成途径包含异源基因,因缺少与之匹配的原生翻译系统,异源基因在底盘宿主内的表达常受到阻碍。即便对于宿主来源的基因,其表达产生蛋白的效率也往往难以达到规模化生产的要求。翻译速率的优化可以使细胞在单位时间内产生更多的酶,从而由生长状态快速投入至生产活动,也能使细胞工厂及时补充生产过程中酶的消耗,确保生产的持续进行[43-44]。
翻译速率的优化可以从翻译的起始和肽链的延伸两个过程入手。前者依赖于对核糖体结合区域(RBS)(原核)或mRNA的5'端帽(真核)的改造,旨在加强mRNA招募核糖体的能力,提升核糖体起始翻译的效率[9]。优化后的RBS可以使蛋白表达量提升数倍,进一步驱动目标化合物的生成[10]。调控肽链延伸的常用手段是密码子优化,它依据宿主的密码子偏好性,将异源基因中的稀有密码子替换为宿主常用的密码子形式,使其更容易被宿主的tRNA读取,从而将核酸信息快速翻译为氨基酸序列。与原始基因相比,优化后的基因在微生物宿主中的表达效率可提高100倍以上[45]。此外,mRNA中的二级结构也会阻碍核糖体的移动并降低翻译速率[46],这一负面作用也可通过密码子优化消除[47]。
3.2 翻译的持续性作为翻译的模板,mRNA的稳定性将直接影响翻译过程的持续时间。稳定的mRNA可供翻译系统反复使用,使细胞以最少的转录投入获得预期的蛋白产量。该策略弥补了基因表达过程中转录环节的限制,可有效提升低拷贝途径基因的表达水平[11]。一般可通过向mRNA的5'或3'端引入发卡或茎环结构,保护片段不被RNA酶降解[12-13]。
对转录和翻译水平的正向调控可以使微生物工厂合成目标代谢物的能力得到提升,而对翻译水平的负调控则为筛选氨基酸高产菌株提供了全新的思路[29, 48]。参与蛋白质合成的20种氨基酸总共由61个密码子编码,除蛋氨酸和色氨酸外,每一种氨基酸至少对应两种以上的密码子,其中精氨酸、亮氨酸和丝氨酸的同义密码子数量可达6个[49]。原核与真核生物均表现出对特定密码子的使用偏好性,其中使用频率较低的一类密码子被称为稀有密码子[50-51]。密码子使用的偏好性因物种而异,一种生物的高频密码子可能是另一种生物的稀有密码子[52]。这一偏好性会影响异源基因的翻译起始[53]、肽链延伸[54]及蛋白折叠[55]等过程。
4 总结与展望目前仍有一些氨基酸缺少能够用于大规模生产的菌株。一方面由于传统筛选方法的局限性,某些途径特殊的氨基酸必须经过多种类似物的轮流筛选,但产量难有显著提升;另一方面是对高产菌株进行进一步基因编辑或整合多种优势突变时难度较大,缺乏对微生物细胞工厂理性设计及优化的技术基础。已经有研究仅对谷氨酸棒状杆菌基因组进行12处编辑,其赖氨酸的产量提高至120 g/L[56]。因此,需要将全新的筛选方法应用于多种氨基酸、不同菌种的筛选中,获得更多的优势突变信息,为分析和构建氨基酸高产菌株提供更多的基因信息;同时也利用简易高效的基因编辑技术,整合多种优势突变信息于单一菌株中,从头构建可用于大规模生产的工业菌株,在转录和翻译水平针对性设计优化胞内代谢流分配,最终将菌株的生产能力和效率提升到新的水平。
| [1] |
Wu G. Functional amino acids in growth, reproduction, and health[J]. Advances in Nutrition, 2010, 1(1): 31-37. DOI:10.3945/an.110.1008 |
| [2] |
Mitsuhashi S. Current topics in the biotechnological production of essential amino acids, functional amino acids, and dipeptides[J]. Curr Opin Biotechnol, 2014, 26: 38-44. DOI:10.1016/j.copbio.2013.08.020 |
| [3] |
Udaka S, Kinoshita S. Studies on L-ornithine fermentation[J]. J Gen Appl Microbiol, 1958, 4(4): 272-282. DOI:10.2323/jgam.4.272 |
| [4] |
Nakayama K, Kitada S, Kinoshita S. Studies on lysine fermentation I[J]. J Gen Appl Microbiol, 1961, 7(3): 145-154. DOI:10.2323/jgam.7.145 |
| [5] |
Adelberg EA. Selection of bacterial mutants which excrete antagonists of antimetabolites[J]. J Bacteriol, 1958, 76(3): 326-326. DOI:10.1128/JB.76.3.326-326.1958 |
| [6] |
Moyed H, Friedman M. Interference with feedback control:a mechanism of antimetabolite action[J]. Science, 1959, 129(3354): 968-969. DOI:10.1126/science.129.3354.968 |
| [7] |
Sano K, Shiio I. Microbial production of L-lysine[J]. J Gen Appl Microbiol, 1970, 16(5): 373-391. DOI:10.2323/jgam.16.5_373 |
| [8] |
Shive W, Skinner CG. Amino acid analogues[M]. New York: Academic Press, 2012: 1-58.
|
| [9] |
Laursen BS, Sørensen HP, Mortensen KK, et al. Initiation of protein synthesis in bacteria[J]. Microbiol Mol Biol Rev, 2005, 69(1): 101-123. |
| [10] |
George KW, Thompson MG, Kang A, et al. Metabolic engineering for the high-yield production of isoprenoid-based C5 alcohols in E. coli[J]. Scientific Reports, 2015, 5(1): 11128. DOI:10.1038/srep11128 |
| [11] |
Carrier TA, Keasling JD. Controlling messenger RNA stability in bacteria:strategies for engineering gene expression[J]. Biotechnol Prog, 1997, 13(6): 699-708. DOI:10.1021/bp970095h |
| [12] |
Hannig G, Makrides SC. Strategies for optimizing heterologous protein expression in Escherichia coli[J]. Trends Biotechnol, 1998, 16(2): 54-60. DOI:10.1016/S0167-7799(97)01155-4 |
| [13] |
Smolke CD, Carrier TA, Keasling JD. Coordinated, differential expression of two genes through directed mRNA cleavage and stabilization by secondary structures[J]. Appl Environ Microbiol, 2000, 66(12): 5399-5405. DOI:10.1128/AEM.66.12.5399-5405.2000 |
| [14] |
Park JH, Jang YS, Lee JW, et al. Escherichia coli W as a new platform strain for the enhanced production of L-valine by systems metabolic engineering[J]. Biotechnol Bioeng, 2011, 108(5): 1140-1147. DOI:10.1002/bit.23044 |
| [15] |
Cusyatiner M, Voroshilova E, Rostova Y, et al. Method for producing L-leucine: USA, US20040091980A1[P]. 2004-5-13.
|
| [16] |
Shiio I, Sasaki A, Nakamori S, et al. Production of L-isoleucine by AHV resistant mutants of Brevibacterium flavum[J]. Agric Biol Chem, 1973, 37(9): 2053-2061. DOI:10.1080/00021369.1973.10860948 |
| [17] |
Nakamori S, Morioak H, Yoshinaga F, et al. Fermentative production of L-proline by DL-3, 4-dehydroproline resistant mutants of L-glutamate producing bacteria[J]. Agric Biol Chem, 1982, 46(2): 487-491. |
| [18] |
Tsuchida T, Matsui H, Enei H, et al. Method of producing L-phenylalanine by fermentation: USA, US3909353A[P]. 1975-9-30.
|
| [19] |
Hagino H, Nakayama K. L-Tryptophan production by analog-resistant mutants derived from a phenylalanine and tyrosine double auxotroph of Corynebacterium glutamicum[J]. Agric Biol Chem, 1975, 39(2): 343-349. |
| [20] |
Suga M, Sugimoto M, Osumi T, et al. Method of producing L-serine by fermentation: USA, US6037154A[P]. 2000-3-14.
|
| [21] |
Hagino H, Nakayama K. L-Tyrosine production by analog-resistant mutants derived from a phenylalanine auxotroph of Corynebacterium glutamicum[J]. Agric Biol Chem, 1973, 37(9): 2013-2023. DOI:10.1080/00021369.1973.10860958 |
| [22] |
Kase H, Nakayama K. L-Methionine production by methionine analog-resistant mutants of Corynebacterium glutamicum[J]. Agric Biol Chem, 1975, 39(1): 153-160. |
| [23] |
Kase H, Nakayama K. Production of L-threonine by analog-resistant mutants[J]. Agric Biol Chem, 1972, 36(9): 1611-1621. DOI:10.1080/00021369.1972.10860451 |
| [24] |
Yoshida H, Araki K, Nakayama K. L-arginine production by arginine analog-resistant mutants of microorganisms[J]. Agric Biol Chem, 1981, 45(4): 959-963. |
| [25] |
Araki K, Nakayama K. Studies on histidine fermentation:Part I. L-histidine production by histidine analog-resistant mutants from several bacteria[J]. Agric Biol Chem, 1971, 35(13): 2081-2088. |
| [26] |
Dong H, Nilsson L, Kurland CG. Co-variation of trna abundance and codon usage in Escherichia coli at different growth rates[J]. J Mol Biol, 1996, 260(5): 649-663. DOI:10.1006/jmbi.1996.0428 |
| [27] |
Burgess-Brown NA, Sharma S, Sobott F, et al. Codon optimization can improve expression of human genes in Escherichia coli:A multi-gene study[J]. Protein Expr Purif, 2008, 59(1): 94-102. DOI:10.1016/j.pep.2008.01.008 |
| [28] |
Sørensen MA. Charging levels of four tRNA species in Escherichia coli Rel+ and Rel- strains during amino acid starvation:a simple model for the effect of ppGpp on translational accuracy[J]. J Mol Biol, 2001, 307(3): 785-798. DOI:10.1006/jmbi.2001.4525 |
| [29] |
Zheng B, Ma X, Wang N, et al. Utilization of rare codon-rich markers for screening amino acid overproducers[J]. Nat Commun, 2018, 9(1): 3616. DOI:10.1038/s41467-018-05830-0 |
| [30] |
Pelicic V, Reyrat JM, Gicquel B. Expression of the Bacillus subtilis sacB gene confers sucrose sensitivity on mycobacteria[J]. J Bacteriol, 1996, 178(4): 1197-1199. DOI:10.1128/JB.178.4.1197-1199.1996 |
| [31] |
Gregg CJ, Lajoie MJ, Napolitano MG, et al. Rational optimization of tolC as a powerful dual selectable marker for genome engineering[J]. Nucleic Acids Res, 2014, 42(7): 4779-4790. DOI:10.1093/nar/gkt1374 |
| [32] |
Wang H, Bian X, Xia L, et al. Improved seamless mutagenesis by recombineering using ccdB for counterselection[J]. Nucleic Acids Res, 2013, 42(5): e37-e37. |
| [33] |
Napolitano MG, Landon M, Gregg CJ, et al. Emergent rules for codon choice elucidated by editing rare arginine codons in Escherichia coli[J]. Proc Natl Acad Sci USA, 2016, 113(38): E5588-E5597. DOI:10.1073/pnas.1605856113 |
| [34] |
Fredrick K, Ibba M. How the sequence of a gene can tune its translation[J]. Cell, 2010, 141(2): 227-229. DOI:10.1016/j.cell.2010.03.033 |
| [35] |
Quax TE, Claassens NJ, Soll D, et al. Codon Bias as a Means to Fine-Tune Gene Expression[J]. Mol Cell, 2015, 59(2): 149-161. DOI:10.1016/j.molcel.2015.05.035 |
| [36] |
Liu W, Brock A, Chen S, et al. Genetic incorporation of unnatural amino acids into proteins in mammalian cells[J]. Nat Meth, 2007, 4(3): 239-244. DOI:10.1038/nmeth1016 |
| [37] |
Lajoie MJ, Rovner AJ, Goodman DB, et al. Genomically recoded organisms expand biological functions[J]. 2013, 342(6156): 357-360.
|
| [38] |
Fredens J, Wang K, De La Torre D, et al. Total synthesis of Escherichia coli with a recoded genome[J]. Nature, 2019, 569(7757): 514-518. DOI:10.1038/s41586-019-1192-5 |
| [39] |
Anderson JC, Wu N, Santoro SW, et al. An expanded genetic code with a functional quadruplet codon[J]. 2004, 101(20): 7566-7571.
|
| [40] |
Liu X, Ding W, Jiang H. Engineering microbial cell factories for the production of plant natural products:from design principles to industrial-scale production[J]. Microb Cell Fact, 2017, 16(1): 125. DOI:10.1186/s12934-017-0732-7 |
| [41] |
Kosuri S, Goodman DB, Cambray G, et al. Composability of regulatory sequences controlling transcription and translation in Escherichia coli[J]. Proceedings of the National Academy of Sciences, 2013, 110(34): 14024-14029. DOI:10.1073/pnas.1301301110 |
| [42] |
Mutalik VK, Guimaraes JC, Cambray G, et al. Precise and reliable gene expression via standard transcription and translation initiation elements[J]. Nat Meth, 2013, 10(4): 354. DOI:10.1038/nmeth.2404 |
| [43] |
Peralta-Yahya PP, Zhang F, Del Cardayre SB, et al. Microbial engineering for the production of advanced biofuels[J]. Nature, 2012, 488(7411): 320. DOI:10.1038/nature11478 |
| [44] |
Seo SW, Yang J, Min BE, et al. Synthetic biology:Tools to design microbes for the production of chemicals and fuels[J]. Biotechnol Adv, 2013, 31(6): 811-817. DOI:10.1016/j.biotechadv.2013.03.012 |
| [45] |
Johansson A, Boltongrob R, Mannervik B. Use of silent mutations in cDNA encoding human glutathione transferase M2-2 for optimized expression in Escherichia coli[J]. Protein Expr Purif, 1999, 17(1): 105-112. DOI:10.1006/prep.1999.1117 |
| [46] |
Tuller T, Waldman YY, Kupiec M, et al. Translation efficiency is determined by both codon bias and folding energy[J]. Proc Natl Acad Sci USA, 2010, 107(8): 3645-3650. DOI:10.1073/pnas.0909910107 |
| [47] |
Wu X, Jörnvall H, Berndt KD, et al. Codon optimization reveals critical factors for high level expression of two rare codon genes in Escherichia coli:RNA stability and secondary structure but not tRNA abundance[J]. Biochem Biophys Res Commun, 2004, 313(1): 89-96. DOI:10.1016/j.bbrc.2003.11.091 |
| [48] |
Huo YX, Zheng B, Wang N, et al. Identifying amino acid overproducers using rare-codon-rich markers[J]. J Vis Exp, 2019(148): e59331. |
| [49] |
Crick FHC. On the genetic code[J]. Science, 1963, 139(3554): 461-464. DOI:10.1126/science.139.3554.461 |
| [50] |
Hershberg R, Petrov DA. Selection on codon bias[J]. Annu Rev Genet, 2008, 42: 287-299. DOI:10.1146/annurev.genet.42.110807.091442 |
| [51] |
Plotkin JB, Kudla G. Synonymous but not the same:the causes and consequences of codon bias[J]. Nat Rev Genet, 2011, 12(1): 32-42. DOI:10.1038/nrg2899 |
| [52] |
Bulmer M. Coevolution of codon usage and transfer RNA abundance[J]. Nature, 1987, 325(6106): 728-730. DOI:10.1038/325728a0 |
| [53] |
Bentele K, Saffert P, Rauscher R, et al. Efficient translation initiation dictates codon usage at gene start[J]. Mol Syst Biol, 2013, 9(1): 675-675. DOI:10.1038/msb.2013.32 |
| [54] |
Chu D, Kazana E, Bellanger N, et al. Translation elongation can control translation initiation on eukaryotic mRNAs[J]. The EMBO journal, 2014, 33(1): 21-34. DOI:10.1002/embj.201385651 |
| [55] |
Pechmann S, Frydman J. Evolutionary conservation of codon optimality reveals hidden signatures of co-translational folding[J]. Nat Struct Mol Biol, 2013, 20(2): 237-243. DOI:10.1038/nsmb.2466 |
| [56] |
Becker J, Zelder O, Häfner S, et al. From zero to hero—Design-based systems metabolic engineering of Corynebacterium glutamicum for L-lysine production[J]. Metab Eng, 2011, 13(2): 159-168. DOI:10.1016/j.ymben.2011.01.003 |