枯草芽孢杆菌与真核系统相比具有生长快,发酵周期短且易于培养等优势;与其他原核系统相比具有较强的分泌表达能力[1-2],能有效避免细胞内蛋白质的积累和不溶性包涵体的形成,其遗传背景清晰,被认为是下一代超高效分泌的细胞工厂[3]并具有开发成底盘细胞的潜力[4]。
利用传统的代谢工程手段可以实现一定时间内目的产物的积累,但是由于局部途径的过表达,往往会造成细胞代谢流的失衡[5]。如何平衡细胞的生长与产物的高效合成是代谢工程的一个重要问题[6]。微生物代谢流的分配在生长过程中并非一成不变,而是随着胞内代谢物水平及环境的变化而发生动态调整。动态调控手段能在保证目的产物高效合成的情况下,同时平衡细胞的生长,实现高产量和高底物转化率的统一。
本文主要从转录水平和转录后水平上综述了近几年来枯草芽孢杆菌动态表达调控工具的开发,并就其调控机制进行了介绍,最后对这些调控工具在产物合成中的应用做了简要概述。
1 转录水平调控目的基因表达的第一步就是转录,在转录水平上进行基因表达调控是最直接和最方便的。基因的转录过程通常与外界物质、转录因子、胞内代谢物以及群体环境等相关[7],通过控制这些影响因子可以实现基因转录水平的调控。
1.1 基于诱导型启动子的调控与组成型启动子相比,诱导型启动子具有可以人为控制基因转录的开启或者关闭等独特优势,从而有效地应用于基因的调控表达。近几年来,多种类型的诱导型启动子被开发,应用于代谢调控中。常见的诱导型启动子有Pgrac(IPTG诱导)、PspaS(枯草菌素诱导)、PxylA(木糖诱导)、Pglv(麦芽糖诱导)和PsacB(蔗糖诱导),这些启动子已被普遍用于产物的代谢调控[8]。Jiao等[9]将启动子PgroE和PsacB融合构建了蔗糖诱导型启动子Pg1,同时将PgroE与lacO融合构建了IPTG诱导型启动子Pg2,并进一步通过在-35与-10区域进行突变获得启动子Pg3,最终使表面活性素高达9.74 g/L。为实现多个基因的表达调控,Zhou等[10]将来源于巨大芽孢杆菌的PxylA木糖诱导启动子和来源于大肠的IPTG诱导型启动子Pgrac组合生产紫穗槐二烯,成功实现了在枯草芽孢杆菌中的调控。最近,Toymentseva等[11]开发了基于lial启动子的LIKE系统,其相似于乳酸菌NICE系统[12]和枯草芽孢杆菌SURE系统[13],其能严格调控基因表达,在非诱导条件下紧密关闭启动子。
考虑到诱导剂的补充和发酵过程的简化性,环境诱导型启动子也逐渐被开发。Welsch等[14]通过将des启动子与冷休克蛋白cspB基因的下游表达框或5'-UTR茎环结构融合构建成冷诱导启动子,在发酵培养过程中,只需要通过温度的控制即可以实现对木聚糖酶表达的调控。Li等[15]从枯草芽孢杆菌中分离和鉴定了两个温度敏感型启动子,构建了有效的温度诱导型表达系统。此外,还有一些环境依赖型的启动子,Yang等[16]选择了枯草芽孢杆菌114个内源启动子,以绿色荧光蛋白(GFP)为报告基因,分别对不同的温度和pH条件下启动子的表达强度进行了表征,通过将不同表达时期的启动子组合可以实现脂酶、角蛋白酶和碱性果胶酶的差异性表达。
1.2 基于核糖开关介导的表达调控2002年核糖开关(Riboswitch)在细菌中被发现,Riboswitch是mRNA上的一段5'非翻译区(5'UTR),包括核糖体结合位点(RBS)、操纵基因等元件,大多数核糖开关由适体和表达平台两个部分组成。适体是其RNA元件最保守的部分,可以与特定的小分子代谢物结合诱发mRNA变构,表达平台作为遗传控制元件将适体中的折叠变化转化为基因表达的变化。
Riboswitch主要有4种调控形式,分别为转录水平和翻译水平的抑制和激活调控,如图 1所示。在转录水平,若适体未结合配体之前处于打开状态,与配体结合后会使表达平台形成不依赖ρ因子(poly U)或依赖ρ因子(rho)的终止结构,表现为关闭状态,从而导致转录终止,如枯草芽孢杆菌的S-腺苷甲硫氨酸(SAM)riboswitch[17]、赖氨酸(lysC)riboswitch[18]和yitJ riboswitch[19]都属于这类调控方式。然而与之调控机制相反的腺嘌呤(pbuE)riboswitch[20],当在胞内存在高浓度的腺嘌呤时,腺嘌呤的结合可稳定核糖开关的适体结构域充当抗终止元件,从而导致下游基因的表达上调。大多数核糖开关可分为适体和表达平台两个结构域,而甘氨酸核糖开关[21]存在两个类似适体的配体结构域,这些结构域以一个短的保守序列连接,与配体结合后激活基因的表达,这种独特结构需确保过量的甘氨酸提供柠檬酸循环中的碳通量,当甘氨酸浓度减小时,核糖开关会快速关闭并保持足够量的氨基酸用于蛋白质合成[22]。除此之外,翻译水平的riboswitch将在下文进行介绍。
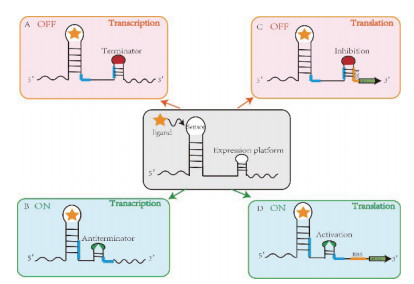
|
| 图 1 核糖开关调控基因表达的机理 |
群体感应(Quorum sensing,QS)是细菌细胞间通讯的一种形式[23],多存在革兰氏阴性菌中,主要是通过响应细胞密度进行基因的表达调控,该过程的调控途径是复杂的,需要多个酶和转录因子的共同作用。依赖于细胞密度调控的电路主要由诱导分子和受体蛋白两部分组成,诱导分子与受体结合激活靶基因,靶基因在细菌中发挥必要的功能,QS系统可以动态平衡目标产物的有效合成与细胞生长之间的关系。
革兰氏阳性菌和革兰氏阴性菌均存在QS系统,革兰氏阴性菌的QS系统已经得到了很好的开发[24],目前在枯草芽孢杆菌中存在的QS主要有ComP-ComA系统[25]和Phr-Rap系统[26],Phr60-Rap60-Spo0A系统是枯草芽孢杆菌中研究较多的群体感应系统,Spo0A是孢子形成的主要调节剂,必须磷酸化才能发挥活性。此外,随着细胞的生长,细胞密度的增加会促使Phr60增加[27],并通过Opp转运体进入细胞,而Phr60抑制Rap60的活性,Rap60通过调节磷酸化基来负调控Spo0A的活性,从而激活Spo0A调控基因的表达。其QS调控途径如图 2所示,Cui等[28]利用Phr60-Rap60-Spo0A系统构建了双功能表达的分子开关,通过增加前体磷酸烯醇丙酮酸(PEP)和庚二烯二磷酸(HDP)的供应,以改善甲基萘醌-7(MK-7)的合成。Spo0A抑制pyk和uppS的表达导致从PEP到TCA途径代谢通量受到限制,IPP的消耗减少,在后期阶段因为丙酮酸的生成,细胞生长迅速下降,其最终MK-7的产量增加,Spo0A上调ispH和hepS / T的表达,将碳通量推向MK-7合成途径,动态平衡有毒物质HMBPP和DMAPP对细胞生长的影响。Boguslawski等[29]确定了Rap蛋白调节ComA活性的新机制,提出了Rap60结合ComA并抑制其活性,而不会干扰ComA与DNA的结合的新研究。Wolf等[30]分析了ComA激活基因的转录机制,通过对比rapA,rapC,srfAA和lutP等启动子序列发现ComA除了结合反向重复序列(Inverted repeat,IR),还会结合直接重复序列(Direct repeat,DR),并以荧光为报告基因验证了DR是ComA与启动子结合的功能序列,同时利用凝胶电泳表明ComA是通过与α亚基相互作用而促进RNAP募集到启动子的过程。但ComP-ComA系统还未见在代谢调控中的应用。
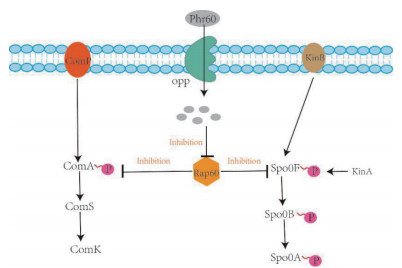
|
| 图 2 QS调控系统原理图 |
转录后水平调控是在mRNA水平上进行的调控,主要有小RNA(small ribonucleic acids,sRNAs)、Ri-boswitch和CRISPR系统。
2.1 基于sRNAs介导的表达调控sRNAs是重要的转录后调节因子,在枯草芽孢杆菌中目前预测和鉴定的sRNAs一共有108个[31],sRNAs通过与mRNA碱基互补配对可改变mRNA的二级结构并激活或抑制翻译,从调节方式上可分为顺式作用(trans-encoded)和反式作用(cis- encoded)。FsrA是枯草芽孢杆菌发现的第一个trans-encoded RNAs,其参与铁代谢相关的靶标mRNA,同时需要伴侣蛋白FbpA、FbpB和FbpC[32]的参与。SR1是枯草芽孢杆菌第一个双功能的sRNAs,一方面可以与靶标ahrC的mRNA碱基配对抑制翻译,还参与精氨酸的分解代谢[33];另一方面也可以充当编码肽SR1P的mRNA。SR1P与糖酵解酶GapA形成GapA/SR1P复合物并与RNase J1相互作用,从而促进RNase J1目标的降解[34]。
Cis-encoded RNAs一般存在毒素-抗毒素(To-xin-Antitoxin)系统中,毒素基因编码一个有毒性蛋白,抗毒素基因转录的sRNAs与毒素基因形成sRNAs-mRNA配对复合物,从而抑制翻译和影响mRNA的稳定性。Yang等[35]以GFP为报告基因,通过对Type I毒素-抗毒素bsrG/sr4进行优化,构建了MS-DOS(Modulation via the sRNA-dependent operation system)转录后调控表达系统,其调控机理如图 3所示,将MS-DOS系统应用于透明质酸(HA)合成过程中,发现通过下调参与磷酸戊糖径途、糖酵解途径和细胞壁多糖合成的9种基因,其抑制zwf、pfkA和galE基因表现了对HA合成明显的积极影响,基因pfkA的下调使得HA的最高产量达1.52 g/L,是原始菌株的1.6倍。编码6-磷酸果糖激酶的pfkA基因是糖酵解途径中的关键酶,pfkA失活会引起严重的生长缺陷[36]。应用MS-DOS,可以在任何指定的时间点通过添加IPTG抑制pfkA基因,以协调细胞生长和HA的合成,此处MS-DOS仅通过引入单个RNA sr4即可实现多个基因的同时精确抑制,这与其他报道的干扰系统完全不同。为进一步促进枯草芽孢杆菌的应用,Yang等[37]构建了新型、稳定的枯草芽孢杆菌食品级表达系统(TypeⅡ毒素-抗毒素系统ydcD/ydcE),该系统在不添加抗生素的条件下,菌株传代100后质粒仍然稳定存在,利用该系统将透明质酸合酶构建于HA合成途径中,同对照相比,获得了更大的生物量(OD600)和更高的HA产量0.6 g/L,与由抗生素维持的常用表达系统相比,该新型表达系统消除了代谢负担并促进了细胞生长。类似的txpA/ratA和bsrE/sr5毒素-抗毒素系统也通过影响mRNA降解的机制达到调控基因的目的[38]。
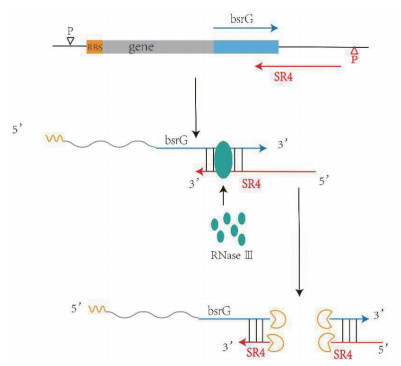
|
| 图 3 bsrG/sr4毒素-抗毒素系统原理图 |
Riboswitch一方面是通过对RBS序列遮蔽和暴露进行调控,枯草芽孢杆菌yjdF riboswitch[39]处于关闭状态,与RBS区域配对遮蔽RBS序列,导致翻译被抑制;其与配体结合后表达平台的茎环结构发生改变,RBS区域被暴露出来进而激活下游基因的表达。许多细菌的mRNA翻译起始取决于同核糖体结合的RBS与起始密码子的距离[40],Suess等[41]创建了一种新型的茶碱核糖开关,通过在距离RBS不同位置处插入茎环结构,该元件可干扰核糖体结合并能将其位置可逆地移动1 nt,配体茶碱结合后诱导结构转变,并调整合适的核糖体结合位点。
Riboswitch还可以通过对mRNA的稳定性进行基因的表达调控,通常与胞内代谢物的浓度相关。枯草芽孢杆菌glmS 核酶riboswitch[42],如图 4所示,其与代谢物6-磷酸氨基葡萄糖(GlcN6P)发生响应,当细胞中过量积累GlcN6P时glmS核酶将被激活,在5'位点附近发生自催化特异性裂解,从而导致glmS mRNA不稳定和表达量降低。Klein等[43]证实了在glmS核酶第40位发现了重要的鸟嘌呤(G)位点,将其突变为腺嘌呤(A)后消除了glmS核酶的催化作用。Winkler等[44]进一步对glmS核酶进行了一系列突变,其中将裂解位点AG突变为CC的M9突变体显示了20倍以上的β-半乳糖苷酶活性,表明glmS核酶活性被抑制。根据这一原理,Niu等[45]设计了GlcN6P响应性glmS核酶开关,动态控制GlcN6P供应和消耗所涉及的pgi、pfkA和glmM基因的表达,使得GlcNAc的产量可达16.26 g/L,形成了GlcNAc合成途径、肽聚糖合成途径(PSP)、糖酵解途径(EMP)和磷酸戊糖途径(HMP)之间的代谢平衡,细胞内GlcN6P浓度的水平影响代谢途径中关键酶的活性调节,其可以形成反馈路径,动态的平衡细胞生长和GlcNAc的合成,减少了副产物乙偶姻的产生。这一glmS核糖开关的调控机制在酿酒酵母和大肠杆菌中也普遍进行了研究。

|
| 图 4 glms mRNA的失稳机制 |
CRISPR系统由核酸内切酶Cas9和sgRNA(single-guide RNA)两部分组成,这个sgRNA可以帮助Cas9定位到目标序列,从而对DNA序列进行定点切割,PAM基序(Proto-spacer adjacent motifs)有利于识别目的序列的位点,位于切割序列的下游。CRISPR系统调控可分为CRISPRi(CRISPR inhibition)和CRISPRa(CRISPR activator)[46],如图 5所示,Cas9的D10A和H840A位点突变形成失活的dCas9(deactivated Cas9),该突变体缺乏核酸内切酶活性,保留了结合DNA的活性[47],将dCas9定位到靶序列会阻碍基因的转录,而与激活因子结合后可以募集RNA聚合酶(RNA polymerase,RNAP),促进基因的转录。
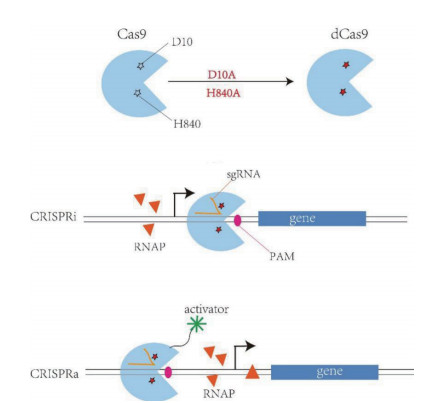
|
| 图 5 CRISPRi和CRISPRa调控机理 |
Wang等[48]为了提高枯草芽孢杆菌中表面活性素的产生,使用CRISPRi技术分别抑制了氨基酸生物合成分支代谢途径上的20个基因,由于细胞会将能量从生长转移到生产,因此高生产率可能会降低细胞生长,然而,其中对单基因yrpC、racE的抑制表现了更高的细胞生物量和较高的表面活性素产量,因为其具有高的底物利用速率。在枯草芽孢杆菌中,由Pxyl启动子介导的dCas9表达比其他启动子具有更高的抑制效率[49],Wu等[50]开发了一种基于木糖诱导的枯草芽孢杆菌CRISPRi系统,当抑制PSP中的glmM时,较高的碳通量被导向EMP,对EMP中pfkA基因抑制可以缓解碳溢出,基因zwf表达被抑制,其利用葡萄糖量减少而木糖增多,说明zwf的缺失抑制了葡萄糖流入HMP,导致大量碳通量导向EMP,同时对PSP、EMP和HMP这三个途径进行抑制,可以减少葡萄糖分解代谢并促进木糖的利用,使GlcNAc产量提高了13.2%。Westbrook等[51]通过CRISPRi减少pfkA和zwf的表达从而减少中间代谢碳的流失,可以将碳通量分别从EMP和HMP途径重新定向到细胞壁的生物合成,并通过适当调节pfkA和zwf的抑制强度可以显著提高HA的产生,而不会影响HA的分子量,同时对细胞生理的影响较小。研究发现RNAP的ω亚基与启动子上游区域的dCas9融合可以实现基因的转录激活(CRISPRa)[52],最近,Lu等[53]首次在枯草芽孢杆菌中应用CRISPRa系统,dCas9-α/ω介导的转录调控高度依赖靶位,因此可以通过设计特定的sgRNA来同时激活和抑制不同基因的表达,结合dCas9-ω介导的转录和新型启动子工程OAPS(Oligonucleotide annealing based promoter shuffling),枯草芽孢杆菌中淀粉酶的产量增加了260倍。
3 总结与展望枯草芽孢杆菌的动态调控工具可以有效的平衡代谢产物的合成和细胞的自身生长,从而维持代谢网络的稳定,避免了细胞受损、中间代谢物积累以及产量低等问题。枯草芽孢杆菌中的动态调控可以通过诱导物的添加、QS系统的响应影响基因的转录,也可以利用sRNAs、Riboswitch和CRISPR调控基因的翻译水平。
然而目前枯草芽孢杆菌的动态调控工具仍然缺乏,且天然的sRNAs、QS系统、Riboswitch代谢因子响应范围较窄、调控元件数量不多、调控机理不清晰,而基于对细胞代谢全局调控需求的增加,开发更多的调控工具去维持枯草芽孢杆菌的动态平衡是有必要的。一方面可以进一步鉴定枯草芽孢杆菌自身代谢物响应的转录因子、Riboswitch、sRNAs等有关遗传调控元件,挖掘更多的枯草芽孢杆菌自身调控机制,明确产物合成的磷酸戊糖径途、糖酵解途径、碳通量流向等代谢网络信息,从而赋予枯草芽孢杆菌更好的动态调控能力;另一方面基于不断发展的系统生物学和合成生物学技术,可以定向设计目标启动子、分析特定的核糖体结合位点、改造sRNAs的功能结构等组合控制基因表达。加之高通量技术的应用,更多潜在的调控元件将会被更加高效地筛选出来,大量遗传调控元件被开发,大大加快了微生物细胞工厂的构建。随着生物技术的发展,枯草芽孢杆菌底盘细胞的潜力逐渐被呈现出来,其底盘细胞是通过基因组最小化、碳分解代谢物阻遏去除、细胞膜工程和蛋白质分泌途径工程获得的,将动态调控工具与人工底盘细胞结合应用可以更加针对性的对目标途径进行动态调控及机制解析。
| [1] |
Kang Z, Yang S, Du G, et al. Molecular engineering of secretory machinery components for high-level secretion of proteins in Bacillus species[J]. Journal of Industrial Microbiology & Biotechnology, 2014, 41(11): 1599-1607. |
| [2] |
Contesini FJ, Melo RR, Sato HH. An overview of Bacillus proteases:from production to application[J]. Critical Reviews in Biotechnology, 2018, 38(3): 321-334. DOI:10.1080/07388551.2017.1354354 |
| [3] |
van Dijl JM, Hecker M. Bacillus subtilis:from soil bacterium to super-secreting cell factory[J]. Microbial Cell Factories, 2013, 12: 3. DOI:10.1186/1475-2859-12-3 |
| [4] |
Liu Y, Liu L, Li J, et al. Synthetic biology toolbox and chassis devel-opment in Bacillus subtilis[J]. Trends in Biotechnology, 2019, 37(5): 548-562. DOI:10.1016/j.tibtech.2018.10.005 |
| [5] |
Wu G, Yan Q, Jones JA, et al. Metabolic burden:Cornerstones in synthetic biology and metabolic engineering applications[J]. Trends in Biotechnology, 2016, 34(8): 652-664. DOI:10.1016/j.tibtech.2016.02.010 |
| [6] |
Colletti PF, Goyal Y, Varman AM, et al. Evaluating factors that influence microbial synthesis yields by linear regression with numerical and ordinal variables[J]. Biotechnology and Bioengineering, 2011, 108(4): 893-901. DOI:10.1002/bit.22996 |
| [7] |
Liu Y, Li J, Du G, et al. Metabolic engineering of Bacillus subtilis fueled by systems biology:Recent advances and future directions[J]. Biotechnology Advances, 2017, 35(1): 20-30. DOI:10.1016/j.biotechadv.2016.11.003 |
| [8] |
Promchai R, Promdonkoy B, Tanapongpipat S, et al. A novel salt-inducible vector for efficient expression and secretion of heterologous proteins in Bacillus subtilis[J]. Journal of Biotechnology, 2016, 222: 86-93. DOI:10.1016/j.jbiotec.2016.02.019 |
| [9] |
Jiao S, Li X, Yu H, et al. In situ enhancement of surfactin biosynthesis in Bacillus subtilis using novel artificial inducible promoters[J]. Biotechnology and Bioengineering, 2017, 114(4): 832-842. DOI:10.1002/bit.26197 |
| [10] |
Zhou K, Zou R, Zhang C, et al. Optimization of amorphadiene synthesis in Bacillus subtilis via transcriptional, translational, and media modulation[J]. Biotechnology and Bioengineering, 2013, 110(9): 2556-2561. DOI:10.1002/bit.24900 |
| [11] |
Toymentseva AA, Schrecke K, Sharipova MR, et al. The LIKE system, a novel protein expression toolbox for Bacillus subtilis based on the liaI promoter[J]. Microbial Cell Factories, 2012, 11: 143. DOI:10.1186/1475-2859-11-143 |
| [12] |
Mierau I, Kleerebezem M. 10 years of the nisin-controlled gene expression system(NICE)in Lactococcus lactis[J]. Applied Microbiology and Biotechnology, 2005, 68(6): 705-717. DOI:10.1007/s00253-005-0107-6 |
| [13] |
Bongers RS, Veening JW, Van Wieringen M, et al. Development and characterization of a subtilin-regulated expression system in Bacillus subtilis:strict control of gene expression by addition of subtilin[J]. Applied and Environmental Microbiology, 2005, 71(12): 8818-8824. DOI:10.1128/AEM.71.12.8818-8824.2005 |
| [14] |
Welsch N, Homuth G, Schweder T. Stepwise optimization of a low-temperature Bacillus subtilis expression system for "difficult to express" proteins[J]. Applied Microbiology and Biotechnology, 2015, 99(15): 6363-6376. DOI:10.1007/s00253-015-6552-y |
| [15] |
Li W, Li HX, Ji SY, et al. Characterization of two temperature-inducible promoters newly isolated from B. subtilis[J]. Biochemical and Biophysical Research Communications, 2007, 358(4): 1148-1153. DOI:10.1016/j.bbrc.2007.05.064 |
| [16] |
Yang S, Du G, Chen J, et al. Characterization and application of endogenous phase-dependent promoters in Bacillus subtilis[J]. Applied Microbiology and Biotechnology, 2017, 101(10): 4151-4161. DOI:10.1007/s00253-017-8142-7 |
| [17] |
Roy S, Hennelly SP, Lammert H, et al. Magnesium controls aptamer-expression platform switching in the SAM-I riboswitch[J]. Nucleic Acids Research, 2019, 47(6): 3158-3170. DOI:10.1093/nar/gky1311 |
| [18] |
Sun Y, Wang Y, Tan ZJ, et al. Regulation mechanism of lysC riboswitch in gram-positive bacterium Bacillus subtilis[J]. Journal of Biomolecular Structure & Dynamics, 2019. DOI:10.1080/07391102.2019 |
| [19] |
Gong S, Wang Y, Wang Z, et al. Co-transcriptional folding and regulation mechanisms of riboswitches[J]. Molecules, 2017, 22(7): pii:E1169. DOI:10.3390/molecules22071169 |
| [20] |
Marcano-Velazquez JG, Batey RT. Structure-guided mutational analysis of gene regulation by the Bacillus subtilis pbuE adenine-responsive riboswitch in a cellular context[J]. Journal of Biological Chemistry, 2015, 290(7): 4464-4475. DOI:10.1074/jbc.M114.613497 |
| [21] |
Babina AM, Lea NE, Meyer MM. In vivo behavior of the tandem glycine riboswitch in Bacillus subtilis[J]. American Society for Microbiology, 2017, 8(5): pii:e01602-17. |
| [22] |
Phan TT, Schumann W. Development of a glycine-inducible expression system for Bacillus subtilis[J]. Journal of Biotechnology, 2007, 128(3): 486-499. DOI:10.1016/j.jbiotec.2006.12.007 |
| [23] |
Bai AJ, Rai VR. Bacterial quorum sensing and food industry[J]. Comprehensive Reviews in Food Science and Food Safety, 2011, 10(3): 183-193. DOI:10.1111/j.1541-4337.2011.00150.x |
| [24] |
Papenfort K, Bassler BL. Quorum sensing signal-response systems in Gram-negative bacteria[J]. Nature Reviews Microbiology, 2016, 14(9): 576-588. DOI:10.1038/nrmicro.2016.89 |
| [25] |
Banerjee G, Ray AK. Quorum-sensing network-associated gene regulation in Gram-positive bacteria[J]. Acta Microbiologica et Immunologica Hungarica, 2017, 64(4): 439-453. DOI:10.1556/030.64.2017.040 |
| [26] |
Smits WK, Bongiorni C, Veening JW, et al. Temporal separation of distinct differentiation pathways by a dual specificity Rap-Phr system in Bacillus subtilis[J]. Molecular Microbiology, 2007, 65(1): 103-120. DOI:10.1111/j.1365-2958.2007.05776.x |
| [27] |
Koetje EJ. A plasmid-borne Rap-Phr system of Bacillus subtilis can mediate cell-density controlled production of extracellular proteases[J]. Microbiology, 2003, 149(1): 19-28. DOI:10.1099/mic.0.25737-0 |
| [28] |
Cui S, Lv X, Wu Y, et al. Engineering a bifunctional Phr60-Rap60-Spo0A quorum-sensing molecular switch for dynamic fine-tuning of menaquinone-7 synthesis in Bacillus subtilis[J]. ACS Synthetic Biology, 2019, 8(8): 1826-1837. DOI:10.1021/acssynbio.9b00140 |
| [29] |
Boguslawski KM, Hill PA, Griffith KL. Novel mechanisms of controlling the activities of the transcription factors Spo0A and ComA by the plasmid-encoded quorum sensing regulators Rap60-Phr60 in Bacillus subtilis[J]. Molecular Microbiology, 2015, 96(2): 325-348. DOI:10.1111/mmi.12939 |
| [30] |
Wolf D, Rippa V, Mobarec JC, et al. The quorum-sensing regulator ComA from Bacillus subtilis activates transcription using topologically distinct DNA motifs[J]. Nucleic Acids Research, 2016, 44(5): 2160-2172. DOI:10.1093/nar/gkv1242 |
| [31] |
Brantl S, Bruckner R. Small regulatory RNAs from low-GC Gram-positive bacteria[J]. RNA Biology, 2014, 11(5): 443-456. DOI:10.4161/rna.28036 |
| [32] |
Gaballa A, Antelmann H, Aguilar C, et al. The Bacillus subtilis iron-sparing response is mediated by a Fur-regulated small RNA and three small, basic proteins[J]. Proceedings of The National Academy of Sciences of the United States of America, 2008, 105(33): 11927-11932. DOI:10.1073/pnas.0711752105 |
| [33] |
Heidrich N, Chinali A, Gerth U, et al. The small untranslated RNA SR1 from the Bacillus subtilis genome is involved in the regulation of arginine catabolism[J]. Molecular Microbiology, 2006, 62(2): 520-536. DOI:10.1111/j.1365-2958.2006.05384.x |
| [34] |
Gimpel M, Maiwald C, Wiedemann C, et al. Characterization of the interaction between the small RNA-encoded peptide SR1P and GapA from Bacillus subtilis[J]. Microbiology, 2017, 163(8): 1248-1259. DOI:10.1099/mic.0.000505 |
| [35] |
Yang S, Wang Y, Wei C, et al. A new sRNA-mediated posttranscriptional regulation system for Bacillus subtilis[J]. Biotechnology and Bioengineering, 2018, 115(12): 2986-2995. DOI:10.1002/bit.26833 |
| [36] |
Munoz-Marquez ME, Ponce-Rivas E. Effect of pfkA chromosomal interruption on growth, sporulation, and production of organic acids in Bacillus subtilis[J]. J Basic Microbiol, 2010, 50(3): 232-240. DOI:10.1002/jobm.200900236 |
| [37] |
Yang S, Kang Z, Cao W, et al. Construction of a novel, stable, food-grade expression system by engineering the endogenous toxin-antitoxin system in Bacillus subtilis[J]. Journal of Biotechnology, 2016, 219: 40-47. DOI:10.1016/j.jbiotec.2015.12.029 |
| [38] |
Meissner C, Jahn N, Brantl S. In vitro Characterization of the type I toxin-antitoxin system bsrE/SR5 from Bacillus subtilis[J]. Journal of Biological Chemistry, 2016, 291(2): 560-571. DOI:10.1074/jbc.M115.697524 |
| [39] |
Gong S, Wang Y, Wang Z, et al. Genetic regulation mechanism of the yjdF riboswitch[J]. Journal of Theoretical Biology, 2018, 439: 152-159. DOI:10.1016/j.jtbi.2017.12.007 |
| [40] |
Kozak M. Regulation of translation via mRNA structure in prokaryotes and eukaryotes[J]. Gene, 2005, 361: 13-37. DOI:10.1016/j.gene.2005.06.037 |
| [41] |
Suess B, Fink B, Berens C, et al. A theophylline responsive riboswitch based on helix slipping controls gene expression in vivo[J]. Nucleic Acids Research, 2004, 32(4): 1610-1614. DOI:10.1093/nar/gkh321 |
| [42] |
Collins JA, Irnov I, Baker S, et al. Mechanism of mRNA destabilization by the glmS ribozyme[J]. Genes & Development, 2007, 21(24): 3356-3368. |
| [43] |
Klein DJ, Been MD, Ferre-D'Amare AR. Essential role of an active-site guanine in glmS ribozyme catalysis[J]. Journal of The American Chemical Society, 2007, 129(48): 14858-14859. DOI:10.1021/ja0768441 |
| [44] |
Winkler WC, Nahvi A, Roth A, et al. Control of gene expression by a natural metabolite-responsive ribozyme[J]. Nature, 2004, 428(6980): 281-286. DOI:10.1038/nature02362 |
| [45] |
Niu T, Liu Y, Li J, et al. Engineering a glucosamine-6-phosphate responsive glmS ribozyme switch enables dynamic control of metabolic flux in Bacillus subtilis for overproduction of N-Acetylglucosamine[J]. ACS Synthetic Biology, 2018, 7(10): 2423-2435. DOI:10.1021/acssynbio.8b00196 |
| [46] |
Cho S, Shin J, Cho BK. Applications of CRISPR/Cas system to bacterial metabolic engineering[J]. International Journal of Molecular Sciences, 2018, 19(4): pii:E1089. DOI:10.3390/ijms19041089 |
| [47] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [48] |
Wang C, Cao Y, Wang Y, et al. Enhancing surfactin production by using systematic CRISPRi repression to screen amino acid biosynthesis genes in Bacillus subtilis[J]. Microbial Cell Factories, 2019, 18(1): 90. DOI:10.1186/s12934-019-1139-4 |
| [49] |
Peters JM, Colavin A, Shi H, et al. A comprehensive, CRISPR-based functional analysis of essential genes in bacteria[J]. Cell, 2016, 165(6): 1493-1506. DOI:10.1016/j.cell.2016.05.003 |
| [50] |
Wu Y, Chen T, Liu Y, et al. CRISPRi allows optimal temporal control of N-acetylglucosamine bioproduction by a dynamic coordination of glucose and xylose metabolism in Bacillus subtilis[J]. Metabolic Engineering, 2018, 49: 232-241. DOI:10.1016/j.ymben.2018.08.012 |
| [51] |
Westbrook AW, Ren X, Oh J, et al. Metabolic engineering to enhance heterologous production of hyaluronic acid in Bacillus subtilis[J]. Metabolic Engineering, 2018, 47: 401-413. DOI:10.1016/j.ymben.2018.04.016 |
| [52] |
Bikard D, Jiang W, Samai P, et al. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system[J]. Nucleic Acids Research, 2013, 41(15): 7429-7437. DOI:10.1093/nar/gkt520 |
| [53] |
Lu Z, Yang S, Yuan X, et al. CRISPR-assisted multi-dimensional regulation for fine-tuning gene expression in Bacillus subtilis[J]. Nucleic Acids Research, 2019, 47(7): e40. DOI:10.1093/nar/gkz072 |




