2. 农业部农业转基因生物安全评价(食用)重点实验室,北京 100083
2. Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety), Ministry of Agriculture, Beijing 100083
生物传感器(Biosensors)是将生物活性物质和物理化学检测器相结合的设备,由固定化的生物敏感材料作为识别元件对生物活性物质进行特异性的识别,然后将生化信号转换为物理化学信号,再将信号进行输出的一类可以用于测定各种生物物质及浓度的设备。至今已有50多年的发展历史。生物传感器根据其识别元件即敏感元件的不同,可分为7类:酶传感器、微生物传感器、细胞传感器、组织传感器、免疫传感器、核酸传感器及分子印迹生物传感器[1]。酶传感器是最早出现的生物传感器,因为具有高灵敏度和高特异性,并且操作简单,反应温度一般在30-60℃之间,因此在生化分析及临床应用中得到了较大的发展。
FEN1酶是一种结构特异性核酸酶,只识别特异性的结构而不依赖序列,并且有着高效稳定的切割效率,已被广泛应用于生物传感器中靶物质的信号循环放大过程中。在FEN1酶所介导的生物传感器中,对三碱基重叠结构进行识别,再通过搭载不同的信号循环和信号放大方式,可实现对靶物质的精准检测。本文首先对FEN1酶的发现过程、性质及作用进行介绍,再根据靶物质的不同对FEN1酶所介导的生物传感器进行分类综述。此外,还对FEN1酶在体内的成像和治疗进行了介绍,旨在使人们更多的了解FEN1酶在生物传感器上的应用及发展现状,为今后所用。
1 FEN1酶的简介 1.1 FEN1酶的发现瓣状核酸内切酶-1(Flap endonuclease 1,FEN1)是1994年Harrington和Libber[2]根据底物的形状特征进行命名的结构特异性核酸酶。其发现可以追溯到1968年,首先由Klett等[3]发现大肠杆菌聚合酶具有5'-3'外切酶活性;次年,Kelly等[4]发现大肠杆菌聚合酶同时具有内切酶活性,并且可以切除由于辐射损伤造成的多聚胸腺区域的错配序列;同年,Lindahl等[5]从兔子组织中分离得到一种能降解双链DNA的外切酶DNase Ⅳ,分子量约为42 000,并且DNase Ⅳ降解合成多核苷酸的速度远快于天然多核苷酸DNA,与大肠杆菌聚合酶有着共同特性;1971年,Setlow等[6]报道大肠杆菌DNA聚合酶在体外可被水解成两个酶活性结构域,较大片段(分子量为76 kD)具有聚合酶和3'-5'外切酶活性,较小片段(分子量为36 kD)具有5'-3'核酸外切酶活性;Masamune和Richardson[7]推测大肠杆菌聚合酶在DNA单链断裂,合成新的序列过程中会产生大量的DNA分枝;到1982年,Lundquist和Olivera[8]发现大肠杆菌聚合酶的外切酶活性中最理想的底物为分枝结构单链。
1.2 FEN1酶的性质FEN1酶是一种金属核酸酶,需要二价阳离子镁(Mg2+)和锰(Mn2+)的参与才能执行功能。在Mg2+浓度为1 mmol/L-10 mmol/L时,FEN1酶可以发挥最佳的切割能力[2]。除此之外,FEN1酶的切割效率会受到盐离子的影响,如当氯化钠浓度为50 mmol/L时,FEN1酶的切割效率受到大大抑制[2]。FEN1酶在25-85℃具有切割活性,哺乳动物FEN1酶在37℃下活性最强[9]。
FEN1酶具有5'分枝结构内切酶活性(Flap endonucleases,FEN)、缺口依赖性核酸内切酶活性(Gap endonuclease,GEN)和5'外切酶活性(Exonuclease,EXO)。
FEN1酶5'分枝结构内切酶活性(FEN)的最佳底物结构(图 1)为双分枝DNA双链结构(Double Flap),并且FEN1上有一疏水口袋恰好容纳3'分枝结构[10]。分枝结构核苷酸在20个以内可以被FEN1酶快速有效剪切,但超过20个核苷酸时,FEN1酶的剪切活性迅速降低,并且分枝结构上结合了DNA双链或者大分子结构也会抑制FEN1酶的剪切活性[11]。

|
| 图 1 分枝结构内切酶(FEN1)活性底物 |
FEN1酶缺口依赖性内切酶活性底物结构(图 2)包括Y型DNA结构、气泡状结构(Bubble)等。其活性相较于分枝结构内切酶活性较低[12]。

|
| 图 2 缺口依赖性内切酶(GEN)活性底物 |
FEN1酶核酸外切酶活性底物结构(图 3)包括缺刻结构(Nick)、缺口结构(Gap)、平末端双链DNA(Blunt)、霍利迪连接体(Holiday junction)等结构[12]。
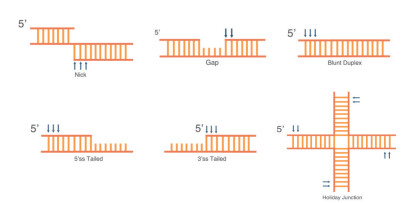
|
| 图 3 核酸外切酶活性(EXO)底物 |
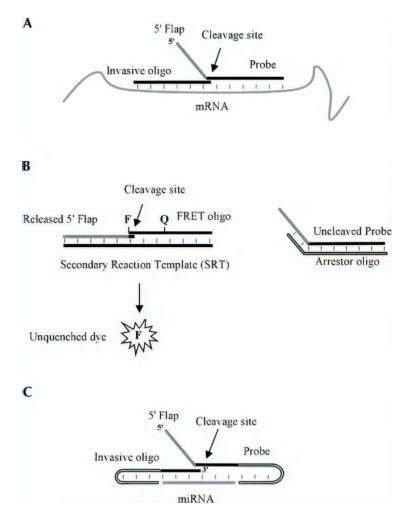
FEN1酶在体外行使其活性使DNA双链断裂的过程[12]:首先FEN1酶识别5'Flap片段并对其切割产生刻痕,当DNA双链上出现缺口时,FEN1酶可以从5'端移除一些核苷酸,然后在模板链上裂解,使最终得双链DNA断裂(图 4)。
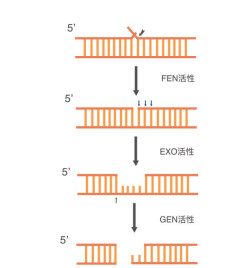
|
| 图 4 FEN1酶体外切割DNA双链过程 |
FEN1酶在生物细胞的DNA代谢中扮演重要角色,参与DNA复制过程和损伤修复过程,不仅如此,FEN1酶在多种肿瘤组织中过度表达,并且通过不同的调控机制促进肿瘤的发展及耐药。
FEN1酶在DNA复制过程中主要参与冈崎片段的RNA引物的过程降解,主要有两种切除方式。第一种方式为在RNA酶H(RNase H)切除大部分的RNA引物后,在DNA-RNA交汇处留下一个RNA残基,此时FEN1酶行使外切酶活性将其切除。第二种方式是RNA引物发生移位,形成一个5'Flap结构,此时FEN1酶行使分枝结构内切酶活性将其切除[13-15]。
DNA损伤可根据来源分为两类,一类源于体内代谢产物的损伤[2];另一类为体外环境及某些分子化合物的攻击。而DNA损伤的修复通路有光复活修复、碱基切除修复、重组修复以及SOS紧急修复等。FEN1酶目前阐述最清楚的是碱基切除修复过程。
FEN1酶多态性改变和基因突变与肿瘤的发生相关。研究发现多肽位点69G→A和4150G→T与肺癌、乳腺癌及消化道肿瘤有关,并且这两种多肽位点的改变与FEN1mRNA表达下降有关[16-17],因此这两种多肽位点的改变可以用做癌症的检测。而改变FEN1酶和Mg2+的结合位点,使得突变频繁发生,无法进行外源性DNA损伤修复,使得小鼠患慢性炎症和癌症[18]。
FEN1酶的表达改变也与肿瘤的发生相关。当单倍体量不足时,FEN1酶的活性降低,促进肿瘤的发生发展。Singh等[19]发现FEN1在乳腺癌、子宫癌、肾癌、卵巢癌及结肠癌组织中表达升高。研究发现FEN1蛋白水平几乎在所有肿瘤中都有所增加,并且FEN1蛋白在大部分神经胶质细胞瘤和星形胶质细胞瘤中表达也升高。
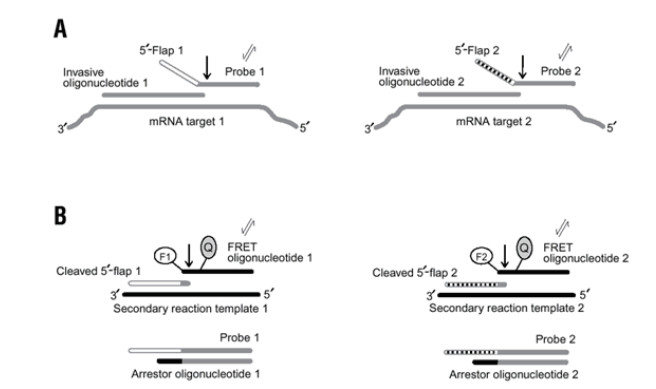
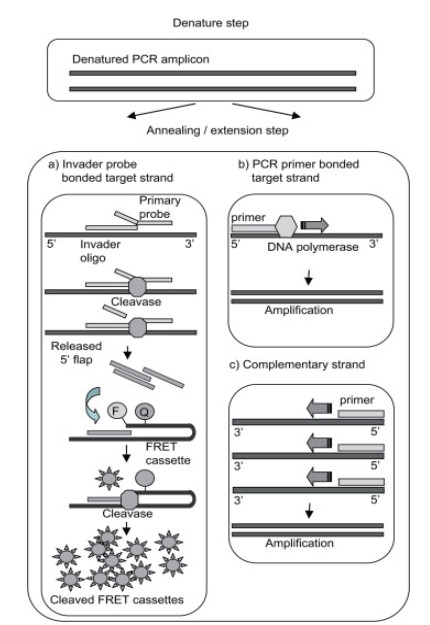
2 基于不同物质与核酸作用的FEN1酶生物传感器FEN1酶所介导的生物传感器多采用核酸侵入反应(Invader assay)来进行核酸的信号放大。该方法的检测原理是基于FEN1酶所具有的结构特异性对错配的序列的高度敏感性。反应过程为FEN1酶识别寡核苷酸与目标DNA形成三碱基的重叠结构,并切割相应的寡核苷酸,将产生的Flaps片段作为识别信号,再通过二次裂解荧光共振转移发卡探针(FRET probe)等方法进行信号放大检测(图 5)[20]。
2.1 单核苷酸多态性检测单核苷酸多态性(Single nucleotide polymorph-ism,SNP)是人类基因组DNA序列中最常见的变异形式,指在基因组水平上由单个核苷酸变异引起的DNA序列多态性,包括置换、颠换、缺失和插入。在过去的几年中,许多大规模的工作已经确定了大量的SNP,并且到目前为止,用于这些多态性的公共存储库db- SNP(http://www.ncbi.nlm.nih.gov/SNP)包含了大约1 000万个SNP的信息[21]。
Hall等[22]将两种侵入性裂解反应结合成单一均匀试验的分析方法,解决了初始侵入者分析方法的困难。如图 6所示,这种连续入侵信号放大反应(Serial invasive signal amplification reaction,SISAR)为目前使用的大多数入侵检测方法奠定了基础。该方法最初应用于单核苷酸反应,要求在单独的反应中研究每个SNP等位基因。随后,使用两个单独的FRET盒式寡核苷酸将该实验转化为单管双重检验,可以得到两个SNP等位基因的荧光信号。
2.2 甲基化检测现代DNA甲基化作为最早发现的修饰途径之一,是指由S-腺苷甲硫氨酸作为甲基供体,DNA甲基转移酶(DNA methyltransferase,DNMT)将其胞嘧啶催化为5-甲基胞嘧啶的过程[23]。
2012年,临床化学上刊登了对粪便中结直肠癌相关甲基化基因的检测方法(图 7),QuARTS法(Quantitative allele-specific real-time target andsignal amplification),该方法将实时荧光PCR与核酸侵入信号扩增反应相结合,利用核酸侵入反应序列识别的高特异性和PCR扩增的高灵敏性提高了检测的特异性。该方法分为3个部分,首先先对靶标进行扩增,随后利用FEN1酶的对所形成三碱基结构进行切割释放Flaps片段,最后再通过荧光能量共振转移的方法获得信号。QuARTS法检测甲基化标记物可检测出大多数大肠癌和腺瘤,特异性阈值高达95%[24]。
2.3 基因型检测现代医学研究证明,人类疾病都直接或间接地与基因有关,利用P53基因来诊断癌症等都表明检测基因型对现代医学的重要性。
FEN1酶所介导的核酸生物传感器是一种快速、精确的对基因型进行检测的方法,具有巨大的医疗价值。为降低FRET的检测成本,促进其更好的被利用,可以采用以下两种方法对其进行替代:第1种方法是采用检测偏振光的变化[25],荧光分子可以被平面偏振光激发产生荧光偏振(Fluorescence polarization FP),而荧光偏振依赖于染料分子的分子量。Hsu等[25]在使用FP进行检测中使用两个不同荧光体的信号探针,荧光素和羧基四甲基罗丹明(Carboxytetramethylrhodamine TAMRA)。因此,可以在一个反应中测定每个样品,从而节省时间和减少基因型杂合。第2种方法是使用基质辅助激光解吸电离飞行时间质谱(Matrix-assisted laser desorption/ ionization time of flight mass spectrometry,MALDI-TOF MS)的方式进行替代[26]。MALDI-TOF质谱法可以对短DNA探针进行温和分析并且具有较高灵敏度的方法,同时飞行时间(TOF)探测器提供了很好的分辨率。并且在该方法中,混合物的每一组分在质谱上几乎都有一个单峰。MALDI-TOF质谱法成为分析复杂混合物的一种选择方法,并且近年来,此技术已广泛应用于DNA、PNA和抗生素的定量分析中[27]。因此,采用MALDI-TOF MS检测反应中产生的Flap分子,可对多个基因同时分型检测。
不仅如此,美国Third Wave Technologies公司(Madison,WI)开发了一种新的基于微滴度板的测定方法[28],称为InvaderTM,可用于基因型的检测。该方法将核酸侵入反应与和使用微滴度板相结合,在发生裂解之后可以采用微滴度板检测荧光标记的产品,基因型由净野生型/突变信号比决定。2000年,Hessner等[29]对1 369个个体进行导致活化蛋白C(Activated protein C,APC)耐药的突变因子(Factor V leiden,FVL)进行基因分型,分型结果与样本结果一致,因此此方法被认为是一种对FVL进行基因分型的快速、可靠的方法。
2.4 RNA检测RNA定量在医学研究上发挥越来越重要的作用。例如,病毒RNA的定量可以预测疾病的进展和治疗效果,RNA几乎密切参与所有的细胞生理机制,随着人类基因组计划的重点转向基因表达分析,该领域将需要一种灵活的RNA分析技术,能够定量检测多种形式的交替转录或处理过的RNA。
2001年,Eis等[30]利用核酸侵入原理来对总RNA和细胞裂解样品中的RNA进行检测。利用此种方法可以对高度同源的RNA进行检测,反应分为两部分。在初级反应中(图 8-A),侵入性寡核苷酸和探针特异性地与它们各自的RNA靶标形成重叠结构,随后进行切割、退火。初级反应完成后,裂解的5'-Flap然后在第二反应中充当侵入性寡核苷酸(图 8-B),与二级反应模板(Secondary-reaction template,SRT)结合,所形成的5'-Flap-SRT复合物切割多个FRET脱氧寡核苷酸时产生荧光信号。
RNA侵入性切割测定的形式与用于基因组DNA检测的主要有以下几点不同。首先,RNA侵入切割测定需要两个寡核苷酸(FRET和STR),DNA检测中只需要一种寡核苷酸。其次,因为RNA侵入切割测定是一种依赖于靶物质和时间的线性扩增信号,所以在测定过程中要注意两个反应的顺序操作,以防止形成X-结构。而对基因组DNA的检测是两个反应同时进行,并将信号放大作为目标水平[22]。
MicroRNA(miRNA)是一类小的内源性非编码RNA分子,大概由21-25个核苷酸组成,可在转录后水平调节基因表达。近年来研究表明,miRNA参与包括发育,细胞增殖,细胞凋亡,脂肪代谢,激素分泌和肿瘤发展等过程[31]。所以可以通过开发研究期新方法,促进人们对miRNA的理解。
Allawi等[32]及其同事对核酸侵入反应进行改进,新开发出一种用于miRNA敏感性和特异性的方法,命名为Invader miRNA。该方法是对先前设计的Invader mRNA的扩展应用[30, 33]。在Invader mRNA中使用Cleavase酶对形成的三碱基重叠结构进行剪切并释放5'Flap片段,而Cleavase酶是FEN1酶家族中的一员[34]。而miRNA因为其较小所以需要对入侵结构和探针进行一些修饰。设计入侵结构和探针在5'和3'末端含有自身互补序列(图 9-C),使miRNA靶特异性区域(Target specific region,TSR)的两侧都形成侧翼发夹结构。这些侧翼结构通过促进三碱基重叠结合的形成,以达到稳定miRNA靶序列与探针系统的作用,并且还提高Cleavase酶反应的效率。待Cleavase酶切割后,与mRNA测定一样,然后释放的5'Flap片段可以结合二级反应模板再次切割反应以产生荧光信号。(图 9-B)。
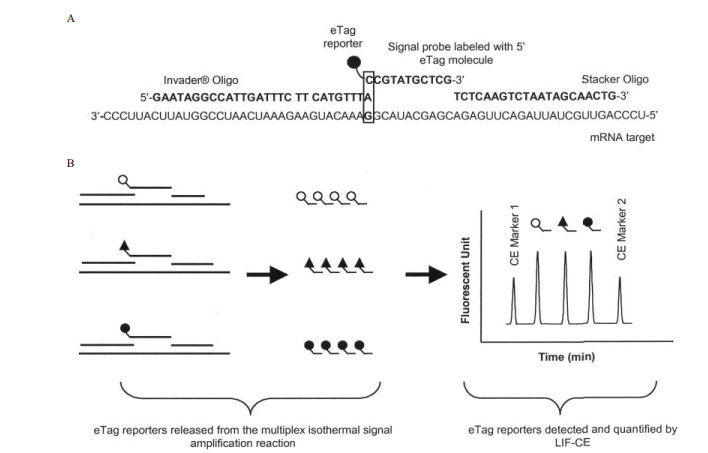
Chenna和Tian等[35]通过对Invader mRNA方法的进一步改进,使其达到多通量检测mRNA的目的。该方法描述了一种使用电泳标记(Electrophoretic tags,eTag)来编码和量化mRNA目标的多路复用方法,eTag是一种小荧光分子库,它们具有相同的荧光性质,但又具有不同的电荷质量比,通过激光诱导荧光(Laser-induced fluorescence,LIF)作用后产生预定义的电泳特性。首先将两个目标特异性的探针与mRNA三碱基重叠结构,信号探针在其5'末端的碱基处连接一个eTag分子(图 10-A)。然后利用Cleavase酶将5'Flap片段与eTag分子一起裂解,释放,再通过激发诱导荧光以及毛细管电泳(Capillary electrophoresis,CE)实现对mRNA的检测(图 10-B)。多路复用是通过将不同的eTag分子分配给多个目标特异性信号探针来实现的。采用LIF结合CE的方式大大提高了反应的灵敏度,并且因为CE的检测限(Limit of detection,LOD)一般在亚皮摩尔范围,因此只需进行一次Invader mRNA便可以直接测定粗裂解液中的mRNA,在eTag多重检测mRNA系统中实现了前所未有的基因表达简化分析。
2.5 病毒检测丙型肝炎病毒(Hepatitis C virus,HCV)感染是一个全球性的公共卫生问题,据世界卫生组织统计,全球HCV的感染率约为3%,每年约有3.5万新发HCV感染病例[36]。在我国,HCV感染病例约2 000万例,HCV感染在我国是继乙型肝炎、肺结核、痢疾之后第四大常见的感染性疾病[37]。HCV感染可能导致慢性肝炎,甚至发展成潜在的致命性肝硬化和肝细胞癌。因此,可以对HCV进行相应的检测。
2013年,Kenichi等[38]采用实时PCR检测核酸侵入反应(Q-Invader法)对HCV-1b核心区域中70/91位的突变进行检测。侵入反应对于检测基因组DNA或PCR产物中的单核苷酸差异具有较高的特异性,再通过实时荧光PCR可以获得高灵敏度的定量分析。通过对上述两个位置(70和91)的野生型/突变序列标记不同的荧光基团,对123名患者进行检测。结果显示Q-Invader法可以对突变体变体的检测低至总量的1%。
2018年,Kenichi Tadokoro团队[39]再次利用Q-Invader法与直接测序法比较测定了HCV感染中的Y93H突变。结果显示Q-Invader测定的灵敏度与用于HCV检测的直接测序分析的灵敏度相同,并且Q-Invader测定可以检测通过直接测序分析无法检测的次要菌株。因此,Q-Invader测定可以用于临床环境中对HCV患者的检测。
2.6 肿瘤检测大肠癌是消化道恶性肿瘤中常见的一种癌,发病率仅次于胃癌和食管癌,死亡率在恶性肿瘤中占第3-6位。大肠癌早期并没有明显症状,不易被发现,一旦发现便血等症状,病程已经达到中晚期。而手术效果与患病阶段有密切关系:早期发现并进行手术患者存活率可高达90%以上;晚期癌即使手术,存活率也不足10%。因此,早期发现,早期治疗是防治大肠癌的关键。
而大肠癌的发生发展与多个基因的甲基化密切相关,而核酸侵入反应偶联PCR定量检测DNA甲基化水平为大肠癌的检测提供了一种新思路。基因启动子区超甲基化造成骨形态发生蛋白3(Bone morphogenetic protein 3,BMP3)基因表达下调,是引发结直肠癌的重要机制之一[40-42]。NDRG4基因也与2014年被美国美国食品药品监督管理局(Food and drug administration,FDA)批准作为大肠癌粪便检测的其中一种靶标[42]。
严志进等[43]对QuARTS法进行了改进,通过△CT法[44]进行甲基化定量。反应中以NDRG4为目的基因,引入ACTB作为参比基因,对检测体系进行优化,使两基因的扩增效率均接近100%,通过测定目的基因与参比基因的CT值的差值,计算出甲基化基因的相对甲基化水平,以此来对甲基化进行定量。刘琳琳[40]等采用相同的核酸侵入反应偶联PCR定量检测DNA甲基化水平的方法对BMP3基因进行了定量检测(图 11)。该方法可以准确检测低至0. 01%的DNA甲基化。并且该方法避免了使用高浓度标准目的基因片段测定标准曲线,降低了污染风险,同时简化了实验操作,在低丰度甲基化基因的临床定量检测中有着巨大的应用前景。
肺癌是当今癌症死亡的主要原因,根据《中国肿瘤年报2015》的数据显示,我国每年肺癌发病78.1万例,死亡病例约62.6万[45]。非小型细胞肺癌(Non-small cell lung cancer,NSCLC)是肺癌中的主要类型,约占肺癌中的80%-85%,通过大规模的基因测序工程后发现,表皮生长因子受体(Epidermal growth factor receptor,EGFR)的突变与肺癌的发生发展有密切关系,EGFR突变已被报道为非小型细胞肺癌治疗中一个非常重要的因素[46]。
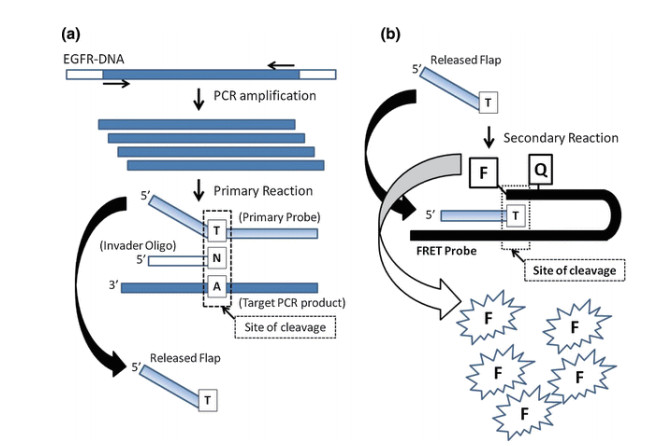
Naoki等[47]通过核酸侵入反应偶联PCR的过程对非小型细胞肺癌中的EGFR突变位点进行检测(图 12),可以检测稀释100-1 000倍的肺癌细胞系的已知EGFR突变,显现了较高的灵敏度,在临床检测上具有较高的应用价值。
2.7 微生物检测细菌(Bacteria)是生物的主要类群之一,也是所有生物中数量最多的一类。细菌对人类活动产生较大的影响,一方面,细菌是许多疾病的病原体:另一方面,人类也利用细菌进行酸奶的制造和部分抗生素的制造。实时PCR是最常用的细菌定量分析方法之一,但是不管是采用荧光探针或者插入荧光染料,都使得对细菌成本的检测较高。针对以上状况,Kenichi等[48]改进了对病毒进行对疱疹病毒的定量的Invader PLUS法,使其用于多种细菌类型,称为m-Invader PLUS(Modification of the invader PLUS)法(图 13)。反应过程中,PCR和核酸侵入反应同时进行,变性PCR产物结合侵入初级探针和延伸引物形成侵入性复合物,释放的5'-Flap(第一反应的产物)促进第二反应中通用FRET盒的裂解。随后对唾液样品进行的6种牙周炎相关细菌测定中,m-Invader PLUS与实时PCR具有良好的相关性。
分枝杆菌是一大类细菌菌种,其中的一些细菌会引起人类疾病,如结核分枝杆菌导致人类罹患结核病,麻风分枝杆菌会引起麻风病等[49]。在临床检测中,急需一种快速、准确的分枝杆菌感染诊断方法。Ichimura等[50]将BACTEC MGIT 960系统与入侵检测方法的结合来对分枝杆菌进行检测。其中使用BACTEC MGIT 960系统可以极大的缩短分枝杆菌检测时间,使用核酸侵入反应不仅具有极高的准确性和特异性,还需要事先进行目标扩增就可以直接在基因组DNA上进行检测。随后他们对122株阳性液体培养物进行检测,检出率达到95.1%,说明该方法快速、简便、准确,是分枝杆菌感染诊断的有效方法。
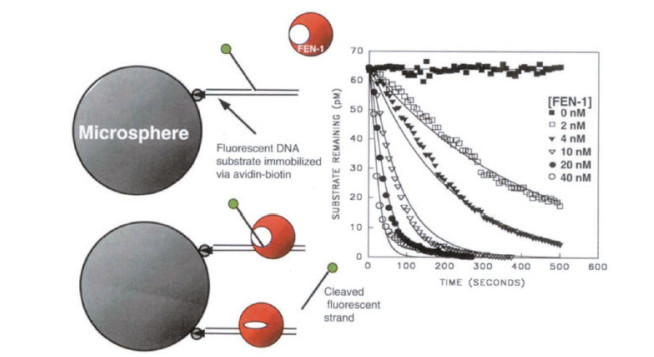
2.8 FEN1酶介导的其他生物传感器流式细胞术(Flow cytometry,FCM)是一种灵敏、定量的测量颗粒或细胞荧光或光散射的方法,它在配体结合和酶动力学、药物筛选、可溶性试剂诊断和检测、DNA序列检测或分析等领域都有所应用[51]。因此可以运用此种方法测定FEN1酶的核酸内切酶活性(图 14)。在链霉亲和素包覆的微球上固定荧光标记的DNA底物,用FEN-I测定DNA的裂解,然后通过快速的动力学仪器和计算机建模可以确定这种相互作用的基本速率常数。此方法提供了一种连续测量底物解离的新方式[52]。
链置换反应是指某些DNA聚合酶在延伸新链的过程中如果遇到下游DNA链,可以继续延伸反应并同时将下游双链剥离而产生游离的单链[53]。2015年,Zou等[54]提出了一种新的策略,将入侵反应与链置换信号扩增耦合(Invasive reaction assisted strand-displacement signal amplification,IRASA),进行目标DNA检测。首先用两个序列特异性探针进行侵入反应扩增目标DNA,然后用链置换反应扩增目标DNA,用此种方法可以检测低至0.2 pmol/L的目标DNA。并且由于二次放大对任何目标都是通用的,因此该方法有效的降低的检测成本,并且该方法可以区分目标DNA之间的单个碱基差异。
FEN1酶所介导的生物传感器除了在动物DNA基因分型上被使用,在植物基因分型上也有所利用。跨国种子公司正转向新的DNA标记技术,以加快植物育种和作物改良的速度。为此,Zec等[55]开发了一种可编程的、基于微流体的玉米基因组DNA基因分型装置(图 15)。微流控平台的一个独特功能是纳米样本处理器,它允许设备仅使用两个输入端连续加载数量不受限制的唯一DNA样本,克服了当前由于设备占用空间小而对样本输入数量的限制。并且所需样本量小,可以在50 nL的液滴中完成基因分型,大大降低了每次反应的试剂消耗。Helen等进行了240次入侵反应(针对10个SNP标记检测8个DNA样本),在单滴实验中对植物DNA样本进行SNP调用的准确率超过93%。
2.9 纳米材料结合检测FEN1酶与纳米材料的结合多是通过纳米金探针的方式。核酸纳米金探针是将寡核苷酸片段修饰在纳米金表面而形成的纳米颗粒,表面的寡核苷酸片段可与待测靶核酸进行特异性杂交,使纳米金颗粒发生聚集,从而导致纳米粒子光散射性质发生改变,造成肉眼可见的颜色变化[56]。
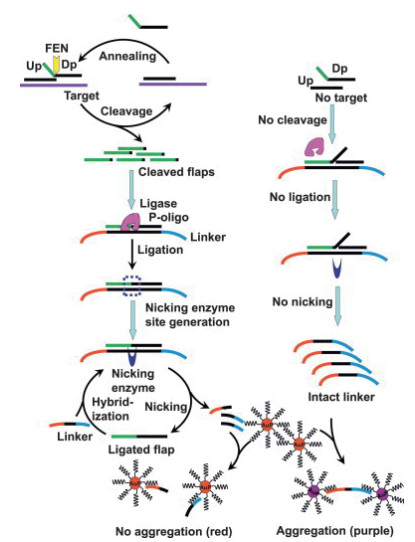
将切口核酸内切酶辅助扩增与基于纳米颗粒的传感器结合用于DNA检测(Novel nicking endonuclease-assisted nanoparticle amplification,NEANA)[57]。但在该方法中,切口核酸内切酶(Nicking endonuclease,NEase)只识别双链DNA的特定核苷酸序列,因此,NEANA无法检测到没有识别序列的目标。为了提高NEANA检测各种序列的真实生物样本的可行性,Zou等[58]提出了通过将核酸侵入反应与NEANA偶联进行比色DNA检测(Invasive reaction with NEANA,IR-NEANA)。靶DNA可通过核酸侵入反应产生许多的Flap片段,然后将Flap片段与5'磷酸化的寡核苷酸连接以产生NEANA的模板,随后进行NEANA反应(图 16)。因为侵入性反应仅依赖于通过上游探针和下游探针与靶DNA的特异性结合形成的重叠结构,与常规NEANA不同,任何DNA序列都可以通过IR-NEANA检测。
除了使用PCR进行扩增外,近年来发展起来并不断完善的环介导等温核酸扩增技术(Loop-mediated isothermal amplification,LAMP)也是一种高灵敏DNA扩增技术。其反应过程始终维持在恒定的温度下,通过添加不同活性的酶和引物来达到快速扩增核酸的目的[59]。为能对LAMP扩增子进行即时检测(Point-of-care test,POST),Lu等[60]提出LAMP基因分型结合核酸侵入,再通过金纳米粒子的显色实现可视化检测(图 17)。由于侵入性反应是高特异性的,因此可以准确区分复杂LAMP扩增子中的一个碱基差异,并且进行核酸扩增过程是在恒定温度下进行的,只需要一个简单的温度控制器,操作更为简便。陈银等[61]也利用此种方法快速检测脑膜炎奈瑟菌。
核酸侵入反应与纳米材料的结合在对蛋白的检测中也表现出巨大潜力。Song等[62]开发了一种核酸侵入反应结合AuNPs辅助酶联免疫吸附实验(Enzyme-linked immunosorbent assay,ELISA)检测方法(图 18),将该方法命名为iaELISA(Invader assisted ELISA)。该方法可以用于疾病生物标志物检测。首先对DNA片段修饰了针对靶向抗原的抗体,然后结合AuNPs进行核酸侵入反应,最后通过金纳米粒子的显色实现可视化检测。并且该方法在对HBV检测中体现了较好的特异性。
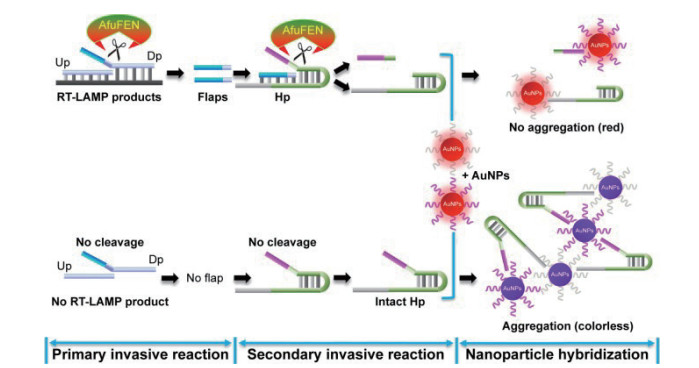
流感病毒主要通过其在全世界引起急性呼吸道疾病的能力对人类健康构成持续威胁。并且流感病毒的不断变异和高速的传播,是发病率和死亡率的最常见原因之一[63]。目前,多采用实时逆转录聚合酶链反应(Reverse transcription-polymerase chain reaction,RT-PCR)对流感病毒感染分子进行检测[64]。相比较而言,LAMP法更适合用于资源有限的区域或用于现场使用。Ge等[65]开发了一种多重逆转录环介导的等温扩增结合使用纳米颗粒作为传感器的级联核酸侵入反应(Multiplex reverse transcription LAMP with cascade invasive reaction using nanoparticles,mRT-LAMP-CIRN)的新方法(图 19),用于同时扩增3种亚型流感病毒(甲型流感/ H1N1pdm09,A / H3和乙型流感),可适用于现场使用和低设备设置实验室使用。
2.10 FEN1酶体内成像和治疗尽管FEN通常被认为是肿瘤抑制基因,但是癌症的发生及癌基因复制过程中FEN1高表达,并且许多研究表明FEN1酶在癌细胞生长和增殖扩散中起到一定作用[66]。FEN1在前列腺癌[67]、胰腺癌[68]、胃癌[69]、肺癌[70]及神经母细胞瘤[71]等肿瘤细胞中高表达,因此FEN1酶可以作为某些癌细胞的潜在生物标记物。
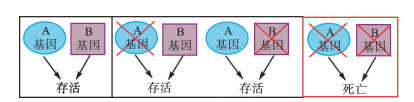
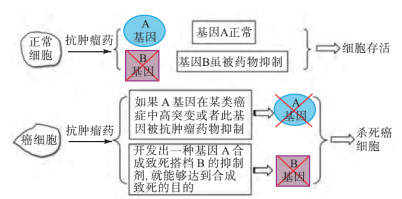
正因为FEN1酶与癌症联系的密切性,实验可以利用合成致死方式来利用FEN1合成抗癌药。合成致死(图 20),简单地说就是指在两个非致死基因中,若仅其中的一个基因发生了变异,细胞仍会存活,若两个基因同时发生变异,则将引起细胞死亡,这两个基因则构成合成致死搭档(partner)。如果发现肿瘤细胞中某个基因失活或者用药物抑制某个基因,与之构成合成致死搭档的另外的一个基因就有可能成为潜在的靶基因,那么对这个靶基因进行抑制就能够特异性的杀死癌细胞,并且不会影响正常细胞的存活(图 21)[72]。
而FEN1酶可以和近20种蛋白发生交互作用进行体内调控,因此认为FEN1可能与DNA损伤修复过程中的多个基因形成合成致死搭档,通过抑制FEN1酶的表达就可以达到合成致死目的。近年来发现姜黄素能够在蛋白表达水平上下调FEN1的表达,这可能是姜黄素抑制乳腺癌细胞增殖的机制之一[73]。中药铁筷子中分离的一种强心苷类成分HTF-1能剂量依赖性地下调FEN1的表达,这也是此化合物能够抑制癌细胞的原因之一[74]。通过基于机器学习的虚拟筛选,发现一种名为JFD00950萘类化合物具有FEN1催化抑制活性[75]。N-羟基脲类化合物也可作为FEN1的抑制剂。[76]
3 总结和展望综上,FEN1酶所介导的生物传感器已经应用于各个检测领域,主要优势FEN1酶对结构的特异性识别,提高了检测的灵敏度。其次,FEN1酶的活度范围广,在较大的温度区间内均具有活性,并且FEN1酶可以与多种信号输出方式相结合(荧光法、微电流法等)以及新兴的纳米材料技术相结合,体现了其良好的兼容性。同时,FEN1酶可以将不同种类的靶物质转换为核酸信号,并进行FEN1酶介导的信号放大,体现了其检测的广泛性。
目前,FEN1酶所介导的生物传感器主要用于DNA、RNA、病毒和细菌检测等方面,但在实际应用中多用于对疾病生物标志物的检测。未来对FEN1酶研究的发展趋势可能为:(1)体内作用研究:FEN1酶在体内不仅具有核酸酶活性,还与多种蛋白发生交互作用,可通过对FEN1酶的进一步研究更深入的了解人类的身体密码。(2)生物传感器的进一步开发:生物传感器的最终目的是进行实际应用,争取用最短的时间最低的成本实现利益的最大化,而FEN1酶作为一种结构特异性核酸酶,所使用的范围大,活度范围广,并且价格低廉,体现了其巨大的使用优势。(3)纳米材料的多样化搭载:纳米材料因为其独特的物理性质和化学性质使得检测的灵敏度大幅提高,并且检测时间大大缩短,目前FEN1酶与纳米材料的研究多集中在与纳米金粒子的结合上,未来在水凝胶、磁珠上可以有更广泛的应用。(4)抗肿瘤药物的研发:FEN1酶在多种肿瘤中的高表达体现了其在抗肿瘤药物研发中的潜力,可以通过利用合成致死原理合成特异性的抗肿瘤药物。
| [1] |
雷娜娜. 生物传感器[J]. 生物技术世界, 2015(6): 266-266. |
| [2] |
Harrington JJ, Lieber MR. The characterization of a mammalian DNA structure-specificendonuclease[J]. EMBO J, 1994, 13(5): 1235-1246. |
| [3] |
Klett RP, Cerami A, Reich E. Exonuclease Ⅵ, a new nuclease activity associated with E. coli DNA polymerase[J]. Proc Natl Acad Sci USA, 1968, 60(3): 943-950. DOI:10.1073/pnas.60.3.943 |
| [4] |
Kelly RB, Atkinson MR, Huberman JA, et al. Excision of thymine dimers and other mismatched sequences by DNA polymerase of Escherichia coli[J]. Nature, 1969, 224(5218): 495-501. DOI:10.1038/224495a0 |
| [5] |
Lindahl T, Gally JA, Edelman GM. Deoxyribonuclease Ⅳ:A new exonuclease from mammalian tissues[J]. Proceedings of the National Academy of Sciences, 1969, 62(2): 597-603. DOI:10.1073/pnas.62.2.597 |
| [6] |
Setlow P, Brutlag D, Kornberg A, et al. Deoxyribonucleic acid poly-merase:Two distinct enzymes in one polypeptide:Ⅰ. A proteolytic fragment containing the polymerase and 3'→5' exonuclease functions[J]. Journal of Biological Chemistry, 1972, 247(1): 224-231. |
| [7] |
Masamune Y, Richardson CC. Strand displacement during deoxyribonucleic acid synthesis at single strand breaks[J]. Journal of Biological Chemistry, 1971, 246(8): 2692-2701. |
| [8] |
Lundquist RC, Olivera BM. Transient generation of displaced single-stranded DNA during nick translation[J]. Cell, 1982, 31(1): 53-60. DOI:10.1016/0092-8674(82)90404-4 |
| [9] |
Lyamichev V, Brow MAD. Structure-specific endonucleolytic cleavage of nucleic acids by eubacterial DNA polymerases[J]. Science, 1993, 260(5109): 778. DOI:10.1126/science.7683443 |
| [10] |
Lee Bi, Nguyen LH, Barsky D, et al. Molecular interactions of human Exo1 with DNA[J]. Nucleic Acids Res, 2002, 30(4): 942-949. DOI:10.1093/nar/30.4.942 |
| [11] |
Erica FH, Ghislaine H, Ulrich H, et al. The acetylatable lysines of human Fen1 are important for endo-and exonuclease activities[J]. J Mol Biol, 2003, 328(1): 73-84. DOI:10.1016/S0022-2836(03)00270-5 |
| [12] |
Shen B, Singh P, Liu R, et al. Multiple but dissectible functions of FEN-1 nucleases in nucleic acid processing, genome stability and diseases[J]. BioEssays, 2005, 27(7): 717-729. DOI:10.1002/bies.20255 |
| [13] |
Henneke G. In vitro reconstitution of RNA primer removal in archaea reveals the existence of two pathways[J]. Biochemical Journal, 2012, 447(2): 271-280. DOI:10.1042/BJ20120959 |
| [14] |
Pike JE, Henry RA, Burgers PMJ, et al. An alternative pathway for okazaki fragment processing:Resolution of fold-back flaps by pif1 helicase[J]. J Biol Chem, 2010, 285(53): 41712-41723. DOI:10.1074/jbc.M110.146894 |
| [15] |
Ryu GH, Tanaka H, Kim DH, et al. Genetic and biochemical analyses of Pfh1 DNA helicase function in fission yeast[J]. Nucleic Acids Res, 2004, 32(14): 4205-4216. DOI:10.1093/nar/gkh720 |
| [16] |
Yang M, Guo H, Wu C, et al. Functional FEN1 polymorphisms are associated with DNA damage levels and lung cancer risk[J]. Human Mutation, 2009, 30(9): 1320-1328. DOI:10.1002/humu.21060 |
| [17] |
Liu L, Zhou C, Zhou L, et al. Functional FEN1 genetic variants contribute to risk of hepatocellular carcinoma, esophageal cancer, gastric cancer and colorectal cancer[J]. Carcinogenesis, 2012, 33(1): 119-123. DOI:10.1093/carcin/bgr250 |
| [18] |
Li Z, Dai HF, Zhou M, et al. Fen1 mutations result in autoimmunity, chronic inflammation and cancers[J]. Nature Medicine, 2007, 13(7): 812-819. DOI:10.1038/nm1599 |
| [19] |
Singh P, Yang M, Dai H, et al. Overexpression and hypomethylation of flap endonuclease 1 gene in breast and other cancers[J]. Molecular Cancer Research, 2008, 6(11): 1710-1717. |
| [20] |
马寅姣, 邹秉杰, 王建平, 等. 基于核酸侵入反应的生物分子检测技术的研究进展[J]. 现代生物医学进展, 2011, 11(12): 2384-2388. |
| [21] |
Olivier M. The Invader® assay for SNP genotyping[J]. Mutation Research, 2005, 573(1): 103-110. |
| [22] |
Hall JG, Eis PS, Law SM, et al. Sensitive detection of DNA polymorphisms by the serial invasive signal amplification reaction[J]. Proceedings of the National Academy of Sciences, 2000, 97(15): 8272-8277. DOI:10.1073/pnas.140225597 |
| [23] |
Feinberg AP, Tycko B. The history of cancer epigenetics[J]. Nature Reviews Cancer, 2004, 4(2): 143-153. DOI:10.1038/nrc1279 |
| [24] |
Zou H, Allawi H, Cao X, et al. Quantification of methylated markers with a multiplex methylation-specific technology[J]. Clinical Chemistry, 2012, 58(2): 375-383. DOI:10.1373/clinchem.2011.171264 |
| [25] |
Hsu TM, Law SM, Duan S, et al. Genotyping single-nucleotide polymorphisms by the invader assay with dual-color fluorescence polarization detection[J]. Clinical Chemistry, 2001, 47(8): 1373-1377. DOI:10.1093/clinchem/47.8.1373 |
| [26] |
Berggren WT, Takova T, Olson MC, et al. Multiplexed gene expressionanalysis using the invader RNA assay with MALDI-TOFmass spectrometry detection[J]. Anal, Chem, 2002, 74: 1745-1750. DOI:10.1021/ac011167t |
| [27] |
Crain PF, McCloskey JA. Applications of mass spectrometry to the characterization ofoligonucleotides and nucleic acids[J]. Current Opinion in Biotechnology, 1998, 9(1): 25-34. DOI:10.1016/S0958-1669(98)80080-3 |
| [28] |
Lyamichev Ⅵ, Kaiser MW, Lyamicheva NE, et al. Experimental and theoretical analysis of the invasive signal amplification reaction[J]. Biochemistry, 2000, 39(31): 9523-9532. DOI:10.1021/bi0007829 |
| [29] |
Hessner MJ, Budish MA, Friedman KD. Genotyping of factor V G1691A(Leiden)without the use of PCR by invasive cleavage of oligonucleotide probes[J]. Clinical Chemistry, 2000, 46(8): 1051-1056. DOI:10.1093/clinchem/46.8.1051 |
| [30] |
Eis PS, Olson MC, Takova T, et al. An invasive cleavage assay for direct quantitation of specific RNAs[J]. Nature Biotechnology, 2001, 19(7): 673-676. DOI:10.1038/90290 |
| [31] |
Almeida MI, Reis RM, Calin GA. MicroRNA history:Discovery, recent applications, and next frontiers[J]. Mutation Research, 2011, 717(1): 1-8. |
| [32] |
Allawi HT, Dahlberg JE, Olson S, et al. Quantitation of microRNAs using a modified Invader assay[J]. RNA, 2004, 10(7): 1153-1161. DOI:10.1261/rna.5250604 |
| [33] |
Arruda Md, Lyamichev Ⅵ, Eis PS, et al. Invader technology for DNA and RNA analysis:principles and applications[J]. Expert Review of Molecular Diagnostics, 2002, 2(5): 487-496. DOI:10.1586/14737159.2.5.487 |
| [34] |
Cobo F. Application of molecular diagnostic techniques for viral testing[J]. Open Virol J, 2012, 6: 104-114. |
| [35] |
Chenna A, Tian H, Cao L, et al. Multiplex mRNA assay using electrophoretic tags for high-throughput gene expression analysis[J]. Nucleic Acids Research, 2004, 32(16): e126. DOI:10.1093/nar/gnh119 |
| [36] |
Ott JJ, Stevens GA, Groeger J, et al. Global epidemiology of hepatitis B virus infection:New estimates of age-specific HBsAg seroprevalence and endemicity[J]. Vaccine, 2012, 30(12): 2212-2219. DOI:10.1016/j.vaccine.2011.12.116 |
| [37] |
乔斌, 李艳. 丙型肝炎病毒实验室检测技术进展[J]. 医学综述, 2016, 22(15): 2997-3000. DOI:10.3969/j.issn.1006-2084.2016.15.025 |
| [38] |
Kenichi T, Mariko K, Fumitaka S, et al. Comparative quantitative analysis of hepatitis C mutations at amino acids 70 and 91 in the core region by the Q-Invader assay[J]. Journal of Virological Methods, 2013, 189(1): 221-227. DOI:10.1016/j.jviromet.2012.10.011 |
| [39] |
Hayashi K, Ishigami M, Ishizu Y, et al. Comparison of direct sequencing and Invader assay for Y93H mutation and response to interferon-free therapy in hepatitis C virus genotype 1b[J]. J Gastroenterol Hepatol, 2018, 33(1): 249-255. DOI:10.1111/jgh.13809 |
| [40] |
刘琳琳, 齐谢敏, 邹秉杰, 等. 基于核酸侵入反应偶联PCR的粪便样本基因甲基化定量分析方法及用于结直肠癌无创筛查[J]. 分析化学, 2018, 46(10): 1552-1559. DOI:10.11895/j.issn.0253-3820.181117 |
| [41] |
Wang RN, Green J, Wang Z, et al. Bone morphogenetic protein(BMP)signaling in development and human diseases[J]. Genes & Diseases, 2014, 1(1): 87-105. |
| [42] |
Loh K, Chia JA, Greco S, et al. Bone morphogenic protein 3 inactivation is an early and frequent event in colorectal cancer development[J]. Genes, Chromosomes and Cancer, 2008, 47(6): 449-460. DOI:10.1002/gcc.20552 |
| [43] |
严志进, 刘云龙, 邹秉杰, 等. 粪便中大肠癌特异性NDRG4基因甲基化水平的超高灵敏定量检测法[J]. 医学研究生学报, 2016, 10: 1031-1035. |
| [44] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [45] |
Chen WQ, Zheng RS, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [46] |
Paez JG, Jänne PA, Lee JC, et al. EGFR mutations in lung cancer:correlation with clinical response to gefitinib therapy[J]. Science, 2004, 304(5676): 1497-1500. DOI:10.1126/science.1099314 |
| [47] |
Naoki K, Soejima K, Okamoto H, et al. The PCR-invader method(structure-specific 5' nuclease-based method), a sensitive method for detecting EGFR gene mutations in lung cancer specimens; comparison with direct sequencing[J]. International Journal of Clinical Oncology, 2011, 16(4): 335-344. DOI:10.1007/s10147-011-0187-5 |
| [48] |
Kenichi T, Toshikazu Y, Katsumi K, et al. Rapid quantification of periodontitis-related bacteria using a novel modification of invader PLUS technologies[J]. Microbiol Res, 2010, 165(1): 43-49. DOI:10.1016/j.micres.2008.06.001 |
| [49] |
李军伟, 李小伟. 分枝杆菌临床检验研究[J]. 深圳中西医结合杂志, 2017, 27(14): 132-133. |
| [50] |
Ichimura S, Nagano M, Ito N, et al. Evaluation of the invader assay with the BACTEC MGIT 960 system for prompt isolation and identification of mycobacterial species from clinical specimens[J]. J Clin Microbiol, 2007, 45(10): 3316-3322. DOI:10.1128/JCM.02289-06 |
| [51] |
Nolan JP, Shen B, Park MS, et al. Kinetic analysis of human flap endonuclease-1 by flow cytometry[J]. Biochemistry, 1996, 35(36): 11668-11676. DOI:10.1021/bi952840+ |
| [52] |
Nolan JP, Sklar LA. The emergence of flow cytometry for sensitive, real-time measurements of molecular interactions[J]. Nature Biotechnology, 1998, 16(7): 633-638. DOI:10.1038/nbt0798-633 |
| [53] |
何艳, 蒋涛. 基于链置换反应的DNA等温扩增技术应用进展[J]. 医学综述, 2010, 19(1): 673-676. |
| [54] |
Zou B, Song Q, Wang J, et al. Invasive reaction assisted strand-dis-placement signal amplification for sensitive DNA detection[J]. Chemical Communications, 2014, 50(89): 13722-13724. DOI:10.1039/C4CC06079B |
| [55] |
Zec HC, Zheng T, Liu L, et al. Programmable microfluidic genoty-ping of plant DNA samples for marker-assisted selection[J]. Microsystems & Nanoengineering, 2018, 4: 17097. |
| [56] |
邹秉杰, 周国华. 基于核酸侵入反应的基因突变检测方法研究进展[J]. 药学进展, 2015, 39(11): 846-854. |
| [57] |
Xu W, Xue X, Li T, et al. Ultrasensitive and selective colorimetric DNA detection by nicking endonuclease assisted nanoparticle amplification[J]. Angewandte Chemie International Edition, 2009, 48(37): 6849-6852. DOI:10.1002/anie.200901772 |
| [58] |
Zou B, Cao X, Wu H, et al. Sensitive and specific colorimetric DNA detection by invasive reaction coupled with nicking endonuclease-assisted nanoparticles amplification[J]. Biosensors and Bioelectronics, 2015, 66: 50-54. DOI:10.1016/j.bios.2014.10.077 |
| [59] |
梁海燕, 刘文鑫, 杨志刚, 等. 等温核酸扩增技术进展[J]. 中国医学创新, 2017, 14(16): 145-148. DOI:10.3969/j.issn.1674-4985.2017.16.042 |
| [60] |
Lu Y, Ma XP, Wang JP, et al. Visualized detection of single-base difference in multiplexed loop-mediated isothermal amplification amplicons by invasive reaction coupled with oligonucleotide probe-modified gold nanoparticles[J]. Biosensors and Bioelectronics, 2017, 90: 388-393. DOI:10.1016/j.bios.2016.12.015 |
| [61] |
陈银, 葛以跃, 赵康辰, 等. 环介导等温扩增结合核酸级联侵入反应和纳米金探针显色技术快速检测脑膜炎奈瑟菌[J]. 江苏预防医学, 2018, 29(6): 607-610. |
| [62] |
Song Q, Qi X, Jia H, et al. Invader Assisted enzyme-linked immun-osorbent assay for colorimetric detection of disease biomarkers using oligonucleotide probe-modified gold nanoparticles[J]. J Biomed Nanotechnol, 2016, 12(4): 831-839. |
| [63] |
Simonsen L, Peter S, Roger L, et al. Global mortality estimates for the 2009 influenza pandemic from the GLaMOR project:a modeling study[J]. PLoS Medicine, 2013, 10(11): e1001558. DOI:10.1371/journal.pmed.1001558 |
| [64] |
Ito M, Nukuzuma S, Sugie M, et al. Detection of pandemic influenza A(H1N1)2009 virus RNA by real-time reverse transcription polymerase chain reaction[J]. Pediatr Int, 2012, 54(6): 959-962. DOI:10.1111/j.1442-200X.2012.03720.x |
| [65] |
Ge Y, Zhou Q, Zhao K, et al. Detection of influenza viruses by coupling multiplex reverse-transcription loop-mediated isothermal amplification with cascade invasive reaction using nanoparticles as a sensor[J]. Int J Nanomedicine, 2017, 12(4): 2645-2656. |
| [66] |
Zheng L, Dai H, Zhou M, et al. Fen1 mutations result in autoimmunity, chronic inflammation and cancers[J]. Nature Medicine, 2007, 13: 812. DOI:10.1038/nm1599 |
| [67] |
LaTulippe E, Satagopan J, Smith A, et al. Comprehensive gene expression analysis of prostate cancer reveals distinct transcriptional programs associated with metastatic disease[J]. Cancer Research, 2002, 62(15): 4499-4506. |
| [68] |
Iacobuzio-Donahue CA, Maitra A, Olsen M, et al. Exploration of global gene expression patterns in pancreatic adenocarcinoma using cdna microarrays[J]. The American Journal of Pathology, 2003, 162(4): 1151-1162. DOI:10.1016/S0002-9440(10)63911-9 |
| [69] |
Kim JM, Sohn HY, Yoon SY, et al. Identification of gastric cancer-related genes using a cDNA microarray containing novel expressed sequence tags expressed in gastric cancer cells[J]. Clin Cancer Res, 2005, 11: 473-482. |
| [70] |
Sato M, Girard L, Sekine I, et al. Increased expression and no mutation of the Flap endonuclease(FEN1)gene in human lung cancer[J]. Oncogene, 2003, 22(46): 7243-7266. DOI:10.1038/sj.onc.1206977 |
| [71] |
Krause A, Combaret V, Iacono I, et al. Genome-wide analysis of gene expression in neuroblastomas detected by mass screening[J]. Cancer Lett, 2005, 225: 111-120. DOI:10.1016/j.canlet.2004.10.035 |
| [72] |
马龙, 王海月, 周津, 等. 基于人皮瓣核酸内切酶抗癌策略的研究进展[J]. 天津科技大学学报, 2019, 3: 1-6. |
| [73] |
Chen B, Zhang Y, Wang Y, et al. Curcumin inhibits proliferation of breast cancer cells through Nrf2-mediateddown-regulation of FEN1 expression[J]. Journal of Steroid Biochemistry & Molecular Biology, 2014, 143(9): 11-18. |
| [74] |
Ma L, Meng Y, Tu C, et al. A cardiac glycoside HTF-1isolated from Helleborusthibetanus Franch displays potentin vitro anti-cancer activity via caspase-9, MAPK and PI3K-Akt-mTOR pathways[J]. Eur J Medl Chem, 2018, 158: 743-752. DOI:10.1016/j.ejmech.2018.09.019 |
| [75] |
Deshmukh AL, Chandra S, Singh DK, et al. Identification of human flap endonuclease 1(FEN1)inhibitors using a machine learning based consensus virtual screening[J]. Molecular Biosystems, 2017, 13(8): 1630-1639. DOI:10.1039/C7MB00118E |
| [76] |
He L, Zhang Y, Sun H, et al. Targeting DNA flap endonuclease1 to impede breast cancer progression[J]. EBioMedicine, 2016, 14: 32-43. DOI:10.1016/j.ebiom.2016.11.012 |