2. 天津化学化工学院,天津 300072
2. School of Chemical Engineering and Technology, Tianjin University, Tianjin 300072
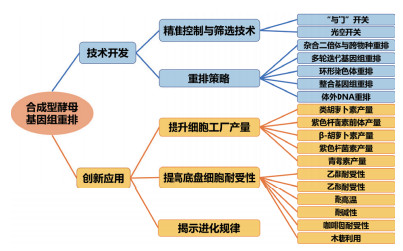
基因组变异为物种进化提供了动力。基因组变异的类型主要有单核苷酸多态性(Single nucleotide polymorphisms,SNPs)、插入缺失(Indels)、结构变异(Structural variation,SVs)。基因组的结构变异包括片段缺失、重复、相互移位和反转等,其包含更多的遗传变异信息。基因组结构变异已经被广泛的认为与物种进化、个体间遗传差异、人类疾病以及大量的表型变异相关[1-4]。设计染色体和基因组的全合成是遗传学和生物学研究的新范式,酿酒酵母是真核生物的模式生物,可以作为真核生物染色体系统研究的平台,以验证和拓宽我们对生物体的认知。全化学合成的酿酒酵母染色体上具有对称的loxP位点,基于Cre-loxP反应的基因组重排技术可以产生DNA片段的删除、反转、复制、移位等不同组合的染色体结构变异,实现全基因组序列的重排,丰富了基因型多样性。本文介绍了最近合成型酵母基因组重排的技术开发和其创新应用的进展(图 1)。
|
| 图 1 合成型酵母基因组重排技术开发与创新应用 |
“人工合成酿酒酵母基因组”计划(Sc2.0)旨在获得第一个完全化学合成的、具有完整生物活性的真核生物基因组[5-12]。通过在合成型染色体非必需基因终止密码子后添加对称的loxP序列(loxPsym),赋予了基因组柔性可变的功能,可使其间的DNA片段发生删除、反转、复制、移位等基因组结构变异。
传统的loxP序列来自P1噬菌体,全长共计34 bp,左右两端为13 bp反向回文序列,中间为8 bp间隔序列。在Cre重组酶诱导下两个loxP序列之间发生位点特异性重组,引起序列间DNA的重排[13-15]。Cre重组酶基因同样来自P1噬菌体,其编码区序列全长1 029 bp,编码由343个氨基酸组成的38 kD单体蛋白,其能够特异性地识别loxP位点。二者共同组成Cre-loxP重组酶系统。
Sc2.0采用对称的loxP序列(LoxPsym)与Cre酶的相互作用构成了基因组重排系统(Synthetic chromosome rearrangement and modification by loxP-mediated evolution,SCRaMbLE)。通过诱导Cre酶的表达,可以实现loxP序列间DNA片段的删除、反转、复制、移位等基因组结构变异。利用基因组重排技术可以实现全基因组范围内的DNA片段重排,从而可以产生大量不同基因型和表型的酵母菌株,快速获得丰富的酵母文库。
2 合成型酵母基因组重排技术进展 2.1 技术开发 2.1.1 基因组重排的精准控制与筛选技术传统的单纯依赖雌激素诱导的Cre重组酶表达系统在实验中被证明了其局限性,在没有雌激素诱导的情况下仍会出现因Cre酶泄露造成的合成型染色体稳定性降低甚至致死现象,因而精准控制基因组重排过程对于产生基因型多样性和具有特定优势的物种至关重要。精准控制基因组重排可以通过“与门”开关或光控制来实现。以合成型酿酒酵母5号染色体(synV)为研究对象,Jia等[16]构建了一个半乳糖驱动的Cre-EBD作为“与门”基因开关,用于精确控制合成型单倍体和二倍体酵母发生基因组重排。“与门”开关pCRE4(pGal1-Cre-EBD-tCYC1)通过组合转录水平和Cre-EBD的细胞核定位水平调节Cre酶活性,精准控制重排系统的开关。只有在半乳糖诱导pGal1启动子表达和雌二醇诱导Cre酶表达同时发生时,“与门”开关pCRE4才能开启基因组的重排,从而达到精准控制的目的[17-18]。Hochrein等[19]开发了一种光控制的Cre重组酶诱导系统(L- SCRaMbLE),并将其应用于控制基因组重排。L- SCRaMbLE技术基于分裂的蛋白而设计,其中Cre酶的N端和C端被分裂并分别与来自拟南芥的发色团结合光感受器(PhyB)及其相互作用因子(PIF3)两种异源蛋白融合,经红光照射后两种植物蛋白相互作用以重建Cre重组酶使其恢复功能。应用L- SCRaMbLE技术可有效应对Cre酶泄露带来的问题,并可通过诱导时间、光剂量和感受器浓度微调Cre重组酶的活性,进而精准控制基因组重排。
基因组重排技术可以产生大量的基因组结构变异并获得庞大的菌株库,有效的筛选方法是获得目标菌株的必要途径。Luo等[20]开发出一种高效筛选重组细胞的报告系统,即ReSCuES。该系统基于loxP介导的两个营养缺陷型标签(URA3和LEU2)的交替“开/关”,两个营养缺陷标签的开放阅读框彼此相邻且对向排列形成一个“URA3-LEU2”模块,其两侧具有两个对称的loxP位点。Cre重组酶诱导一次基因组重排导致模块倒置,打开LEU2的表达同时关闭URA3的表达。借助该报告系统并利用相应的缺陷型培养基可高效筛选已发生基因组重排菌,缩短了实验时间并节省劳力,并且随着菌株中合成型染色体数目的增多系统优势有更佳的体现。
2.1.2 合成型酵母基因组重排策略合成型酵母基因组重排新策略包括杂合二倍体与跨物种基因组重排、多轮迭代基因组重排、环形染色体重排、体外DNA重排、整合基因组重排等。
Shen等开发了杂合二倍体与跨物种基因组重排策略,通过将含有单条合成型染色体(synX)和含有两条合成型染色体(synV and synX)的单倍体酵母与来自酵母菌株库(SGRP)的野生型酿酒酵母(Saccharomyces cerevisiae)和野生型奇艺酵母(Saccharomyces paradoxus)单倍体交配,获得了杂合二倍体和跨物种二倍体[9-10, 21-23]。通过基因组重排技术的驱动进行基因组重排,获得了多达100株的杂合二倍体和跨物种二倍体菌株库,结果表明在二倍体中进行基因组重排操作,菌株的存活率比在单倍体中更高。
为了持续驱动酿酒酵母表型进化,Jia等[16]将酿酒酵母二倍体和单倍体交替的生活史与基因组重排技术相结合开发了一种多轮迭代的基因组重排技术(MuSIC)。通过一轮重排后筛选得到的高产二倍体酵母的高产孢子与其他合成型单倍体酵母交配,再进行下一轮基因组重排,这样的多轮循环可以快速积累大量重排。多轮迭代技术的建立实现了基因组结构的持续进化。
此外,染色体拓扑结构也对染色体重排起着重要作用,Xie等[9]合成了环形酿酒酵母5号染色体(ring_synⅤ),其全长534 637 bp,端粒被去除并被170个loxPsym位点分割成170个片段。Wang等[24]对ring_synⅤ应用基因组重排技术进行基因组重排,发现环形染色体可以发生大规模复制,经过5轮迭代基因组重排技术,合成型环形染色体增加的长度多达原长的一倍以上。此外,合成型环形染色体在基因组重排过程中还能够发生染色体数目变异,形成了染色体数目非整倍的菌株。借助合成型环形染色体的基因组重排技术,可以驱动染色体连续地产生复杂的基因型并得到相应表型,从而可以有效扩大结构变异的规模和数量。
随着DNA合成和组装技术的快速发展,合成型DNA在异源代谢通路和合成型基因组的从头设计和构建方面有了新的应用[7, 25]。Wu等[26]设计开发了一种体外DNA基因组重排技术,利用Cre重组酶与包含多个loxPsym位点DNA的相互作用,可以在试管内实现DNA的高效重排。研究者提出了“自上而下”和“自下而上”的两种代谢通路优化策略。“自上而下”的基因组重排技术采用纯化的DNA重组酶对编码多个loxPsym位点的DNA结构进行基于重排的优化。通过对基因组重排技术后的pLM495文库进行PacBio测序,共确定了94个独特的通路结构,这些通路展现了crtI、crtE、crtYB和tHMG1四个基因基于多个loxPsym位点发生的可测得的重排事件;将pLM495直接转化进酿酒酵母进行表型检测,得到了包括删除、反转和复制在内的17种独特的β-胡萝卜素代谢途径结构。借助体外DNA重排技术可实现在体外建立结构变异文库,为快速实现异源代谢通路的优化提供了一种有效方法。Liu等[27]开发了一种基于重组酶的整合基因组重排技术(称为“SCRaMbLE-in”)。在体外将调节元件、选定的重组酶和靶途径DNA混合后,调节元件可通过重组酶整合进靶重组位点上以产生一个组装代谢路径库。随后借助基因组重排技术将组装路径整合到合成型酵母基因组中,同时重组酶可诱导底盘细胞发生基因组重排。利用SCRaMbLE-in可以只通过两步反应,达到同时实现基因表达的多样化和底盘细胞的工程化的效果,快速优化宿主以更好地适应异源途径以及优化外源途径在细胞内表达,最终实现快速代谢工程。
2.2 合成型酵母基因组重排技术的应用 2.2.1 合成型酵母基因组重排技术提升细胞工厂产量不同的基因组重排技术策略(体内、体外;线型、环型;单倍体、二倍体)驱动的合成型染色体重排可产生大量的基因组结构变异,推动酵母菌株库的产生,进而从中筛选出细胞工厂产量提升的酵母菌株。目前已经可以利用基因组重排技术提升类胡萝卜素、紫色杆菌素及青霉素等的产量[16, 24, 26, 28]。
Jia等[16]利用独特的“与门”基因开关调节Cre酶的活性,对单倍体菌株使用精准控制基因组重排系统生产类胡萝卜素,通过全基因组测序证明了合成型五号染色体上YEL013W的缺失可以提升类胡萝卜素的产量。进一步对二倍体酵母应用MuSIC,经过5轮迭代重排累积多个染色体重排使生产菌株快速进化,最终使类胡萝卜素产量提升38.8倍。Wu等[26]利用体外DNA重排系统来优化酵母细胞内源表达的β-胡萝卜素代谢通路,使β-胡萝卜素产量增加了5.1倍,并发现了CrtI基因是该代谢通路的关键基因,该基因的复制和反转都可以使β-胡萝卜素产量提高。
Wang等[24]对含有合成型环形五号染色体的紫色杆菌素前体物(PDV)生产菌进行多轮诱导重排,一共得到53个新的结构连接。将其中29个新的连接位点整合到酿酒酵母中使PDV产量增加了约3.48倍,表明环形染色体重排产生的全新结构变异与PDV的产量上升有关。Blount等[28]将外源代谢通路整合进酵母细胞中,通过合成型五号染色体的体内基因组重排得到紫色杆菌素高产菌株。此外,还得到了青霉素高产菌株,将青霉素产量提高了近2倍。研究者将基因组重排技术与纳米孔测序技术相结合,从而快速鉴定基因组结构变异,获得了更高的产率。
2.2.2 合成型酵母基因组重排技术提高底盘细胞耐受性使用基因组重排技术可以驱动菌株快速进化,在不同的环境压力中筛选获得适应性菌株。可以增加底盘细胞对乙醇、乙酸、咖啡因、木糖等物质的耐受性,或增强其在碱性、高温等环境下的生活能力[20-21, 28-29]。
Luo等[20]对分别含有合成型12号和3号染色体的酵母细胞进行基因组重排,利用ReSCuES筛选策略,最终得到了乙醇耐受性增加且遗传稳定的菌株,证明了ACE2的丢失导致乙醇耐受性的增加。此外,研究者还利用ReSCuES筛选出了耐乙酸的优势菌株。Shen等[21]基于奇艺酵母CBS5829对咖啡因的相对耐受性,通过跨物种二倍体CBS5829-synX和CBS5829-synVsynX的基因组重排,成功获得十株咖啡因耐受性提升的重排菌株,全基因组测序和验证实验显示POL32基因拷贝数的增加与咖啡因耐受性的提升有关。Blount等[28]将木糖利用的异源途径引入带有合成型五号染色体的酿酒酵母,通过基因组重排成功获得一株在以木糖为唯一碳源的培养基上生长增强的菌株。
Ma等[29]以含有一条合成型5号染色体的酵母菌株和同时含有5号、10号两条合成型染色体的酵母菌株为研究对象,通过Cre-EBD重组酶诱导进行多次独立的基因组重排,驱动合成型酵母菌株的快速进化。在pH为8.0的碱性环境下筛选到了7株耐碱性提高的菌株,全基因组测序并通过实验验证发现YER161C的缺失与菌株耐碱性的提高有关。Shen等[21]以酿酒酵母Y12为研究对象,通过杂合二倍体Y12-synX的基因组重排,成功获得了两株耐受42℃高温的菌株。Luo等[20]利用ReSCuES筛选出了耐高温的优势菌株。
2.2.3 合成型酵母基因组重排技术揭示进化规律遗传变异对于揭示进化规律和遗传改造具有重要意义,但目前我们对遗传变异的认识主要局限于SNPs和indels,对于大尺度的基因组结构变异特别是复制、反转和移位的研究还相对不足[1]。Li等[30]以含有合成型酿酒酵母和奇艺酵母染色体的杂合二倍体菌株为研究对象,使用基因组重排技术驱动合成型杂合酵母菌株重排,通过全基因组测序分析,在进化菌株中检测到不同尺度的杂合性缺失事件。实验结果揭示出合成型五号染色体上基因GLN3的缺失,以及合成型十号染色体全染色体杂合性缺失与酵母雷帕霉素耐受性相关。研究者通过快速适应性进化同样获得了雷帕霉素耐受菌株,发现了八号染色体的拷贝数增加。这表明基因组重排为基因组结构变异与进化的研究提供了一种新思路。
3 展望到目前为止,合成酿酒酵母基因组计划已经完成了6条酵母染色体的化学合成,未来将得到含有完整的16条合成型染色体的人工酵母基因组[32]。全合成型的人工酵母基因组进行基因组重排后,将会产生更多具有不同基因型的酵母菌株库,为提升生产菌种性能、优化底盘细胞和异源途径的适配提供更多可能。基因组重排技术方兴未艾,在技术和应用方面还在不断拓展。目前的基因组重排技术仅限于合成型酿酒酵母菌株,因而将基因组重排技术的研究对象从酿酒酵母这一模式生物扩展到其他工业相关的酵母菌、原核乃至高等真核生物是未来基因组重排技术发展的重要方向。另外,基因组重排技术与其他技术(如CRISPR-Cas9技术、传统的诱变技术)的结合,可以提供多尺度的基因组变异的方式,在基础研究、医疗健康、工业生产、生物资源等领域拥有巨大的发展前景。
| [1] |
Stankiewicz P, Lupski JR. Structural variation in the human genome and its role in disease[J]. Annu Rev Med, 2010, 61: 437-455. DOI:10.1146/annurev-med-100708-204735 |
| [2] |
Korbel JO, Urban AE, Affourtit JP, et al. Paired-end mapping reveals extensive structural variation in the human genome[J]. Science, 2007, 318(5849): 420-426. DOI:10.1126/science.1149504 |
| [3] |
MacDonald JR, Ziman R, Yuen RK, et al. The database of genomic variants: a curated collection of structural variation in the human genome[J]. Nucleic Acids Res, 2014, 42(Database issue): D986-D992.
|
| [4] |
Rodriguez-Revenga L, Mila M, Rosenberg C, et al. Structural variation in the human genome:the impact of copy number variants on clinical diagnosis[J]. Genet Med, 2007, 9(9): 600-606. DOI:10.1097/GIM.0b013e318149e1e3 |
| [5] |
Dymond JS, Richardson SM, Coombes CE, et al. Synthetic chromosome arms function in yeast and generate phenotypic diversity by design[J]. Nature, 2011, 477(7365): 471-476. DOI:10.1038/nature10403 |
| [6] |
Annaluru N, Muller H, Mitchell LA, et al. Total synthesis of a functional designer eukaryotic chromosome[J]. Science, 2014, 344(6179): 55-58. DOI:10.1126/science.1249252 |
| [7] |
Richardson SM, Mitchell LA, et al. Design of a synthetic yeast genome[J]. Science, 2017, 355(6329): 1040-1044. DOI:10.1126/science.aaf4557 |
| [8] |
Shen Y, Wang Y, Chen T, et al. Deep functional analysis of synⅡ, a 770-kilobase synthetic yeast chromosome[J]. Science, 2017, 355(6329): eaaf4791. DOI:10.1126/science.aaf4791 |
| [9] |
Xie ZX, Li BZ, et al. "Perfect" designer chromosome V and behavior of a ring derivative[J]. Science, 2017, 355(6329): eaaf4704. DOI:10.1126/science.aaf4704 |
| [10] |
Mitchell LA, Wang A, Stracquadanio G, et al. Synthesis, debugging, and effects of synthetic chromosome consolidation:synVI and beyond[J]. Science, 2017, 355(6329): eaaf4831. DOI:10.1126/science.aaf4831 |
| [11] |
Wu Y, Li BZ, Zhao M, et al. Bug mapping and fitness testing of chemically synthesized chromosome X[J]. Science, 2017, 355(6329): eaaf4706. DOI:10.1126/science.aaf4706 |
| [12] |
Zhang W, Zhao G, Luo Z, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome[J]. Science, 2017, 355(6329): eaaf3981. DOI:10.1126/science.aaf3981 |
| [13] |
Hoess RH, Wierzbicki A, Abremski K. The role of the loxP spacer region in P1 site-specific recombination[J]. Nucleic Acids Res, 1986, 14(5): 2287-2300. DOI:10.1093/nar/14.5.2287 |
| [14] |
Shen Y, Stracquadanio G, Wang Y, et al. SCRaMbLE generates designed combinatorial stochastic diversity in synthetic chromosomes[J]. Genome Res, 2016, 26(1): 36-49. DOI:10.1101/gr.193433.115 |
| [15] |
Kannan K, Gibson DG. Yeast genome, by design[J]. Science, 2017, 355(6329): 1024-1025. DOI:10.1126/science.aam9739 |
| [16] |
Jia B, Wu Y, et al. Precise control of SCRaMbLE in synthetic hapl-oid and diploid yeast[J]. Nat Commun, 2018, 9(1): 1933. DOI:10.1038/s41467-018-03084-4 |
| [17] |
Johnston M, Flick JS, Pexton T. Multiple mechanisms provide rapid and stringent glucose repression of GAL gene expression in Saccharomyces cerevisiae[J]. Mol Cell Biol, 1994, 14(6): 3834-3841. DOI:10.1128/MCB.14.6.3834 |
| [18] |
Lindstrom DL, Gottschling DE. The mother enrichment program: a genetic system for facile replicative life span analysis in Saccharomyces cerevisiae[J]. Genetics, 2009, 183(2): 413-422, 1S-13S.
|
| [19] |
Hochrein L, Mitchell LA, Schulz K, et al. L-SCRaMbLE as a tool for light-controlled Cre-mediated recombination in yeast[J]. Nat Commun, 2018, 9(1): 1931. DOI:10.1038/s41467-017-02208-6 |
| [20] |
Luo Z, Wang L, Wang Y, et al. Identifying and characterizing SCRaMbLEd synthetic yeast using ReSCuES[J]. Nat Commun, 2018, 9(1): 1930. DOI:10.1038/s41467-017-00806-y |
| [21] |
Shen MJ, Wu Y, et al. Heterozygous diploid and interspecies SCRaMbLEing[J]. Nat Commun, 2018, 9(1): 1934. DOI:10.1038/s41467-018-04157-0 |
| [22] |
Liti G, Carter DM, et al. Population genomics of domestic and wild yeasts[J]. Nature, 2009, 458(7236): 337-341. DOI:10.1038/nature07743 |
| [23] |
Cubillos FA, Louis EJ, Liti G. Generation of a large set of genetically tractable haploid and diploid Saccharomyces strains[J]. Fems Yeast Res, 2009, 9(8): 1217-1225. DOI:10.1111/j.1567-1364.2009.00583.x |
| [24] |
Wang J, Xie ZX, Ma Y, et al. Ring synthetic chromosome V SCRaMbLE[J]. Nat Commun, 2018, 9(1): 3783. DOI:10.1038/s41467-018-06216-y |
| [25] |
Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast[J]. Science, 2015, 349(6252): 1095-1100. DOI:10.1126/science.aac9373 |
| [26] |
Wu Y, Zhu RY, Mitchell LA, et al. In vitro DNA SCRaMbLE[J]. Nat Commun, 2018, 9(1): 1935. DOI:10.1038/s41467-018-03743-6 |
| [27] |
Liu W, Luo Z, Wang Y, et al. Rapid pathway prototyping and engineering using in vitro and in vivo synthetic genome SCRaMbLE-in methods[J]. Nat Commun, 2018, 9(1): 1936. DOI:10.1038/s41467-018-04254-0 |
| [28] |
Blount BA, Gowers GF, Ho J, et al. Rapid host strain improvement by in vivo rearrangement of a synthetic yeast chromosome[J]. Nat Commun, 2018, 9(1): 1932. DOI:10.1038/s41467-018-03143-w |
| [29] |
Ma L, Li Y, Chen X, et al. SCRaMbLE generates evolved yeasts with increased alkali tolerance[J]. Microb Cell Fact, 2019, 18(1): 52. DOI:10.1186/s12934-019-1102-4 |
| [30] |
Li Y, Wu Y, Ma L, et al. Loss of heterozygosity by SCRaMbLEing[J]. Sci China Life Sci, 2019, 62(3): 381-393. DOI:10.1007/s11427-019-9504-5 |
| [31] |
van der Sloot A, Tyers M. Synthetic genomics:Rewriting the genome chromosome by chromosome[J]. Mol Cell, 2017, 66(4): 441-443. DOI:10.1016/j.molcel.2017.05.007 |




