酿酒酵母被用于制作面包和酿酒已有数千年的历史,是与人类关系最密切的微生物之一。由于其具有遗传背景清晰、生长迅速、抗逆性高、无噬菌体污染等优势,在生物制药、食品、化工和能源等领域均有广泛应用[1-2]。同时,酿酒酵母还是现代分子和细胞生物学研究的真核模式生物。
基因编辑和转录调控工具在酿酒酵母的基础研究和工业应用中起着十分重要的作用。细菌和古细菌中存在CRISPR/Cas9(Clustered regularly interspaced short palindromic repeats/CRISPR-associated protein 9)系统,可以特异性地降解外源遗传物质,进行适应性免疫防御。近年来,该系统被广泛开发应用于包括酿酒酵母在内不同物种的基因编辑[3-6]。CRISPR/Cas9系统由核酸内切酶Cas9(CRISPR-associated protein 9)、CRISPR RNA(crRNA)和反式激活RNA(trans-activating crRNA,tracrRNA)组成。其中,crRNA与tracrRNA形成二级结构并招募Cas9蛋白;crRNA和tracrRNA也可以融合为向导RNA(guide RNA,gRNA)以简化操作。Cas9蛋白与核酸结合的复合体可以特异性结合到PAM(Photospacer adjacent motif,NGG)位点相邻并与crRNA互补的DNA序列。接着,Cas9内切酶的HNH结构域切割与crRNA互补的DNA链,RuvC结构域切割另一条DNA链,形成DNA双链断裂(double-strand break,DSB),断裂位点位于PAM位点上游三个核苷酸的位置[7]。同源重组(Homologous recombination,HR)是酿酒酵母中主要的DNA修复方式。同源片段存在时,通过CRISPR/Cas9特异性引入DNA双链断裂,可对酿酒酵母基因组进行精准的敲除、敲入和突变[8-9]。
除基因编辑外,CRISPR系统在转录调控、表观遗传编辑、碱基编辑和细胞成像等方面也有广泛应用[10-12]。这些应用主要基于核酸内切酶活性缺陷的dCas9(Nuclease-deficient Cas9)蛋白,而这一突变体通过在Cas9的RuvC和HNH结构域分别引入D10A和H840A突变获得[13]。酿酒酵母中,将dCas9靶向基因的启动子区域,即有一定的转录抑制作用;但通过融合或募集不同类型的转录调控结构域,如转录抑制因子、激活因子或表观遗传修饰酶等[13-17],并结合精巧的gRNA设计[18-19],可以进一步实现多模式(Multi-mode)、多位点(Multiplex)、正交性(Orthogonal)的基因转录调控。
本文针对酿酒酵母中基于CRISPR/dCas9的基因转录调控工具的开发和应用进行综述。介绍了通过与不同转录调节结构域的结合来调节dCas9或gRNA的活性,概括了dCas9和gRNA表达的设计与优化方法;讨论了影响CRISPR/dCas9体系转录调控效率、特异性和通量的靶向性因素;总结了该工具在酿酒酵母代谢工程中的应用;最后,对该工具的未来研究方向提出了展望。
1 酿酒酵母中CRISPR/dCas9工具的开发 1.1 针对基因转录调控模式的设计与优化转录调控因子通常由DNA结合结构域和转录调控结构域组成。研究人员将dCas9或gRNA进行改造,使CRISPR/dCas9系统与转录抑制因子、激活因子或表观遗传修饰酶等相结合,从而将这些转录调控结构域靶向目的基因的特定位置,实现不同模式的基因调控[20](图 1)。

|
| A:gRNA引导与转录抑制或激活结构域融合的dCas9蛋白靶向目的位点调控基因转录水平。Mxi1:MAX-interacting protein 1;KRAB:Krüppel associated box;MIG1:Multicopy Inhibitor of GAL gene expression;TUP1:dTMP-UPtake;UME6:Unscheduled Meiotic gene Expression;CRT1(RFX1):Regulatory Factor X;XTC1(MHR1):Mitochondrial Homologous Recombination;B:将RNA适配子与gRNA融合,与dCas9共同招募与RNA结合蛋白融合的转录调控结构域至目的基因,从而进行靶基因的转录调控 图 1 通过融合或招募转录调节结构域调整dCas9或gRNA的活性 |
由于空间位阻效应,当gRNA引导dCas9靶向目的基因的特定位置时,能起到转录抑制作用,被称为CRISPR抑制(CRISPR interference,CRISPRi)[13, 21]。在细菌中单纯使用dCas9进行基因表达抑制的效率可高达99.9%[13],而在酿酒酵母中,单纯利用dCas9通常仅能起到50%-60%的抑制效果[18, 21-22],这样的抑制水平与其它真核生物中的报道一致[23-24]。这些实验结果表明,单独使用dCas9无法在真核细胞中高效抑制转录起始或阻挡RNA聚合酶的前进。另一方面,当dCas9与转录抑制结构域融合时,可显著提高其在真核体系中的转录抑制能力[25](图 1-A)。Gilbert等[23]将dCas9与哺乳动物来源的Mxi1蛋白的转录抑制结构域[26]相融合,发现对报告基因的表达抑制效果从单纯使用dCas9时的18倍提高至53倍。在解脂耶氏酵母中,dCas9-Mxi1相比dCas9把Ku80基因的转录抑制效率从38%提高至87%[27]。为了进一步优化CRISPRi在酿酒酵母中的效率,Lian等筛选了多个内源转录抑制因子[28-32],发现在与dCas9结合时,酿酒酵母TUP1、MIG1、XTC1、UME6等蛋白的结构域的转录抑制水平高于哺乳细胞Mxi1。他们同时发现将多个内源抑制结构域同时与dCas9融合时,可以进一步提高转录抑制效率。例如,当靶向酿酒酵母的组成型PTEF1启动子时,dCas9-Mix1、dCas9-TUP1和dCas9-TUP1-MIG1-UME6对于荧光报告基因表达的抑制效率分别为65%、74%和82%[19]。综合以上结果表明,与转录抑制结构域的融合能够显著提升dCas9在酿酒酵母中的转录抑制效率,并且可以利用内源转录因子、融合多重结构域等方式进一步提升抑制效率。
dCas9与转录激活结构域融合可实现对靶基因表达的上调,被称为CRISPR激活(CRISPR activation,CRISPRa)[33]。常用的转录激活结构域有VP16、VP64、p65AD和Rta等[34-36](图 1-A)。Farzadfard等[21]在酿酒酵母中将dCas9与VP64融合并靶向最小化PCYC1启动子,使报告基因的荧光强度提高了3倍左右。在dCas9-VP64的基础上融合多个转录激活结构域,可以进一步增强基因上调效率[36-38]。如Chavez等利用融合了3个转录激活结构域的dCas9-VPR(VP64-p65AD-Rta),使HED1和GAL7的表达分别提高了38倍和78倍,而dCas9-VP64仅使二者分别提高了9倍和14倍[36]。此外,对于不同的转录激活结构域,融合不同来源的CRISPR蛋白也会导致激活效率的差异。Lian等测试了4种不同来源的内切酶活性缺失Cas蛋白(dSpCas9,dSaCas9,dSt1Cas9,dLbCpf1)与3种转录激活结构域(VP64(V),VP64-p65AD(VP),VP64-p65AD-Rta(VPR))的所有组合,发现不同的dCas蛋白最优的激活结构域组合是不同的:对于dSpCas9,激活效率最好的是VPR;对于dSt1Cas9,激活效率最好的是V;对于dLbCpf1,激活效率最好的是VP;而对于dSaCas9,不同组合激活效果均比较微弱[19]。以上结果表明,dCas9与转录激活结构域的融合蛋白可有效上调靶基因转录水平,但不同dCas蛋白和激活结构域的组合需要分别进行优化。
1.1.2 gRNA融合适配子除了将转录调控结构域与dCas9蛋白融合外,还可以改造gRNA结构使其同时具有目的基因靶向序列和效应蛋白招募序列,从而利用RNA-蛋白质相互作用招募转录调控结构域[25]。Zalatan等和Kiani等将RNA适配子(RNA aptamer)与gRNA的3'末端的tracrRNA融合,另外将转录抑制或激活结构域与RNA适配子结合蛋白融合,通过RNA适配子与其结合蛋白的相互作用招募转录调控结构域,实现了靶基因的特异性转录调控(图 1-B)[39-40]。利用Scaffold RNA(scRNA)策略与融合VP64的RNA结合蛋白,Zalatan等在酿酒酵母中将一个合成型报告启动子激活了50倍,而dCas9-VP64的策略仅实现了2-3倍的激活[39]。与dCas9融合多个转录抑制或激活结构域策略类似,作者将多个适配子融合在同一个scRNA中,从而可招募多个RNA结合蛋白以增强其转录调控的能力[39]。另外,通过系统性测试,Zalatan等[39]发现了特异性的3个RNA适配子—结合蛋白的配对(MS2:MCP、PP7:PCP和com:COM,图 1-B),为多个结合蛋白与不同调控结构域融合构建正交性的转录调控因子奠定基础。综上,scRNA技术提供了一种与dCas9融合蛋白技术相互补的策略,可以对多基因靶标进行多模式调控,在基因互作研究、代谢通路组合式优化等方面有重要的应用价值。
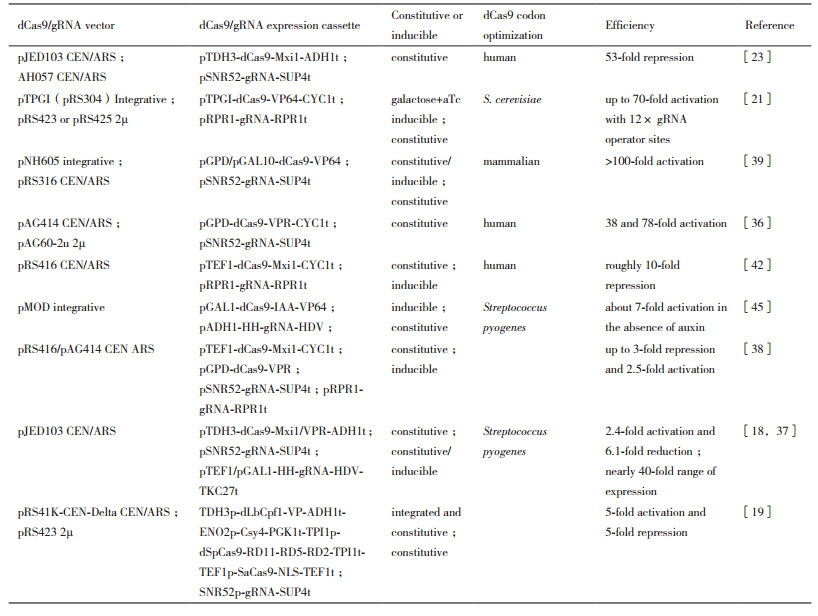
1.2 针对dCas9或gRNA表达的设计与优化优化dCas9蛋白的定位、表达水平和诱导表达等性质,可以提高CRISPR/dCas9系统的转录调控效率。dCas9需要定位到细胞核中以发挥转录调控的功能,通常情况下,会在dCas9的氨基端或羧基端融合一段SV40核定位序列(Nuclear localization sequence,NLS)[21, 41-42],也可在两端同时融合一段NLS序列[43]或在羧基端融合两段NLS序列[23]。优化dCas9蛋白表达量的手段包括调节密码子偏好、启动子强度和表达载体拷贝数等。由于不同物种同义密码子偏好性不同,密码子优化是提高重组蛋白异源表达效率的可能途径[44];酿酒酵母中,基于化脓性链球菌野生型、人或酿酒酵母密码子偏好性优化的dCas9序列均有所应用[21, 23, 38, 42, 45]。通常情况下,转录调控因子的表达量越高,对靶基因的抑制或激活的效率也越高[46]。因此,大部分研究使用了比较强的组成型启动子或糖酵解途径启动子来驱动dCas9的表达。例如,PTEF1、PTDH3、PGPD1和PPDC1等[23, 36, 42](表 1),但是也有研究表明强启动子驱动的dCas9的表达会降低酿酒酵母的适应度和生长速度[47]。对于dCas9基因的表达载体,既可将dCas9表达框整合到酿酒酵母的基因组中[21],也可将其克隆到自主复制质粒上[23, 38, 42, 45](表 1)。除了dCas9的组成型表达外,还可以使用诱导型启动子使其在化学小分子或光诱导条件下表达。例如,通过利用半乳糖和无水四环素诱导合成型启动子驱动dCas9-VP64的基因转录,Farzadfard等在酿酒酵母中将最小化PCYC1启动子驱动的荧光报告基因的转录水平提高了70倍[21, 48]。在蛋白稳定性方面,Khakhar等[45]通过融合降解子序列,实现了植物生长素诱导的SCF(Skp1-Cullin-F-box)泛素连接酶复合物依赖的dCas9蛋白降解。这些条件性表达或稳定性调控的策略进一步提高了CRISPR/dCas9系统在转录调控中应用的可控性[49-51]。
调节gRNA的转录水平也是优化CRISPR/dCas9转录调控性能的重要方法。首先,利用不同类型的天然启动子可以用来调节gRNA的转录水平。与RNA聚合酶相对应,真核生物启动子可分为Ⅰ型(rRNA)、Ⅱ型(mRNA)和Ⅲ型(snoRNA、tRNA等)。由于Ⅱ型启动子会在gRNA的5'和3'端添加额外的核苷酸从而干扰其正常功能,因此一般利用Ⅲ型启动子驱动gRNA的表达,如PSNR52和PRPR1等[6, 21, 23](表 1)。然而,由于Ⅲ型启动子的转录本长度较短,为了提高gRNA设计和表达的灵活性,Gao等[52]通过在gRNA两端分别融合锤头型核酶(Hammerhead-type ribozymes)和HDV核酶(Hepatitis delta virus)在酿酒酵母细胞中实现了由Ⅱ型PADH1启动子驱动的gRNA的表达(表 1)。这种含有核酸酶的gRNA称为RGR(Ribozyme-flanked gRNA)。基于该设计,Deaner等[18]利用Ⅱ型的PTEF1启动子将gRNA的转录水平比Ⅲ型的PSNR52启动子提高了3.88倍,显著提高了dCas9-VPR介导的转录抑制效率(gRNA靶点的位置效应,见下文)。而Gander等[53]利用最小化PCYC1启动子驱动RGR的转录,基于dCas9-Mxi1建立的非逻辑门显示了最小的泄漏并具有数字响应,为更加复杂的遗传电路研究奠定了基础。除组成型启动子外,还可以改造天然的Ⅲ型启动子使其具有对环境因素变化响应的能力,从而增强dCas9调控工具的可控性。例如,研究人员通过在Ⅲ型PRPR1启动子中整合Tet操纵子结合位点,实现无水四环素对gRNA的诱导转录,以及2-20倍的转录调控动态范围[21, 38, 42, 54]。这些不同启动子驱动的多种设计方案为gRNA的表达量、通量和对gRNA表达的控制提供了有力的工具和多样的选择。
1.3 针对靶向性能的设计与优化除了dCas9和gRNA元件的表达外,靶向性能也是决定CRISPR/dCas9基因调控能力的重要因素。具体而言,dCas9/gRNA复合体能否容易且准确地接近单个或多个目的区域的特定位点,决定了转录调控的有效性、准确性和多重性。以下将从gRNA的序列特征、靶点位置与可接近性、靶基因数量等3个方面总结了影响CRISPR/dCas9工具靶向性能的关键因素。
1.3.1gRNA的长度、序列特异性和二级结构gRNA中的向导序列(Guide sequence)与DNA靶序列互补配对,其长度和序列特异性对于CRISPR/dCas9系统的靶向特异性有重要影响(图 2-A)。在大肠杆菌的CRISPRi研究中,Qi等[13]对向导序列的长度进行了截短或延长,发现20 nt的原始长度是转录抑制效率最高的,并且随着该区域长度的截短导致转录抑制效率快速下降,最短的有效长度是12 nt;在哺乳动物细胞中,Kiani等[40]发现使用10-20 nt的向导序列均能使dCas9-VPR有效地发挥转录激活的功能。但是,在向导序列长度小于18 nt时Cas9介导的基因编辑功能基本缺失,表明转录调控相比基因编辑对向导序列的长度变化有更大的耐受性。对于向导序列的特异性,Qi等[13]对向导序列单点突变的筛选结果表明,PAM及距其最近的7个碱基对转录抑制至关重要。Smith等[42]在酿酒酵母中的研究结果与此一致,无论是全长还是截短型向导序列,距PAM最近的10 nt对dCas9-Mxi1介导的转录抑制效率影响较大。因此,虽然PAM远端的核苷酸为向导序列设计提供了一定的灵活性,但截短或错配均会降低CRISPR/dCas9介导的转录调控的效率。另外,Smith等[55]利用dCas9-Mxi1对靶向1 631个基因的超过18 000个gRNA的筛选实验发现,gRNA的靶向效率还与其二级结构相关(斯皮尔曼相关性系数为-0.11),即gRNA中向导序列、骨架序列和PRPR1启动子前导序列(Leader sequence)之间的碱基配对会降低gRNA的靶向效率,因此在设计gRNA时应尽量避免。

|
| A:gRNA向导序列的长度和序列特异性影响CRISPR/dCas9的转录调控效率;B:gRNA的靶点与TATA框和转录起始位点TSS的距离、所处的基因结构域影响CRISPR/dCas9的转录调控效率;C:gRNA的靶点附近是否有核小体占据影响CRISPR/dCas9的转录调控效率 图 2 影响CRISPR-dCas9系统靶向的因素 |
gRNA靶向的DNA单链和位点gRNA靶点也会对CRISPR/dCas9的转录调控效率产生影响,这些因素包括不同的DNA链、基因结构域、靶点与TATA框(TATA box)或转录起始位点(Transcription start site,TSS)的距离等。在酿酒酵母中,Farzadfard等[21]将dCas9-VP64靶向不同DNA链的相近位置,发现二者具有相似的转录调控水平。高通量的gRNA文库筛选研究也表明,gRNA的效率并没有DNA链的偏好性[33, 42]。在作用机制方面,dCas9-gRNA复合体不仅是作为转录过程的路障,更是共同建立了促进转录起始或终止的新环境[56]。
与DNA链特异性效应不同,gRNA靶点的位置效应非常明显(图 2-B)。对于dCas9-VP64或VPR等介导的转录激活,通常将gRNA靶向TATA框和TSS的上游;而对于dCas9融合蛋白介导的转录抑制,通常将gRNA靶向TATA框附近或其下游[21, 37]。在酿酒酵母中当gRNA靶向TSS上游约50-400 bp区域时dCas9-VPR介导的转录激活效率较高,与哺乳动物细胞中dCas9-SunTag:scFV-VP64的实验结果一致[33, 37]。当dCas9-VPR靶向TATA框上游50 bp之内时,可能由于抑制了RNA聚合酶与靶基因的结合,转录激活的效率急剧下降[37]。值得注意的是,当dCas9-VPR靶向开放阅读框(Open reading frame,ORF)区域时,反而起到了转录抑制的作用[18]。另一方面,Smith等利用dCas9-Mxi1测试了靶向20个酿酒酵母基因的约1 000个gRNA。结果显示,gRNA靶点位于TSS上游200 bp范围时转录抑制效率较高[42],进一步对超过1 500个基因的筛选将该区域缩小到TSS上游125 bp范围[55]。与之相比,Gilbert等[33]发现哺乳动物细胞中dCas9-KRAB依赖的最大转录抑制效率的靶点区域为TSS -50到+300 bp。利用gRNA靶点位置效应的特性,Deaner和Alper综合利用dCas9-VPR和dCas9-Mxi1对目标基因表达进行不同程度的下调和上调,从而实现了基因转录的梯度调控(Graded modulation)[37]。例如,针对PGPD1启动子,调控后报告荧光蛋白yECitrine的表达水平是对照组的0.2、0.6、0.7、3、4和8.5倍,动态范围超过40倍。然而,Jensen等[38]在TSS上游200 bp范围内设计了靶向12个酿酒酵母启动子的88个gRNA,其中一些gRNA对dCas-VPR或Mxi1介导的转录激活或抑制是无效的。因此,在选择CRISPR/dCas9系统的靶点时,除了宿主细胞的不同,还需在合适区域内设计多个gRNA进行具体的测试。
1.3.3 gRNA靶点的可接近性除了gRNA序列的本体特征和gRNA靶点所处的基因结构域外,gRNA靶点的可接近性也是CRISPR/dCas9系统有效发挥转录调控作用的重要因素,如染色质高级结构和核小体占据等(图 2-C)。酿酒酵母等真核生物中染色质以DNA缠绕组蛋白形成的核小体形式存在并具有复杂和动态的高级结构[57-58]。之前研究表明,靶向同一个启动子临近位置的不同gRNA具有不同的转录调控效率[21, 38, 42]。利用酿酒酵母高分辨率的核小体占据谱数据[59-60],Smith等发现在TSS上下游400 bp范围内gRNA的转录抑制效率与染色质可接近性呈正相关[42]。体外实验表明,核小体的占据抑制了Cas9蛋白与PAM的相互作用,当在体系中加入染色质重塑蛋白SNF2h或RSC复合物后,该抑制作用被削弱[61-62]。特别的是,在人类细胞系中CRISPR/dCas9依赖的转录抑制研究表明,有效的gRNA定位还与开放的染色质环境和转录活跃相关的组蛋白修饰如H3K9ac、H3K27ac、H3K4me2、H3K4me3、H3K79me2等有关[63-64]。综上,基于CRISPR/dCas9转录调控的效率受到靶点附近染色质微环境的影响,设计gRNA时应参考核小体占据和DNA可接近性的高分辨率图谱[59-60, 65-66],避免靶向核小体DNA的核心区域。
1.3.4 gRNA的数量与dCas9融合多个转录调控蛋白结构域或gRNA融合多个适配子的思路类似,靶向同一目的基因的多条gRNA能够协同调控靶基因的转录(图 3-A)。Farzadfard等[21]组合使用两条gRNA时将转录抑制的效率从2倍提高到了7倍,Deaner等[18]同样利用两条gRNA提高了dCas9-VPR介导的转录抑制水平。此外,还可以通过在启动子内人工合成相同的靶序列,实现多条单一gRNA介导的协同调控。Farzadfard等[21]利用含有12个gRNA结合位点的合成型启动子,将dCas9-VP64介导的转录激活提高了70倍,Gilbert等[23]利用含有7个Tet操纵子结合位点的合成型启动子,将dCas9介导的转录抑制提高了115倍。

|
| A:通过作用于同一靶点的多条gRNA实现协同性转录调控,提高CRISPR/dCas9的转录调控效率;B:利用scRNA及其正交性的RNA结合蛋白,同时实现多重靶基因的转录抑制和激活;C:利用正交性的Cas蛋白,在多个靶基因同时实现基因编辑、转录抑制和激活的多种功能 图 3 利用多条gRNA进行协同、多重或多模式转录调控 |
利用靶向多个目的基因的多条gRNA或scRNA可实现多重性(Multiplex)的转录调控(图 3-B)。Jensen等[38]利用dCas9与scRNA将Mxi1和VPR等转录调控结构域靶向类胡萝卜素和三酰甘油生物合成途径的多个基因,显著增加了相关产物的产量。Ferreira等[54]利用来自绿脓假单胞菌的核糖核酸内切酶Csy4,实现了PRPR1启动子驱动单条转录本中3个gRNA的转录和释放,实现了同时对OLE1,HGM1和ACS1等多个基因进行转录调控。此外,基于CRISRP/dCas9工具中组成元件相互作用的特异性,可以设计彼此正交的gRNA,在同一细胞中对多个目标基因实现不同模式的基因调控。例如,利用不同RNA适配子与其结合蛋白的特异性识别,Zalatan等[39]设计彼此正交、与不同转录调控因子结合的scRNA序列(图 3-B),在同一菌株中对紫色杆菌素合成通路的不同分支的基因进行转录抑制和转录激活,通过调控代谢流实现了不同产物的定向合成。利用不同Cas蛋白识别不同PAM序列的特点,Lian等[19]测试了不同来源的dCas蛋白和多种转录激活或抑制结构域的组合,构建了基于dLbCpf1-VP、dSpCas9-RD1152和SaCas9的酿酒酵母三相基因调控策略,称为CRISPR-AID(Transcriptional Activation,transcriptional Interference,and gene Deletion)。作者设计了彼此正交、与不同Cas蛋白对应的gRNA序列(图 3-C),在同一细胞中可以实现对多个目的基因进行转录抑制、转录激活、基因敲除等不同模式的调控。综上,多重gRNA的设计与使用能够提高单个靶基因的转录调控能力,实现多个靶基因的单向多重调控,还能够利用正交性设计系统实现靶基因的多重和多模式调控。通过增加可同时操作的基因靶点和调控模式,有助于从多个工程化改造靶点中快速找到最优的调控策略及其组合,即利用组合式优化为复杂基因线路、合成途径和细胞代谢的设计和改造提供有力工具。
2 酿酒酵母中CRISPR/dCas9工具在代谢工程中的应用在酿酒酵母细胞工厂的优化过程中,平衡内源或外源代谢通路的基因表达水平是提高目的产物产量的重要手段。CRISPR/dCas9转录调控工具可对多重基因的表达进行精细和多模式调控,从而通过代谢工程的理性设计和代谢靶点的高通量筛选等手段优化酿酒酵母的物质和能量流动,提高目的产物的产量和效率等。
首先,利用CRISPR/dCas9转录调控工具的多重基因靶向和多模式调控的优点,可对酿酒酵母代谢通路多种关键酶的基因表达进行理性设计。例如,上调或下调甲羟戊酸(Mevalonate,MVA)途径的关键基因HMG1、ERG9和ERG20等,能够提高类异戊二烯的产量[67-68]。Jensen等[38]利用dCas9和scRNA将转录调控结构域Mxi1或VPR靶向HMG1、HMG2、ERG9、ERG20、PGK1或TDH3等基因的启动子区域,通过测试多种转录调控的组合提高了类胡萝卜素的产量。而Lian等[19]利用CIRSPR-AID方法对酿酒酵母中甲羟戊酸途径限速酶基因HMG1的表达进行了上调,对处于β-胡萝卜素和甾醇生物合成分支点的必需基因ERG9的表达进行了下调,同时敲除了转录调节因子ROX1基因,使得β-胡萝卜素的产量提高了2.8倍。另外,Jensen等[38]将dCas9-VPR同时靶向OLE1和DGA1基因的启动子区域,使得三酰基甘油的产量提高了2倍以上。
其次,揭示基因型与表型的关系仍是代谢工程的一大挑战,由于具有易于设计使用和高通量的优点,CRISPR/dCas9可用于代谢通路靶基因表达强度与表型关系的的高通量筛选和研究。例如,Deaner和Alper利用dCas9-VPR或dCas9-Mxi1以及靶向启动子不同位置的gRNA文库建立了一种梯度式的基因转录调控工具,实现不同水平的基因表达上调和下调。他们将该方法用于系统性测试代谢通路中不同催化步骤对于调控干扰的敏感性(Systematically Test Enzyme Perturbation Sensitivities,STEPS),构建了甘油、3-脱氢莽草酸发酵和木糖分解的代谢通量敏感性图谱(Flux sensitivity map)[37]。通过对通量敏感性图谱进行迭代分析,依次鉴定了甘油发酵中GPD1、TPI1等基因编码的酶是合成通路中的限速步骤,并通过工程化改造菌株使甘油的产量从4.89g/L提高至28g/L。通过类似的方法,又将3-脱氢莽草酸的产量提高至126.4mg/L。Ferreira等[69]利用dCas9-VPR和靶向168个基因的3194个gRNA组成的文库,结合基于转录因子FapR的丙二酰辅酶A生物传感器,利用流平衡分析筛选了促进代谢流向丙二酰辅酶A的靶基因和gRNA。利用筛选的结果,使3-羟基丙酸的产量得到显著提高。
如上所述,由于具有多重靶基因多模式转录调控和易于高通量等优势,基于CRISPR/dCas9的基因转录调控工具在代谢靶点筛选、机制研究和代谢通路的工程化改造等方面具有广泛的应用,是基因编辑工具的重要补充。随着该工具的进一步开发和优化,基于CRISPR/dCas9的转录调控工具定能在微生物细胞工厂及其它领域有更加广泛的应用。
3 结论与展望综上所述,CRISPR/dCas9系统在酿酒酵母中提供了一种高效率和可编程的靶向基因转录调控策略。该策略包括如下优点:(1)gRNA易于设计和使用;(2)可实现多重靶基因转录调控;(3)可通过改变gRNA靶点的具体位置等实现梯度式基因转录调控;(4)可通过组合正交性的scRNA或dCas系统同时实现基因转录的激活和抑制。但是,目前酿酒酵母中CRISPR/dCas9介导的转录调控的效率普遍仍比大肠杆菌或哺乳动物细胞偏低[13, 23, 36],相关调控工具还需要进一步开发和优化。
就dCas元件而言,可以扩展dCas蛋白的类型、物种来源以及转录调控结构域类型等。除了广泛使用的SpCas9外,Lian等[19]测试了其它物种来源的Cas9蛋白,如NmCas9、St1Cas9、SaCas9,以及两种物种来源的Cpf1,即AsCpf1、LbCpf1,并系统的比较了它们在基因编辑、转录抑制或激活方面的性能。这不仅扩大了PAM位点的选择范围,还为转录调控的优化提供了借鉴。此外,除了常用的Mxi1、KRAB和VPR外,Lian等测试并发现来源于UME6、MIG1和TUP1的结构域组合具有更强的转录抑制作用。Keung等[70]基于可编程的锌指蛋白测试了213种酿酒酵母染色质调节因子,并对它们的转录调控作用模式进行了系统分析,为多样化转录调控结构域提供了参考。而且,DNA甲基化、组蛋白修饰等表观遗传学机制同样在基因转录等生物学过程中起着多样化而且重要的作用[71]。利用dCas9与表观遗传修饰酶全长或活性结构域的融合进行靶向表观遗传修饰的编辑,可对基因表达进行调控以实现特定的生物学效应,在深化理解哺乳动物细胞的生物学过程、开发基因治疗或细胞治疗的工具中有广泛应用[72-74]。酿酒酵母作为单细胞真核生物,有丰富的组蛋白修饰和组蛋白修饰酶,并广泛参与到DNA转录调控中[75]。然而,虽然在哺乳动物中通过表观遗传编辑进行转录调控已有很多应用案例,但在酿酒酵母中的研究仍十分匮乏,这也为酿酒酵母转录调控的表观遗传编辑工具开发和应用指明了一个重要方向。
就gRNA元件而言,虽然CHOPCHOP[76]等计算机辅助设计工具被应用于转录调控gRNA的设计当中,但相关算法以基因编辑为核心,对CRISPR/dCas9介导转录调控效率的影响因素并未有充分考虑。针对这一局限,Smith等[42]开发了用于酵母CRISPRi的gRNA设计工具(http://lp2.github.io/yeast-crispri/),算法原理涉及基因组位置、染色质可接近性、核小体占据、gRNA长度与序列和转录因子占据等。未来的设计算法开发还应综合考虑各类转录调节结构域及结合方式、不同dCas蛋白及PAM序列、靶点染色质微环境、菌株背景等因素,且需要依据全基因组范围大规模筛选等实验结果进行优化。目前,dCas9转录激活在酿酒酵母中尚无gRNA辅助设计工具。另外值得注意的是,通常CRISPR/dCas9系统的转录激活效率与靶基因的基础表达呈负相关,即靶基因基础表达水平越高,对其转录增强的效率越低[36, 38]。因此,还需针对每个靶基因设计特异性的、更为有效的转录调控方法。
| [1] |
Nielsen J, Larsson C, van Maris A, et al. Metabolic engineering of yeast for production of fuels and chemicals[J]. Current Opinion in Biotechnology, 2013, 24: 398-404. DOI:10.1016/j.copbio.2013.03.023 |
| [2] |
Kavscek M, Strazar M, Curk T, et al. Yeast as a cell factory:current state and perspectives[J]. Microbial Cell Factories, 2015, 14: 94. DOI:10.1186/s12934-015-0281-x |
| [3] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337: 816-821. DOI:10.1126/science.1225829 |
| [4] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339: 819-823. DOI:10.1126/science.1231143 |
| [5] |
Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339: 823-826. DOI:10.1126/science.1232033 |
| [6] |
DiCarlo JE, Norville JE, Mali P, et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J]. Nucleic Acids Research, 2013, 41: 4336-4343. DOI:10.1093/nar/gkt135 |
| [7] |
Jiang F, Doudna JA. CRISPR-Cas9 structures and mechanisms[J]. Annual Review of Biophysics, 2017, 46: 505-529. DOI:10.1146/annurev-biophys-062215-010822 |
| [8] |
Tarasava K, Oh EJ, Eckert CA, et al. CRISPR-enabled tools for engineering microbial genomes and phenotypes[J]. Biotechnology Journal, 2018, 13: e1700586. DOI:10.1002/biot.201700586 |
| [9] |
Mitsui R, Yamada R, Ogino H. CRISPR system in the yeast Saccharomyces cerevisiae and its application in the bioproduction of useful chemicals[J]. World Journal of Microbiology & Biotechnology, 2019, 35: 111. |
| [10] |
Dominguez AA, Lim WA, Qi LS. Beyond editing:repurposing CRISPR-Cas9 for precision genome regulation and interrogation[J]. Nature Reviews Molecular Cell Biology, 2016, 17: 5-15. DOI:10.1038/nrm.2015.2 |
| [11] |
Fu Y, Rocha PP, Luo VM, et al. CRISPR-dCas9 and sgRNA scaffolds enable dual-colour live imaging of satellite sequences and repeat-enriched individual loci[J]. Nature Communications, 2016, 7: 11707. DOI:10.1038/ncomms11707 |
| [12] |
Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J]. Science, 2016, 53(6305): pii:aaf8729. |
| [13] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152: 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [14] |
Larson MH, Gilbert LA, Wang X, et al. CRISPR interference(CRISPRi)for sequence-specific control of gene expression[J]. Nature Protocols, 2013, 8: 2180-2196. DOI:10.1038/nprot.2013.132 |
| [15] |
Perez-Pinera P, Kocak DD, Vockley CM, et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors[J]. Nature Methods, 2013, 10: 973-976. DOI:10.1038/nmeth.2600 |
| [16] |
Maeder ML, Linder SJ, Cascio VM, et al. CRISPR RNA-guided activation of endogenous human genes[J]. Nature Methods, 2013, 10: 977-979. DOI:10.1038/nmeth.2598 |
| [17] |
Cheng AW, Wang H, Yang H, et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system[J]. Cell Research, 2013, 23: 1163-1171. DOI:10.1038/cr.2013.122 |
| [18] |
Deaner M, Mejia J, Alper HS. Enabling Graded and Large-Scale Multiplex of Desired Genes Using a Dual-Mode dCas9 Activator in Saccharomyces cerevisiae[J]. ACS Synthetic Biology, 2017, 6: 1931-1943. DOI:10.1021/acssynbio.7b00163 |
| [19] |
Lian J, HamediRad M, Hu S, et al. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system[J]. Nature Communications, 2017, 8: 1688. DOI:10.1038/s41467-017-01695-x |
| [20] |
Khalil AS, Lu TK, Bashor CJ, et al. A synthetic biology framework for programming eukaryotic transcription functions[J]. Cell, 2012, 150: 647-658. DOI:10.1016/j.cell.2012.05.045 |
| [21] |
Farzadfard F, Perli SD, Lu TK. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas[J]. ACS Synthetic Biology, 2013, 2: 604-613. DOI:10.1021/sb400081r |
| [22] |
Vanegas KG, Lehka BJ, Mortensen UH. SWITCH:a dynamic CRISPR tool for genome engineering and metabolic pathway control for cell factory construction in Saccharomyces cerevisiae[J]. Microbial Cell Factories, 2017, 16: 25. DOI:10.1186/s12934-017-0632-x |
| [23] |
Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell, 2013, 154: 442-451. DOI:10.1016/j.cell.2013.06.044 |
| [24] |
Lawhorn IE, Ferreira JP, Wang CL. Evaluation of sgRNA target sites for CRISPR-mediated repression of TP53[J]. PLoS One, 2014, 9: e113232. DOI:10.1371/journal.pone.0113232 |
| [25] |
Jensen MK. Design principles for nuclease-deficient CRISPR-based transcriptional regulators[J]. FEMS Yeast Research, 2018, 18. |
| [26] |
Schreiber-Agus N, Chin L, Chen K, et al. An amino-terminal domain of Mxi1 mediates anti-Myc oncogenic activity and interacts with a homolog of the yeast transcriptional repressor SIN3[J]. Cell, 1995, 80: 777-786. DOI:10.1016/0092-8674(95)90356-9 |
| [27] |
Schwartz C, Frogue K, Ramesh A, et al. CRISPRi repression of nonhomologous end-joining for enhanced genome engineering via homologous recombination in Yarrowia lipolytica[J]. Biotechnology and Bioengineering, 2017, 114: 2896-2906. DOI:10.1002/bit.26404 |
| [28] |
Edmondson DG, Smith MM, Roth SY. Repression domain of the yeast global repressor Tup1 interacts directly with histones H3 and H4[J]. Genes & Development, 1996, 10: 1247-1259. |
| [29] |
Ostling J, Carlberg M, Ronne H. Functional domains in the Mig1 repressor[J]. Molecular and Cellular Biology, 1996, 16: 753-761. DOI:10.1128/MCB.16.3.753 |
| [30] |
Kadosh D, Struhl K. Repression by Ume6 involves recruitment of a complex containing Sin3 corepressor and Rpd3 histone deacetylase to target promoters[J]. Cell, 1997, 89: 365-371. DOI:10.1016/S0092-8674(00)80217-2 |
| [31] |
Zhang Z, Reese JC. Molecular genetic analysis of the yeast repressor Rfx1/Crt1 reveals a novel two-step regulatory mechanism[J]. Molecular and Cellular Biology, 2005, 25: 7399-7411. DOI:10.1128/MCB.25.17.7399-7411.2005 |
| [32] |
Traven A, Staresincic L, Arneric M, et al. The yeast protein Xtc1 functions as a direct transcriptional repressor[J]. Nucleic Acids Research, 2002, 30: 2358-2364. DOI:10.1093/nar/30.11.2358 |
| [33] |
Gilbert LA, Horlbeck MA, Adamson B, et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation[J]. Cell, 2014, 159: 647-661. DOI:10.1016/j.cell.2014.09.029 |
| [34] |
Beerli RR, Segal DJ, Dreier B, et al. Toward controlling gene expression at will:specific regulation of the erbB-2/HER-2 promoter by using polydactyl zinc finger proteins constructed from modular building blocks[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95: 14628-14633. DOI:10.1073/pnas.95.25.14628 |
| [35] |
Schmitz ML, Baeuerle PA. The p65 subunit is responsible for the strong transcription activating potential of NF-kappa B[J]. EMBO Journal, 1991, 10: 3805-3817. DOI:10.1002/j.1460-2075.1991.tb04950.x |
| [36] |
Chavez A, Scheiman J, Vora S, et al. Highly efficient Cas9-mediated transcriptional programming[J]. Nature Methods, 2015, 12: 326-328. DOI:10.1038/nmeth.3312 |
| [37] |
Deaner M, Alper HS. Systematic testing of enzyme perturbation sensitivities via graded dCas9 modulation in Saccharomyces cerevisiae[J]. Metabolic Engineering, 2017, 40: 14-22. DOI:10.1016/j.ymben.2017.01.012 |
| [38] |
Jensen ED, Ferreira R, Jakociunas T, et al. Transcriptional reprogramming in yeast using dCas9 and combinatorial gRNA strategies[J]. Microbial Cell Factories, 2017, 16: 46. DOI:10.1186/s12934-017-0664-2 |
| [39] |
Zalatan JG, Lee ME, Almeida R, et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds[J]. Cell, 2015, 160: 339-350. DOI:10.1016/j.cell.2014.11.052 |
| [40] |
Kiani S, Chavez A, Tuttle M, et al. Cas9 gRNA engineering for genome editing, activation and repression[J]. Nature Methods, 2015, 12: 1051-1054. DOI:10.1038/nmeth.3580 |
| [41] |
Reider Apel A, d'Espaux L, Wehrs M, et al. A Cas9-based toolkit to program gene expression in Saccharomyces cerevisiae[J]. Nucleic Acids Research, 2017, 45: 496-508. DOI:10.1093/nar/gkw1023 |
| [42] |
Smith JD, Suresh S, Schlecht U, et al. Quantitative CRISPR interference screens in yeast identify chemical-genetic interactions and new rules for guide RNA design[J]. Genome Biology, 2016, 17: 45. DOI:10.1186/s13059-016-0900-9 |
| [43] |
Bao Z, Xiao H, Liang J, et al. Homology-integrated CRISPR-Cas(HI-CRISPR)system for one-step multigene disruption in Saccharomyces cerevisiae[J]. ACS Synthetic Biology, 2015, 4: 585-594. DOI:10.1021/sb500255k |
| [44] |
Juturu V, Wu JC. Heterologous Protein Expression in Pichia pastoris:Latest Research Progress and Applications[J]. Chembiochem, 2018, 19: 7-21. DOI:10.1002/cbic.201700460 |
| [45] |
Khakhar A, Bolten NJ, Nemhauser J, et al. Cell-Cell Communication in Yeast Using Auxin Biosynthesis and Auxin Responsive CRISPR Transcription Factors[J]. ACS Synthetic Biology, 2016, 5: 279-286. DOI:10.1021/acssynbio.5b00064 |
| [46] |
Skjoedt ML, Snoek T, Kildegaard KR, et al. Engineering prokaryotic transcriptional activators as metabolite biosensors in yeast[J]. Nature Chemical Biology, 2016, 12: 951-958. DOI:10.1038/nchembio.2177 |
| [47] |
Ryan OW, Skerker JM, Maurer MJ, et al. Selection of chromosomal DNA libraries using a multiplex CRISPR system[J]. eLife, 2014, 3. |
| [48] |
Ellis T, Wang X, Collins JJ. Diversity-based, model-guided construction of synthetic gene networks with predicted functions[J]. Nature Biotechnology, 2009, 27: 465-471. DOI:10.1038/nbt.1536 |
| [49] |
Polstein LR, Gersbach CA. A light-inducible CRISPR-Cas9 system for control of endogenous gene activation[J]. Nature Chemical Biology, 2015, 11: 198-200. DOI:10.1038/nchembio.1753 |
| [50] |
Gao Y, Xiong X, Wong S, et al. Complex transcriptional modulation with orthogonal and inducible dCas9 regulators[J]. Nature Methods, 2016, 13: 1043-1049. DOI:10.1038/nmeth.4042 |
| [51] |
Oakes BL, Nadler DC, Flamholz A, et al. Profiling of engineering hotspots identifies an allosteric CRISPR-Cas9 switch[J]. Nature Biotechnology, 2016, 34: 646-651. DOI:10.1038/nbt.3528 |
| [52] |
Gao Y, Zhao Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing[J]. Journal of Integrative Plant Biology, 2014, 56: 343-349. DOI:10.1111/jipb.12152 |
| [53] |
Gander MW, Vrana JD, Voje WE, et al. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates[J]. Nature Communications, 2017, 8: 15459. DOI:10.1038/ncomms15459 |
| [54] |
Ferreira R, Skrekas C, Nielsen J, et al. Multiplexed CRISPR/Cas9 Genome Editing and Gene Regulation Using Csy4 in Saccharomyces cerevisiae[J]. ACS Synthetic Biology, 2018, 7: 10-15. DOI:10.1021/acssynbio.7b00259 |
| [55] |
Smith JD, Schlecht U, Xu W, et al. A method for high-throughput production of sequence-verified DNA libraries and strain collections[J]. Molecular Systems Biology, 2017, 13: 913. DOI:10.15252/msb.20167233 |
| [56] |
Howe FS, Russell A, Lamstaes AR, et al. CRISPRi is not strand-specific at all loci and redefines the transcriptional landscape[J]. eLife, 2017, 6.
|
| [57] |
Luger K, Mader AW, Richmond RK, et al. Crystal structure of the nucleosome core particle at 2. 8 A resolution[J]. Nature, 1997, 389: 251-260. DOI:10.1038/38444 |
| [58] |
Yadav T, Quivy JP, Almouzni G. Chromatin plasticity:A versatile landscape that underlies cell fate and identity[J]. Science, 2018, 361: 1332-1336. DOI:10.1126/science.aat8950 |
| [59] |
Yuan GC, Liu YJ, Dion MF, et al. Genome-scale identification of nucleosome positions in S. cerevisiae[J]. Science, 2005, 309: 626-630. DOI:10.1126/science.1112178 |
| [60] |
Lee W, Tillo D, Bray N, et al. A high-resolution atlas of nucleosome occupancy in yeast[J]. Nature Genetics, 2007, 39: 1235-1244. DOI:10.1038/ng2117 |
| [61] |
Hinz JM, Laughery MF, Wyrick JJ. Nucleosomes Inhibit Cas9 Endonuclease Activity in Vitro[J]. Biochemistry, 2015, 54: 7063-7066. DOI:10.1021/acs.biochem.5b01108 |
| [62] |
Isaac RS, Jiang F, Doudna JA, et al. Nucleosome breathing and remodeling constrain CRISPR-Cas9 function[J]. eLife, 2016, 5. |
| [63] |
Horlbeck MA, Gilbert LA, Villalta JE, et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation[J]. eLife, 2016, 5. |
| [64] |
Radzisheuskaya A, Shlyueva D, Muller I, et al. Optimizing sgRNA position markedly improves the efficiency of CRISPR/dCas9-mediated transcriptional repression[J]. Nucleic Acids Research, 2016, 44: e141. DOI:10.1093/nar/gkw583 |
| [65] |
Jiang C, Pugh BF. A compiled and systematic reference map of nucleosome positions across the Saccharomyces cerevisiae genome[J]. Genome Biology, 2009, 10: R109. DOI:10.1186/gb-2009-10-10-r109 |
| [66] |
Schep AN, Buenrostro JD, Denny SK, et al. Structured nucleosome fingerprints enable high-resolution mapping of chromatin architecture within regulatory regions[J]. Genome Research, 2015, 25: 1757-1770. DOI:10.1101/gr.192294.115 |
| [67] |
Xie W, Ye L, Lv X, et al. Sequential control of biosynthetic pathways for balanced utilization of metabolic intermediates in Saccharomyces cerevisiae[J]. Metabolic Engineering, 2015, 28: 8-18. DOI:10.1016/j.ymben.2014.11.007 |
| [68] |
Verwaal R, Wang J, Meijnen JP, et al. High-level production of beta-carotene in Saccharomyces cerevisiae by successive transformation with carotenogenic genes from Xanthophyllomyces dendrorhous[J]. Applied and Environmental Microbiology, 2007, 73: 4342-4350. DOI:10.1128/AEM.02759-06 |
| [69] |
Ferreira R, Skrekas C, Hedin A, et al. Model-Assisted Fine-Tuning of Central Carbon Metabolism in Yeast through dCas9-Based Regulation[J]. ACS Synthetic Biology, 2019, 8: 2457-2463. DOI:10.1021/acssynbio.9b00258 |
| [70] |
Keung AJ, Bashor CJ, Kiriakov S, et al. Using targeted chromatin regulators to engineer combinatorial and spatial transcriptional regulation[J]. Cell, 2014, 158: 110-120. DOI:10.1016/j.cell.2014.04.047 |
| [71] |
Kouzarides T. Chromatin modifications and their function[J]. Cell, 2007, 128: 693-705. DOI:10.1016/j.cell.2007.02.005 |
| [72] |
Thakore PI, Black JB, Hilton IB, et al. Editing the epigenome:technologies for programmable transcription and epigenetic modulation[J]. Nature Methods, 2016, 13: 127-137. DOI:10.1038/nmeth.3733 |
| [73] |
Pulecio J, Verma N, Mejia-Ramirez E, et al. CRISPR/Cas9-Based Engineering of the Epigenome[J]. Cell Stem Cell, 2017, 21: 431-447. DOI:10.1016/j.stem.2017.09.006 |
| [74] |
Pickar-Oliver A, Gersbach CA. The next generation of CRISPR-Cas technologies and applications[J]. Nature Reviews Molecular Cell Biology, 2019, 20: 490-507. DOI:10.1038/s41580-019-0131-5 |
| [75] |
Grunstein M, Gasser SM. Epigenetics in Saccharomyces cerevisiae[J]. Cold Spring Harbor Perspectives in Biology, 2013, 5. |
| [76] |
Labun K, Montague TG, Krause M, et al. CHOPCHOP v3:expanding the CRISPR web toolbox beyond genome editing[J]. Nucleic Acids Research, 2019, 47: W171-W174. DOI:10.1093/nar/gkz365 |