2. 吉林省农业科学院农业生物技术研究所,长春 130033;
3. 吉林省农业生物技术重点实验室 吉林省农业科学院,长春 130033;
4. 吉林师范大学生命科学学院,四平 136000
2. Agro-Biotechnology Research Institute, Jilin Academy of Agricultural Sciences, Changchun 130033;
3. Jilin Provincial Key Laboratory of Agricultural Biotechnology, Changchun 130033;
4. School of Life Science, Jilin Normal University, Siping 136000
香味品质已经成为消费者选择稻米的重要指标。然而,市售优质香稻品种较少,特别是在口感良好的粳稻品种中,香味品质突出的则更为稀缺。因此,对优质粳稻材料进行改良,提升其香味品质,将具有广阔的市场前景。1979年Yajima等[1]在米饭中检测到114种具有香味的挥发性化合物。其中,2-乙酰基-1-吡咯啉(2-acetyl-1-pyrroline,2-AP)是影响稻米香味的主要化合物[2]。2-AP前体物质Δ-1-吡咯啉(1-pyrroline)的代谢是由甜菜醛脱氢酶Ⅱ(Betaine aldehyde dehydrogenase 2,BADH2)催化的,水稻内源BADH2活性降低会导致2-AP含量增加,进而会提升水稻的香味[3]。水稻OsBADH2基因现已成为改良水稻香味品质的一个关键目标基因。
CRISPR/Cas9(Clustered regularly interspaced short palindromic repeats/CRISPR-associated nuclease 9,Cas9)技术是目前应用最为广泛的基因组编辑技术[4]。因其具有准确性高、细胞毒性小、可同时对多个目标基因进行编辑等优势,已被应用在水稻等主要作物的遗传改良中[5]。目前已有报道使用CRISPR/Cas9技术在水稻中编辑OsBADH2基因,并获得了badh2突变体材料[6-9]。这些研究均采用了农杆菌介导的稳定遗传转化方法,这种方法需要进行4-5周的抗性筛选,而利用这一方法获得T0材料后,至少需经过一次自交才能在后代中获得无转基因成分的基因编辑后代材料,并且仍需要进行一代甚至多代的回交,才能最终获得编辑靶点纯合的突变材料,突变材料创制和筛选耗时较长。2016年,高彩霞课题组利用基因枪介导的瞬时转化方法,在小麦中建立了高效的CRISPR/Cas9基因编辑体系[10]。该体系为快速获得基因组编辑粳稻材料提供了新思路。另外,基因编辑后代材料的分子检测也是基因组编辑体系中的重要环节。目前,利用PCR扩增、限制酶酶切和测序相结合的检测方法应用较多,但仍存在着耗时长、成本高等不足。而随着高分辨率溶解曲线(High resolution melting,HRM)技术被引入到在基因编辑后代材料的分子检测[11-12],有效的降低了成本,提高了检测效率。因此,本研究通过基因枪介导的瞬时转化体系对“吉粳88”进行基因编辑,敲除调控水稻香味的OsBADH2基因,并利用HRM技术对基因编辑后代材料进行分子检测,预期在T1代获得无转基因成分且编辑位点纯合的“吉粳88”badh2突变体,为香味粳稻育种提供新的种质资源。同时,也对基于瞬时表达的粳稻基因组编辑技术体系进行探索和完善。
1 材料与方法 1.1 材料以粳稻“吉粳88”为转化材料,转化实验于2017-2018年在吉林省农业科学院完成。基因编辑后代材料在吉林省农业科学院公主岭院区温室中盆栽种植。大肠杆菌(DH5α)感受态细胞,基因编辑载体均来源于吉林省农业科学院。实验所用引物的合成以及PCR产物测序工作由吉林省库美生物科技有限公司完成。
1.2 方法 1.2.1 OsBADH2靶点的选择首先,对“吉粳88”的OsBADH2基因进行全长扩增(引物见表 1)并测序,测序结果与NCBI数据库中的OsBADH2基因参考序列(Gene ID:105128600)比对,确定保守区域;进而使用在线工具CRISPR-GE(http://skl.scau.edu.cn/)在基因保守区域中选择候选靶点;最后,为避免出现脱靶,在水稻全基因组数据库RADB(https://rapdb.dna.affrc.go.jp)以及NCBI数据库中对候选靶点进行特异性检验,最终确定靶点。
分别构建含有不同靶点序列的gRNA表达盒,表达盒中的靶点序列由U6启动子驱动,并在sgRNA scaffold后添加NOS终止密码。Xba Ⅰ和Pst Ⅰ双酶切后,将gRNA表达盒连入pJSCRISPR/Cas9载体(图 2)。
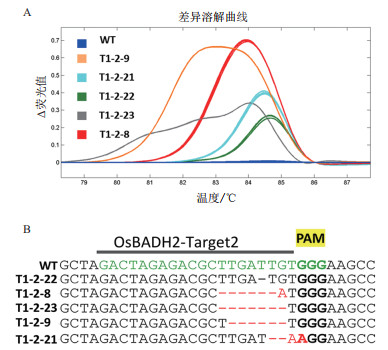
|
| A:差异溶解曲线;B:编辑后OsBADH2靶点序列 图 2 突变材料的HRM分析与测序验证 |
去颖壳的“吉粳88”籽粒经1 ‰的升汞溶液浸泡处理8 min后,无菌水清洗5次。消毒后的种子在NS和NS1培养基(表 2)中分别进行愈伤组织诱导和继代。使用基因枪法对所获愈伤组织进行遗传转化。转化后的愈伤组织在NS1培养基(表 2)上恢复培养48 h。恢复处理后的愈伤组织于MD培养基(表 2)上诱导成苗。将株高约1.5-2 cm的T0代再生苗转至MR培养基(表 2)诱导生根。参照李楠等[13]的粳稻愈伤组织的培养方法,培养条件为全程光照,光照强度1 600 lux,室温29℃。挑选根系茁壮的T0植株在温室中炼苗2-3 d后移栽。
为检测靶点的编辑情况,针对不同靶点,设计特异的HRM检测引物(表 1),并进行PCR扩增。PCR反应体系为:2× Phanta Max Master mix 10 μL;上下游引物各0.5 μL;EvaGreen 0.5 μL;DNA模板1 μL;水7.5 μL。PCR反应程序为:95℃ 3 min;95℃ 10 s,58℃ 10 s,72℃ 10 s,共35个循环;72℃ 5 min;95℃ 30 s后,以每秒0.1℃降温至40℃;4℃保存。使用LightScanner(Idaho Technology,美国)进行HRM分析,根据溶解曲线差异,将鉴定为序列发生变化的PCR扩增产物,进行测序验证,确定靶点编辑类型。
2 结果 2.1 基因编辑靶点的选择使用CRISPR-GE最终在OsBADH2基因的第2、第3和第4外显子区选择了3个编辑靶点(图 1-A),并分别合成了gRNA表达盒连接入pJSCRISPR/Cas9载体中(图 1-B)。
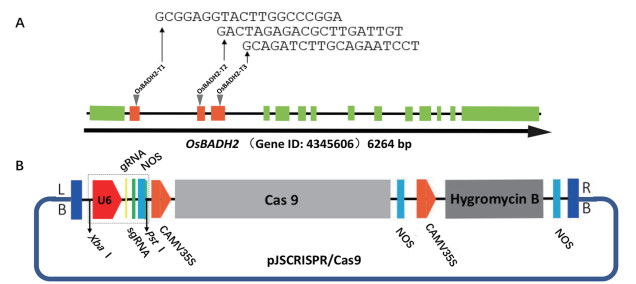
|
| A:OsBADH2中的3个靶位点;B:靶点gRNA表达盒与pJSCRISPR/Cas9表达载体 图 1 pJSCRISPR/Cas9表达载体与基因编辑靶位点 |
通过对李楠等[13]的粳稻组培再生体系进行优化,建立了一套仅需约70 d,即可完成从成熟胚诱导愈伤到最终获得T0代阳性植株的组培再生体系。该体系仅使用了NS、NS1、MD、MR四种培养基(表 2)。首先,成熟胚在NS培养基中诱导培养7-10 d即可获得淡黄色且蓬松的愈伤组织;再经过7-10 d的继代培养,愈伤组织可增殖一倍以上。在MD培养基上仅需14 d的分化培养即可获得分化苗,每皿12块初始愈伤经分化培养后,可获得8-20株分化苗;在MR培养基上,分化苗的生根率为100%,且易于移栽,移栽成活率也可达到100%。利用基因枪法介导的瞬时转化,组培过程中省略了抗生素的筛选,因此获得分化苗的周期并未增长。最终,获得靶点1的T0代基因编辑再生苗270株,靶点2的基因编辑T0代再生苗296株,靶点3的基因编辑T0代再生苗281株。
2.3 基因编辑材料的分子检测为快速检测所获得的后代材料是否在设计靶点位置发生了编辑,本研究利用HRM检测技术对T0和T1代材料进行了分子鉴定。HRM检测结果(图 2)显示,后代材料的目标片段PCR扩增产物的溶解曲线可清晰的区分突变体与野生型。经测序验证,HRM检测鉴定出的突变体,其靶点序列均发生了编辑。编辑类型主要为单碱基或多碱基缺失,以及部分碱基的替换。
为获得编辑位点纯合突变且无转基因成分的基因编辑后代材料,我们对T1代材料进行了潮霉素抗性基因的PCR检测。潮霉素检测为阴性的样品,进一步利用HRM分析结合测序验证,检测编辑靶点的纯合性。结果显示3个编辑靶点的T1代材料中,均获得了纯合靶点编辑且无转基因成分的基因编辑材料,其中靶点1编辑有3类,靶点2编辑有2类,靶点3编辑有2类(图 3)。HRM检测不但能将编辑靶点上发生多碱基缺失的材料(如:T1-C-2-16)与野生型材料清晰的区分,而且对编辑靶点只发生单碱基缺失的材料(如T1-C-2-22),也能明确的检出(图 4)。说明HRM检测非常适用于基因编辑后代材料的检测。
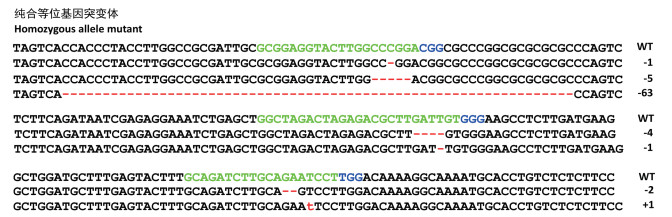
|
| Cas9识别靶点(绿色),PAM(protospacer-adjacent motif)序列(蓝色),删除和插入分别用红色“-”和红色小写字母表示 图 3 T1纯合等位基因突变体比对分析 |
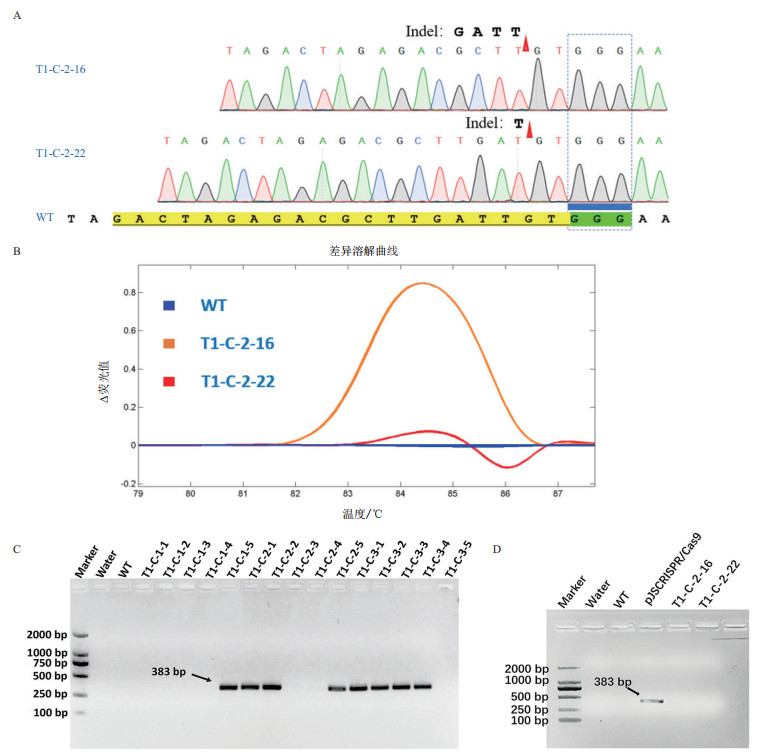
|
| A:纯合编辑位点的测序峰图;B:差异溶解曲线;C:潮霉素抗性基因PCR检测结果;D:纯合样品的潮霉素抗性基因PCR检测结果 图 4 纯合无转基因成分编辑材料的分子验证 |
“吉粳88”是稻米品质达到国家一级标准的粳稻品种。2005年至今,由其作为育种亲本而获得认定的粳稻优质品种共有72个。进一步改良吉粳88的香味品质,迎合市场对稻香米的需求,具有重要的育种价值。CRISPR/Cas9技术相对于锌指蛋白(Zinc-finger nucleases,ZFNs)技术和TALEN核酸酶(Transcription activator like effector nucleases,TALENs)技术,更为简单、高效,在植物基因组编辑的研究领域已得到了广泛的应用[15]。目前,对水稻OsBADH2基因进行CRISPR/Cas9基因编辑的研究已有报道[6-9],而这些研究均选择了农杆菌介导的稳定遗传转化方案。农杆菌介导的遗传转化,必须要经过转化后的抑菌以及对愈伤组织的抗性筛选等过程,最终获得基因编辑突变体通常需要2-3年的时间。相对于农杆菌转化而言基因枪转化容易导入多个拷贝,因此在转基因转化中不被广泛使用。鉴于CRISPR/Cas9技术不需要载体整合到基因组持续发挥功能这一特点,在转化阶段多拷贝的导入反而增大导入片段发挥功能的概率,因此我们认为基因枪转化与植物基因组编辑具有更高的契合度。2016年,高彩霞课题组在小麦中建立了高效的CRISPR/Cas9基因编辑体系,该体系采用了基于基因枪转化的瞬时表达体系,且减少了抗性筛选过程,大大缩短了成苗时间[10]。本研究借鉴了该研究成果的成功经验,采用基因枪介导的瞬时转化体系,针对3个OsBADH2基因编辑靶点分别进行了编辑,在50-60 d即可获得T0代材料。分子检测结果发现OsBADH2基因3处靶点均获得了发生序列变异的后代材料。OsBADH2基因靶点的编辑类型主要以小片段的碱基缺失为主。在T1代材料中,成功的筛选出了badh2突变体。
在规模化的基因编辑育种工作中,后代材料的分子检测是重要的一环。尽管RFLP分析(Restric- tion fragment length polymorphism,RFLP)[15-16]、dC-APS衍生分析(derived cleaved amplified polymorphic sequence,dCAPS)[17-18]、限制性酶切PCR法(Res-triction enzyme-PCR,RE-PCR)[19-20]以及测序分析能够满足编辑后代材料的鉴定需求[21]。然而,这些方法耗时长、成本高,不适合规模化基因编辑育种工作。因此如何提高基因编辑后代材料的检测效率、降低检测成本,成为了优化基因编辑育种体系的重要方面。
HRM是一种针对小片段核酸多态性进行精准分型的检测技术。Andrew等[22]在2014年就应用HRM检测成功鉴别出基因编辑果蝇的突变体;随后Thomas等[23]将该技术系统的应用在基因组编辑的斑马鱼突变体的检测中;Chen等[11]使用HRM技术鉴别番茄的不同基因编辑突变体。上述研究表明HRM技术可应用于动植物基因编辑后代材料的分子检测。2010年,Li等[24]建立的基于EvaGreen的HRM体系,将HRM检测成本降低至96个反应2美金,推动了HRM的广泛应用。为了进一步的加快粳稻基因编辑效率,降低编辑材料鉴定成本,本研究引入了HRM技术对基因编辑后代材料进行鉴别。结果表明HRM技术可以在1.5 h内完成检测,大大缩短了检测时间。
评价稻米香味品质是培育香稻的重要环节,但目前尚未有评价稻米香味的通用标准。以往的研究中对香米的评价方式主要为KOH法检测水稻分蘖期叶片的气味,口嚼法检测水稻种子的味道,以及GC-MS联用检测主要香味成分2-AP的含量等[25]。由于本研究的所获得突变体材料有限,尚无法对突变体中2-AP进行检测,本研究仅对叶片的香味表型做了感官性评价。通过采用KOH法对T1代基因编辑材料的叶片进行检测,发现OsBADH2基因编辑材料与野生型对照相比,大部分badh2突变体的叶片香味增加。我们也发现了5株badh2不具有明显的香味变化。Jang等[26]在OsIAA23中的研究发现,CRISPR基因编辑可诱发后代材料中的可变剪切,进而补偿性的修复编辑位点的功能突变。因此,后续研究中我们将通过2-AP的含量的测定更准确的分析badh2突变体预期表型与基因型的对应关系,并解析部分badh2突变体不具备香味表型的机理。
4 结论利用CRISPR/Cas9技术创制了一批OsBADH2基因编辑的“吉粳88”后代材料,并筛选获得了香味性状改良的后代编辑材料。建立了一套基于基因枪瞬时转化的粳稻CRISPR/Cas9编辑体系和后代材料HRM检测技术体系。
| [1] |
Yajima I, Yanai T, Nakamura M, et al. Volatile flavor components of cooked kaorimai(scented rice, O. sativa japonica)[J]. Journal of the Agricultural Chemical Society of Japan, 1979, 43(12): 2425-2429. |
| [2] |
Buttery RG, Ling LC, Juliano BO, et al. Cooked rice aroma and 2-acetyl-1-pyrroline[J]. Journal of Agricultural and Food Chemistry, 1983, 31(4): 823-826. |
| [3] |
Mathure SV, Jawali N, Thengane RJ, et al. Comparative quantitative analysis of headspace volatiles and their association with BADH2 marker in non-basmati scented, basmati and non-scented rice(Oryza sativa L.)cultivars of India[J]. Food Chemistry, 2014, 142(142): 383-391. |
| [4] |
刘妮, 陆沁, 刘娟, 等. CRISPR/Cas系统最新研究进展[J]. 生物技术通报, 2017, 33(2): 53-58. |
| [5] |
瞿礼嘉, 郭冬姝, 张金喆, 等. CRISPR/Cas系统在植物基因组编辑中的应用[J]. 生命科学, 2015, 27(1): 64-70. |
| [6] |
沈兰, 华宇峰, 付亚萍, 等. 利用CRISPR/Cas9多基因编辑系统在水稻中快速引入遗传多样性[J]. 中国科学:生命科学, 2017(11): 62-71. |
| [7] |
邵高能, 谢黎虹, 焦桂爱, 等. 利用CRISPR/CAS9技术编辑水稻香味基因Badh2[J]. 中国水稻科学, 2017, 31(2): 216-222. |
| [8] |
周文甲, 田晓杰, 任月坤, 等. 利用CRISPR/Cas9创造早熟香味水稻[J]. 土壤与作物, 2017, 6(2): 146-152. |
| [9] |
王付华, 薛华政, 王亚, 等. 利用CRISPR/CAS9基因编辑技术创制香型郑稻19新种质[J]. 作物杂志, 2018, 187(6): 42-48. |
| [10] |
Zhang Y, Zhen L, Zong Y, et al. Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA[J]. Nature Communications, 2016, 7(1): 12617. |
| [11] |
Chen L, Li W, Katingrazzini L, et al. A method for the production and expedient screening of CRISPR/Cas9-mediated non-transgenic mutant plants[J]. Horticulture Research, 2018, 5(1): 13. |
| [12] |
Li S, Liu SM, Liu YH, et al. HRM-facilitated rapid identification and genotyping of mutations induced by CRISPR/Cas9 mutagenesis in rice[J]. Crop Breeding and Applied Biotechnology, 2018, 18(2): 184-191. |
| [13] |
李楠, 柳青, 蒋世翠, 等. 超级稻吉粳88再生体系的建立[J]. 作物杂志, 2010(2): 71-75. |
| [14] |
丁莉萍, 张杰伟, 马艳, 等. CRISPR/Cas基因组编辑技术研究进展及其在植物中的应用[J]. 植物生理学报, 2019, 55(4): 28-41. |
| [15] |
Srivastava D, Shamim M, Kumar M, et al. Current status of conventional and molecular interventions for blast resistance in rice[J]. Methods Rice Science, 2017, 24(6): 299-321. |
| [16] |
张科, 郭鑫鑫, 刘西岗, 等. 植物花分生组织终止发育机制的研究进展[J]. 中国生态农业学报, 2018, 26(10): 154-165. |
| [17] |
Shibuya K, Watanabe K, Ono M. CRISPR/Cas9-mediated mutagenesis of the EPHEMERAL1 locus that regulates petal senescence in Japanese morning glory[J]. Plant Physiology and Biochemistry, 2018, 131: 53-57. |
| [18] |
Hamada H, Liu Y, Nagira Y, et al. Biolistic-delivery-based transient CRISPR/Cas9 expression enables in planta genome editing in wheat[J]. Scientific Reports, 2018, 8: 14422. |
| [19] |
Nadakuduti SS, Starker CG, Ko DK, et al. Evaluation of methods to assess activity of engineered genome-editing nucleases in protoplasts[J]. Frontiers in Plant Science, 2019, 10: 110. |
| [20] |
Chen X, Lu X, Shu N, et al. Targeted mutagenesis in cotton(Gossypium hirsutum L.)using the CRISPR/Cas9 system[J]. Scientific Reports, 2017, 7(1): 44304. |
| [21] |
刘春霞, 耿立召, 许建平. 植物基因组编辑检测方法[J]. 遗传, 2018, 40(12): 1075-1091. |
| [22] |
Andrew RB, Charlotte T, Chris PP, et al. Highly efficient targeted mutagenesis of drosophila with the CRISPR/Cas9 System[J]. Cell Reports, 2014, 546: 377-413. |
| [23] |
Thomas HR, Percival SM, Yoder BK, et al. High-throughput genome editing and phenotyping facilitated by high resolution melting curve analysis[J]. PLoS One, 2014, 9(12): e114632. |
| [24] |
Li YD, Chu ZZ, Liu XG, et al. A cost-effective high-resolution melting approach using the evagreen dye for DNA polymorphism detection and genotyping in plants[J]. Journal of Integrative Plant Biology, 2010, 52(12): 1036-1042. |
| [25] |
邹茜, 蒋聪, 寇姝燕, 等. 云南粳稻主栽品种的香味基因鉴定和不同检测方法的比较分析[J]. 分子植物育种, 2017(10): 43-51. |
| [26] |
Jiang MM, Hu HY, Kai J, et al. Different knockout genotypes of OsIAA23 in rice using CRISPR/Cas9 generating different phenotypes[J]. Plant Molecular Biology, 2019, 100: 467-479. |






