2. 重庆大学医学院,重庆 400044
2. Medical College of Chongqing University, Chongqing 400044
CRISPR/Cas技术作为一种革命性的基因编辑工具,被广泛应用于基因编辑、基因组成像等领域。因其独特的精准识别能力,CRISPR/Cas系统在生物传感领域也展现出广阔的应用前景。近年来,通过与核酸扩增技术联用,CRISPR生物传感系统已将检测特异性提高到单碱基水平,并在不同亚型的病毒鉴别领域取得了突破性的进展,正日趋成为临床核酸检测的有利工具。本文主要就CRISPR生物传感系统的最新研究进展进行综述。
1 常见CRISPR/Cas系统及其工作原理 1.1 CRISPR/Cas系统简介CRISPR/Cas系统由簇状规则间隔短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeats,CRISPR)和CRISPR关联蛋白(CRISPR associated,Cas)组成,是一种古生菌和细菌的适应性免疫系统[1-2]。CRISPR/Cas系统主要包括Cas基因序列、前导区、重复序列区和间隔区(图 1)[3]。CRISPR/Cas系统种类繁多,根据发挥作用的蛋白类型,大致可分为两类:1类为多蛋白效应复合物;2类为单一效应蛋白。目前常见的Cas9、Cas12a、Cas13a均属于第2类(表 1)[4]。近年来,CRISPR/Cas技术发展迅速,已被大量应用于基因修饰[5]、基因治疗[6]、生物传感[7]等领域。

|
| 图 1 CRISPR/Cas系统示意图 |
CRISPR/Cas9是第一个应用于基因工程的CRISPR/Cas系统。Cas9蛋白含有HNH和RuvC两个核酸酶结构域,能够对双链DNA(double-stranded DNA,dsDNA)进行定点编辑[8]。其原理是CRISPR系统在转录并处理后形成由CRISPR RNA(crRNA)和反式作用crRNA(tran-activating CRISPR RNA,tracrRNA)构成的单导RNA(single guide RNA,sgRNA)。Cas9蛋白在sgRNA引导下形成Cas9- tracrRNA-crRNA复合物,通过扫描外源DNA序列,定位到前间隔序列邻近基序(Protospacer adjacent motif,PAM)位点(通常为5′-NGG)。随后,双链DNA被部分打开,crRNA与互补链杂交,Cas9蛋白中的HNH结构域部分切割杂交DNA链,而RuvC结构域部分切割游离链,造成目标DNA双链断裂[9-10]。2013年,Gilbert等[11]通过引入催化失活突变,使Cas9的RuvC和HNH两个结构域失去剪切dsDNA能力,形成Cas9的突变体dCas9。dCas9既能特异性靶向dsDNA,又保留了dsDNA结构的完整。通过在dCas9上标记各种功能蛋白,可构建相应的生物传感器以检测靶标序列,较传统的原位杂交技术具有更高的特异性[12-13],进一步拓展了CRISPR/Cas系统在生物传感中的应用。
1.3 CRISPR/Cas12a系统CRISPR/Cas12a系统是由RNA引导的DNA靶向CRISPR系统。Cas12a又名Cpf1,是一种由RNA介导的核酸内切酶,包含一个RuvC核酸酶结构域,由张锋团队于2015年发现。与Cas9相比,Cas12a具有3个特性:(1)Cas12a能催化其自身的crRNA成熟,不需要tracrRNA参与;(2)Cas12a-crRNA复合物通过靶向一个短的T核苷酸丰富的PAM位点实现对靶DNA序列的精准切除;(3)Cas12a切割dsDNA后,会产生4-5个核苷酸的5′缺口[14-15]。2018年,Jennifer Dounda团队[16]在研究Lachnospiraceae bacterium ND2006 Cas12a(LbCas12a)和Streptococcus pyogenes Cas9(SpCas9)时发现,在sgRNA引导下,LbCas12a能够快速的降解单链M13DNA噬菌体而SpCas9不能。由此提出了LbCas12a在与靶DNA结合后具有强大的非特异性裂解单链DNA(single-stranded DNA,ssDNA)的能力,即“附属切割”活性,为CRISPR/Cas系统在生物传感中的应用提供了一个新的思路。
1.4 CRISPR/Cas13a系统CRISPR/Cas13a系统是由RNA引导的RNA靶向CRISPR系统。Cas13a又名C2c2,含有两个HEPN结构域,由Broad研究所最早发现[17]。Cas13a与Cas12a相似,都只需要crRNA引导,但CRISPR/Cas9和Cas12a在序列特定位点附近与crRNA互补的位置切割DNA,而Cas13a是在crRNA互补位置外的侧翼序列附近切割靶RNA。2016年,Abudayyeh等[18]在进行Leptotrichia shahii C2c2(LshC2c2)的体外切割实验时发现,在靶RNA存在的情况下,LshC2c2-crRNA复合物不仅可以切割靶RNA,还可以非特异性地切割侧枝单链RNA(single-stranded RNA,ssRNA)。Cas13a的“附属切割”活性具有应用于快速、高灵敏度、高特异性的生物传感器的潜力。
2 CRISPR/Cas系统在生物传感中的应用生物传感器是将生物信号转化为其他可定量的信号的检测技术,主要可以分为核酸传感器、酶传感器、免疫传感器等,近年来发展迅速。CRISPR/Cas系统作为基因编辑工具,大多应用于分子传感器,解决其特异性问题。目前已建立的一系列分子传感器大多是将扩增技术与CRISPR/Cas系统进行整合,但各传感器在靶标分子类型、Cas效应器类别、扩增方法上存在根本差别。本文主要综述了CRISPR/Cas系统在DNA、RNA及其他分子传感中的应用。
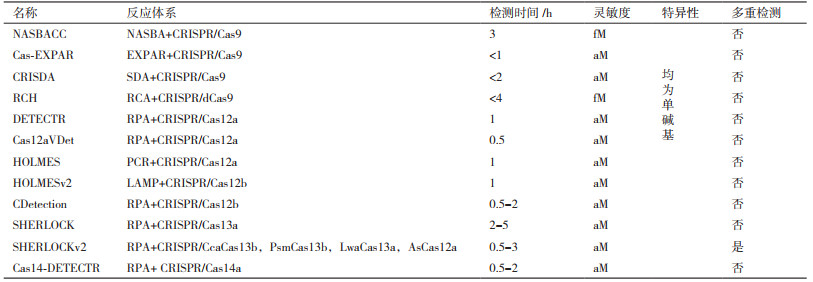
2.1 DNA传感传统的生物传感器在检测靶标分子时,通常需要扩增以实现低浓度检测。PCR是一种经典核酸扩增技术,但因扩增时间长,逐渐被扩增时间更短,效率更高的等温扩增替代。喻学锋团队[19]开发了一种CRISPR/Cas9触发的内切酶介导的链置换扩增(CRISDA)检测系统,具有aM灵敏度和单碱基特异性。CRISDA使用等温扩增反应链替代扩增(Strand displacement amplification,SDA)作为扩增手段,利用HNH结构域失活的Cas9切割打开靶DNA的一条链,随后引物结合启动SDA反应,利用肽核酸探针进行读数。CRISDA可用于全基因组定点检测,在复杂的背景干扰下仍然具有高特异性和灵敏度。除此之外,CRISDA通过引入等温扩增技术,打破了传统DNA传感器对于设备的依赖性,在核酸的现场即时检测中拥有巨大的应用潜力。同时简化了操作步骤,只需进行简单的引物设计即可在一个溶液体系里完成所有操作,缩短了检测时间,在特异性上也有所提升。但受限于SDA的扩增效率,CRISDA在灵敏度上具有一定的提升空间。
指数扩增反应(Exponential amplification reaction,EXPAR)是一种极为快速的核酸等温扩增技术,可在0.5 h内扩增到106-108倍。但EXPAR非特异性扩增明显,限制了其应用。Huang等[20]将CRISPR/Cas9与EXPAR联用开发了Cas-EXPAR检测系统用于ssDNA的快速超敏检测,具有aM灵敏度和单碱基特异性。该系统通过一个独立的反义PAM寡核苷酸来激活CRISPR/Cas9靶向dsDNA特定位点并切割。切割产物可以触发EXPAR,产生大量靶标dsDNA片段,并与SYBR Green Ⅰ染料结合显色(图 2)。Cas-EXPAR充分利用了CRISPR/Cas系统精准识别并切割的特性,除去体系中不需要的多余片段,解决了EXPAR的非特异性扩增问题,极大的提升了检测速度和检测效率。但该检测方法需要对dsDNA进行预处理,仍难以应用于现场即时检测。
Cas12a是由RNA引导的核酸酶,具有结合靶标后非特异性裂解ssDNA的能力。王金团队[21]首次将CRISPR/Cas12a应用于DNA传感,将CRISPR/Cas12a与PCR联用开发了快速灵敏的HOLMES(an one-Hour Low-cost Multipurpose highly Efficient System,HOLMES)系统。随后对其进行优化,将Cas12b与扩增效率更高的环介导等温扩增(Loop-mediated isothermal amplification,LAMP)结合开发了HOL-MES version2(HOLMESv2)系统[22]。Cas12b同样具有结合dsDNA靶标后非特异性裂解ssDNA的能力,且Cas12b切割特异性更高,但临床样本的检测仍具有一定难度。
基于此,Jennifer Doudna团队[16]开发了DET-ECTR(DNA Endonuclease-Targeted CRISPR Trans Reporter,DETECTR)生物传感平台,可准确和快速的检测人乳头瘤病毒(HPV),且能鉴别不同HPV亚型,具有aM灵敏度。DETECTR使用重组酶聚合酶扩增(Recombinase polymerase amplification,RPA)扩增含有HPV16的质粒,将扩增后的HPV16质粒、以HPV16为靶标的LbCas12a-crRNA复合物、ssDNA荧光猝灭基团共培养。LbCas12a-crRNA复合物结合靶标DNA后,激活了其“附属切割”活性,切割体系中的ssDNA荧光猝灭基团,产生荧光信号。但用以HPV18为靶标的LbCas12a-crRNA复合物时,不能检测到荧光信号。与其他新型DNA传感器相比,DETECTR不仅做到了单一病毒的检测,还能够区分出序列极为类似的病毒不同亚型,使现场即时检测结果的判断更为准确。同时DETECTE具有检测复杂样本的能力,在临床样本检测时,识别HPV16样本检测准确率达到100%,HPV18样本达到92%,在临床诊断、现场检测中拥有巨大的应用前景。然而DETECTR存在扩增和切割步骤需分离开的弊端,增加了检测时间。Wang等[23]提出了一种基于CRISPR/Cas12a的快速核酸传感策略(Cas12aVDet),将Cas12a附着在管壁上,在RPA扩增后通过离心的方式使Cas12a加入体系,实现一体化检测。该方法将检测时间缩短到了0.5 h左右,超过了目前已知的绝大部分检测方法。CRISPR芯片的出现进一步扩展了CRISPR-Cas9技术在DNA传感中的应用,使CRISPR传感器在无需扩增的情况下仍然具有较高灵敏度[24-25]。
近年来,不断有新的Cas蛋白被发现,如Cas12b、Cas14a等。这些新型Cas蛋白被报道同样具有识别靶标后的“附属切割”活性。Teng等[26]开发了一种Cas12b介导的DNA检测系统(CDetection),同样用于HPV16和HPV18的鉴别。CDetection检测原理与DETECTR类似,但相比于Cas12a和Cas12b对dsDNA的切割具有更高的活性和特异性,因此CDetection在检测灵敏度上优于DETECTR。而与同样用Cas12b的HOLMESv2相比,CDetection使用了更为高效的RPA作为扩增手段,且使用了dsDNA激活剂辅助Cas12b激活其“附属切割”活性,提高了检测的灵敏度。CDetection在分子诊断和临床研究具有应用潜力,但是其临床样本检测效果较差,仍需进一步优化提高检测准确性。
Cas14蛋白是一种RNA引导的ssDNA靶向蛋白,其大小仅为目前已知的2类Cas效应器的一半,但其独特的ssDNA靶向性为DNA传感提供了新的思路。Jennifer Doudna团队[27]在DETECTR的基础上,将Cas12a换成Cas14a,开发了一种单核苷酸多态性基因分型系统(Cas14-DETECTR)。Cas14-DETECTR用含磷酸的引物扩增DNA底物,以保护单链不被外切酶降解。扩增完成后,添加T7外切酶,未经修饰的链会被降解,留下可由Cas14a检测的ssDNA底物。该方法选择了人类HERC2基因上一个负责眼睛颜色的单核苷酸多态位点作为检测对象,可鉴别蓝眼和褐眼两种不同类型的基因。Cas14蛋白的高保真度使Cas14-DETECTR能更为真实的反应结果。除此之外,Cas14蛋白在直接检测ssDNA病毒也拥有广阔的应用前景。
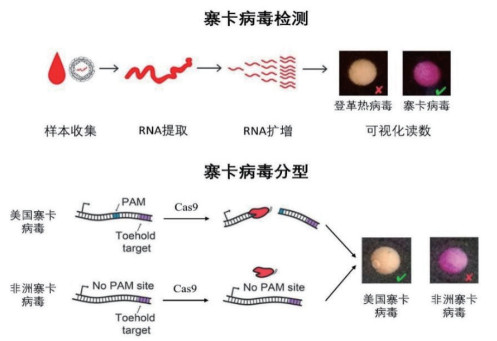
2.2 RNA传感RNA病毒占据了病毒的大部分,如何快速检测RNA病毒成为了急需解决的问题。然而目前大多数RNA传感系统缺乏高特异性,在实际应用中受到阻碍。2016年,Collins教授[28]等首次将CRISPR/Cas9系统应用于RNA传感,开发了纸基寨卡病毒不同亚型检测传感器(NASBA/CRISPR Cleavage,NASBACC),实现了不同寨卡病毒亚型的区分。利用依赖核酸序列的扩增技术(Nucleic acid sequence-based amplification,NASBA)对目标RNA进行高效扩增,扩增产物可打开试纸条上toehold生物传感器的发卡结构,激活Lacz酶合成,通过生化反应产生肉眼可见的颜色变化,以检测寨卡病毒。进一步将样本与CRISPR/Cas9结合,利用CRISPR/Cas9系统对PAM序列精准的特异性识别,鉴别寨卡病毒不同亚型(图 3),可在3 h内读出结果,检出限达到fM水平。NASBACC是第一个使用CRISPR/Cas系统进行病毒亚型鉴别的传感器,通过设计特异的sgRNA,可使Cas9对不同病毒亚型序列展现出不同的切割活性,解决了寨卡病毒现场即时检测的难题。但Cas9仅仅只能对靶标序列进行切割,当靶标浓度过低时,往往无法产生可读出的信号结果。
Cas13a是一种由RNA引导的核酸酶,在crRNA引导下可特异性结合ssRNA,并展现出惊人的附属ssRNA切割活性。目前的Cas效应器大多是以DNA为靶向目标,因此在RNA传感器构建时需要额外的逆转录步骤,使操作更为繁琐,而Cas13a独特的ssRNA靶向性解决了此问题。基于此,张锋团队[29]开发了鉴别寨卡病毒不同亚型的SHERLOCK(Spe-cific high-sensitivity enzymatic reporter unLOCKing)传感系统。该系统使用RPA对靶标进行扩增,可在短时间内扩增出大量目的片段,极大地提高了其检测的灵敏度。并利用Cas13a的“附属切割”活性切割体系中的荧光报告基团,产生可定量的信号(图 4)。SHERLOCK展现出aM灵敏度及单碱基的特异性,远远超出了临床检测要求。但仍然存在一定的局限性:(1)RPA扩增后目标DNA拷贝数过多,导致荧光报告基团饱和;(2)需额外的辅助设备。
为了解决上述问题,张锋团队[30]开发出超敏、便携、可多重检测的SHERLOCK version 2(SHERLOCKv2)传感系统。相较于SHERLOCK,SHERLOCKv2具有以下优点:(1)将辅助性CRISPR酶Csm6与Cas13a耦合实现信号增强,将灵敏度提高了3.5倍;(2)减少了等温扩增时引物的加入量,避免荧光报告基团饱和;(3)简单便携;(4)筛选了4种不同的Cas效应器分别切割各自特异性的ssDNA荧光报告基团,可同时检测4种不同类型的病毒。但SHERLOCKv2无法直接检测人的体液样本,限制了其在实际生活中的应用。Myhrvold等[31]通过将SHERLOCK与不经核酸提取热处理样品灭活核酸酶技术相结合,可以在不经DNA/RNA提取和纯化步骤的情况下,直接从临床样本中检测病毒核酸,极大地简化了操作步骤,使SHERLOCK技术在生物传感的应用得到进一步的加强。
除了检测寨卡病毒RNA,CRISPR传感器还被用于检测多种微小RNA(microRNA,miRNA)。miRNA是许多癌症的生物标志物,通过胞外囊泡传递[32],若能在癌症早期实现精准、超敏检测,将具有广阔的应用前景[33]。Qiu等[13]将滚环扩增技术(Rolling circle amplification,RCA)、辣根过氧化物酶(Horseradish peroxidase,HRP)和CRISPR/dCas9系统结合开发了RCA-CRISPR-split-HRP(RCH)平台,低成本、高效检测miRNA。RCA扩增后,将dCas9蛋白预先与分离的HRP报告片段结合。在sgRNA的引导下,可在扩增产物的茎环结构周围重新聚集HRP蛋白,从而激活HRP活性。利用3,3′,5,5′-四甲基联苯胺显色反应检测靶miRNA(图 5)。该方法可在4 h内读出结果,灵敏度达到fM级别,且能够检测出具有单碱基特异性的miRNA。RCH通过RCA的特点将靶标RNA转化成dCas9可识别的茎环结构,并利用dCas9蛋白的可修饰性使信号读数方式更加简便。
除了miRNA传感外,CRISPR系统也可应用于mRNA传感。Li等[34]开发出一种基于逻辑门的mRNA传感系统。该系统设计了一种具有茎环结构的mRNA传感的向导RNA(sgRNA),只有在同时存在mRNA和Cas9时才能触发其切割活性,产生可读出的荧光信号。通过直接对sgRNA进行修饰使其作为一种传感分子,为CRISPR传感系统提供了一个新的思路。
2.3 其他分子传感虽然目前已发现的CRISPR/Cas系统都是靶向核酸,但通过一定的信号转换也可实现其他分子的检测。Liang等[35]利用细菌变构转录因子(aTFs)对小分子的敏感性,开发了一种可检测小分子的CRISPR传感策略(CaT-SMelor)。aTFs通常包括DNA结合域和效应小分子结合域,将aTFs与dsDNA连接,当靶标小分子存在时,小分子会与aTFs结合并改变其构象,使dsDNA脱落从而触发Cas12a的“附属切割”活性切割荧光报告集团产生荧光信号。CaT-SMelor已成功实现了对人血液样本中尿酸的检测,且通过选择不同的aTFs,有望实现其他多种小分子的检测。现目前用于核酸检测的CRISPR传感器种类繁多且各有特点(表 2),但用于检测其他生物分子的传感器数量仍然非常少,有待进一步开发。
近年来,CRISPR传感器逐渐往更便携、快速、灵敏的方向发展,且如今的CRSIPR传感器已不再局限于单一病毒DNA或RNA的传感,而是更偏向于多重检测及亚型鉴别。基于CRISPR/Cas9的生物传感器,如Cas-EXPAR、CRISDA等,在特异性上高于传统生物传感器。但由于受到Cas9蛋白自身特性的限制,其读出信号的高低完全取决于靶标DNA的浓度,难以在灵敏度上有进一步的提升。基于Cas12a,Cas13a,Cas14的生物传感器,如SHERLOCK,DETECTR等,利用Cas蛋白的“附属切割”活性对信号进行了第二次放大,使其在灵敏度、特异性、便携性上均更有提升,在未来也将拥有更好的发展前景。
尽管CRISPR/Cas系统在生物传感领域已被成功应用,但仍存在许多共性问题亟待解决:(1)不能实时监测:现有的基于荧光或显色反应的生物传感策略只能在反应结束后通过肉眼或仪器读出结果,无法观测整个过程;(2)无法做到真正的通用检测平台:CRISPR/Cas系统中sgRNA不同则其靶向的目标也不同,且不同的靶标结合能力也不同;(3)环境差异可能影响检测结果:这些检测系统是在实验室条件下开发出来的,若今后应用于与实验室相比差异较大的环境时,其效果有待观察。因此,CRISPR生物传感系统仍然有较大的提升空间。
在过去的几年中,陆续发现了许多多功能的Cas蛋白酶(Cas12a、Cas13a等),未来也将会有更多这样的Cas酶被发现。这些酶或许可以再次提高CRISPR生物传感器的灵敏度等。而各种等温扩增技术的不断改进,也为CRISPR生物传感器的发展提供了另一个思路。这些新的CRISPR/Cas生物传感器或将彻底改变人们对传染病和遗传病快速、敏感和准确诊断的认识。
| [1] |
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nat Rev Microbiol, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [2] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [3] |
Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [4] |
Shmakov S, Smargon A, Scott D, et al. Diversity and evolution of class 2 CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2017, 15(3): 169-182. DOI:10.1038/nrmicro.2016.184 |
| [5] |
Roth TL, Puig-Saus C, Yu R, et al. Reprogramming human T cell function and specificity with non-viral genome targeting[J]. Nature, 2018, 559(7714): 405-409. DOI:10.1038/s41586-018-0326-5 |
| [6] |
Haapaniemi E, Botla S, Persson J, et al. CRISPR-Cas9 genome editing induces a p53-mediated DNA damage response[J]. Nat Med, 2018, 24(7): 927-930. DOI:10.1038/s41591-018-0049-z |
| [7] |
Wang M, Sintim HO. Discriminating cyclic from linear nucleotides - CRISPR/Cas-related cyclic hexaadenosine monophosphate as a case study[J]. Anal Biochem, 2019, 567: 21-26. DOI:10.1016/j.ab.2018.11.022 |
| [8] |
Adli M. The CRISPR tool kit for genome editing and beyond[J]. Nat Commun, 2018, 9(1): 1911. |
| [9] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [10] |
Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proc Natl Acad Sci USA, 2012, 109(39): E2579-E2686. DOI:10.1073/pnas.1208507109 |
| [11] |
Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell, 2013, 154(2): 442-451. DOI:10.1016/j.cell.2013.06.044 |
| [12] |
Zhang Y, Qian L, Wei W, et al. Paired design of dCas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains[J]. Acs Synth Biol, 2017, 6(2): 211-216. |
| [13] |
Qiu XY, Zhu LY, Zhu CS, et al. Highly effective and low-cost microRNA detection with CRISPR-Cas9[J]. Acs Synth Biol, 2018, 7(3): 807-813. DOI:10.1021/acssynbio.7b00446 |
| [14] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-cas system[J]. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [15] |
Chylinski K, Le Rhun A, Charpentier E. The tracrRNA and Cas9 families of type Ⅱ CRISPR-Cas immunity systems[J]. Rna Biol, 2013, 10(5): 726-737. DOI:10.4161/rna.24321 |
| [16] |
Chen JS, Ma E, Harrington LB, et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity[J]. Science, 2018, 360(6387): 436-439. DOI:10.1126/science.aar6245 |
| [17] |
Shea MJ, King DL, Conboy MJ, et al. Proteins that bind to drosophila chorion cis-regulatory elements:a new C2H2 zinc finger protein and a C2C2 steroid receptor-like component[J]. Genes Dev, 1990, 4(7): 1128-1140. DOI:10.1101/gad.4.7.1128 |
| [18] |
Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [19] |
Zhou W, Hu L, Ying L, et al. A CRISPR-Cas9-triggered strand displacement amplification method for ultrasensitive DNA detection[J]. Nat Commun, 2018, 9(1): 5012. |
| [20] |
Huang M, Zhou X, Wang H, et al. CRISPR/Cas9 triggered isothermal amplification for site-specific nucleic acid detection[J]. Anal Chem, 2018, 90(3): 2193-2200. DOI:10.1021/acs.analchem.7b04542 |
| [21] |
Li SY, Cheng QX, Wang JM, et al. CRISPR-Cas12a-assisted nucleic acid detection[J]. Cell Discov, 2018, 4: 20. |
| [22] |
Li L, Li S, Wu N, et al. HOLMESv2:A CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation[J]. Acs Synth Biol, 2019, 8(10): 2228-2237. DOI:10.1021/acssynbio.9b00209 |
| [23] |
Wang B, Wang R, Wang D, et al. Cas12aVDet:A CRISPR/Cas12a-based platform for rapid and visual nucleic acid detection[J]. Anal Chem, 2019, 91(19): 12156-12161. DOI:10.1021/acs.analchem.9b01526 |
| [24] |
Hajian R, Balderston S, Tran T, et al. Detection of unamplified target genes via CRISPR-Cas9 immobilized on a graphene field-effect transistor[J]. Nat Biomed Eng, 2019, 3(6): 427-437. DOI:10.1038/s41551-019-0371-x |
| [25] |
Shao N, Han X, Song Y, et al. CRISPR-Cas12a coupled with platinum nanoreporter for visual quantification of SNVs on a volumetric bar-chart chip[J]. Anal Chem, 2019, 91(19): 12384-12391. DOI:10.1021/acs.analchem.9b02925 |
| [26] |
Teng F, Guo L, Cui T, et al. CDetection:CRISPR-Cas12b-based DNA detection with sub-attomolar sensitivity and single-base specificity[J]. Genome Biol, 2019, 20(1): 132. |
| [27] |
Harrington LB, Burstein D, Chen JS, et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes[J]. Science, 2018, 362(6416): 839-842. DOI:10.1126/science.aav4294 |
| [28] |
Pardee K, Green AA, Takahashi MK, et al. Rapid, low-cost detection of zika virus using programmable biomolecular components[J]. Cell, 2016, 165(5): 1255-1266. DOI:10.1016/j.cell.2016.04.059 |
| [29] |
Gootenberg JS, Abudayyeh OO, Lee JW, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-442. DOI:10.1126/science.aam9321 |
| [30] |
Gootenberg JS, Abudayyeh OO, Kellner MJ, et al. Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6[J]. Science, 2018, 360(6387): 439-444. DOI:10.1126/science.aaq0179 |
| [31] |
Myhrvold C, Freije CA, Gootenberg JS, et al. Field-deployable viral diagnostics using CRISPR-Cas13[J]. Science, 2018, 360(6387): 444-448. DOI:10.1126/science.aas8836 |
| [32] |
Qing GC, Gong NQ, Chen XH, et al. Natural and engineered bacterial outer membrane vesicles[J]. Biophysics Reports, 2019, 5(4): 184-198. DOI:10.1007/s41048-019-00095-6 |
| [33] |
Qiu JH, Xu JX, Zhang K, et al. Refining cancer management using integrated liquid biopsy[J]. Theranostics, 2020, 10(5): 2374-2384. DOI:10.7150/thno.40677 |
| [34] |
Li Y, Teng X, Zhang K, et al. RNA strand displacement responsive CRISPR/Cas9 system for mRNA sensing[J]. Anal Chem, 2019, 91(6): 3989-3996. DOI:10.1021/acs.analchem.8b05238 |
| [35] |
Liang M, Li Z, Wang W, et al. A CRISPR-Cas12a-derived biosensing platform for the highly sensitive detection of diverse small molecules[J]. Nat Commun, 2019, 10(1): 3672. DOI:10.1038/s41467-019-11648-1 |