精准高效整合技术是将外源DNA插入到目的细胞基因组特定位置的一种转基因技术。为了实现精准高效外源DNA整合, 基因编辑技术不断发展[1]。从第一代组装复杂的锌指核酸酶(Zinc finger nuclease, ZFN)技术[2]到第二代脱靶严重的转录激活因子样效应物核酸酶(Transcription activator-like effector nuclease, TALEN)技术[3], 再到第三代操作简单的CRISPR/Cas9(Clustered regularly interspaced short palindromic repeats-CRISPR associated system)技术[4], 基因编辑技术的效率和精准程度都有了很大的提高[5]。但CRISPR/Cas9的脱靶效应仍是困扰基因编辑安全应用的瓶颈, 更精准可靠的靶向整合技术仍是这些技术应用到人医临床和转基因动物生产的前提[6]。如今通过改造Cas9氨基酸残基序列(优化后的spCas9)[7]、修饰sgRNA五碳糖[8]和sgRNA结构改造(改造后catRNA)等均提高了系统稳定性[9]; 基于CRISPR/Cas9衍生的先导编辑器(Primer Editor, PE)[10]与CRISPR-转座酶CRISPR-associated Transposase, CAST)技术[11]及CRISPR家族发现的新成员Cas12a(Cpf1)蛋白[12], 极大提升了该系统的精准靶向和整合效率[7]。此外, 双链DNA断裂修复方式与基因靶向整合效率息息相关[13]。其中同源重组(Homologous recombination, HR)修复只发生在细胞周期的G2/S期[14], 而非同源末端连接(Non-homologous end joining, NHEJ)的修复方式, 发生在整个细胞周期, 效率更高[15]。其中基于NHEJ的微同源介导的末端连接(Microhomology-mediated end joining, MMEJ)、同源非依赖性靶向整合(Homology-independent targeted integration, HITI)和HR介导的末端连接(Homology-mediated end joining, HMEJ)等修复方式在外源DNA整合效率上各有不同, 通过干预细胞DNA断裂的修复方式可以显著提高外源DNA整合效率[16]。本文将围绕基因编辑研究进展、引导RNA修饰技术、单碱基整合技术、转座子技术和外源DNA整合效率等方面对精准靶向和高效整合技术进行综述, 以期促进外源DNA精准整合技术的利用和推广。
1 基因编辑技术概述1974年, Jaenisch等[17]把猿猴病毒(SV40)注入小鼠囊胚腔中, 得到部分组织含有SV40 DNA的嵌合体小鼠, 开启了外源DNA整合研究的大门(图 1); 1984年, Kucherlapati等[18]利用HR技术将外源质粒DNA插入到人的基因组内, 基因敲入的技术问世; 2002年, 利用Fok Ⅰ Ⅱ型限制性核酸内切酶与锌指蛋白结合, 对目的基因组DNA进行识别和切割, 开发出了一种ZFN技术[4], 但是ZFN的设计和构建程序太复杂, 极大地限制了其应用[19]; 2011年, 在植物病原体黄单胞菌属中发现了可以特异性识别核苷酸序列的转录因子样效应蛋白, 开发出TALEN技术[19], 由特异性较强的TALE蛋白代替锌指蛋白, 具有相对准确的基因组识别和切割作用, 但其存在较高的脱靶效应, 难以广泛应用[20]; 2012年, Jinek等[21]证实Cas9核酸酶能在小的引导RNA(Small guide RNA, sgRNA)指引下, 靶向切割基因组DNA, 拉开了CRISPR/Cas9基因编辑技术的序幕。利用该技术使同源染色体DNA双链断裂(Double strand break, DSB), 通过DNA损伤修复机制, 实现了特定位点的外源DNA整合和基因敲除[22]。与ZFN和TALEN相比, CRISPR/Cas9系统是通过RNA与DNA的碱基互补配对作用靶向识别的技术, 设计简单、操作容易, 并且具有效率高和特异性强的特点[23]。随后又发现具有切割黏性末端的CRISPR/Cpf1系统[9], 但随着研究深入发现这些系统也存在严重脱靶效应[24]。为了解决这个问题, CRISPR系统不断被优化和更新, 如catRNA技术[9]、PE技术[10]和CAST技术[11]的开发和应用(图 1), 这些技术均促进了外源DNA整合技术的发展。

|
| 图 1 基因编辑技术的发展历程 |
CRISPR家族问世以来, 家族成员不断地被发现, 具有RNA切割能力的Ⅱ类Ⅵ型核酸酶Cas13[25]和DNA切割作用Ⅱ类Ⅴ型的Cas12(Cpf1)[12]广泛被关注。其中Cpf1蛋白(包括FnCpf1, LbCpf1和AsCpf1)[26], 在单链向导RNA(crRNA)引导下与靶DNA特定位点结合并切割, 简化了实验的设计方案, 广泛应用到动物基因编辑中。Cpf1相比Cas9蛋白要小, 更容易操作, 能识别5'-TTTN-3'(AsCpf1和LbCpf1)和5'-TTTN-3'(FnCpf1)两种PAM序列, 切割DNA形成一个有5个核苷酸突出的黏性末端[27], 极大地扩充了基因组编辑的范围, 增加HR修复DSB的比例[28]。此外, 通过温度控制还有效地实现了LbCpf1和AsCpf1对斑马鱼胚胎基因组编辑[29]。
3 Cas9和sgRNA的改造提高外源DNA精准整合效率张锋课题组[7](2015)根据Cas9蛋白与基因结合的结构域特点, 将Cas9蛋白的第848(赖氨酸)、1003(赖氨酸)、1060(精氨酸)位替换成非极性的丙氨基酸, 增强了蛋白与核酸之间的连接强度, 大大减少Cas9的“脱靶”效应, 命名为spCas9。
最近研究显示, 对sgRNA组成中的五碳糖修饰[8]或sgRNA结构进行改造[7], 也可以增加基因编辑的效率和降低脱靶效应。在sgRNA五碳糖的2号位点引入其它的原子或某种化学基团如戊糖分子[8], 可以提高sgRNA稳定性, 促进基因编辑进行(图 2-A); 根据细胞内大量稳定存在tRNA现象, 开发出了一种耐RNase的笼状tRNA样crRNA(catRNA, 图 2-B)[9], 与传统crRNA相比, catRNA非常稳定, 介导外源DNA整合研究中发现整合效率增加了4.3倍[9]。
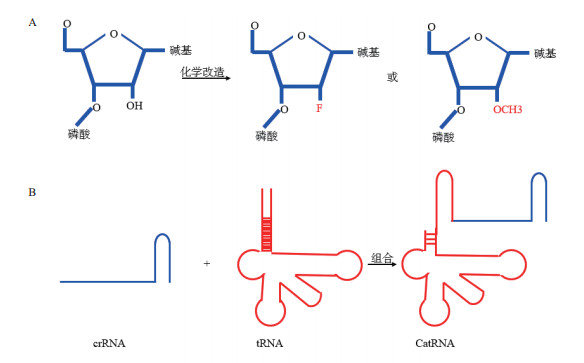
|
| 图 2 化学修饰RNA和catRNA结构 |
单碱基编辑技术是基因编辑技术发展和应用的一项目标, 体现了基因编辑的精准性。最早是哈佛大学Broad研究所科研人员利用CRISPR/Cas9和胞嘧啶脱氨酶(APOBEC)联合建立胞嘧啶碱基编辑器(CBE), 可实现胞嘧啶向胸腺嘧啶的转换[30-31]。在此基础上开发出Cas9碱基编辑器(hA3A-BE)和Cpf1碱基编辑器(dCpf1-BE), 分别在G/C富集区域、高甲基化区域和A/T富集区域内开展碱基替换。该研究所于2017年又开发出腺嘌呤碱基器(ABE), 能将A-T碱基对转换为G-C碱基对, 且在人类细胞中的编辑效率高达50%, 脱靶效率低, 扩展了单碱基编辑的范围[32]。迄今为止, 优化的碱基编辑器(包括BE4-G、xCas9、NG-Cas9、A3A-Cas9和AID-Cas9等)[33]显著提高了点突变效率及靶向突变的精确性[34]。
2019年开发了一种无需双链断裂或供体DNA, 直接搜索和替换基因组碱基的新编辑方法, 即PE技术[10], 其几乎可以编辑核酸序列的任何位点, 且不需要DSB和供体模板, 对于小片段DNA序列插入或单碱基突变, 具有明显的优势, 安全性更强。该技术以CRISPR/Cas9系统为基础, 首先在sgRNA的3'末端增加了一段RNA序列, 起到模板的作用, 可以加入要突变的碱基, 这种新的向导RNA(Prime editing extended guide RNA, pegRNA)既能引导Cas9到目标位点进行切割, 又含有修复模板序列; 其次, Cas9蛋白发生H840A突变, 只能切断含PAM的靶点DNA链, 并且与逆转录酶M-MLV融合获得新的PE蛋白。
以插入3个碱基为例(图 3), 在pegRNA的引导下, Cas9 H840切口酶切断含PAM的靶点DNA单链, 断裂的靶DNA链与pegRNA的3'末端序列互补并结合, 在逆转录酶作用下开始逆转录, pegRNA另一段则是逆转录的模板, 其上携带有目标插入、突变或缺失碱基以实现基因编辑。随后3' flap结构的DNA链携带有目标突变, 而5' flap结构的DNA链则无任何突变。细胞内5' flap结构易被结构特异性内切酶识别并切除, 之后经DNA连接酶连接后便实现了精准的小片段DNA整合。

|
| A: pegRNA与Cas9结合, 靶向切割基因组; B: DNA含PAM的链断裂; C:在逆转录酶的作用下开始逆转录; D:在切口处形成了3'-flap, 形成与pegRNA模板互补配对的DNA链; E:蛋白和sgRNA脱离的DNA链, 细胞内核酸内切酶启动剪切修复 图 3 PE整合技术原理 |
转座子是可以在原核和真核生物基因组上进行位置转换的DNA片段, 最初由美国生物学家在玉米中发现[35]。通过在原始位置上进行剪切或复制, 在转座酶辅助下插入到宿主基因组的其他位置, 转座子发生位置转移的过程即为转座[36]。根据转座机制的不同, 可以分为逆转录转座子和DNA转座子[37]。现在应用最广泛的是PB转座子, PB转座子系统显示在小鼠和人细胞中, 能够高效转入并稳定表达外源基因, 具有高效、安全、负载容量大和插入位点为TTAA的特异性特点, 但仍然存在整合错误和基因毒性的缺点, 限制了其广泛应用[37]。
转座子与基因编辑技术的结合可以提高外源DNA靶向整合效率, PB转座子结合ZFN和TELEN技术, 实现了外源DNA特定位点的高效整合[37]。目前, PB转座子和CRISPR/Cas9相结合使用, 实现了对人诱导多能干细胞的靶向整合[37]。
2019年, 在蓝细菌中发现了一种新型的转座酶(Tn-7), 它的3个亚基(TnsB、TnsC和TniQ), 可以和CRISPR效应物(Cas12K)相关联, 形成CRISPR相关转座酶(CAST)技术[11]。同时, 哥伦比亚大学研究团队在霍乱弧菌中也发现了Tn7-like转座子(Tn-6677), 由4个亚基(TnsA、TnsB、TnsC和TniQ)组成, 联合Cas家族蛋白(Cas6、Cas7和Cas8), 可以在其基因组识别目标DNA后, TniQ蛋白引导转座子插入到基因组中的特定位置[38]。目前, 已开发出蓝藻菌CAST技术(ShCAST)和鱼腥蓝菌(AcCAST)两套系统, 该技术利用转座子将目的基因片段高效精准整合到基因组DNA特定位点, 而且不依赖DNA断裂, 安全性很高[11]。在大肠杆菌靶位点插入2.5 kb长度的DNA片段, 成功率高达80%[11], 远超于以往任何外源DNA整合方法(图 4)。研究还发现两臂长度影响外源DNA整合效率:左臂约250 bp, 右臂200 bp时, 其整合效率较高。这些新技术显示出了在精准靶向和高效整合上的极大优势, 在哺乳动物细胞精准高效外源DNA整合应用中具有巨大潜力。
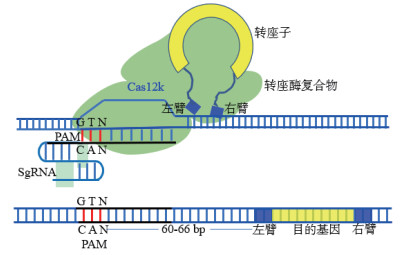
|
| 图 4 CAST转座子介导的外源DNA整合 |
HR技术需要在外源DNA两侧各引入一段与整合位点序列相同的类似手臂一样的结构, 称同源臂, 然后与细胞基因组DNA发生HR, 实现外源DNA靶向定点整合。HR发生效率极低, 极大限制了外源DNA整合的研究和应用[19]。结合基因编辑技术, 人工地在靶位点附近造成DSB, 通过引入人工合成的同源DNA供体模板, 可显著提高重组效率[39-40]。
外源DNA整合效率和DNA断裂修复方式有[16], DNA断裂修复方式主要包括HR和NHEJ修复方式[41]。HR需要利用一个完整姐妹染色单体或供体DNA(>400 bp)作为修复模板[16], 因此仅发生在S和G2期。主要通过两条同源染色体DNA发生联会, 其中一条DNA分子发生双链断裂, 形成的3'端入侵双链DNA并在同源区配对结合, 断裂缺口封闭后形成中间体, 然后完成HR途径修复[42]。研究显示CRISPR/Cas9联合HR的修复方式, 重组率仅有10-5[43], 而NHEJ修复途径可发生在整个细胞周期, 拥有较高的基因修复效率[15, 44], 相当于HR途径的1 000倍[45]。但NHEJ修复途径具有随机性, 使DSB修复时引入了各种类型的插入或缺失, 难以达到精确整合目的[46-47]。在DNA修复过程中, NHEJ修复途径会对HR途径产生竞争性抑制, 使HR效率降低。通过减少NHEJ途径发生可以提高HR效率[48]。Gutschner等[49]将Cas9蛋白修饰为细胞周期依赖性表达, 使Cas9仅在G2/S期表达, HR效率显著提高。还有学者过表达细胞周期素D1和使用一种微管解聚剂(Nocodazole), 使细胞周期处于G2、S和M期的细胞比例增高, HR修复效率提高了近1倍[50], 但效率仍然很低, 远不能满足靶向整合的要求。此外, 通过改变同源臂长度也能提高外源DNA整合的效率[51]。
6.1 同源臂长短对外源DNA整合的效率影响同源臂长度对HR修复效率有显著的影响, 早期使用一系列50-1 049 bp同源臂长度的供体, 应用ZFN技术在CHO细胞的目标位点进行切割, 发现同源臂长度为50 bp时, HR效率为0.8%;当同源臂长度增加到1 000 bp以上时, HR效率可达23.15%[51]。研究显示一系列供体DNA的同源臂长度为150-2 000 bp, 利用CRISPR/Cas9技术对靶向位点进行切割, 同源臂长度在150 bp时, 在人诱导多能干细胞中重组效率仅有0.7%, 同源臂长度在300 bp时, 重组效率仅有0.22%, 同源臂长2 000 bp的时可达到10.4%[50], 说明应用较长同源臂能够提高HR效率。但是在一些物种中, sgRNA的靶向效率很低(如猴胚胎中), 导致HR效率仍然很低[44], 基于HR途径的靶向整合依然有很大局限性。相比之下, NHEJ途径的靶向整合更有应用潜力[52]。
6.2 非同源性末端接合(NHEJ)途径外源DNA整合效率NHEJ途径在修复DSB中效率高, 具有随机性[53]。根据NHEJ修复途径分为两种, 一种为依赖于Ku蛋白的经典NHEJ途径(Canonical-NHEJ, c-NHEJ), 另一种为不依赖Ku蛋白的替代NHEJ途径(Alternative-NHEJ, a-NHEJ)[54]。c-NHEJ的发生是由异源二聚体Ku70/80分别结合到两个断裂的DNA末端, 在断裂处形成套环结构向内移动[55], 然后招募DNA依赖蛋白激酶催化亚基与DNA末端结合, 最后在聚合酶、核酸酶和DNA连接酶Ⅳ的作用下, 完成DSB修复[52]。一定程度上NHEJ途径难以做到精确外源DNA整合。但根据NHEJ的特点, 利用同源重组介导的末端连接(Microhomology-mediated end joining, MMEJ)和同源非依赖性靶向整合(Homology-independent targeted integration, HITI)修复方式也可以实现外源DNA序列的精准靶向整合, 目前已成功将外源DNA序列插入到斑马鱼[56]、鼠[47]和人胚胎干细胞(Human embryonic stem cell, hESC)[57]等。
6.2.1 同源非依赖性靶向整合(HITI)在外源DNA整合中的应用HITI依赖于c-NHEJ修复途径[47]。将环状DNA供体加入单个sgRNA靶向序列(表 1), 利用Cas9蛋白同步切割环状供体DNA和宿主基因组DNA, 将HITI-DNA片段高效插入到DSB的位置, 从而成功地将外源DNA整合到细胞中基因组的精确位点上, 这个方法可以发生在非分裂细胞, 并且表现出了高效的整合效率。在分裂细胞中的效率比其它方法高10倍以上; 体内无分裂的神经元(眼睛和大脑)靶向外源DNA整合能力是前所未有的[58]。
c-NHEJ途径的核心蛋白因子(如Ku70/80)发生突变时, 细胞不能利用c-NHEJ途径进行DSB修复, 将通过a-NHEJ途径进行修复。MMEJ是a-NHEJ的一种亚型[59], 通过微同源序列(3-30 bp)[16]介导外源DNA整合(表 1)。此修复方式可以有效地靶向整合大片段外源DNA[60], 但只发生在细胞周期的G1和S期早期[61]。与c-NHEJ途径相比, 其发生频率较低, 外源DNA整合的效率依然很低。
6.2.3 同源重组介导的末端连接(HMEJ)外源DNA整合使用800 bp长度的同源臂, 并在同源臂两端加入sgRNA靶向序列, 建立了HMEJ系统(表 1)[44]。研究将HR(只有同源臂)、MMEJ(两端加入sgRNA靶向序列)、NHEJ(两端加入sgRNA靶向序列)和HMEJ 4种供体DNA片段转染到ESC、N2a细胞、293T细胞、星形胶质细胞、成体小鼠肝细胞和胚胎(小鼠和猴), 结果显示在ESC和N2a细胞内, HMEJ供体的HR率远大于NHEJ和MMEJ供体效率, 但与HR途径相比, 未见显著提高[44]; 在293T细胞、原代星形胶质细胞、成体肝细胞以及神经元中, HMEJ供体比HR、MMEJ和NHEJ供体的同源重组率显著提高[44]。HMEJ修复机制可能在不同细胞周期同时利用两种路径进行修复DSB, 在S/G2期, HR路径介导DSB修复; 在G1/S早期, 可能是利用MMEJ路径进行DSB修复。
7 问题与展望精准靶向和高效整合技术一直是基因编辑技术的难点, 尽管基因编辑技术在基因治疗和许多研究领域已取得了不少突破性的进展, 但是在临床应用方面仍然存在很多困难与挑战。在近期的研究中, 我们看到了新型基因编辑技术正在逐渐走向成熟, 如基于转座子的基因编辑技术和单碱基编辑的PE新工具的开发和应用, 在不产生DSB和不需要供体修复模板的条件下, 达到高效的外源DNA整合, 对于许多点突变造成的遗传疾病具有很大的应用潜能, 同时对人类的疾病治疗和转基因育种呈现了更光明的前景, 基因编辑技术一直在不断创造着不可估量的社会经济价值。
| [1] |
陈楠楠. CRISPR/Cas9基因编辑技术研究进展[J]. 生物化工, 2019, 5(5): 140-143. DOI:10.3969/j.issn.2096-0387.2019.05.040 |
| [2] |
Bibikova M, Golic M, Golic KG, et al. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases[J]. Genetics, 2002, 161(3): 1169-1175. |
| [3] |
Ding Q, Lee YK, Schaefer EA, et al. A TALEN genome-editing system for generating human stem cell-based disease models[J]. Cell Stem Cell, 2013, 12(2): 238-251. |
| [4] |
张启超, 刘龙海, 陈旭, 等. 新型CRISPR/Cas基因编辑系统的研究与应用进展[J]. 蚕业科学, 2018, 44(3): 474-480. |
| [5] |
张玉苗, 李蓉, 鲁瑶, 等. 基于提高CRISPR/Cas基因编辑效率的研究进展[J]. 热带作物学报, 2019, 40(10): 2006-2015. DOI:10.3969/j.issn.1000-2561.2019.10.013 |
| [6] |
谢芳艳. 基因编辑技术在基因治疗中的应用进展[J]. 生物化工, 2019, 01(5): 162-163. |
| [7] |
Slaymaker IM, Gao L, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity[J]. Science, 2016, 351(6268): 84-88. |
| [8] |
Cai W, Wang M. Engineering nucleic acid chemistry for precise and controllable CRISPR/Cas9 genome editing[J]. Sci Bull, 2019, 64(24): 1841-1849. DOI:10.1016/j.scib.2019.07.035 |
| [9] |
Zhang X, Xu L, Fan R, et al. Genetic editing and interrogation with Cpf1 and caged truncated pre-tRNA-like crRNA in mammalian cells[J]. Cell Discov, 2018, 4(1): 36. DOI:10.1038/s41421-018-0035-0 |
| [10] |
Anzalone AV, Randolph PB, Davis JR, et al. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576(7785): 149-157. DOI:10.1038/s41586-019-1711-4 |
| [11] |
Strecker J, Ladha A, Gardner Z, et al. RNA-guided DNA insertion with CRISPR-associated transposases[J]. Science, 2019, 365(6448): 48-53. DOI:10.1126/science.aax9181 |
| [12] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-Guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771. |
| [13] |
Kass EM, Lim PX, Helgadottir HR, et al. Robust homology-directed repair within mouse mammary tissue is not specifically affected by Brca2 mutation[J]. Nat Commun, 2016, 7: 13241. DOI:10.1038/ncomms13241 |
| [14] |
Iliakis G, Murmann T, Soni A. Alternative end-joining repair pathways are the ultimate backup for abrogated classical non-homologous end-joining and homologous recombination repair: Implications for the formation of chromosome translocations[J]. Mutation Research/Genetic Toxicology and Environmental Mutagenesis, 2015, 793: 166-175. DOI:10.1016/j.mrgentox.2015.07.001 |
| [15] |
Rothkamm K, Kruger I, Thompson LH, et al. Pathways of DNA double-strand break repair during the mammalian cell cycle[J]. Mol Cell Biol, 2003, 23(16): 5706-5715. DOI:10.1128/MCB.23.16.5706-5715.2003 |
| [16] |
Morita H, Taimatsu K, Yanagi K, et al. Exogenous gene integration mediated by genome editing technologies in zebrafish[J]. Bioengineered, 2017, 8(3): 287-295. DOI:10.1080/21655979.2017.1300727 |
| [17] |
Jaenisch R. Infection of mouse blastocysts with SV40 DNA: Normal development of the infected embryos and persistence of SV40-specific DNA sequences in the adult animals[J]. Cold Spring Harb Symp Quant Biol, 1975, 39: 375-380. |
| [18] |
Kucherlapati RS, Eves EM, Song KY, et al. Homologous recombination between plasmids in mammalian cells can be enhanced by treatment of input DNA[J]. Proc Natl Acad Sci USA, 1984, 81(10): 3153-3157. DOI:10.1073/pnas.81.10.3153 |
| [19] |
肖安, 张博. 人工核酸内切酶介导的新一代基因组编辑技术进展[J]. 生物工程学报, 2015, 31(6): 917-928. |
| [20] |
Wright DA, Li T, Yang B, et al. TALEN-mediated genome editing: Prospects and perspectives[J]. Biochem J, 2014, 462(1): 15-24. DOI:10.1042/BJ20140295 |
| [21] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [22] |
陈一欧, 宝颖, 马华峥, 等. 基因编辑技术及其在中国的研究发展[J]. 遗传, 2018, 40(10): 900-915. |
| [23] |
刘玉彪, 许馨, 曹山虎, 等. 基因编辑技术最新研究进展[J]. 生物技术通报, 2017, 33(6): 39-44. |
| [24] |
Pattanayak V, Lin S, Guilinger JP, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J]. Nat Biotechnol, 2013, 31(9): 839-843. DOI:10.1038/nbt.2673 |
| [25] |
Cox D, Gootenberg JS, Abudayyeh OO, et al. RNA editing with CRISPR-Cas13[J]. Science, 2017, 358(6366): 1019-1027. DOI:10.1126/science.aaq0180 |
| [26] |
杨帆, 李寅. 新一代基因组编辑系统CRISPR/Cpf1[J]. 生物工程学报, 2017, 33(3): 361-371. |
| [27] |
Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [28] |
Thyme SB, Schier AF. Polq-Mediated end joining is essential for surviving DNA Double-Strand breaks during early zebrafish development[J]. Cell Rep, 2016, 15(7): 1611-1613. DOI:10.1016/j.celrep.2016.04.089 |
| [29] |
Moreno-Mateos MA, Fernandez JP, Rouet R, et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing[J]. Nat Commun, 2017, 8(1): 2024. |
| [30] |
McGrath E, Shin H, Zhang L, et al. Targeting specificity of APOBEC-based cytosine base editor in human iPSCs determined by whole genome sequencing[J]. Nat Commun, 2019, 10(1): 5353. |
| [31] |
Eid A, Alshareef S, Mahfouz MM. CRISPR base editors: Genome editing without double-stranded breaks[J]. Biochem J, 2018, 475(11): 1955-1964. DOI:10.1042/BCJ20170793 |
| [32] |
Kankowski S, Forstera B, Winkelmann A, et al. A novel RNA editing sensor tool and a specific agonist determine neuronal protein expression of RNA-Edited glycine receptors and identify a genomic APOBEC1 dimorphism as a new genetic risk factor of epilepsy[J]. Front Mol Neurosci, 2017, 10: 439. |
| [33] |
Zeng D, Li X, Huang J, et al. Engineered Cas9 variant tools expand targeting scope of genome and base editing in rice[J]. Plant Biotechnol J, 2019. DOI:10.1111/pbj.13293 |
| [34] |
Liu Z, Chen M, Chen S, et al. Highly efficient RNA-guided base editing in rabbit[J]. Nat Commun, 2018, 9(1): 2717. |
| [35] |
Sun W, Teng J, Zeng J, et al. The piggyBac-based double-inducible binary vector system: A novel universal platform for studying gene functions and interactions[J]. Plasmid, 2019, 105: 102420. DOI:10.1016/j.plasmid.2019.102420 |
| [36] |
于正洪, 姜恩泽, 张杰明, 等. PiggyBac转座子:人类基因编码研究的新工具[J]. 医学研究生学报, 2014, 27(2): 199-202. |
| [37] |
张文豪, 李旭, 帅领. PiggyBac转座系统的发展及应用[J]. 发育医学电子杂志, 2018, 6(3): 149-153. DOI:10.3969/j.issn.2095-5340.2018.03.004 |
| [38] |
Klompe SE, Vo P, Halpin-Healy TS, et al. Transposon-encoded CRISPR-Cas systems direct RNA-guided DNA integration[J]. Nature, 2019, 571(7764): 219-225. DOI:10.1038/s41586-019-1323-z |
| [39] |
Urnov FD, Miller JC, Lee YL, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases[J]. Nature, 2005, 435(7042): 646-651. DOI:10.1038/nature03556 |
| [40] |
Bibikova M, Beumer K, Trautman JK, et al. Enhancing gene targeting with designed zinc finger nucleases[J]. Science, 2003, 300(5620): 764. DOI:10.1126/science.1079512 |
| [41] |
Shrivastav M, De Haro LP, Nickoloff JA. Regulation of DNA double-strand break repair pathway choice[J]. Cell Res, 2008, 18(1): 134-147. DOI:10.1038/cr.2007.111 |
| [42] |
Lieber MR. NHEJ and its backup pathways in chromosomal translocations[J]. Nat Struct Mol Biol, 2010, 17(4): 393-395. DOI:10.1038/nsmb0410-393 |
| [43] |
Merkle FT, Neuhausser WM, Santos D, et al. Efficient CRISPR-Cas9-mediated generation of knockin human pluripotent stem cells lacking undesired mutations at the targeted locus[J]. Cell Rep, 2015, 11(6): 875-883. DOI:10.1016/j.celrep.2015.04.007 |
| [44] |
Yao X, Wang X, Hu X, et al. Homology-mediated end joining-based targeted integration using CRISPR/Cas9[J]. Cell Res, 2017, 27(6): 801-814. DOI:10.1038/cr.2017.76 |
| [45] |
Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes[J]. Nat Biotechnol, 2014, 32(4): 347-355. DOI:10.1038/nbt.2842 |
| [46] |
Maresca M, Lin VG, Guo N, et al. Obligate ligation-gated recombination(ObLiGaRe): Custom-designed nuclease-mediated targeted integration through nonhomologous end joining[J]. Genome Res, 2013, 23(3): 539-546. DOI:10.1101/gr.145441.112 |
| [47] |
Suzuki K, Tsunekawa Y, Hernandez-Benitez R, et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration[J]. Nature, 2016, 540(7631): 144-149. DOI:10.1038/nature20565 |
| [48] |
Heyer W, Ehmsen KT, Liu J. Regulation of homologous recombination in eukaryotes[J]. Annu Rev Genet, 2010, 44(1): 113-139. DOI:10.1146/annurev-genet-051710-150955 |
| [49] |
Gutschner T, Haemmerle M, Genovese G, et al. Post-translational Regulation of Cas9 during G1 Enhances Homology-Directed Repair[J]. Cell Rep, 2016, 14(6): 1555-1566. DOI:10.1016/j.celrep.2016.01.019 |
| [50] |
Zhang JP, Li XL, Li GH, et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage[J]. Genome Biol, 2017, 18(1): 35. |
| [51] |
聂宇, 乔艳乐, 陈瑶生, 等. 供体同源臂长度对ZFN介导的同源重组效率的影响[J]. 中山大学学报:自然科学版, 2016, 55(4): 100-107. |
| [52] |
朱娉慧, 罗群, 王曜峰, 等. 同源重组及非同源末端连接修复途径介导的基因编辑: CRISPR技术的认知、应用及展望[J]. 生命科学, 2018, 30(9): 1003-1009. |
| [53] |
Jiricny J. The multifaceted mismatch-repair system[J]. Nat Rev Mol Cell Biol, 2006, 7(5): 335-346. |
| [54] |
Nakade S, Tsubota T, Sakane Y, et al. Microhomology-mediated end-joining-dependent integration of donor DNA in cells and animals using TALENs and CRISPR/Cas9[J]. Nat Commun, 2014, 5(1): 1. |
| [55] |
Britton S, Coates J, Jackson SP. A new method for high-resolution imaging of Ku foci to decipher mechanisms of DNA double-strand break repair[J]. J Cell Biol, 2013, 202(3): 579-595. DOI:10.1083/jcb.201303073 |
| [56] |
Auer TO, Del BF. Homology-Independent Integration of Plasmid DNA into the Zebrafish Genome[J]. Methods Mol Biol, 2016, 1451: 31-51. |
| [57] |
He X, Tan C, Wang F, et al. Knock-in of large reporter genes in human cells via CRISPR/Cas9-induced homology-dependent and independent DNA repair[J]. Nucleic Acids Rre, 2016, 44(9): e85. DOI:10.1093/nar/gkw064 |
| [58] |
Shi M, Kawabe Y, Ito A, et al. Targeted knock-in into the OVA locus of chicken cells using CRISPR/Cas9 system with homology-independent targeted integration[J]. J Biosci Bioeng, 2020, 129(3): 363-370. DOI:10.1016/j.jbiosc.2019.09.011 |
| [59] |
McVey M, Lee SE. MMEJ repair of double-strand breaks(director's cut): Deleted sequences and alternative endings[J]. Trends Genet, 2008, 24(11): 529-538. DOI:10.1016/j.tig.2008.08.007 |
| [60] |
Sakuma T, Nakade S, Sakane Y, et al. MMEJ-assisted gene knock-in using TALENs and CRISPR-Cas9 with the PITCh systems[J]. Nat Protoc, 2016, 11(1): 118-133. DOI:10.1038/nprot.2015.140 |
| [61] |
Taleei R, Nikjoo H. Biochemical DSB-repair model for mammalian cells in G1 and early S phases of the cell cycle[J]. Mutat Res, 2013, 756(1): 206-212. |





