2. 加拿大阿尔伯塔大学农业食品营养科学系, 埃德蒙顿, 加拿大 T6G 2P5
2. Department of Agricultural, Food & Nutritional Science, University of Alberta, Edmonton T6G 2P5, Canada
奶及奶制品是人类最重要的食用蛋白质来源之一。奶牛作为奶及奶制品的供应源, 起着关键作用。虽然当前奶牛的产奶性能已经达到了较高水平, 如美国奶牛的年平均产奶量超过了10 t[1], 但随着世界人口的持续增长以及人均牛奶需求量的逐渐增加, 如何进一步提高奶牛生产效率和环境可持续性成为了当前紧迫的任务。目前, 该方面的研究已经从关注传统的泌乳调控的外部要素(如改变日粮的营养成分等)逐渐转变为揭示泌乳调节的生物学过程[2]。奶牛的瘤胃微生物包括细菌、古菌、真菌和原虫4大类, 它们负责降解和发酵不同类型的饲料, 从而为宿主的生长、繁殖、维持和泌乳提供大量营养物质, 这就为揭示相关的生物学过程提供了巨大的潜力[3]。瘤胃中的微生物通过发酵合成挥发性脂肪酸(Volatile fatty acid, VFA), 微生物蛋白(Microbial crude protein, MCP)和维生素等养分与宿主建立联系, 协助宿主完成多种生理活动[4], 这为研究和调控奶牛的泌乳生物学过程提供了帮助, 有利于实现更加高效和可持续的奶业。
1990年前, 奶牛瘤胃微生物的研究主要依靠基于培养的方法, 但由于大多数瘤胃微生物是不可培养的, 因此得到的信息非常有限[5]。之后出现了一系列新技术, 并逐渐应用于奶牛瘤胃微生物领域。以"奶牛"和"瘤胃微生物"的英文翻译为关键词在Web of Science数据库中进行检索, 结果显示从1990-2019年, 共有2 052篇相关SCI文章发表, 2013年以后文章数量增长较快(图 1)。使用一个词云来说明这2 052篇论文摘要中出现的高频词, 其中最常见的有"甲烷"、"瘤胃"、"发酵"、"MCP"、"VFA"、"奶产量"、"16S"和"宏基因组学"(图 1)。这表明在奶牛瘤胃微生物领域的已发表研究中, 与效率和生产有关的参数受到了更多的关注。此外, 近年来瘤胃微生物对犊牛健康和发育的影响也开始受到了重视[6-7]。越来越多的研究强调了二代测序(Nest generation sequence, NGS)技术在检测和分析微生物中的重要性, 如扩增子测序、宏基因组学、宏转录组学和宏蛋白组等。16S rRNA基因等扩增子测序、宏基因组学和宏转录组学分别用于识别操作分类单元(Operational taxonomic units, OTU)/扩增子序列变异体(Amplicon sequence variants, ASV)、预测功能信息和活性微生物/代谢通路(图 1)。这些基于NGS的研究需要扎实的综合知识, 包括生物信息学、分析化学、生物学等。最近一项基于283个牛瘤胃微生物宏基因组二、三代混合测序的研究将宏基因组数据比对率从15%提高至50%-70%, 同时也提供了宏基因组分箱、下游分析的全套流程和方法标准[8]。即便NGS高通量分析方法已经被广泛接受并形成标准流程, 但在应用过程中的技术性操作和数据处理分析产生的变异也必须最小化[9]。在NGS分析流程中, 如样品DNA/RNA的提取[10]、分类学数据库的比对[11]、数据归一化[12], 任何不当的处理都将导致微生物检测的结果出现显著偏差和错误。
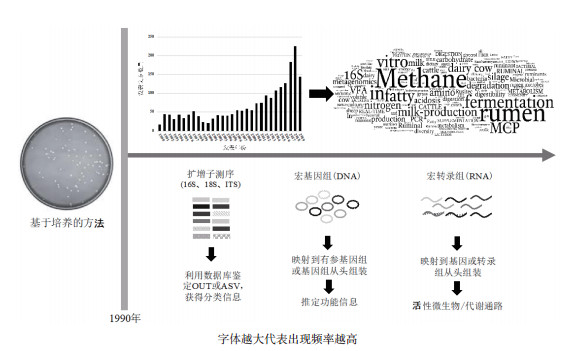
|
| 图 1 奶牛瘤胃微生物领域论文发表、研究热点和研究方法 |
奶牛瘤胃微生物研究正处于快速发展阶段, 但同时也面临着巨大挑战。瘤胃微生物可以在多大程度上帮助人们揭示奶牛泌乳的基本生物学机制仍然是一个问题。为了更好理解瘤胃微生物与奶牛生长发育和重要表型的关系, 以期从瘤胃微生物调控策略角度提高奶牛生产效率和环境可持续性, 本文系统综述了瘤胃微生物在产奶效率、甲烷排放和瘤胃发育方面的研究进展, 详细归纳了瘤胃微生物与奶牛主要生产性状关系, 同时指出了奶牛瘤胃微生物未来的研究趋势。
1 瘤胃微生物与奶牛产奶效率目前, 全球奶牛养殖业面临的重要挑战之一是如何提高动物对饲料能量的利用效率[13]。产奶效率是指产奶动物研究上所使用的饲料效率的概念, 反映了牧场盈利能力和对环境的影响[14]。产奶效率的不同通常归因于先进的遗传选育、饲养和管理策略[15-16]。但是, 产奶效率是一个复杂的性状, 除了遗传因素外, 奶牛个体之间的差异还受许多因素的影响[17]。有证据表明, 瘤胃微生物的组成、菌群内部以及与宿主的相互作用与乳成分和饲料效率有关[18-19]。作为瘤胃中微生物群发酵的最终产物, VFA可以提供奶牛70%以上的能量需要[19], MCP可以提供60%的小肠可吸收非氨态氮[20]。VFA和MCP给奶牛提供了大量的养分, 并显著影响营养状况, 最终导致饲料效率变化。加拿大Leluo Guan教授实验室发现肉牛的饲料效率与瘤胃微生态[21]、细菌菌群[22-23]和甲烷菌群有关[24], 且最新的试验表明某些与饲料效率相关的瘤胃微生物特征可遗传[25]。其他研究人员也发现了相似的结果[26-27]。相比之下, 奶牛在一个泌乳周期不同的生理阶段(如泌乳早期、泌乳中期、泌乳后期、干奶期和围产期), 能量需求存在较大波动, 这为类似的研究带来了更多挑战。
Jami等[4]对15头奶牛的泌乳性能参数与瘤胃细菌群落进行相关性分析发现, 产奶量和乳成分与各种瘤胃细菌的丰度密切相关, 如Adlercreutzia与产奶量呈正相关(R=0.57, P=0.027), Firmicutes与Bacteroidetes的比率和乳脂产量之间也存在很强的相关性(R=0.72, P=0.002)。但是, 这项研究并未考虑泌乳阶段的影响, 因此, 他们实验室使用454焦磷酸测序进一步表征了14头荷斯坦奶牛在前两个泌乳周期的早期、中期和后期的细菌群落组成[28]。结果表明, 即使在相同的日粮条件下, 瘤胃微生物菌群在不同泌乳期和年龄中也是动态的, 低效率奶牛的Anaerovibrio和Butyrivibrio的丰度较高, 而Coprococcus的丰度较低, 这表明瘤胃微生物菌群在影响奶牛产奶效率方面可能起着至关重要的作用。本课题组最近对334头奶牛瘤胃微生物的16S rRNA基因进行测序发现, 瘤胃菌群与产奶能力之间存在显著相关, 其中核心微生物群对关联的贡献为52.9%[29]。Shabat等[30]比较了40头泌乳中期的高效率奶牛和38头低效率奶牛的瘤胃细菌和古菌发现, 高饲料效率奶牛有着较低的瘤胃微生物组基因含量和OTU丰度, 他们还提出了Megasphaera elsdenii和Coprococcus catus是产奶效率的关键决定因素。Xue等[31]比较了20头低产奶量、低乳蛋白产量(LL)奶牛和20头高产奶量、高乳蛋白含量(HH)奶牛的瘤胃细菌菌群发现, HH组奶牛瘤胃的Sharpea属相对丰度显著高于LL组, 而Succinivibrio显著低于LL组。这些结果表明, 瘤胃中的少数微生物种类可能会在一定程度上影响微生物菌群的结构和功能, 最终改变宿主的生产效率。因此, 对瘤胃微生物作用机制的理解可以潜在地应用于实现高效和环保的畜牧业。在围产期, 适应日粮的变化对瘤胃微生物来说是一个很大的挑战[32]。Lima等[33]使用MiSeq对115头高产奶牛产前和产后瘤胃微生物的16S rRNA和18S rRNA进行了测序, 结果显示, 与产后奶牛相比, 产前真菌的丰富度较高, 而纤毛虫的丰富度较低, 产前和产后瘤胃微生物菌群的变化曲线可用于准确预测宿主的生产性状。Jewell等[28]对奶牛连续3个泌乳周期进行研究发现, 较高丰度的Christensenellaceae家族与产奶效率也呈负相关。Derakhshani等[34]研究发现, 在产前, 当奶牛的自由干物质采食量减少时, 瘤胃微生物的个体间差异大大降低, 在产后, 当自由干物质采食量增加时, 蛋白降解菌、淀粉分解菌和乳酸产生菌的丰富度较高, 这表明瘤胃微生物能够从高浓度碳水化合物的产后泌乳日粮中获取能量。Indugu等[35]使用16S rRNA基因扩增子序列比较了泌乳中期的47头高产奶牛和38头低产奶牛的瘤胃细菌组成, 结果发现在高产奶牛中, Succinivibrionaceae丰度较高, Prevotella、S24-7和Succinivibrionaceae与产奶量呈正相关, 但是该研究并没有排除日粮因素对产奶量的影响。为了研究其中的作用机制, 而不仅仅是瘤胃微生物群与产奶效率之间的相关性, Weimer等[14]把高低效率奶牛的瘤胃内容物几乎完全互换, 结果表明瘤胃细菌群落对产奶效率有明显的贡献, 并提示移植和重塑整个瘤胃微生物菌群可以改变宿主表型。通过评估低、中、高产奶量和干奶期的荷斯坦奶牛的瘤胃细菌、古菌和真菌对甲烷排放量和生产性能的影响, Cunha等[36]的研究发现, 瘤胃中高丰度的Christensenel-laceae, Mogibacteriaceae, S24-7, Butyrivibrio, Shwartzia, Treponema和Methanosphaera可能会减少甲烷排放并提高产奶量。
与产奶效率相关的瘤胃微生物的研究工作主要是在近几年完成的, 这些研究大大拓宽了我们对瘤胃微生物与宿主生产性能之间的相互作用的理解[37]。根据已有的研究结果, 对瘤胃微生物进行调控的可行性越来越大, 如直接给奶牛口服活性微生物对提高产奶量有很好的效果[38]。然而, 目前对瘤胃微生物菌群的认知还非常有限。对瘤胃中特定的微生物和微生物间复杂的相互作用的进一步探索需要更先进的技术, 能在种或菌株水平来准确鉴定微生物[39]。与目前依赖相关性和推测分析的研究相比, 建立一个更可靠的模型来评估瘤胃微生物菌群对产奶量的影响, 并预测瘤胃微生物对日粮调控的反应, 更具挑战性[40]。在未来, 将微生物这个因素纳入"遗传-环境-瘤胃-微生物菌群"的相互作用模型中, 来加深对奶牛泌乳机制和调控的了解是件值得期待的事情。在微生物分析中除了微生物多样性和预测功能外, 关注直接反映微生物代谢活性的下游代谢物, 对了解瘤胃发生的生物学机制也具有重要作用[41-42]。利用有效途径改善瘤胃功能只是时间问题, 但选择调控方法时, 必须考虑瘤胃微生物群代谢引起的环境问题, 特别是来自甲烷代谢的环境问题[43]。
2 瘤胃古菌的甲烷排放与减排的研究进展家畜胃肠道甲烷排放约占全球甲烷排放量的17%[44]。奶牛排放的甲烷占其能量损失的5.3%- 6.1%[45-46], 甲烷的增温潜力是二氧化碳的25倍[47], 减少胃肠道甲烷的排放对经济和环境都有积极的影响。
瘤胃中的古菌是主要产甲烷菌。近20年来, 对瘤胃古菌的研究蓬勃发展, 其主要集中于瘤胃古菌群落的组成及开发调控甲烷菌的方法的研究。一般来说, 泌乳的反刍动物每克瘤胃内容物中含有107- 108个古菌。甲烷菌群通常由3个主要的系统发育群组成, 包括Methanobrevibacter、Methanomicrobium和Methanomassiliicoccales[48]。
降低动物胃肠道甲烷排放的研究主要集中在降低古菌总数上, 如添加食用油和脂肪酸[49-50], 甲烷菌的免疫抑制[51]、清除原虫[52-54]和调整日粮配方[55]等措施。然而, 无论古菌总数是否受到影响, 这些方法都没有实现长期有效地减少甲烷排放。于是有人提出了一个问题:古菌总数是否重要。事实上, 后来的研究已经证明, 古细总数量与宿主甲烷排放量无关[24, 56-57]。相反, 古菌群落的结构被认为与宿主甲烷排放有关。与Methanobrevibacter gottschalkii相关的古菌系统发育群和荷斯坦奶牛甲烷排放量呈正相关[58]。Danielsson等[59]根据King等[51]和Danielsson等[56]的研究, 通过将系统发育群划分为SGMT组(Methanobrevibacter smithii、Methanobrevibacter gottschalkii、Methanobrevibacter millerae和Methanobrevibacter thaueri)和RO组(Methanobrevibacter ruminantium和Methanobrevibacter olleyae)来研究瘤胃的古菌群落, 部分解释了奶牛胃肠道甲烷排放的变化。RO组相对丰度越高, 奶牛甲烷排放量越低[59]。一项基于超过1 000头奶牛基因型和16S rRNA和18S rRNA测序的研究, 发现奶牛具有的核心微生物群落对决定甲烷排放有显著作用[60]。因此, 减少动物胃肠道甲烷排放的有效方法可能依赖个别古菌而非整个古菌群落。
不同的饮食结构可以改变宿主瘤胃古菌群落的组成, 但在之前的研究中, 饮食干预并没有实现长期减少甲烷排放[61-62]。2014年有学者开发的3-硝基丙醇(3-NOP), 是一种结构上类似于甲基辅酶M的化合物, 在对多个奶牛研究中证明其可以有效地减少胃肠道甲烷的排放[63-64]。这种化合物针是对甲烷生成的最后一个步骤来抑制瘤胃甲烷的产生[65]。有研究证明海藻也可以减少胃肠道甲烷的排放。用海藻喂养肉牛时, 农场内的甲烷排放量大大降低(Kiney等, 未发表)。在最近的一项研究中, 用风干的棕色海藻饲喂公羊, 古菌数量减少了50%以上[66]。之前的任何研究均未实现如此显著的古菌种群减少, 这需要进一步研究饲喂海藻减少动物胃肠道甲烷排放的机制。
改变宿主幼年时的瘤胃古菌群落的组成可能是另一种甲烷减排的方法。断奶前的犊牛胃肠道几乎不产生甲烷, 但甲烷菌群在犊牛幼年时期就已发育。Zhou等[67]对3周龄的犊牛胃肠道的微生物进行了研究发现, 尽管个体间的菌群相对丰度和组成差异很大, 但在提供固体饲料之前, 甲烷菌群已形成。正如Yáñez-Ruiz[68]等提出的, 在反刍动物的幼年时期, 特别是在瘤胃微生物群完全建立之前, 是"预先规划"瘤胃微生物区系的理想时间窗口。因此, 有必要进一步研究如何应用早期干预方法建立一个既能产生较少甲烷又能有效处理氢气的古菌群落。
很多研究都是基于基因组DNA和RNA的分析方法开展的, 在对微生物DNA样品进行检测的研究中, 无论是采用传统的分析方法(如克隆文库、PCR-DGGE、T-RFLP), 还是高通量测序方法(如扩增子测序、宏基因组), 人们一直认为与Methanobrevibacter相关的古菌是瘤胃中产甲烷的主要的微生物。然而, 一项基于宏转录组分析活性微生物群的研究发现, 相对丰度最高的古菌转录本与Methanomassillicoccales有关[69]。这一结果提示需要重新考虑瘤胃古菌群落研究方法的重要性以及不同方法在多大程度上反映出"真实的微生物群落"。瘤胃古菌群落研究的局限性还表现在所参考数据库的选择和分类学方法的选择上。当使用基于16S rRNA基因序列的方法和宏基因组时, 从DNA样本中发现了不同的古菌组成。不同数据库(SILVA、RDP、Greengenes、NCBI和OTT)中包含的不同参考序列也导致相同样本的不同分类归属[11]。因此, 对瘤胃古菌群落的研究迫切需要规范的工作流程。但新的分析和测序方法的出现将有助于更好地了解包括古菌在内的瘤胃微生物的组成及功能。
3 瘤胃微生物在瘤胃发育中的作用 3.1 组学和瘤胃发育早期微生物近年来, 随着NGS的广泛应用, 对反刍动物建立反刍行为前的瘤胃微生物研究成为了热点。在这些NGS方法中, 最常用的方法是16S rRNA基因的扩增子测序。有研究利用扩增子测序发现瘤胃微生物定植始于犊牛出生的第1天[70]。瘤胃细菌的复杂性(多样性)随着犊牛年龄的增长而增加, 同时个体间的异质性减少[70]。Rey等[71]对同一组犊牛从出生到第83天进行研究, 以探索瘤胃微生物定植的时间动态变化, 尽管他们没有对犊牛出生第1天的瘤胃细菌群落进行分析, 但成功地展示了不同分类水平下细菌群落的时间变化, 特别是这项研究清楚地表明, 优势菌群的丰度随犊牛年龄及其采食量的变化而变化。例如, 随着固体饲料的引入, 犊牛瘤胃中相对丰度最高的细菌属之一Prevotella的丰度显著增加[70, 72-73]。Jami等[70]和Rey等[71]使用瘤胃液来研究瘤胃细菌群落, 通过比较瘤胃内容物与瘤胃黏膜表面附着细菌的含量, 发现两个微生物群落之间存在显著差异[73], 因此在研究幼龄反刍动物的瘤胃微生物组成时, 这两个微生物群都应该纳入考虑。
扩增子测序主要是产生关于分类组成及其相对丰度的数据, 值得一提的是, 利用一些生物信息学分析方法, 如PICRUSt[74]、Tax4Fun[75]、PICRUSt2[76]等可以根据扩增子测序数据预测微生物功能。与扩增子测序相比, 宏基因组测序允许通过对微生物的全基因组测序来探索菌群分类和功能组成。用宏基因组评估动物建立反刍行为前的微生物区系的报道较少[72, 77]。但这些研究已经报道了在建立反刍行为前, 甚至在引入固体食物之前, 瘤胃中就存在水解复杂碳水化合物中糖苷键的糖苷水解酶。扩增子测序显示, 在建立反刍行为前, 瘤胃中就存在类似成年反刍动物的菌群, 而宏基因组测序显示, 类似成年反刍动物的菌群包含复杂的碳水化合物发酵所需的基因。尽管基因的存在并不一定意味着它们有活性, 但基于宏基因组测序的方法为探索反刍动物建立反刍行为前的瘤胃微生物提供了在功能层面的线索。因此, 在未来, 使用宏转录组的测序方法将有助于更好地了解反刍动物建立反刍行为前的瘤胃微生物群的活性/功能。这对于预测早期微生物在瘤胃发育过程中的作用是至关重要的。
3.2 影响早期发育瘤胃微生物组成的因素对早期发育瘤胃微生物的研究提示了很多可能影响微生物区系的因素, 这些因素主要分为生物学因素(年龄、饮食、断奶方法/年龄、个性差异)和技术因素(取样方法/类型、测序方法、16S靶区、数据库/质量控制/生物信息学工具、环境污染)。探索瘤胃微生物定植过程的研究[70-71, 77-78]清楚地证明了瘤胃微生物组的年龄依赖性变化, 还需要注意的是, 这些研究中的年龄依赖性变化总是与饮食变化相结合。在Li等[72]的研究中, 犊牛出生后只饲喂代乳品, 并在第2周和第6周采集瘤胃液进行16S rRNA和全基因组鸟枪法测序。因此, 观察到的微生物群落的组成和功能的变化仅代表瘤胃微生物的年龄依赖性变化。
Abecia等[79-80]使用双胞胎羔羊进行两种饮食干预, 这样可以使遗传变异的影响最小化, 从而充分反映出断奶前饮食对瘤胃微生物群的影响。这些研究表明, 早期饮食干预的变化(代乳品与哺乳)影响瘤胃微生物群[79-80]及其定植过程[81]。因此, 即使在断奶后, 早期喂养也是调节瘤胃微生物定植过程和微生物组成的重要因素之一。然而, 这种饲养方式是如何影响与早期瘤胃发育过程相关的瘤胃微生物群功能的问题仍未得到解答。
影响早期瘤胃微生物的生物学因素可以真实地解释瘤胃微生物定植过程, 但技术因素可能会使结果出现偏差。特别是, 在扩增子测序中使用16S rRNA基因高变区和引物会引入假象[82-83]。Jovel等[79]清楚地展示了基于NGS技术层面影响肠道微生物数据解释的因素。他们在宏基因组和扩增子测序中使用了模拟(人工)菌群, 并比较了数据处理和分类的可用方法以评估最适合的方法。虽然扩增子测序存在很多问题, 但宏基因组测序因为缺乏参考基因组而受到影响。特别是, 他们观察到, 在没有生物参考基因组的情况下, 序列会被分配到最接近的参考基因组。此外, 环境污染也是影响群落组成的重要因素。因此, 样品制备过程使用阴性对照可以避免污染造成的偏差。
3.3 瘤胃微生物在瘤胃早期发育中的作用目前很多研究关注反刍动物断奶过渡期瘤胃的生理(瘤胃乳头长度、瘤胃大小)、代谢(发酵能力)和转录(转录组、microRNA组)的变化。虽然固体饲料的引入有助于瘤胃的生理和代谢发育, 但瘤胃发育背后的分子机制是个体发育控制的[85]。然而, 有研究试图揭示早期瘤胃发育背后的分子机制[86-89]。Connor及其同事还提出, 潜在的分子标记物(转化生长因子-β1和雌激素相关受体-α)与瘤胃发育过程相关[87]。此外, 瘤胃细菌与microRNAs表达之间的关联性表明, 早期微生物群可能参与瘤胃早期发育过程中的转录调控[88]。近期Malmuthuge等[90]通过整合犊牛在出生30 min, 第1周、第3周和第6周的瘤胃微生物(组织和内容物)宏基因组和宿主瘤胃组织转录组数据发现, 早期微生物群可能通过锌的吸收影响瘤胃发育, 这种相互作用可能通过miRNA进行调节。
人们对肠道微生物菌群在哺乳动物胃肠道发育中的作用已有较为充分的研究[86], 但对瘤胃微生物群(一种研究时间最长和最多的宿主相关微生物群)对个体发育的调控机制的认识依然非常有限。Malmuthuge等[92]研究了瘤胃微生物组、转录组和microRNA组之间的相互作用, 并利用一个综合的网络分析来揭示潜在的微生物驱动瘤胃发育的分子机制。这项研究进一步表明, 组织发育和代谢相关基因和microRNAs(26.3%的mRNAs和46.4%的microRNAs)与微生物代谢产物浓度相关, 这意味着并非所有与瘤胃发育相关的机制都是个体发育调节的。然而, 这些机制仍然只是基于关联分析, 有待进一步通过设计试验和功能验证来证实。利用免疫染色在蛋白质水平上确认瘤胃微生物驱动瘤胃发育的变化, 可能为验证基于关联分析的预测和鉴定微生物驱动的瘤胃发育提供更多的机会。
4 小结与展望瘤胃微生物种类繁多、功能强大, 其参与或影响宿主生理功能及表型的特征和机制逐渐被认识。在奶牛研究中, 有关瘤胃微生物如何影响产奶性能、甲烷排放和瘤胃发育的研究被相继报道, 一些关键的微生物种类和代谢通路也被揭示。为了更加系统便捷的展示相关结果, 我们整理了瘤胃微生物与奶牛主要生产性状的关系(表 1), 其中涵盖了具体性状、各项研究中的技术方法和主要发现。这些研究结果有助于我们理解瘤胃自身发育、调控甲烷产生以及提高牛奶产量和效率, 对将来通过微生物调控策略实现奶业高效可持续发展也提出新的见解。
同时我们也应该认识到, 奶牛瘤胃微生物的研究还远远不够。例如, 对于瘤胃微生物成员的解析还需要大量的工作, 尽管目前基于培养的方法, 对瘤胃细菌和古菌的研究取得了一些进展[93], 但更多的细菌和古菌仍需要建立培养方法在实验室进行研究。近期基于283个牛瘤胃微生物宏基因组二、三代混合测序的研究大幅度提高了宏基因组数据比对率[8], 但据估计至少还有1 000种以上瘤胃微生物有待测序和组装, 尤其是对瘤胃中真核生物(如厌氧真菌、原虫)和病毒组的测序仍然存在巨大的技术挑战。长读长的三代测序和单细胞微生物测序将有助于探索新型功能基因和物种丰度极小的未被培养的瘤胃微生物。此外, 在功能层面, 功能基因的表达和编码蛋白的活性研究还需要借助宏转录组和宏蛋白组开展大量工作。目前对瘤胃微生物代谢过程中代谢丰富度的探究方法依然不足, 因此对瘤胃微生物代谢过程的研究可能只是盲人摸象, 有学者指出通过使用功能组(Functional group)整合瘤胃微生物物种分类(因为不同的物种分类的微生物在功能上也可能相同, 同种物种分类的微生物功能也存在差异)来解决由于水平基因转移事件以及功能冗余而造成的分类混乱, 进而更好地解析瘤胃中的具体代谢途径[94]。奶牛瘤胃微生物功能的研究还需要逐渐从描述性向机制性转变, 将来微生物组和其他组学及宿主功能基因组的联合分析将有助于识别和构建瘤胃微生物的功能性和生态性网络, 进而通过网络调控减少甲烷排放和提高泌乳效率。
| [1] |
Baumgard L, Collier R, Bauman D. A 100-year review:regulation of nutrient partitioning to support lactation[J]. Journal of Dairy Science, 2017, 100(12): 10353-10366. DOI:10.3168/jds.2017-13242 |
| [2] |
Bradford BJ, Yuan K, Ylioja C. Managing complexity:Dealing with systemic crosstalk in bovine physiology[J]. Journal of Dairy Science, 2016, 99(6): 4983-4996. DOI:10.3168/jds.2015-10271 |
| [3] |
Jami E, Mizrahi I. Composition and similarity of bovine rumen microbiota across individual animals[J]. PLoS One, 2012, 7(3): e33306. DOI:10.1371/journal.pone.0033306 |
| [4] |
Jami E, White BA, Mizrahi I. Potential role of the bovine rumen microbiome in modulating milk composition and feed efficiency[J]. PLoS One, 2014, 9(1): e85423. DOI:10.1371/journal.pone.0085423 |
| [5] |
Hungate RE. The rumen and its microbes[M]. Elsevier, 2013.
|
| [6] |
Malmuthuge N, Griebel P, Guan L. The gut microbiome and its potential role in the development and function of newborn calf gastrointestinal tract[J]. Frontiers in Veterinary Science, 2015, 2: 36. |
| [7] |
Malmuthuge N. Understanding the gut microbiome of dairy calves:Opportunities to improve early-life gut health[J]. Journal of Dairy Science, 2017, 100(7): 5996-6005. DOI:10.3168/jds.2016-12239 |
| [8] |
Stewart RD, Auffret MD, Warr A, et al. Compendium of 4, 941 rumen metagenome-assembled genomes for rumen microbiome biology and enzyme discovery[J]. Nature Biotechnology, 2019, 37(8): 953-961. DOI:10.1038/s41587-019-0202-3 |
| [9] |
Pfeifer S. From next-generation resequencing reads to a high-quality variant data set[J]. Heredity, 2017, 118(2): 111. DOI:10.1038/hdy.2016.102 |
| [10] |
Costea PI, Zeller G, Sunagawa S, et al. Towards standards for human fecal sample processing in metagenomic studies[J]. Nature Biotechnology, 2017, 35(11): 1069. DOI:10.1038/nbt.3960 |
| [11] |
Balvočiūtė M, Huson DH. SILVA, RDP, Greengenes, NCBI and OTT-how do these taxonomies compare?[J]. BMC Genomics, 2017, 18(2): 114. |
| [12] |
Weiss S, Xu Z, Peddada S, et al. Normalization and microbial differential abundance strategies depend upon data characteristics[J]. Microbiome, 2017, 5(1): 27. DOI:10.1186/s40168-017-0237-y |
| [13] |
Connor E. Invited review:improving feed efficiency in dairy production:challenges and possibilities[J]. Animal, 2015, 9(3): 395-408. DOI:10.1017/S1751731114002997 |
| [14] |
Weimer PJ, Cox MS, Vieira de Paula TV, et al. Transient changes in milk production efficiency and bacterial community composition resulting from near-total exchange of ruminal contents between high-and low-efficiency Holstein cows[J]. Journal of Dairy Science, 2017, 100(9): 7165-7182. DOI:10.3168/jds.2017-12746 |
| [15] |
Arndt C, Powell JM, Aguerre M, et al. Feed conversion efficiency in dairy cows:Repeatability, variation in digestion and metabolism of energy and nitrogen, and ruminal methanogens[J]. Journal of Dairy Science, 2015, 98(6): 3938-3950. DOI:10.3168/jds.2014-8449 |
| [16] |
VandeHaar M, Armentano L, Weigel K, et al. Harnessing the genetics of the modern dairy cow to continue improvements in feed efficiency[J]. Journal of Dairy Science, 2016, 99(6): 4941-4954. DOI:10.3168/jds.2015-10352 |
| [17] |
Berry DP, Coffey M, Pryce J, et al. International genetic evaluations for feed intake in dairy cattle through the collation of data from multiple sources[J]. Journal of Dairy Science, 2014, 97(6): 3894-3905. DOI:10.3168/jds.2013-7548 |
| [18] |
Rius A, Kittelmann S, Macdonald K, et al. Nitrogen metabolism and rumen microbial enumeration in lactating cows with divergent residual feed intake fed high-digestibility pasture[J]. Journal of Dairy Science, 2012, 95(9): 5024-5034. DOI:10.3168/jds.2012-5392 |
| [19] |
France J, Dijkstra J. Volatile fatty acid production[J]. Quantitative Aspects of Ruminant Digestion and Metabolism, 2005, 2: 157-175. |
| [20] |
Clark J, Klusmeyer T, Cameron M. Microbial protein synthesis and flows of nitrogen fractions to the duodenum of dairy cows[J]. Journal of Dairy Science, 1992, 75(8): 2304-2323. DOI:10.3168/jds.S0022-0302(92)77992-2 |
| [21] |
Guan L, Nkrumah JD, Basarab JA, et al. Linkage of microbial ecology to phenotype:correlation of rumen microbial ecology to cattle's feed efficiency[J]. FEMS Microbiology Letters, 2008, 288(1): 85-91. DOI:10.1111/j.1574-6968.2008.01343.x |
| [22] |
Hernandez-Sanabria E, Goonewardene LA, Li M, et al. Correlation of particular bacterial PCR-denaturing gradient gel electrophoresis patterns with bovine ruminal fermentation parameters and feed efficiency traits[J]. Applied Environmental Microbiology, 2010, 76(19): 6338-6350. DOI:10.1128/AEM.01052-10 |
| [23] |
Hernandez-Sanabria E, Goonewardene LA, Wang Z, et al. Impact of feed efficiency and diet on adaptive variations in the bacterial community in the rumen fluid of cattle[J]. Applied Environmental Microbiology, 2012, 78(4): 1203-1214. DOI:10.1128/AEM.05114-11 |
| [24] |
Zhou M, Hernandez-Sanabria E. Characterization of variation in rumen methanogenic communities under different dietary and host feed efficiency conditions, as determined by PCR-denaturing gradient gel electrophoresis analysis[J]. Applied Environmental Microbiology, 2010, 76(12): 3776-3786. DOI:10.1128/AEM.00010-10 |
| [25] |
Li F, Li C, Guan L, et al. Host genetics influence the rumen microbiota and heritable rumen microbial features associate with feed efficiency in cattle[J]. Microbiome, 2019, 7: 92. DOI:10.1186/s40168-019-0699-1 |
| [26] |
Carberry CA, Kenny DA, Han S, et al. Effect of phenotypic residual feed intake and dietary forage content on the rumen microbial community of beef cattle[J]. Applied Environmental Microbiology, 2012, 78(14): 4949-4958. DOI:10.1128/AEM.07759-11 |
| [27] |
Myer PR, Smith TP, Wells JE, et al. Rumen microbiome from steers differing in feed efficiency[J]. PLoS One, 2015, 10(6): e0129174. DOI:10.1371/journal.pone.0129174 |
| [28] |
Jewell KA, McCormick CA, Odt CL, et al. Ruminal bacterial community composition in dairy cows is dynamic over the course of two lactations and correlates with feed efficiency[J]. Applied and Environmental Microbiology, 2015, 81(14): 4697-4710. DOI:10.1128/AEM.00720-15 |
| [29] |
Xue M, Sun H, Liu J, et al. Host genetics influence the rumen microbiota and heritable rumen microbial features associate with feed efficiency in cattle[J]. Environmental Microbiology, 2018, 84: 19. |
| [30] |
Shabat SKB, Sasson G, Doron-Faigenboim A, et al. Specific microbiome-dependent mechanisms underlie the energy harvest efficiency of ruminants[J]. The ISME Journal, 2016, 10(12): 2958-2972. DOI:10.1038/ismej.2016.62 |
| [31] |
Xue M, Sun H, Liu J, et al. Assessment of rumen bacteria in dairy cows with varied milk protein yield[J]. Journal of Dairy Science, 2019, 102(6): 5031-5040. DOI:10.3168/jds.2018-15974 |
| [32] |
Nocek J, Kautz W, Leedle J, et al. Direct-fed microbial supplementation on the performance of dairy cattle during the transition period[J]. Journal of Dairy Science, 2003, 86(1): 331-335. DOI:10.3168/jds.S0022-0302(03)73610-8 |
| [33] |
Lima FS, Oikonomou G, Lima SF, et al. Prepartum and postpartum rumen fluid microbiomes:characterization and correlation with production traits in dairy cows[J]. Applied and Environmental Microbiology, 2015, 81(4): 1327-1337. DOI:10.1128/AEM.03138-14 |
| [34] |
Derakhshani H, Tun HM, Cardoso FC, et al. Linking peripartal dynamics of ruminal microbiota to dietary changes and production parameters[J]. Frontiers in Microbiology, 2017, 7: 2143. |
| [35] |
Indugu N, Vecchiarelli B, Baker LD, et al. Comparison of rumen bacterial communities in dairy herds of different production[J]. BMC microbiology, 2017, 17(1): 190. |
| [36] |
Cunha CS, Veloso CM, Marcondes, M I, et al. Assessing the impact of rumen microbial communities on methane emissions and production traits in Holstein cows in a tropical climate[J]. Systematic and Applied Microbiology, 2017, 40(8): 492-499. DOI:10.1016/j.syapm.2017.07.008 |
| [37] |
Pitta D, Indugu N, Vecchiarelli B, et al. Alterations in ruminal bacterial populations at induction and recovery from diet-induced milk fat depression in dairy cows[J]. Journal of Dairy Science, 2018, 101(1): 295-309. DOI:10.3168/jds.2016-12514 |
| [38] |
Khan RU, Naz S, Dhama K, et al. Direct-fed microbial:beneficial applications, modes of action and prospects as a safe tool for enhancing ruminant production and safeguarding health[J]. International Journal of Pharmacology, 2016, 12(3): 220-231. DOI:10.3923/ijp.2016.220.231 |
| [39] |
Weimer PJ. Redundancy, resilience, and host specificity of the ruminal microbiota:implications for engineering improved ruminal fermentations[J]. Frontiers in Microbiology, 2015, 10(6): 296. |
| [40] |
Bickhart D, Weimer P. Host-rumen microbe interactions may be leveraged to improve the productivity of dairy cows[J]. Journal of Dairy Science, 2017, 101(8): 7680-7689. |
| [41] |
Sun H, Wang D, Wang B, et al. Metabolomics of four biofluids from dairy cows:potential biomarkers for milk production and quality[J]. Journal of Proteome Research, 2015, 14(2): 1287-1298. DOI:10.1021/pr501305g |
| [42] |
Zhang J, Shi H, Wang Y, et al. Effect of dietary forage to concentrate ratios on dynamic profile changes and interactions of ruminal microbiota and metabolites in Holstein heifers[J]. Frontiers in Microbiology, 2017, 8: 2206. DOI:10.3389/fmicb.2017.02206 |
| [43] |
De Mulder T, Peiren N, Vandaele L, et al. Impact of breed on the rumen microbial community composition and methane emission of Holstein Friesian and Belgian Blue heifers[J]. Livestock Science, 2018, 207: 38-44. DOI:10.1016/j.livsci.2017.11.009 |
| [44] |
Knapp J, Laur G, Vadas P, et al. Invited review:Enteric methane in dairy cattle production:Quantifying the opportunities and impact of reducing emissions[J]. Journal of Dairy Science, 2014, 97(6): 3231-3261. DOI:10.3168/jds.2013-7234 |
| [45] |
Benchaar C, Hassanat F, Gervais R, et al. Effects of increasing amounts of corn dried distillers grains with solubles in dairy cow diets on methane production, ruminal fermentation, digestion, N balance, and milk production[J]. Journal of Dairy Science, 2013, 96(4): 2413-2427. DOI:10.3168/jds.2012-6037 |
| [46] |
Hassanat F, Gervais R, Julien C, et al. Replacing alfalfa silage with corn silage in dairy cow diets:Effects on enteric methane production, ruminal fermentation, digestion, N balance, and milk production[J]. Journal of Dairy Science, 2013, 96(7): 4553-4567. DOI:10.3168/jds.2012-6480 |
| [47] |
Stocker TF, Qin D, Plattner GK, et al. Climate change 2013: The physical science basis. contribution of working group I to the fifth assessment report of IPCC the intergovernmental panel on climate change[M]. Cambridge: Cambridge University Press, 2014.
|
| [48] |
Bulumulla A, Zhou M, Guan L. Achieving sustainable production of cow's milk[M]. 3rd ed. Alberta: Burleigh Dodds Science Publishing, 2017.
|
| [49] |
Ding X, Long R, Zhang Q, et al. Reducing methane emissions and the methanogen population in the rumen of Tibetan sheep by dietary supplementation with coconut oil[J]. Tropical Animal Health and Production, 2012, 44(7): 1541-1545. DOI:10.1007/s11250-012-0103-7 |
| [50] |
Hristov A, Callaway T, Lee C, et al. Rumen bacterial, archaeal, and fungal diversity of dairy cows in response to ingestion of lauric or myristic acid[J]. Journal of Animal Science, 2012, 90(12): 4449-4457. DOI:10.2527/jas.2011-4624 |
| [51] |
King EE, Smith RP, St-Pierre B, et al. Differences in the rumen methanogen populations of lactating Jersey and Holstein dairy cows under the same diet regimen[J]. Applied Environmental Microbiology, 2011, 77(16): 5682-5687. DOI:10.1128/AEM.05130-11 |
| [52] |
Jouany J, Demeyer D, Grain J. Effect of defaunating the rumen[J]. Animal Feed Science and Technology, 1988, 21(2-4): 229-265. DOI:10.1016/0377-8401(88)90105-8 |
| [53] |
Morgavi D, Jouany JP, Martin C. Changes in methane emission and rumen fermentation parameters induced by refaunation in sheep[J]. Australian Journal of Experimental Agriculture, 2008, 48(2): 69-72. DOI:10.1071/EA07236 |
| [54] |
Mosoni P, Martin C, Forano E, et al. Long-term defaunation increases the abundance of cellulolytic ruminococci and methanogens but does not affect the bacterial and methanogen diversity in the rumen of sheep[J]. Journal of Animal Science, 2011, 89(3): 783-791. DOI:10.2527/jas.2010-2947 |
| [55] |
Beauchemin KA, McGinn SM. Reducing methane in dairy and beef cattle operations:what is feasible[J]. Prairie Soil Crop, 2008, 1: 17-21. |
| [56] |
Danielsson R, Schnürer A, Arthurson V, et al. Methanogenic population and CH4 production in Swedish dairy cows fed different levels of forage[J]. Applied Environmental Microbiology, 2012, 78(17): 6172-6179. DOI:10.1128/AEM.00675-12 |
| [57] |
Shi W, Moon CD, Leahy SC, et al. Methane yield phenotypes linked to differential gene expression in the sheep rumen microbiome[J]. Genome Research, 2014, 24(9): 1517-1525. DOI:10.1101/gr.168245.113 |
| [58] |
Zhou M, Chung YH, Beauchemin K, et al. Relationship between rumen methanogens and methane production in dairy cows fed diets supplemented with a feed enzyme additive[J]. Journal of Applied Microbiology, 2011, 111(5): 1148-1158. DOI:10.1111/j.1365-2672.2011.05126.x |
| [59] |
Danielsson R, Dicksved J, Sun L, et al. Methane production in dairy cows correlates with rumen methanogenic and bacterial community structure[J]. Frontiers in Microbiology, 2017, 8: 226. |
| [60] |
Wallace RJ, Sasson G, Garnsworthy PC, et al. A heritable subset of the core rumen microbiome dictates dairy cow productivity and emissions[J]. Science Advances, 2019, 5: 78391. |
| [61] |
Chung YH, Zhou M, Holtshausen L, et al. A fibrolytic enzyme additive for lactating Holstein cow diets:Ruminal fermentation, rumen microbial populations, and enteric methane emissions[J]. Journal of Dairy Science, 2012, 95(3): 1419-1427. DOI:10.3168/jds.2011-4552 |
| [62] |
Duthie CA, Troy SM, Hyslop JJ, et al. The effect of dietary addition of nitrate or increase in lipid concentrations, alone or in combination, on performance and methane emissions of beef cattle[J]. Animal, 2018, 12(2): 280-287. |
| [63] |
Haisan J, Sun Y, Guan L, et al. The effects of feeding 3-nitrooxypropanol on methane emissions and productivity of Holstein cows in mid lactation[J]. Journal of Dairy Science, 2014, 97(5): 3110-3119. DOI:10.3168/jds.2013-7834 |
| [64] |
Reynolds CK, Humphries DJ, Kirton P, et al. Effects of 3-nitrooxypropanol on methane emission, digestion, and energy and nitrogen balance of lactating dairy cows[J]. Journal of Dairy Science, 2014, 97(6): 3777-3789. DOI:10.3168/jds.2013-7397 |
| [65] |
Duval S, Kindermann M. Use of nitrooxy organic molecules in feed for reducing enteric methane emissions in ruminants, and/or to improve ruminant performance[J]. International Patent Application, 2012, 84629:A1.
|
| [66] |
Zhou M, Hünerberg M, Guan L, et al. Air-dried brown seaweed, ascophyllum nodosum, alters the rumen microbiome in a manner that changes rumen fermentation profiles and lowers the prevalence of foodborne pathogens[J]. Applied and Environmental Science, 2018, 3: 17-18. |
| [67] |
Zhou M, Chen Y, Griebel PJ. Methanogen prevalence throughout the gastrointestinal tract of pre-weaned dairy calves[J]. Gut Microbes, 2014, 5(5): 628-638. DOI:10.4161/19490976.2014.969649 |
| [68] |
Yáñez-Ruiz DR, Abecia L, Newbold CJ. Manipulating rumen microbiome and fermentation through interventions during early life:a review[J]. Frontiers in Microbiology, 2015, 6: 1133. |
| [69] |
Li F, Guan LL. Metatranscriptomic profiling reveals linkages between the active rumen microbiome and feed efficiency in beef cattle[J]. Applied Environmental Microbiology, 2017, 83(9): e00061-00017. |
| [70] |
Jami E, Israel A, Kotser A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [71] |
Rey M, Enjalbert F, Combes S, et al. Establishment of ruminal bacterial community in dairy calves from birth to weaning is sequential[J]. Journal of Applied Microbiology, 2014, 116(2): 245-257. DOI:10.1111/jam.12405 |
| [72] |
Li R, Connor EE, Li C, et al. Characterization of the rumen microbiota of pre-ruminant calves using metagenomic tools[J]. Environmental Microbiology, 2012, 14(1): 129-139. DOI:10.1111/j.1462-2920.2011.02543.x |
| [73] |
Malmuthuge N, Griebel PJ. Taxonomic identification of commensal bacteria associated with the mucosa and digesta throughout the gastrointestinal tracts of preweaned calves[J]. Applied Environmental Microbiology, 2014, 80(6): 2021-2028. DOI:10.1128/AEM.03864-13 |
| [74] |
Langille MG, Zaneveld J, Caporaso JG, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [75] |
Aβhauer KP, Wemheuer B, Daniel R, et al. Tax4Fun:predicting functional profiles from metagenomic 16S rRNA data[J]. Bioinformatics, 2015, 31(17): 2882-2884. DOI:10.1093/bioinformatics/btv287 |
| [76] |
Douglas GM, Maffei VJ, Zaneveld J, et al. Langille PICRUSt2:An improved and extensible approach for metagenome inference[J]. BioRxiv, 2019, 67: 2295. |
| [77] |
Malmuthuge N, Guan L. Gut microbiome and omics:a new definition to ruminant production and health[J]. Animal Frontiers, 2016, 6(2): 8-12. DOI:10.2527/af.2016-0017 |
| [78] |
Dill-McFarland KA, Breaker JD, Suen G. Microbial succession in the gastrointestinal tract of dairy cows from 2 weeks to first lactation[J]. Scientific Reports, 2017, 7: 40864. DOI:10.1038/srep40864 |
| [79] |
Abecia L, Martín-García A, Martínez G, et al. Nutritional intervention in early life to manipulate rumen microbial colonization and methane output by kid goats postweaning[J]. Journal of Animal Science, 2013, 91(10): 4832-4840. DOI:10.2527/jas.2012-6142 |
| [80] |
Abecia L, Ramos-Morales E, Martínez-Fernandez G, et al. Feeding management in early life influences microbial colonisation and fermentation in the rumen of newborn goat kids[J]. Animal Production Science, 2014, 54(9): 1449-1454. DOI:10.1071/AN14337 |
| [81] |
Abecia L, Jiménez E, Martínez-Fernandez G, et al. Natural and artificial feeding management before weaning promote different rumen microbial colonization but not differences in gene expression levels at the rumen epithelium of newborn goats[J]. PLoS One, 2017, 12(8): e0182235. DOI:10.1371/journal.pone.0182235 |
| [82] |
Chakravorty S, Helb D, Burday M, et al. A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria[J]. Journal of Microbiological Methods, 2007, 69(2): 330-339. DOI:10.1016/j.mimet.2007.02.005 |
| [83] |
Frank JA, Reich CI, Sharma S, et al. Critical evaluation of two primers commonly used for amplification of bacterial 16S rRNA genes[J]. Applied Environmental Microbiology, 2008, 74(8): 2461-2470. DOI:10.1128/AEM.02272-07 |
| [84] |
Jovel J, Patterson J, Wang W, et al. Characterization of the gut microbiome using 16S or shotgun metagenomics[J]. Frontiers in Microbiology, 2016, 7: 459. |
| [85] |
Baldwin R, McLeod K, Klotz J, et al. Rumen development, intestinal growth and hepatic metabolism in the pre-and postweaning ruminant[J]. Journal of Dairy Science, 2004, 87: 55-65. DOI:10.3168/jds.S0022-0302(04)70061-2 |
| [86] |
Connor EE, Baldwin RL, Li CJ, et al. Gene expression in bovine rumen epithelium during weaning identifies molecular regulators of rumen development and growth[J]. Functional & Integrative Genomics, 2013, 13(1): 133-142. |
| [87] |
Connor E, Baldwin R, Walker M, et al. Transcriptional regulators transforming growth factor-β1 and estrogen-related receptor-αidentified as putative mediators of calf rumen epithelial tissue development and function during weaning[J]. Journal of Dairy Science, 2014, 97(7): 4193-4207. DOI:10.3168/jds.2013-7471 |
| [88] |
Liang G, Malmuthuge N, McFadden TB, et al. Potential regulatory role of microRNAs in the development of bovine gastrointestinal tract during early life[J]. PLoS One, 2014, 9(3): e92592. DOI:10.1371/journal.pone.0092592 |
| [89] |
Naeem A, Drackley JK, Lanier JS, et al. Ruminal epithelium transcriptome dynamics in response to plane of nutrition and age in young Holstein calves[J]. Functional & Integrative Genomics, 2014, 14(1): 261-273. |
| [90] |
Malmuthuge N, Liang G, Guan L. Regulation of rumen development in neonatal ruminants through microbial metagenomes and host transcriptomes[J]. Genome Biology, 2019, 20: 172. DOI:10.1186/s13059-019-1786-0 |
| [91] |
Sommer F, Bäckhed F. The gut microbiota-masters of host development and physiology[J]. Nature Reviews Microbiology, 2013, 11(4): 227-238. DOI:10.1038/nrmicro2974 |
| [92] |
Malmuthuge N. Role of commensal microbiota in neonatal calf gut development[D]. Edmonton: University of Alberta, 2016.
|
| [93] |
Seshadri R, Leahy SC, Attwood GT, et al. Cultivation and sequencing of rumen microbiome members from the Hungate1000 Collection[J]. Nature Biotechnology, 2018, 36(4): 359-367. DOI:10.1038/nbt.4110 |
| [94] |
Moraïs S, Mizrahi I. The road not taken:the rumen microbiome, functional groups, and community states[J]. Trends in Microbiology, 2019, 27(6): 538-549. DOI:10.1016/j.tim.2018.12.011 |





