解淀粉芽孢杆菌(Bacillus amyloliquefaciens)属于芽孢菌属,该菌株在自然界中的分布中极其广泛,例如土壤、秸秆、植物根际、甚至湖泊污水中均可分离得到。解淀粉芽孢杆菌形态、培养特征以及生理生化、16S rRNA基因等方面均与枯草芽孢杆菌(Bacillus subtilis)、贝莱斯芽孢杆菌(Bacillus velezensis)等极其相似,常规鉴定方法难以确定相关类群的分类地位。随着鉴定检测技术的发展,持家基因如gyrA、gyrB、rpoB等片段[1]分析以及全基因组测序等方法应用于解淀粉芽孢杆菌及其相近种类的比较鉴定[2-3]。
2008年Wang等[4]通过DNA-DNA的相关性分析得出贝莱斯芽孢杆菌(B. velezensis)是解淀粉芽孢杆菌的同物异名(B. amyloliquefaciens)。2016年Dunlap等[5]通过对“ B. oryzicola”的基因组进行测序后发现,贝莱斯芽孢杆菌并不是解淀粉芽孢杆菌的同物异名,经过基因组比较分析显示甲基营养芽孢杆菌(B. methylotrophicus)、解淀粉芽孢杆菌植物亚种和贝莱斯芽孢杆菌的基因组仅有微小差异,模式菌株间DNA-DNA杂交值均大于84%,远高于标准物种阈值70%。因此,将甲基营养芽孢杆菌与解淀粉芽孢杆菌植物亚种FZB42应重新归类于贝莱斯芽孢杆菌的类群中。
另外,解淀粉芽孢杆菌、贝莱斯芽孢杆菌相关类群具有广泛的生物学功能,该类群具有丰富的次生代谢产物,其分泌的抗生素以及抗菌蛋白等物质有抑制细菌和真菌的能力[6],可以起到较好的生物防治效果;还能作为一种天然的防腐剂,并将其应用于各种食品行业当中,可以抑制腐败真菌和致病菌的生长[7]。此外,该类群还发现能够消除残留的化学农药[8]、作为动物的益生菌等功能[9]。据“中国农业微生物菌种保藏管理中心(ACCC)”数据库显示,该菌株ACCC 19742对茄子灰霉病、黄瓜枯萎病的病原菌有一定的抑制作用,其发酵产物还可以促进作物根系的生长,可用于制备微生物肥料和生物有机肥等。
中国农业微生物菌种中心库藏菌株ACCC 19742是2014年从山东蔬菜大棚的土壤中分离并保藏,在ACCC数据库中分类地位为解淀粉芽孢杆菌(B. amyloliquefaciens)。在菌种中心定期转接保藏过程中,发现该菌株在同一培养基上出现两种不同的菌落形态,两株菌的16S rRNA基因相似性为100%,因此推测为同一株菌的不同形态变异型。经16S rRNA基因和基因组比较,并结合API 20NE、BIOLOG及脂肪酸组成等表型特征分析,确定该菌株的分类地位,并确定不同形态菌落的关系。
1 材料与方法 1.1 材料 1.1.1 供试菌株及培养基供试菌株:解淀粉芽孢杆菌ACCC 19742-1和ACCC 19742-2来源于中国农业微生物菌种保藏管理中心。培养基:BUG合成培养基(Biolog,Inc.);TSA合成培养基(DifcoTM)[10];牛肉膏蛋白胨培养基(北京奥博星生物技术有限责任公司);R2A琼脂培养基(DifcoTM),配方见参照文献[10]。
1.1.2 主要试剂及仪器API20 NE试验条及相关试剂耗材购自生物梅里埃公司;DNA提取试剂盒购自北京天根生化科技有限公司;革兰氏染料试剂盒购自北京索莱宝科技有限公司等。
1.2 方法 1.2.1 革兰氏染色法分别在R2A、TSA、BUG、牛肉膏蛋白胨培养基上接种19742-1和19742-2菌株,30℃培养过夜后观察菌落形态;菌体使用革兰氏染色试剂盒法进行染色,参照文献[11]。
1.2.2 脂肪酸分析法具体方法参照Ichihara K等[12]文献。
1.2.3 BIOLOG GEN- Ⅲ微孔板检测将19742-1和19742-2菌株在BUG培养基上划线接种,30℃过夜培养;按照要求将菌液浊度调到90%后混匀,制成接种液;使用排枪将菌悬液按每孔100 μL的量加入到Gen-III微孔板的96孔内;适温培养4-6 h,12-24 h后通过BIOLOG鉴定系统(Microlog-M,MicroStation,Omnilog®)中的软件读取Gen-III微孔板上所表现出的表型图谱来对细菌进行鉴定。
1.2.4 API 20NE试剂条法首先在牛肉膏蛋白胨培养基上接种19742-1和19742-2菌株,30℃培养过夜,使用无菌棉签挑取单菌落,逐量加入灭菌的0.85%生理盐水,使用比浊管调节菌悬液的浊度为0.5麦氏单位。按说明书接种,30℃培养24 h,参考说明表判读最终结果。
1.2.5 16S rRNA及持家基因gyrB扩增与分析(1)DNA提取:按照北京天根细菌基因组DNA试剂盒中的说明书提取;通用引物:27F(5'-AGAGTTTGATCCTGGCTCAG-3'),1492R(5'-GGTTACCTTGTTACGACTT-3');PCR反应体系和反应条件参照文献中[10]方法。(2)持家基因gyrB提取:运用试剂盒法提取总基因组DNA获得模板;上游引物(5'-GA AGTCATCATGATGACCGTTCTGCA-3')、下游引物(5'-AGCAGGGTACGGATG TGCGAGCC-3');PCR扩增体系和反应条件见参考文献[13]。将扩增完成的目的片段送往上海生工公司进行测序,序列结果在Ezbiocloud网站上(www.ezbiocloud.net/eztaxon)进行比对,整理后的序列最后用Mega 7.0软件进行系统发育分析。
1.2.6 19742-1和19742-2菌株基因组拼接与注释(1)基因组拼接:基因组DNA提取按照北京天根细菌基因组DNA试剂盒说明书进行,纯度和浓度符合测序标准后,送至上海派森诺生物科技股份有限公司进行基因组框架图测定。采用FastQC(http//www.bioinformatics.babraham.ac.uk/projects/fastqc)首先对数据进行质量控制;使用AdapterRemoval(ver. 2.1.7)[14]将低质量的reads和污染的接头去除;采用A5-miseq v2015052[14]软件对去除接头序列的测序数据进行从头拼装,构建contigs和scaffolds。(2)基因组预测与注释:通过KEGG[15](https://www.kegg.jp/)、GO注释[16](http://www.geneontology.org/)以及使用在线软件tRNAscan-SE 1.21[17]和RNAmmer 1.2[18]对tRNA及rRNA操纵子等进行预测和注释
1.2.7 ANI和DDH值分析使用JSpeciesWS软件[19]计算ANI值,网站(http://ggdc.dsmz.de/ggdc.php)得到菌株的DDH值,并上传菌株相应的fasta或fna文件后进行分析。
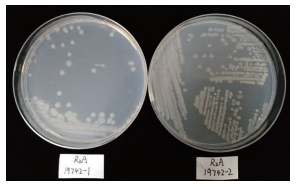
2 结果 2.1 菌落形态比较分析分别在R2A、BUG、TSA、牛肉膏蛋白胨培养基上连续进行3次划线培养后,发现19742-1和19742-2的菌落形态存在一定差别。如表 1汇总所示。
|
| 图 1 19742-1(左)和19742-2(右)菌落形态 |
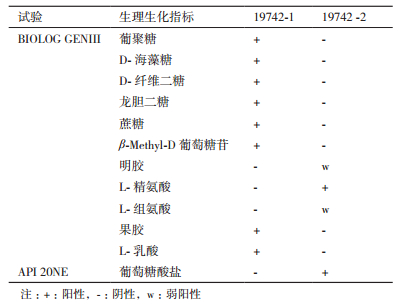
BIOLOG碳源利用:通过实验得知19742-1能利用25种碳源,19742-2能利用20种碳源,其中有17种碳源被两种菌共同利用。对于α-D-葡萄糖、D-甘露糖、D-果糖、1%乳酸钠、D-山梨醇、D-甘露醇、L-谷氨酸、L-天冬氨酸、L-丙氨酸等反应两者均呈阳性;对于D-松二糖、水苏糖、α-D-乳糖、N-Acetyl-β-D甘露糖胺、D-半乳糖、D-海藻糖、肌苷、D-阿拉伯醇、D-阿拉伯醇等反应两者均呈阴性。不同的是,葡聚糖、D-海藻糖、D-纤维二糖、龙胆二糖、蔗糖、β-Methyl-D葡萄糖苷、果胶和L-乳酸这8个反应中,19742-1菌株呈阳性,而19742-2则呈阴性;明胶、L-精氨酸和L-组氨酸这3个反应中,19742-2菌株呈阳性或弱阳性,而19742-1呈阴性。以上结果说明19742-1与19742-2利用碳源的能力相似,但存在一定的差别。
API 20NE试剂条:结果分析发现吲哚产生、精氨酸双水介酶、脲酶、半乳糖甙酶、癸酸、乙二酸、苯乙酸同化反应19742-1和19742-2均为阴性,其余均为阳性。仅葡萄糖酸盐同化反应19742-1显示为阴性,而19742-2显示为阳性反应。具体如表 2所示。
脂肪酸测定结果如表 3所示,菌株19742-1与19742-2均含有Bacillus属中主要的脂肪酸成分,其主要脂肪酸均为anteiso-C15:0,iso-C15:0,anteisoC 17:0和iso-C17:0,由此可以看出这两株菌的脂肪酸主要组成相同,只是含量方面略微有差异。
19742-1和19742-2的16S rRNA基因片段长度分别为1 547 bp和1 547 bp,Genbank登录号分别为MK779999和MK780002,将两者测序得到的基因序列在NCBI网站上进行比对,表明两株菌间的16S rRNA基因序列相似性为100%,与B. velezensis CR-502T相似度最高,分别为99.86%和99.79%。同时,19742-1和19742-2菌株的gyrB基因片段长度分别是1 156 bp和1 163 bp,gyrB基因序列相似性为99.4%,登录号分别为MK792247和MK792248。构建基于16S rRNA和gyrB基因序列的系统发育树(图 2)。结果可以看出,19742-1和19742-2均与B. velezensis CR-502T菌株亲缘关系最近,因此进一步说明二者可能是同一株菌的不同形态型。

|
| 图 2 邻接法构建19742-1和19742-2菌株16S rRNA和gyrB序列系统发育树 |
19742-1的基因组全长为3 883 546 bp,GC含量为46.41%,含有3 897个基因,可编码3 897个蛋白,Genbank登录号为SSNF00000000;19742-2基因组全长为3 882 439 bp,GC含量46.59%,含有3 896个基因,可编码3 896个蛋白,Genbank登录号为SSNG00000000,二者基因组大小并无很大差别。另外,19742-1基因组中有2161个蛋白序列能与KEGG数据库匹配并得到功能分类;有2 741个基因得到GO分类注释;基因组中预测到71个tRNA,5个rRNA。而19742-2的基因组中有2 162个蛋白序列能与KEGG数据库匹配并得到功能分类;有2 742个基因得到GO分类注释;基因组中预测到66个tRNA,4个rRNA。此外,从基因组分析来看,19742-1菌株特有的基因是12个,而19742-2菌株特有的基因是10个。综上可以看出,19742-1和19742-2菌株在基因组预测方面并无很大差别。
2.6 ANI和DDH值分析19742-1和19742-2及其与相关模式菌种的ANI和DDH值见表 4所示。根据计算结果可知,19742-1和19742-2菌株的ANI值为99.95%,DDH值为99.62%;两株菌与B. amyloliquefaciens DSM 7T的ANI值分别为93.53%和93.54%,DDH值分别为66.42%和66.43%;与B. velezensis NRRL_B 41580T的ANI值分别为97.01%和97.03%,DDH值分别为77.22%和77.28%。一般来讲,同种之间ANI值≥ 95%,DDH值≥ 70%[20]。19742-1和19742-2菌株与B. velezensis NRRL_B 41580T的ANI及DDH值均大于种分类水平的临界值,而与B. amyloliquefaciens DSM 7T的ANI及DDH值均小于种分类水平的临界值,因此ACCC 19742属于贝莱斯芽孢杆菌而并非解淀粉芽孢杆菌。
在细菌分类学上,当同种或同亚种内不同菌株之间的性状差异不足以分为新的亚种时,可以细分为不同的“型”,但他们不是正式的分类等级[21]。从表型来看,两株菌具有不同的形态,19742-2菌落粘性相对较强,推测其可能含有更多的糖类物质,而19742-1菌株表面相对干燥,出现这样的现象可能是由于某个特异性基因突变或重组而引起在形态特征和生理类型等方面的差异[22]。同时,已报道的贝莱斯芽孢杆菌菌落形态也存在着明显差异。如王伟等[23]筛选到一株可拮抗大丽轮枝菌的B. velezensis 12-51,其菌落形态呈白色,边缘整齐且光滑,中间略微凸起,菌体生长物可向四周呈云雾状扩散。而连彩等[24]分离的抗兰花枯萎病贝莱斯芽孢杆菌单菌落形态呈圆形,表面褶皱,菌落中央凹陷呈火山口状。可见,不同的B. velezensis菌株形态不尽相同。
根据API 20NE、BIOLOG以及脂肪酸分析等生理生化分析结果,19742-1和19742-2十分相似,结合16S rRNA基因、gyrB以及基因组分析,可以确定二者是同一株菌不同的形态型;从目前的结果也可以看出,这两株菌在功能方面并没有很大的分化,只是形态结构有所差异。进一步确定了该菌株属于贝莱斯芽孢杆菌而并非解淀粉芽孢杆菌。基因组分析来看,19742-1与19742-2分别具有12个和10个特有基因,可能是由于进化过程中的基因突变和自然选择适应[25]造成的,具体原因还有待我们后续深入进行探究。
菌落形态特征是判断菌种是否污染的最直接、最快速且一直以来认为是无争议的一种方法,本研究的结果旨在阐明在微生物检测、生产及菌种资源的保藏过程中,应结合不同的方法判断菌种的纯度。解淀粉芽孢杆菌及贝莱斯芽孢杆菌均为微生物饲料和微生物肥料重要的生产菌株,本研究修正了ACCC 19742的分类地位,从不同角度证明了同一株菌存在不同的菌落和菌体形态。本研究为微生物肥料以及饲料等相关微生物产品的检测,尤其对于菌种保藏中心保藏及共享工作提供了重要参考价值。
4 结论本文通过形态学、生理生化、基因组等多方面综合分析,证实19742-1和19742-2为来源于同一菌株的不同形态,而并非污染造成;同时根据系统发育进化树、ANI和DDH值分析得知,该菌株ACCC 19742的分类地位属于贝莱斯芽孢杆菌而并非解淀粉芽孢杆菌,研究结果对菌种保藏工作具有一定指导意义。
| [1] |
喻国辉, 牛春艳, 陈远凤, 等. 利用16S rDNA结合gyrA和gyrB基因对生防芽孢杆菌R31的快速鉴定[J]. 中国生物防治学报, 2010, 26(2): 160-166. |
| [2] |
Gamez RM, Rodríguez F, Ramírez S, et al. Genome sequence of the banana plant growth-promoting rhizobacterium Bacillus amyloliquefaciens BS006[J]. Genome Announcements, 2015, 3(6): e01391-15. |
| [3] |
Qin YX, Han YZ, Shang QM, et al. Complete genome sequence of Bacillus amyloliquefaciens L-H15, a plant growth promoting rhizobacteria isolated from cucumber seedling substrate[J]. Journal of Biotechnology, 2015, 200: 59-60. DOI:10.1016/j.jbiotec.2015.02.020 |
| [4] |
Wang LT, Lee FL, Tai CJ, et al. Bacillus velezensis is a later heterotypic synonym of Bacillus amyloliquefaciens[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(3): 671-675. DOI:10.1099/ijs.0.65191-0 |
| [5] |
Dunlap CA, Kim SJ, Kwon SW, et al. Bacillus velezensis is not a later heterotypic synonym of Bacillus amyloliquefaciens; Bacillus methylotrophicus, Bacillus amyloliquefaciens subsp. plantarum and " Bacillus oryzicola" are later heterotypic synonyms of Bacillus velezensis based onphylogenomics.[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(3): 1212-1217. DOI:10.1099/ijsem.0.000858 |
| [6] |
张娟, 杨彩梅, 曹广添, 等. 解淀粉芽孢杆菌及其作为益生菌的应用[J]. 动物营养学报, 2014, 26(4): 863-867. DOI:10.3969/j.issn.1006-267x.2014.04.005 |
| [7] |
梁才. 现代食品中防腐剂的应用与展望[J]. 食品安全导刊, 2016(6): 76-77. |
| [8] |
Chen XH, Scholz R, Borriss M, et al. Difficidin and bacilysin produced by plant-associated Bacillus amyloliquefaciens are efficient in controlling fire blight disease[J]. Journal of Biotechnology, 2009, 140(1/2): 38-44. |
| [9] |
李卫芬, 邓斌, 陈南南, 等. 芽孢杆菌对草鱼生长和肠粘膜抗氧化功能及养殖水质的影响[J]. 水生态学杂志, 2012, 33(1): 65-70. |
| [10] |
曹艳花, 徐凤花, 陈小忠, 等. 水稻种子相关细菌的系统发育分析与促生能力评价[J]. 中国土壤与肥料, 2011(5): 83-87. |
| [11] |
吴红萍, 王陈仪, 宋晶霞, 等. 微生物学实验教学—细菌的革兰氏染色经典法和三步法的比较与分析[J]. 高校实验室工作研究, 2017(3): 56-59. |
| [12] |
Ichihara K, Kohsaka C, Tomari N, et al. Fatty acid analysis of triacylglycerols :Preparation of fatty acid methyl esters for gas chromatography[J]. Analytical Biochemistry, 2016, 495: 6-8. DOI:10.1016/j.ab.2015.11.009 |
| [13] |
高健, 康健, 邱冠周. 嗜铁钩端螺旋菌gyrB基因的PCR扩增、克隆与序列分析[J]. 激光生物学报, 2011, 20(3): 367-372. |
| [14] |
Schubert M, Lindgreen S, Orlando L. Adapter Removal v2 :rapid adapter trimming, identification, and read merging[J]. BMC Research Notes, 2016, 9(1): 88. DOI:10.1186/s13104-016-1900-2 |
| [15] |
李向真, 刘子朋, 李娟, 等. KEGG数据库的进展及其在生物信息学中的应用[J]. 药物生物技术, 2012, 19(6): 535-539. |
| [16] |
刘粉香, 杨文国, 孙勤红. 基于转录组测序数据分析及高通量GO注释理论的研究[J]. 安徽农业科学, 2018, 46(31): 88-91, 100. |
| [17] |
Lewis RE, Zanarini MC, Williams AA. Reported pathological childhood experiences associated with the development of borderline personality disorder[J]. The American Journal of Psychiatry, 1997, 154: 1101-1106. DOI:10.1176/ajp.154.8.1101 |
| [18] |
Lagesen K, Hallin P, Rodland EA, et al. RNAmmer :consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Res, 2007, 35: 3100-3108. DOI:10.1093/nar/gkm160 |
| [19] |
刘波.基于全基因组的芽孢杆菌平均核苷酸同源性(ANI)分析[C].中国微生物学会微生物资源专业委员会、国家微生物资源平台.第五届全国微生物资源学术暨国家微生物资源平台运行服务研讨会论文摘要集, 2013.
|
| [20] |
Kim M, Oh HS, Park SC, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(2): 346-351. |
| [21] |
杨苏声. 细菌分类学[M]. 北京: 中国农业大学出版社, 1997.
|
| [22] |
杜思, 曾小波, 李友国. 大豆快生根瘤菌SfHH103_03182基因突变体的构建和共生固氮表型分析[J]. 华中农业大学学报, 2019, 38(1): 35-42. |
| [23] |
王伟, 李术娜, 李红亚, 等. 大丽轮枝菌拮抗细菌菌株12-51的筛选鉴定与抗菌物性质分[J]. 中国农学通报, 2009, 25(19): 14-19. |
| [24] |
连彩, 郭晓军, 朱宝成, 等. 兰花枯萎病拮抗细菌的筛选与鉴定[J]. 华北农学报, 2017, 27(2): 222-225. |
| [25] |
钟振晖.稻瘟病菌不同宿主来源菌株的比较基因组学研究[D].福州: 福建农林大学, 2014.
|