2. 中国农业科学院饲料研究所,北京 10081
2. Animal Food Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081
对畜禽及鱼类等单胃动物而言,磷是其生长的必要元素。在谷物类及粕类饲料中,总磷的60%- 70%是以植酸的形式存在的[1]。植酸又名肌醇六磷酸,在单胃动物(如人、猪和鸡和鱼类等)体内,缺乏有效降解植酸的消化酶类,因此对植酸磷的利用率极低,不被消化的植酸磷排出体外导致土地和水资源污染;另一方面,植酸往往与蛋白类、糖类及有益矿物质如铁、锌、钙等鳌合,也是一种抗营养因子,不利于营养物质及矿物质的消化和吸收[2]。因此,为了避免磷缺乏,饲料中往往还要额外添加无机磷(如磷酸氢钙),这导致饲料成本提高,也造成磷矿这种不可再生资源的浪费[3]。
植酸酶(Myo-inositol hexakisphosphate phosphohydrolase)是指能够水解植酸及其盐类物质中磷酸单酯键而生成低级磷酸肌醇和无机磷的一类酶的总称[4]。饲料用植酸酶的商业化是在1991年荷兰关于减少磷对环境污染立法的背景下启动的[5],在随后的几十年中,研究者们对植酸酶进行了深入广泛的研究,一些综述文章总结了植酸酶的发展过程[6-12]。植酸酶可以在单胃动物体内水解植酸释放无机磷,作为饲料添加剂,能够明显提高植酸磷的利用率,降低动物饲料中无机磷的补充量,同时降低排泄物中的磷含量,减少环境污染,而且植酸酶降解植酸并释放出鳌合的有益矿物质,可有效提高饲料中可吸收铁、锌、钙的含量[13]。目前,植酸酶已经成为在单胃动物(如猪、鸡、鱼)饲料中应用最广的酶制剂之一,其在单胃动物日粮中的添加效果也已经得到了广泛的认同[14],实现了生态效益和经济效益的“双赢”。
植酸酶的作用场所主要是单胃动物的胃及肠道,对于鱼类而言,其消化道组成具有很大的多样性,有的有胃,有的无胃,并且鱼类的消化道相对较短,消化道的pH一般为6.5-7.5,接近中性,与猪和鸡酸性的消化道有很大区别,因此,水产饲料里的植酸酶制剂要求在中性pH下具有较高的酶活性;另外由于鱼类养殖的水体温度一般在0-30℃之间,因此,投放到养鱼池的植酸酶需在低温环境中具有较高的活性。而到目前为止,在饲料上应用的植酸酶最适pH值主要是酸性的,只能在酸性条件下发挥水解植酸的能力,在pH高于6.0时,酶活性损失严重[15]。但在中性pH值及低温条件下具有高酶活性的植酸酶报道还很少,因此,开发中性低温植酸酶在水产养殖中具有较大的应用潜力和商业价值。
本研究通过兼并引物和Tail-PCR技术克隆了来自青霉的中性植酸酶基因[16],并成功将其在毕赤酵母中进行异源表达,并对其酶学性质和动力学参数进行了分析,为植酸酶在水产养殖中的应用提供了新的候选基因。
1 材料与方法 1.1 材料 1.1.1 质粒与菌株从自云南锡矿的酸性废水中分离的青霉C1菌株(Penicillium sp. C1)[17],保存于中国普通微生物菌种保藏管理中心,编号为:CGMCC No.4432。pEASY-blunt克隆载体及克隆宿主菌大肠杆菌XL-10购自北京全式金生物技术有限公司;表达宿主菌株毕赤酵母(Pichia pastoris)GS115由本实验室保存,表达载体pPIC9 γ由本实验室构建并保存。
1.1.2 试剂植酸钠购自Sigma公司;Genome Walking Kit(TaKaRa code:D316)试剂盒购自TaKaRa公司;实验所用的限制性内切酶SnaB Ⅰ和NotⅠ为TaKaRa公司产品;DNA回收试剂盒购自北京天根生物科技有限公司;Fly DNA聚合酶及pEASY系列克隆载体购自北京全式金生物技术有限公司;脱糖基化酶Endo-[β] -Nacetylglucosaminidase H(Endo H)以及T4 DNA连接酶为New England Biolabs公司产品;真菌DNA提取试剂盒购自OMEGA公司(Fungal DNA Mini Kit,D3390-01);蛋白质分子量标准为上海生化研究所产品;蛋白纯化层析介质为Amersham Pharmacia产品;其它所用试剂为国产分析纯试剂。
1.1.3 培养基LB培养基:0.5%酵母提取物,1%NaCl,1%蛋白胨;MD固体培养基:葡萄糖20 g/L,琼脂粉20 g/L,生物素4×10-4 g/L,YNB 13.4 g/L;BMGY培养基:酵母提取物10 g/L,蛋白胨20 g/L,甘油10 mL,生物素4×10-4 g/L,YNB 13.4 g/L;BMMY培养基:酵母提取物10 g/L,蛋白胨20 g/L,生物素4×10-4 g/L,YNB 13.4 g/L,甲醇0.5%。
1.2 方法 1.2.1 PhyC1成熟蛋白编码区的克隆根据NCBI数据库中的真菌HAP植酸酶保守序列RHGVRXPT和HDTN,设计简并引物HAP-FF和HAP-FR(表 1),用于植酸酶基因片段的扩增。
提取青霉C1的基因组DNA(详见Fungal DNA Mini Kit说明书),并以此为模板,以HAP-FF和HAP-FR为上下游引物,Fly DNA聚合酶扩增植酸酶保守区DNA序列,用胶回收试剂盒回收该PCR产物,克隆到pEASY-blunt载体上,进行DNA测序,根据测序结果,设计扩增植酸酶保守基因的侧翼序列的特异性引物,5'侧翼序列的特异性引物为usp1/2/3;3'侧翼序列的特异性引物为dsp1/2/3。特异性引物设计及PCR扩增程序详见Genome Walking Kit试剂盒说明书。扩增得到的PCR产物经过1%的琼脂糖凝胶电泳分离,特异性扩增的DNA条带经过琼脂糖凝胶回收试剂盒回收,送至华大基因进行测序,直至获得编码PhyC1成熟蛋白的全长基因序列。
1.2.2 真核表达载体pPIC9 γ-phyC1的构建以青霉C1的基因组DNA为模板,以phyC1-mF和phyC1- mR为上下游引物,用Fly DNA聚合酶扩增植酸酶phyC1基因全长序列,PCR产物带有限制性内切酶SnaB Ⅰ和Not Ⅰ的酶切位点(表 1黑色斜体),用胶回收试剂盒回收该PCR产物,将其克隆到pEASYblunt-simple载体上,经过DNA测序,获得正确的带有phyC1基因的重组质粒,重组质粒经SnaB Ⅰ和Not Ⅰ双酶切,与经过同样双酶切的pPIC9γ表达载体连接,获得重组表达载体pPIC9 γ-phyC1。
1.2.3 毕赤酵母的转化和阳性转化子筛选毕赤酵母GS115菌株划线获得单克隆,并在YPD液体培养基中过夜生长至OD600=1.6左右,然后根据Invitrogen手册的方法制备毕赤酵母感受态细胞。重组表达载体pPIC9γ-phyC1用Bgl Ⅱ酶切线性化后,取2 μg线性化质粒及80 μL感受态细胞加入电击杯(0.2 cm Bio Rad)中,电击(2.1 kV、25 μF、200Ω)转化,然后涂布在MD平板上,于30℃恒温箱中培养至长出转化子,挑选生长旺盛的转化子在小管中用3 mL BMGY培养基培养2 d后离心弃去培养基,再加入1.5 mL BMMY液体培养基诱导2 d,测定各管的植酸酶活性,挑选活性高的单克隆作为种子,诱导PhyC1的大量表达。
1.2.4 PhyC1蛋白的诱导表达将产植酸酶的菌株接种到装有300 mL BMGY液体培养基的1 L三角瓶中,在30℃、220 r/min摇床中培养48 h用以增加菌体量,然后将培养液在7 000 r/min离心10 min,弃上清,取沉淀(尽量除尽上清),再用100 mL含有0.5%甲醇的BMMY液体培养基重悬,继续在30℃诱导培养,每隔12 h补加甲醇,使培养基中的甲醇浓度保持在0.5%的终浓度,并且每隔12 h取样进行酶活测定。
1.2.5 重组植酸酶的纯化将获取的菌液在4℃、8 000 r/min下离心10 min,收集上清液,对其进行50%-70%硫酸铵沉淀,然后13 000 r/min、4℃离心15 min,收集沉淀,将沉淀的蛋白溶于20 mmol/L pH 8.0的Tris-HCl缓冲液中,并将其在相同的缓冲液中进行透析,透析后的酶液用PEG 6000进行浓缩。浓缩后的酶液通过阴离子交换层析(HiTrap Q Sepharose XL 5 mL)和分子筛凝胶层析(Sephacryl S-200 HR FPLC column)进行纯化,得到电泳纯的蛋白样品。
1.2.6 脱糖基化及SDS-PAGE分析毕赤酵母表达的异源蛋白往往会被糖基化修饰,造成异源表达的蛋白分子量与理论分子量不符的情况,因此,我们对纯化的PhyC1进行脱糖基化处理及蛋白电泳验证,分析其糖基化情况和实际分子量,脱糖基化反应体系如下:向18 μL纯化的酶液中加入2 μL 10×Glycoprotein变性缓冲液,100℃煮沸10 min,使酶蛋白充分变性;再加入2.4 μL 10×G 5缓冲液、2 μL Endo H,37℃水浴处理1 h。而后通过SDSPAGE电泳分析重组酶的实际和理论分子量。SDSPAGE浓缩胶和分离胶分别是5%和12%的丙烯酰胺凝胶溶液。
1.2.7 植酸酶活性测定将纯化的酶液用NaAc-HAc缓冲液进行稀释一定倍数(0.25 mol/L;并含有0.05% BSA和0.05% Triton X-100;pH 4.5),将50 μL稀释后的酶液加入到950 μL 1.5 mmol/L植酸钠底物(用上述NaAc-HAc缓冲液配制)中,在37℃水浴锅中反应15 min,加入1 mL 10%(W/V)TCA终止反应,最后加入2 mL显色液(1%(W/V)四水合钼酸铵,3.2%(W/V)浓硫酸,7.32%(W/V)硫酸亚铁)进行显色。对照管是先加入TCA混匀后再加酶液,其它步骤相同。显色后,在700 nm光吸收下测定OD值,并计算酶活。
1.2.8 重组植酸酶的酶学性质分析 1.2.8.1 最适pH和pH稳定性最适pH测定:将纯化后的酶液在不同的pH(2.0-12.0)条件下进行酶促反应,所使用的缓冲液如下:0.1 mol/L甘氨酸-盐酸缓冲液,pH 2.0-4.0;0.1 mol/L醋酸钠-醋酸缓冲液,pH 4.0-7.0;0.1 mol/L Tris-盐酸缓冲液,pH 7.0-9.0;0.1 mol/L甘氨酸-氢氧化钠缓冲液,pH 9.0-12.0。酶促反应在37℃下进行。
pH稳定性测定:将纯化后的酶液用不同pH缓冲液稀释,使其在不同pH、37℃处理1 h,然后用最适pH缓冲液稀释,使其在最适pH、37℃下进行酶促反应测定酶活性研究酶的pH稳定性。每个pH下的测定重复3次,取平均值。
1.2.8.2 最适反应温度和热稳定性在最适pH条件下,使酶促反应在不同温度(0℃-70℃)条件下进行,通过测定酶活性来确定最适温度。热稳定性测定是在40℃、45℃、50℃条件下将酶液分别处理2-60 min后,在37℃、最适pH条件下进行酶活性测定。每个温度下的测定重复3次,取平均值。
1.2.8.3 比活性测定酶活性单位定义:在一定条件下,每分钟释放出1 μmol无机磷所需的酶量为一个酶活性单位(U)。比活力单位定义是:每毫克酶蛋白所含有的酶活力单位。用结晶牛血清白蛋白(BSA)配置标准蛋白质溶液,并绘制蛋白含量标准曲线。通过考马斯亮蓝G-250试剂测定样品中酶蛋白的含量,同时在37℃下测得重组植酸酶的酶活,由此得到酶的比活。
1.2.8.4 不同化学试剂及相关金属离子对重组植酸酶PhyC1酶活性影响测定在酶促反应体系中加入相应的金属离子及相关化学试剂,研究其对酶活性的影响,各种物质的终浓度为1 mmol/L,在最适pH、37℃条件下测定。以没有加金属离子和化学试剂的反应为对照。
1.2.8.5 PhyC1动力学常数Km值及Vmax的测定在PhyC1的最适反应条件下,以0.0625 mmol/L的植酸钠为底物,依次在酶促反应进行1、3、5、7、10、15 min时加入1 mL 10% TCA试剂终止反应,测定酶活性,计算出酶活性与时间的比值,在一定时间内比值呈一常数,则在此时间内的酶促反应为一级反应,此时间即可做为测定Km和Vmax的反应时间。在一级反应时间内,用不同浓度的植酸钠(0.0625、0.1、0.125、0.2、0.25、0.5、1.0和1.5 mmol/L)为底物,在最适条件下测定酶活,计算相应的酶促反应速度,按照双倒数作图法(Lineweaver-Burk法)测定Km值及Vmax。
1.2.8.6 蛋白酶耐受性的测定用0.2 mol/L、pH 7.0的Tris-Hcl缓冲液配制0.1 mg/mL的胰蛋白酶。按照蛋白酶与纯化植酸酶质量比为1:1的比例在植酸酶中加入胰蛋白酶,在37℃分别保温0、5、10、20、30、60 min后取样放置于冰上,然后对蛋白酶处理后的样品用最适pH缓冲液稀释,在37℃、最适pH条件下进行酶活性测定,研究蛋白酶对植酸酶活性的影响。
2 结果 2.1 植酸酶基因phyC1的克隆通过简并引物的扩增及染色体步移方法,最终获得了来源于青霉的植酸酶基因phyC1的ORF序列,如图 1所示:phyC1的结构基因全长1 477 bp,有意思的是58 bp的内含子序列(黑体加下划线),正处于phyC1信号肽的内部。PhyC1成熟蛋白编码449个氨基酸,理论分子量为50 kD,预测等电点4.73,含有6个潜在的N糖基化位点。将phyC1的结构基因在NCBI数据库中进行序列比对,PhyC1属于典型的组氨酸植酸酶家族HAP(Histidine Acid Phosphatase),在N端和C端分别含有该家族保守的RHG和HD基序,其中两个组氨酸和天冬氨酸是植酸酶催化残基。PhyC1与来源于Talaromycesverruculosus(KUL82701.1)的植酸酶的氨基酸序列最为相似,相似性约为92%;与来源于Talaromycescellulolyticus(GAM38406.1)的植酸酶的氨基酸序列相似性约为90%;与来源于Penicillium sp.‘ occitanis’(PCG95438.1)的植酸酶的氨基酸序列相似性约为87%。

|
| 图 1 植酸酶phyC1的核酸序列 |
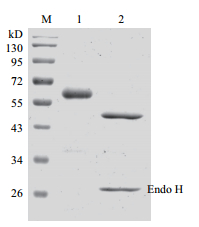
phyC1基因通过SnaB Ⅰ和Not Ⅰ双酶切位点与pPIC9γ表达载体连接,获得重组表达载体pPIC9 γ-phyC1,线性化的重组载体经电激转化,导入酵母GS115菌株,经过平板筛选和小管筛选,选取植酸酶活性最高的转化子,在装有200 mL培养基的1 L摇瓶中进行诱导表达,96 h左右发酵液酶活达到最高,离心收集发酵液,对其进行50%-70%硫酸铵沉淀、透析、浓缩,浓缩的酶液利用阴离子交换层析和分子筛凝胶层析进行纯化,以纯化的样品进行酶学性质的分析。将纯化的样品10 μL进行SDSPAGE蛋白电泳分析(图 2),结果表明,重组酶纯化条带单一,分子量约为60 kD左右,经Endo H脱糖基化处理后,重组酶的分子量下降至50 kD左右,说明在GS115菌株中PhyC1蛋白被糖基化。
|
| M:预染蛋白分子量标准;1:纯化的PhyC1;2:N-端去糖基化的PhyC1和Endo H 图 2 重组酶PhyC1的SDS-PAGE分析 |
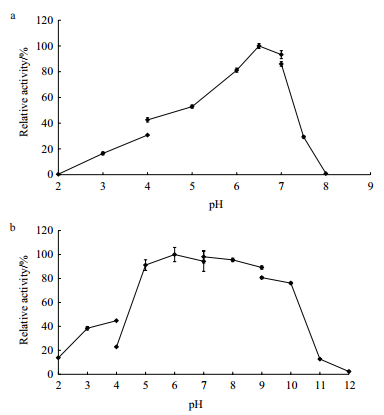
将纯化的酶液在37℃下置于pH 2.0-8.0的缓冲液中测定酶活力,结果显示重组植酸酶PhyC1的最适pH为6.5;在pH值为7.0时酶活性也很高,剩余酶活为90%,说明该酶是一个中性的植酸酶(图 3-a)。
|
| a:37℃条件下,pH值对PhyC1活性的影响;b:37℃,在2-12的pH值缓冲液中处理1 h后,PhyC1的pH稳定性,剩余酶活在最适pH值、37℃条件下检测。每个实验数据有3个重复 图 3 PhyC1的最适pH(a)和pH稳定性(b) |
将PhyC1分别在不同pH缓冲液(pH 2.0-12.0)中于冰上处理60 min,在最适pH6.5,37℃的条件下测定不同pH处理后的酶活力。结果显示在pH 2.0-4.0的条件下,剩余酶活为15%-40%,而在pH 5.0-10.0条件下,剩余酶活为80%-100%,pH 11.0- 12.0条件下,剩余酶活仅为10%以下(图 3-b)。可见PhyC1在pH5.0-10.0范围内稳定性很好。
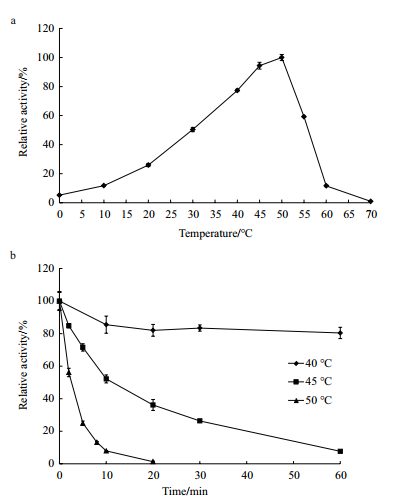
2.4 最适温度和温度稳定性在最适pH为6.5的条件下,不同温度下测定PhyC1的活性,结果如图 4-a所示,PhyC1的最适反应温度为50℃,在0-20℃时,该酶仍然保持一定的酶活性。热稳定性测定时,40℃保温60 min后,其相对酶活力仍然保留80%以上,说明PhyC1在40℃条件下热稳定性较好;在45℃保温20 min后,其剩余酶活为40%左右,但在50℃保温20 min后,酶活完全丧失,说明该酶在50℃条件下热稳定性较差,如图 4-b所示。
|
| a:最适pH值条件下,温度对PhyC1活性的影响;b:PhyC1的热稳定性。PhyC1在40℃、45℃和50℃条件下处理不同的时间,剩余酶活在最适pH值、37℃条件下检测。每个实验数据有3个重复 图 4 PhyC1的最适温度和温度稳定性 |
纯化的重组酶,经过蛋白浓度测定和酶活性测定,计算出PhyC1的比活性为16 U/mg;在37℃,pH 6.5条件下,纯化的重组植酸酶PhyC1对植酸钠的动力学参数Km和Vmax分别为0.11 mmol/L和21.83 μmol/ min/mg。
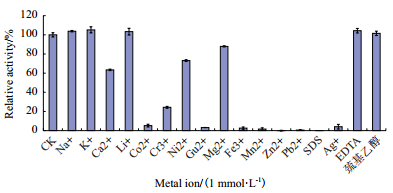
2.6 PhyC1对不同金属离子和化学试剂的耐受性在最适pH、37℃条件下,测定在标准反应总体系中加入终浓度为5 mmol/L的不同金属离子与化学试剂,结果如图 5所示,Na+、K+、Li+、EDTA、巯基乙醇在此条件下对酶活性无影响,也有部分金属离子对酶产生抑制作用,对酶活抑制影响效果排列为:Mg2+ < Ni2+ < Ca2+ < Cr3+ < Co2+ < Ag+ < Cu2+ < Fe3+ < Mn2+ < Pb2+ < Zn2+,其中Cu2+、Fe3+、Mn2+、Pb2+、Zn2+的抑制作用最强,剩余酶活力极低,而化学试剂中SDS对酶的抑制作用最强,酶活完全受到抑制。
|
| 图 5 PhyC1对化学试剂和金属离子的耐受性 |
如图 6所示,植酸酶PhyC1在胰蛋白酶处理60 min后,剩余酶活达70%以上,说明该植酸酶有很强的抗胰蛋白酶水解的能力。
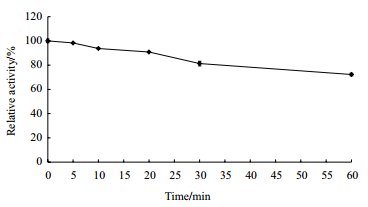
|
| 图 6 PhyC1的胰蛋白酶耐受性 |
植酸酶广泛存在于植物、动物和微生物中,目前,应用最广泛的是来源于黑曲霉Aspergillus niger的PhyA[18-19]和来源于大肠杆菌Escherichia coli的AppA[20-21]。但以上植酸酶均为酸性植酸酶,其最适pH值为4.0-6.0,使其在鱼类和虾类饲料中的应用受到很大的限制。已有研究表明,在饲料中补充酸性植酸酶对斑节对虾、日本对虾和南亚野鲮的生长性能并无影响,但补充中性植酸酶却改善了异育银鲫的生长性能和营养物质消化率[22],因此在植酸酶的选择上要依据不同动物的消化道酸碱性而定。
目前,青霉来源的植酸酶已经有一些报道,最适pH值均在4.0-5.5之间[23-27],本研究获得的植酸酶phyC1是来源于本实验室分离保存的青霉菌Penicillium pinophilum[17],植酸酶基因是首次在该菌种中报道,依据植酸酶分类方法,phyC1属于组氨酸酸性植酸酶HAP家族[6],因其最适pH为6.5,偏中性,与现有的组氨酸酸性植酸酶有一定差异,因此属于HAP家族中一个新的成员,并且phyC1在pH为5-10的范围内维持80%以上的活性,而且在较低的温度下保持更高的活性,这些性质特征都表明植酸酶PhyC1在低温及pH偏中性的环境下具有较高的植酸酶活性和稳定性,非常适宜在鱼类的消化道内发挥作用。但该酶的热稳定性较差,可以通过水产饲料加工中近几年发展起来的后喷涂工艺,得到缓解。另外在今后的研究中,通过蛋白质工程的手段,对该基因进行酶学性质改良,因此该低温中性植酸酶PhyC1可以作为储备的水产用功能性基因资源,为中性植酸酶的研发和应用提供参考。
目前,酶制剂在鱼虾饲料中的应用已经取得了一定成效,为了使植酸酶得到更加广泛的应用,科学家们通过遗传工程和蛋白质工程等技术手段获得在酶学性质方面具有更加广泛pH作用范围、蛋白酶抗性、热稳定性提高的植酸酶,以适应动物消化道内pH条件的变化、增加蛋白酶抗性、减少饲料制粒过程中的损失[28]。随着符合水产动物生理特点的酶制剂的开发,植酸酶将在水产饲料中得到更多的应用,从而提高饲料利用率,节约饲料资源,促进水产养殖业的可持续发展。
| [1] |
Reddy NR, Sathe SK, Salunkhe DK. Phytases in tegumes and cereals[J]. Adv Food Rev, 1982, 28: 1-92. DOI:10.1016/S0065-2628(08)60110-X |
| [2] |
Shanmugam G. Characteristics of phytase enzyme and its role in animal nutrition[J]. Int J Curr Microbiol Appl Sci, 2018, 7(3): 1006-1013. DOI:10.20546/ijcmas.2018.703.120 |
| [3] |
Menezes-Blackburn D, Gabler S, Greiner R. Performance of seven commercial phytases in an in vitro simulation of poultry digestive tract[J]. J Agric Food Chem, 2015, 63(27): 6142-6149. DOI:10.1021/acs.jafc.5b01996 |
| [4] |
Wyss M, Brugger R, Kronenberger A, et al. Biochemical characterization of fungal phytases(myo-inositol hexakisphosphate phosphohydrolases):catalytic properties[J]. Appl Environ Microbiol, 1999, 65: 367-373. DOI:10.1128/AEM.65.2.367-373.1999 |
| [5] |
Selle PH, Ravindran V. Microbial phytase in poultry nutrition[J]. Anim Feed Sci Technol, 2007, 135: 1-41. DOI:10.1016/j.anifeedsci.2006.06.010 |
| [6] |
Mullaney EJ, Daly CB, Ullah AH. Advances in phytase research[J]. Adv Appl Microbiol, 2000, 47: 157-199. DOI:10.1016/S0065-2164(00)47004-8 |
| [7] |
Konietzny U, Greiner R. Molecular and catalytic properties of phytate-degrading enzymes(phytases)[J]. Int J Food Sci Technol, 2002, 37: 791-812. DOI:10.1046/j.1365-2621.2002.00617.x |
| [8] |
Haefner S, Knietsch A, Scholten E, et al. Biotechnological production and applications of phytases[J]. Appl Microbiol Biotechnol, 2005, 68: 588-597. DOI:10.1007/s00253-005-0005-y |
| [9] |
Lei XG, Porres JM, Mullaney EJ, et al. Phytase : source, structure and application. In : Polaina J, MacCabe AP(eds)Industrial enzymes : source, structure and application[M], 1st edn. Springer, Netherlands, 2007 : 505-529.
|
| [10] |
Yao MZ, Zhang YH, Lu WL, et al. Phytases :crystal structures, protein engineering and potential biotechnological applications[J]. J Appl Microbiol, 2012, 112(1): 1-14. |
| [11] |
Rebello S, Jose L, Sindhu R, et al. Molecular advancements in the development of thermostable phytases[J]. Appl Microbiol Biotechnol, 2017, 101(7): 2677-2689. DOI:10.1007/s00253-017-8195-7 |
| [12] |
Mrudula Vasudevan U, Jaiswal AK, Krishna S, et al. Thermostable phytase in feed and fuel industries[J]. Bioresour Technol, 2019, 278: 400-407. DOI:10.1016/j.biortech.2019.01.065 |
| [13] |
Troesch B, Jing H, Laillou A, et al. Absorption studies show that phytase from Aspergillus niger significantly increases iron and zinc bioavailability from phytate-rich foods[J]. Food Nutr Bull, 2013, 34(S2): 90-101. |
| [14] |
Odle J, Jacobi SK, Boyd RD, et al. The potential impact of animal science research on global maternal and child nutrition and health :A Landscape Review[J]. Adv Nutr, 2017, 8(2): 362-381. DOI:10.3945/an.116.013896 |
| [15] |
Mullaney EJ, Ullah AH. The term phytase comprises several different classes of enzymes[J]. Biochem Biophys Res Commun, 2003, 312(1): 179-184. DOI:10.1016/j.bbrc.2003.09.176 |
| [16] |
Huang H, Luo H, Wang Y, et al. A novel phytase from Yersinia rohdei with high phytase hydrolysis activity under the low pH and strong pepsin conditions[J]. Appl Microbiol Biotechnol, 2008, 80: 417-426. DOI:10.1007/s00253-008-1556-5 |
| [17] |
Cai H, Shi P, Luo H, et al. Acidic β-mannanase from Penicillium pinophilum C1 :Cloning, characterization and assessment of its potential for animal feed application[J]. J Biosci Bioeng, 2011, 112(6): 551-557. DOI:10.1016/j.jbiosc.2011.08.018 |
| [18] |
Krishna C, Nokes S. Predicting vegetative inoculum performance to maximize phytase production in solid-state fermentation using response surface methodology[J]. Journal of Industrial Microbiology and Biotechnology, 2001, 26(3): 161-170. DOI:10.1038/sj.jim.7000103 |
| [19] |
Singh B, Satyanarayana T. Fungal phytases :characteristics and amelioration of nutritional quality and growth of nonruminants[J]. J Anim Physiol Anim Nutr, 2015, 99(4): 646-660. DOI:10.1111/jpn.12236 |
| [20] |
罗会颖, 姚斌, 袁铁铮, 等. 来源于Escherichia coli的高比活植酸酶基因的高效表达[J]. 生物工程学报, 2004, 20(1): 78-84. DOI:10.3321/j.issn:1000-3061.2004.01.017 |
| [21] |
Lim D, Golovan S, Forsberg C, et al. Crystal structures of Escherichia coli phytase and its complex with phytate[J]. Nat Struct Biol, 2000, 7: 108-113. DOI:10.1038/72371 |
| [22] |
冷向军. 水产饲料中酶制剂的研究与应用[J]. 饲料工业, 2018, 39(22): 1-7. |
| [23] |
Kim BH, Lee JY, Lee PC. Purification, sequencing and evaluation of a divergent phytase from Penicillium oxalicum KCTC6440[J]. J Gen Appl Microbiol, 2015, 61(4): 117-123. DOI:10.2323/jgam.61.117 |
| [24] |
Ribeiro Corrêa TL, de Queiroz MV, et al. Cloning, recombinant expression and characterization of a new phytase from Penicillium chrysogenum[J]. Microbiol Res, 2015, 170: 205-212. DOI:10.1016/j.micres.2014.06.005 |
| [25] |
Lee SH, Cho J, Bok J, et al. Characterization, gene cloning, and sequencing of a fungal phytase, PhyA, from Penicillium oxalicum PJ3[J]. Prep Biochem Biotechnol, 2015, 45(4): 336-347. DOI:10.1080/10826068.2014.923446 |
| [26] |
Zhao Q, Liu H, et al. Engineering of protease-resistant phytase from Penicillium sp. :high thermal stability, low optimal temperature and pH[J]. J Biosci Bioeng, 2010, 110(6): 638-645. DOI:10.1016/j.jbiosc.2010.08.003 |
| [27] |
Tseng YH, Fang TJ, Tseng SM. Isolation and characterization of a novel phytase from Penicillium simplicissimum[J]. Folia Microbiol(Praha), 2000, 45(2): 121-127. DOI:10.1007/BF02817409 |
| [28] |
Ushasree MV, Shyam K, Vidya J, et al. Microbial phytase :Impact of advances in genetic engineering in revolutionizing its properties and applications[J]. Bioresour Technol, 2017, 245(Pt B): 1790-1799. |





