天然橡胶(Natural rubber,NR)是植物类异戊二烯代谢途径中一种重要的次生代谢产物,其弹性、延展性、散热性等性能都优于合成橡胶。因此,NR作为一种重要的能源物质,在国民经济和国防建设中有至关重要的作用。巴西橡胶树(Hevea brasiliensis)是NR原料的主要来源,但存在种植地域局限性、真菌易感性及收割工作的繁琐性等问题。为满足天然橡胶逐年上升的需求量,各国政府和跨国公司都将寻找NR替代资源列入战略计划中[1]。
在已探明的2 500多种产胶植物中,橡胶草(Taraxacum kok-saghyz Rodin)和银色橡胶菊(Parthenium argentatum L.)产胶的相对分子质量较大,能够满足工业应用需求。其中,橡胶草适应性强、生长周期短、适合机械化种植和采收,是理想的天然橡胶替代资源,具有极高的开发价值;又因其基因组相对简单,遗传转化与基因编辑较为容易,可作为科学研究的模式植物。李家洋研究组运用PacBio单分子实时测序技术(SMRT)组装完成了高质量的橡胶草基因组草图,为天然橡胶研究奠定了坚实的基础[2]。
橡胶粒子是天然橡胶生物合成与贮存的重要场所,根据其直径的不同可分为小橡胶粒子(Small rubber particles,SRPs)和大橡胶粒子(Large rubber particles,LRPs)。橡胶粒子的膜蛋白,如橡胶转移酶(Rubber transferase,HRT)、橡胶延伸因子(rubber elongation factor,REF)和小橡胶粒子蛋白(SRP protein,SRPP),是橡胶生物合成的关键酶或蛋白因子[3-6]。所有的REF和SRPP蛋白都有一个REF保守结构域,不同的是大部分SRPPs家族成员位于SRPs,SRPPs蛋白家族N端较短,且有一个可变C端。橡胶草基因组共编码五个TkSRPP蛋白,相关研究表明,TkSRPP3、TkSRPP4和TkSRPP5基因在胶乳中的表达量较高[7]。通过抑制TkSRPP3基因的表达,橡胶草根中的乳胶含量和橡胶相对分子质量均显著降低[8]。由此可见,SRPP在维持橡胶粒子稳定和天然橡胶合成等方面都有重要作用。
SRPP基因在橡胶树乳管细胞中高水平表达,SRPP是催化和调控橡胶生物合成的重要蛋白因子[9]。为探究SRPP2蛋白在橡胶草中的作用,本研究通过提取橡胶草RNA,反转录得到橡胶草cDNA全长,克隆橡胶草小橡胶粒子蛋白(SRPP2)基因,并命名为TkSRPP2。运用生物信息学的方法对该基因序列进行同源性分析、亚细胞定位预测、蛋白质结构预测,构建细胞融合表达载体,并进行烟草亚细胞定位预测,为深入研究TkSRPP2基因的功能奠定基础。
1 材料与方法 1.1 材料新疆野生型(Wild Type,WT)橡胶草(Taraxacum kok-saghyz Rodin)无菌苗由石河子大学农业生物技术重点实验室培养。
1.2 方法 1.2.1 TkSRPP2的克隆利用李家洋课题组公布的橡胶草基因组数据,运用Primer Premier 5.0设计TkSRPP2基因引物,用TkSRPP2-F和TkSRPP2-R(表 1);以橡胶草cDNA为模板,克隆橡胶草TkSRPP2基因。PCR体系:10 μL Mix;8 μL ddH2O;1 μL cDNA;0.5 μL TkSRPP2-F;0.5 μL TkSRPP2-R。PCR反应程序:94℃预变性1 min;94℃变性15 s,57℃退火15 s,72℃延伸60 s,35个循环。
利用NCBI中的Blast数据库,获得15条相似度较大的CDS序列,利用MTGA7.0对所得序列进行聚类分析并构建系统进化树。利用Vector NTI Advance 11获得TkSRPP2基因的氨基酸序列,使用在线生物软件ExPASY ProtParam tool、SOPMA secondary structure prediction、SWISS-MODEL等在线软件分析其蛋白质理化性质并构建其三维蛋白模型。利用SignalP3.0Server(http://http://www.cbs.dtu.dk/services/SignalP-3.0/)预测信号肽,使用在线软件WoLF PSORT(http://psort.hgc.jp/form.html)对TkSRPP2蛋白亚细胞定位进行预测。通过NCBI中的Blastp工具对TkSRPP2蛋白的保守结构域进行分析。利用在线软件TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)预测分析TkSRPP2蛋白的跨膜区。
1.2.3 细胞融合表达载体构建设计引物并命名为Y-TkSRPP2-F和Y-TkSRPP2-R(表 1),克隆去终止子且带有XhoLI和BamHI两个酶切位点的TkSRP-P2基因。PCR反应条件为94℃预变性1 min;94℃变性15 s,62℃退火15 s,72℃延伸60 s,35个循环。回收得到的目的片段与细胞融合表达载体pCAMBIA2300-35S-GFP连接,连接产物转入大肠杆菌DH5α感受态细胞并进行重组子筛选鉴定,获得细胞融合表达载体pCAMBIA2300-35S-GFP-TkSRPP2。
1.2.4 烟草表皮细胞的瞬时转化参照于小帆等[10]的植物亚细胞定位方法,采用注射法进行烟草叶片瞬时表达的亚细胞定位。烟草注射菌液后在22-24℃下培养48 h。将处理后烟草叶片使用酶解法制备原生质体,共聚焦显微镜观察TkSRPP2蛋白质的定位。
1.2.5 橡胶草TkSRPP2基因的时空表达分析研究基于已公布的橡胶草基因组数据,选取稳定的GAPDH基因为内参基因[11],用Primer5.0设计引物,分别命名为q-TkSRPP2-F、q-TkSRPP2-R(表 1)。提取6月龄橡胶草不同组织及不同生长期橡胶草植株根部RNA并反转录为cDNA,采用实时荧光定量PCR,检测6月龄橡胶草不同组织及不同生长期橡胶草TkSRPP2基因表达量的差异。
2 结果 2.1 TkSRPP2基因的克隆与分析以橡胶草cDNA为模板,根据橡胶草全基因组数据设计特异引物TkSRPP2-F和TkSRPP2-R,进行PCR扩增,经1%琼脂糖凝胶电泳检测,结果(图 1)显示:在大约633 bp处有单一的特异条带,片段大小与理论值相符,将所得片段测序,测序结果与已公开基因序列相符,将其命名为TkSRPP2。
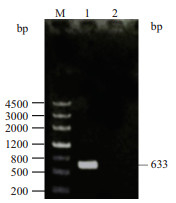
|
| M:DNA Maker3;1:TkSRPP2 图 1 TkSRPP2基因的克隆 |
通过NCBI中的Blastp工具对TkSRPP2氨基酸序列进行分析,结果显示TkSRPP2蛋白均具有一个REF超家族保守结构域。经在线生物软件ExPASY ProtParam tool(http://web.expasy.org/protparam/)分析可以得出,TkSRPP2蛋白共有210个氨基酸;理论分子量为23.15 kD;理论等电点为8.51,属于碱性蛋白;不稳定性系数为41.02,属于不稳定蛋白;平均亲水性为-0.180,为亲水性蛋白。在线软件SOPMA预测TkSRPP2蛋白质的二级结构,发现其主要由66.19% α螺旋构成,其次为26.19%无规则卷曲、4.29% β-转角。利用SWISS-MODEL软件构建其三维蛋白模型(图 2)。TkSRPP2蛋白没有跨膜区,不属于跨膜蛋白。SignalP3.0Sever预测TkSRPP2蛋白质没有信号肽,不属于分泌蛋白。亚细胞定位预测结果表明TkSRPP2蛋白主要分布于细胞质中。
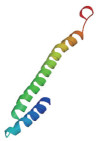
|
| 图 2 TkSRPP2蛋白的三级结构预测 |
TkSRPP2基因编码的氨基酸序列经BLAST比对,与莴苣(Lactuca sativa)、黄花蒿(Artemisia annua)、向日葵(Helianthus annuus)、洋蓟(Cynara cardunculus var. scolymus)、短角蒲公英(Taraxacum brevicorniculatum)等的抗胁迫蛋白或小橡胶粒子蛋白具有同源性,相似度均在90%以上。运用MEFA7.0构建系统进化树,结果(图 3)表明,TkSRPP2氨基酸序列与莴苣SRPP3亲缘关系最近。

|
| 图 3 TkSRPP2系统进化树分析 |
将去终止子的目的基因片段与克隆载体pMD19T连接,以此获得重组质粒pMD19-T-TkSRPP2。将测序鉴定正确的pMD19-T-TkSRPP2重组质粒和细胞融合表达载体pCAMBIA2300-35S-GFP同时用XbaⅠ和BamHⅠ进行双酶切,连接并转化大肠杆菌DH5α感受态细胞,挑取抗性筛选平板上的单菌落,做菌落PCR鉴定;PCR鉴定为阳性的菌落扩繁提取质粒,并做双酶切鉴定(图 4),成功构建细胞融合表达载体pCAMBIA2300-35S-GFP-TkSRPP2。

|
| 图 4 pCAMBIA2300-35S-GFP-TkSRPP2双酶切鉴定图 |
在模式植物烟草体内进行瞬时表达,用酶解法处理侵染后的叶片,成功制备原生质体后,在激光扫描共焦显微镜下观察,分析橡胶草TkSRPP2蛋白的亚细胞定位情况(图 5)。显微镜下多数细胞中均有荧光,橡胶草TkSRPP2蛋白定位于植物细胞质内,与其功能及预测结果相符。
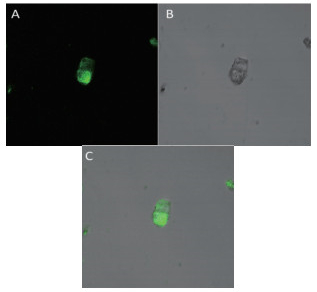
|
| A:绿色荧光;B:明场;C:合成 图 5 TkSRPP2亚细胞定位图 |
采用qRT-PCR分析橡胶草TkSRPP2基因的时空表达特异性,检测TkSRPP2基因在6月龄橡胶草根、叶、花、花葶等不同组织表达情况以及在1月龄、3月龄、5月龄、6月龄、7月龄、8月龄不同生长期橡胶草根部的表达量。结果(图 6)表明,TkSRPP2基因在6月龄橡胶草根部表达量最高;6月龄橡胶草根部TkSRPP2基因表达量到达峰值。
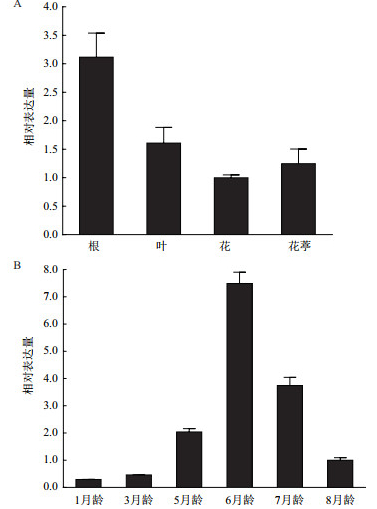
|
| A:6月龄橡胶草不同组织;B:橡胶草根部不同生长时期 图 6 TkSRPP2基因时空表达分析 |
天然橡胶是一类有经济价值的聚异戊二烯链,存在于约2 500种植物的胶乳细胞中。虽然不同物种胶乳中的化学成分存在一定差异,但天然橡胶生物合成的机制相似,植物体内主要通过MVA的类异戊二烯代谢途径完成天然橡胶的合成,都涉及酶与胶乳中橡胶粒子结合的过程[12-13]。橡胶粒子中含有300种以上蛋白质,其中橡胶延伸因子(REF)和小橡胶粒子蛋白(SRPP)含量最高[14]。经系统进化关系研究发现,REF蛋白和SRPP蛋白为同源蛋白,都属于植物应激相关蛋白超家族[4],在橡胶树乳管细胞中,REF和SRPP家族基因均有很高的表达量[9]。在银胶菊、橡胶草等产胶植物的乳胶中均发现了SRPP蛋白,短角蒲公英的SRPP蛋白与橡胶草中的极为相似,与橡胶粒子密切相关[15-16]。由此可见,这些基因与橡胶的生物合成相关。SRPP蛋白属于植物抗逆蛋白家族中的一员,已有研究表明,在环割胁迫后,SRPP蛋白出现高表达和大量累积[17];研究者在巴西橡胶树SRPP基因启动子的研究中发现,SRPP基因启动子存在热响应元件、脱落酸响应元件等顺式作用元件,据此推测SRPP基因在抵御逆境胁迫的生理过程中可能具有重要的功能[18]。
本研究从橡胶草植株中克隆得到全长633 bp的编码序列,并将其命名为TkSRPP2。实时荧光定量分析结果显示,该基因主要在橡胶草根中表达,与其协助橡胶草产胶的功能一致;6月龄橡胶草根部TkSRPP2基因表达量达到峰值,此时橡胶草乳管分化最为完全。由此可见,TkSRPP2基因功能与乳管分化息息相关。运用NCBI中的Blastp功能进行结构域预测,发现TkSRPP2蛋白有一个REF保守结构域,符合SRPP家族的基本特征。通过相关软件预测TkSRPP2蛋白结构及性质,结果表明,该蛋白属于碱性、亲水性、不稳定蛋白,没有信号肽及跨膜结构域。亚细胞定位预测结果表明TkSRPP2蛋白主要位于细胞质中,这一预测结果与烟草亚细胞定位结果相符。已有研究表明,SRPP蛋白可能会通过渗透作用从橡胶粒子中释放,在橡胶粒子膜脂质的头部基团起到类似于“覆盖”的作用[19],本研究的预测结果符合这一特征。在橡胶树中,SRPP蛋白被认为存在于橡胶粒子膜上,起到维持胶体稳定性的作用,在调控胶乳形成过程中起着重要作用[20]。进化树分析结果表明,产胶植物的REF/SRPP家族成员分布于一大支,其中橡胶草TkSRPP2蛋白与莴苣SRPP3蛋白有最近的亲缘关系,橡胶树SRPP家族成员分布于另一小支,这也表明橡胶草TkSRPP2蛋白与橡胶树SRPP蛋白具有相似功能。这些研究为TkSRPP2基因功能的探索提供了重要依据。
4 结论以橡胶草cDNA为模板,克隆得到TkSRPP2基因。该基因主要在根部表达,在6月龄橡胶草根部表达量达到峰值,TkSRPP2蛋白定位于细胞质中。
| [1] |
Beilen JBV, Poirier Y. Establishment of new crops for the production of natural rubber[J]. Trends in Biotechnology, 2007, 25(11): 522-529. DOI:10.1016/j.tibtech.2007.08.009 |
| [2] |
Lin T, Xu X, Ruan J, et al. Genome analysis of Taraxacum kok-saghyz Rodin provides new insights into rubber biosynthesis[J]. National Science Review, 2018, 5: 78-87. DOI:10.1093/nsr/nwx101 |
| [3] |
Dennis MS, Light DR. Rubber elongation factor from Hevea brasiliensis. Identification, characterization, and role in rubber biosynthesis[J]. Journal of Biological Chemistry, 1989, 264(31): 18608. |
| [4] |
Berthelot K, Lecomte S, Estevez Y, et al. Rubber particle proteins, HbREF and HbSRPP, show different interactions with model membranes[J]. Biochimica et Biophysica Acta(BBA)- Biomembranes, 2014, 1838(1): 287-299. DOI:10.1016/j.bbamem.2013.08.025 |
| [5] |
Berthelot K, Lecomte S, Estevez Y, et al. Homologous Hevea brasiliensis REF(Hevb1)and SRPP(Hevb3)present different auto-assembling[J]. Biochimica et Biophysica Acta(BBA)- Proteins and Proteomics, 2014, 1844(2): 473-485. DOI:10.1016/j.bbapap.2013.10.017 |
| [6] |
Laibach N, Hillebrand A, Twyman RM, et al. Identification of a, Taraxacum brevicorniculatum, rubber elongation factor protein that is localized on rubber particles and promotes rubber biosynthesis[J]. The Plant Journal, 2015, 82(4): 609-620. DOI:10.1111/tpj.12836 |
| [7] |
Amerik AY, Martirosyan YT, Gachok IV. Regulation of natural ru-bber biosynthesis by proteins associated with rubber particles[J]. Russian Journal of Bioorganic Chemistry, 2018, 44(2): 140-149. DOI:10.1134/S106816201801003X |
| [8] |
Men X, Wang F, Chen G, et al. Biosynthesis of natural rubber:Current state and perspectives[J]. Int J Mol Sci, 2019, 20. DOI:10.3390/ijms20010050 |
| [9] |
Oh SK, Kang H, Shin DH, et al. Isolation, characterization, and functional analysis of a novel cDNA clone encoding a small rubber particle protein from Hevea brasiliensis[J]. Journal of Biological Chemistry, 1999, 274(24): 17132-17138. DOI:10.1074/jbc.274.24.17132 |
| [10] |
于一帆, 朱小彬, 葛会敏, 等. 基于绿色荧光蛋白瞬时表达的植物亚细胞定位方法[J]. 江苏农业科学, 2014, 42(12): 58-61. |
| [11] |
覃碧, 潘敏, 余海洋, 等. 橡胶草实时荧光定量PCR内参基因评价[J]. 植物生理学报, 2016(7): 1059-1065. |
| [12] |
Bushman BS, Scholte AA, Cornish K, et al. Identification and comparison of natural rubber from two Lactuca species[J]. Phytochemistry, 2006, 67(23): 2590-2596. DOI:10.1016/j.phytochem.2006.09.012 |
| [13] |
Cornish K. Similarities and differences in rubber biochemistry among plant species[J]. Phytochemistry(Oxford), 2001, 57(7): 1123-1134. DOI:10.1016/S0031-9422(01)00097-8 |
| [14] |
Wang X, Shi M, Lu X, et al. A method for protein extraction from different subcellular fractions of laticifer latex in Hevea brasiliensis compatible with 2-DE and MS[J]. Proteome Science, 2010, 8(1): 35. DOI:10.1186/1477-5956-8-35 |
| [15] |
Ko JH, Chow KS, Han KH. Transcriptome analysis reveals novel features of the molecular events occurring in the laticifers of Hevea brasiliensis(para rubber tree)[J]. Plant Molecular Biology, 2003, 53(4): 479-492. DOI:10.1023/B:PLAN.0000019119.66643.5d |
| [16] |
Collins JE. The role of small rubber particle proteins in rubber biosynthesis[D]. Reno: University of Nevada, 2009.
|
| [17] |
Fricke J, Hillebrand A, Twyman RM, et al. Abscisic acid-dependent regulation of small rubber particle protein gene expression in Taraxacum brevicorniculatum is mediated by TbbZIP1[J]. Plant & Cell Physiology, 2013, 54(4): 448-464. |
| [18] |
李先昆, 段翠芳, 聂智毅, 等. 巴西橡胶树小橡胶粒子蛋白(SRPP)基因启动子的克隆及其功能分析[J]. 中国农学通报, 2009(1): 241-247. |
| [19] |
Li JF, Norville JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nature Biotechnology, 2013, 31(8): 688-691. DOI:10.1038/nbt.2654 |
| [20] |
冯伟强, 仝征, 靳翔, 等. 巴西橡胶树REF/SRPP家族基因的克隆及表达分析[J]. 分子植物育种, 2016, 15(11): 3024-3032. |





