基因突变通常指基因在碱基对上发生的组成或排列顺序的改变,主要的突变类型分为碱基置换突变(点突变),移码突变,缺失突变,插入突变等。外显子编码基因发生突变后通过转录、翻译使多肽(链)中氨基酸组成或顺序发生改变,进而影响蛋白质或酶的生物功能,使机体的表型出现异常。碱基突变是癌症发生与发展的重要物质基础,也是遗传毒性评价的重要的检测终点之一。Pig-a基因编码形成糖磷脂酰肌醇(GPI)锚定生物合成所需的N-乙酰葡糖胺基转移酶的催化亚基,在不同种属之间具有结构、功能的高度保守性[1]。Pig-a基因位于X染色体,其片段中单一突变即可影响GPI锚的合成并导致细胞表面GPI锚链蛋白的缺失,从而可通过检测细胞膜表面锚链蛋白表达水平评价受试物的潜在遗传毒性风险。Araten等[2]最初于1999年建立以Pig-a基因为报告基因的体内基因突变试验方法。基于流式细胞术测定GPI锚链蛋白缺失的体内Pig-a基因突变试验近年来得到迅速的发展,可使用小鼠、大鼠、非人灵长类、人类的造血细胞检测受试物的体内致突变性,且基于大鼠的检测方法已通过联合验证证明其重复性及可转移性[3-6]。
基于Pig-a基因在不同种属之间高度的结构、功能保守性,以人类B淋巴细胞样细胞系TK6、MCL-5以及L5178Y等哺乳动物细胞系开展的体外Pig-a基因突变试验也获得一些进展。McKinzie等[7]对L5178Y进行全基因组测序研究,参与GPI锚链蛋白合成的基因测序研究结果显示发现所有编码基因均存在于L5178Y中,且没有任何一个基因表现出明显的有害突变,在所有编码GPI锚链蛋白基因中只有Pig-a基因存在纯和突变,其他基因均为杂合突变,预测L5178Y细胞适用于体外Pig-a基因突变研究。Wang等[8]首次报道采用L5178Y细胞进行体外Pig-a突变研究,随后David等[9]对该方法进行了验证研究,采用不同作用机制的8种化合物研究结果表明,体外Pig-a基因突变试验不但可以进行基因突变突变诱导检测,更重要的是可以区分不同作用机制遗传毒性物质,此外还证明了GPI锚链蛋白CD90的缺失不是通过环乙亚胺处理抑制蛋白合成。以TK6细胞为试验体系的研究中,2015年Kruger等[10]建立了基于TK6细胞的体外Pig-a基因突变检测方法。在前期实验基础上,Kruger[11]又进行了相关基因型与TK6细胞表型的关系。国内李若婉等[12]建立基于TK6细胞的体外Pig-a基因突变方法。就上述各种报道研究采用不同的细胞系而言,全基因组测序证实L5178Y细胞中不携带Pig-l基因突变[7, 10-11],故试验前期无需进行自发突变清除,但TK6细胞中Pig-l基因的缺失造成自发突变清除前TK6细胞中观察到的高水平Pig-a背景突变,在进行正式实验之前需要预清除自发突变细胞。此外L5178Y细胞还开展tk/hprt基因突变研究,对同一物质可进行不同基因水平致突变联合研究。
本研究在国内首次使用小鼠淋巴瘤(L5178Y)细胞建立和验证体外Pig-a基因突变实验,以期在体内Pig-a试验之前进行体外Pig-a基因突变遗传毒性评价,提供可能的体外结果指示。本方法的建立对Ames试验弱阳性结果以及在药物代谢产物、杂质的遗传毒性研究上具有很大的实用价值,为化合物体外遗传毒性评价,药物研发早期遗传毒性筛选提供新选择[13-14]。
1 材料与方法 1.1 材料 1.1.1 药物与试剂二甲基亚砜(DMSO)(批号:#BCBW5664),G-6-P(批号:#WXBC7942V),NADP(批号:18E2156560),青链霉素混合液(批号:2019313),N-乙基-N-亚硝基脲(N-nitroso-N-ethylure,ENU)(批号:MKCD2123),甲基磺酸乙酯(Ethylmethylsulfone,EMS)(批号:126K0758),4-硝基喹啉-N-氧化物(4-nitroquinoline N-oxide,4-NQO)(批号:89C-0710),苯并芘(BenzoaPyrene,B(a)P)(批号:SLBF45532)购自Sigma。葡萄糖(glucose,Glc)(批号:20181019),氯化钠(Sodium chloride,NaCl)(批号:20190121),多聚甲醛(批号:20141218),丙酮酸钠(批号:20160612)购自国药集团化学试剂有限公司。RPMI1640培养基(批号:2025394)购自Gibco。马血清(批号:AC10235369),PBS(批号:AD19942268)购自Hyclone。APC-Anti-CD45(批号:7166674),PE-Anti-CD90.2(批号:7110569)购自BD biosciences。Anti-PE MicroBeads(批号:51810917330),LS Columns(批号:5180614049)购自Miltenyi。肝匀浆S9(批号:20181105)购自北京安保迪科技有限公司。山羊血清(批号:428E051)购自索莱宝科技有限公司。Rat mAb to CD90(批号:GR3226793-2),RbPAb to CD4(GR3240865-1),山羊抗兔IgG H & L(Alexa Fluor® 568)(批号:GR3230171-3),山羊抗大鼠IgG H & L(Alexa Fluor® 488)(批号:GR3240085-1),DAPI Staining Solution(批号:GR3253253-2)购自Abcam。TRNzol总RNA提取试剂(批号:DP405-02)购自天根生化科技(北京)有限公司。PrimeScriPtTM RT reagent Kit with gDNAEraser(批号:RR047B),2xEs TaqMasterMix(批号:CW0718)购自TaKaRa(宝生物)。引物由Invitrogen合成。
1.1.2 细胞小鼠淋巴瘤细胞L5178Ytk+/--3.7.2C,引自日本国立医药品食品卫生研究所,支原体检查后于液氮长期保存,研究所用细胞为复苏传代后10代以内。
1.1.3 仪器流式细胞仪(FACSCalibur,BD biosc-ience),高速冷冻离心机(Centrifuge 5810R,Epp-endorf),倒置荧光显微镜(IX71,Olympus),二氧化碳培养箱(HERA cell VIOS 160i,Thermo Fisher),生物安全柜(NU-543-400S,Nuair),倒置显微镜(CKX31,Nikon),免疫磁性分离架(QuadroMACSTMSeparator,Miltenyi),分光光度计(NANODROP 2000,Thermo scientific),凝胶成像系统(Tanon 1600,上海天能科技有限公司),PCR仪(ABI,Applied Bio-systems)。
1.2 方法将L5178Y细胞解冻复苏后置于完全培养基R10(含有10%马血清,1%青链霉素混合液,0.2 mg/mL丙酮酸钠的RPMI 1640)在37℃、5% CO2的培养箱内培养,培养基隔天更换,待细胞增殖至适宜密度后进行试验。
1.2.1 细胞毒性通过细胞相对倍增速率(Relative population doubling,RPD)进行评价:
细胞倍增速率(Population doubling,PD)=[log(受试物处理后细胞量/初始细胞量)]/log 2;RPD=(受试物组PD/对照组PD)×100%。
1.2.2 连续(24 h)处理组细胞达对数生长期后,更换R0培养基(含有1%青链霉素混合液,0.2 mg/mL丙酮酸钠RPMI 1640),并添加不同浓度的Glc(0、5.625、11.25、22.5、45 μg/mL),NaCl(0、1.8、3.75、7.5、15、30、60 μg/mL),EMS(0、10、100、200、300、400、500 μg/mL),ENU(0、25、50、75、100、125、250 μg/mL),调整细胞密度为5×105个细胞/mL于10 mL培养基中,细胞于37℃,5%CO2条件下与受试物作用24 h。处理结束后收集细胞并计数,细胞经离心(1 000 r/min,5 min,室温),PBS洗涤两次,细胞重悬于10 mL完全培养基R10,表达期细胞密度维持在1.0×106-2.0×106个细胞/mL。
1.2.3 短时(4 h)处理组细胞达对数生长期后,更换R0培养基(含有1%青链霉素混合液,0.2 mg/mL丙酮酸钠的RPMI 1640),并添加不同浓度的4-NQO(0、0.01、0.05、0.075、0.10、0.15、0.20 μg/mL),B(a)P(0、0.5、1.0、2.0、3.0、4.0、5.0 μg/mL)和2% S9mix,调整细胞密度为5×105个细胞/mL于50 mL离心管中。37℃,20 r/min振摇4 h。细胞经离心(1 000 r/min,5 min,室温)后,PBS洗涤两次,重悬于10 mL完全培养基R10。给药后24 h后作细胞计数,表达期细胞密度维持在1.0×106-2.0×106个细胞/mL。
1.2.4 流式门控模板调试取2×106个细胞离心(1 000 r/min,4℃,5 min),每孔加入200 μL APC-anti-CD45和PE-anti-CD90.2混合工作液,终浓度为1 μg/mL,2-8℃避光孵育30 min,后置于室温继续孵育10 min,离心(340×g,4℃,3 min),收集细胞进行流式检测,初步确定适宜电压及补偿策略。
在EMS给药表达后第21天收集5×108个细胞离心(1 000 r/min,4℃,5 min),加入2.5 mLAPC-Anti-CD45和PE-Anti-CD90抗体工作液,2-8℃避光孵育30 min,后置于室温继续孵育10 min,离心(300×g,4℃,5min),加入120 μL anti-PE磁珠于500 μL工作液(2% FBS+PBS)混匀,置于2-8℃,避光孵育15 min,离心(340×g,5 min,4℃),加入5 mL工作液(2% FBS+PBS)混悬均匀。提前准备工作液(2% FBS+PBS)润洗LS柱子,过柱分离,收集流出组分,离心(340×g,5 min,4℃),收集细胞进行流式检测,用于确定流式检测模板。
1.2.5 Pig-a基因突变检测周期的确定L5178Y细胞分别经不同浓度EMS处理后,于给药后第4、8、12、16、20天检测Pig-a基因突变频率,以确定最大突变频率发生时间点。
1.2.6 免疫荧光成像分析取野生型细胞和突变型细胞混合,置于4%多聚甲醛室温固定20 min,5%山羊血清37℃封闭30 min,一抗RbpAbCD45(1μg/mL)和Rat mAb to CD90(1 μg/mL)4℃孵育过夜,二抗Goat-anti-Rat(AlexaFluor 488)和Goat-anti-Rb(AlexaFluor 568)及DAPI 4℃避光孵育1 h,荧光显微镜成像分析。
1.2.7 突变位点检测富集ENU致突变型细胞,进行PCR方法检测组织样本中目的基因序列突变位点。从NCBI数据库确认小鼠Pig-a基因序列,引物设计见表 1。
采用TRIzol总RNA提取试剂进行样本RNA提取,PrimeScriPtTM RT reagent Kit with gDNA Eraser进行cDNA反转录。
PCR样本检测:将所有cDNA样品分别配置RealtimePCR反应体系。按以下程序进行:95℃,3 min;30个PCR循环(95℃,30 s,58℃,30 s,72℃,40 s);72℃,5min。扩增结束后取5 μL产物用1.5%琼脂糖凝胶进行电泳,切胶回收目的片段,经DNA凝胶回收试剂盒纯化后,用ABI PRISM TM377XL测序仪以末端标记双脱氧法测序,Chromas软件(TechnelysiumPly Ltd. Version 2.6.2)分析测序结果。
1.2.8 数据处理试验数据采用GraphpadPrism7(USA,Graphpad Software公司)进行数据统计,采用One Way ANOVA检验将各受试物处理组的突变率与溶媒对照组作比较,组间比较P < 0.05视为差异存在统计学意义。
2 结果 2.1 细胞毒性测定结果不同受试物给药24 h或4 h后的RPD结果如图 1所示。给药24 h:非S9代谢活化条件下:Glc,NaCl,EMS,ENU,给药4 h组:S9代谢活化条件下B(a)P,4-NQO所设浓度组RPD均大于50%。提示本研究受试物在给药浓度范围内均未见无明显细胞毒性作用,排除试验中假阳性结果。

|
| ENU:给药24 h;EMS:给药24 h;B(a)P:给药4 h;4-NQO:给药4 h;NaCl:给药24 h;Glucose:给药24 h 图 1 不同受试物给药24 h或4 h后的RPD(±s,n=3) |
初步流式门控调试如图 2所示。分别对细胞进行空白对照、单染APC-anti-CD45、PE-anti-CD90.2和双染进行流式电压、补偿等调试至合适的图谱,初步确定合适的流式门控策略,收集突变体细胞对突变域进一步完善确认。
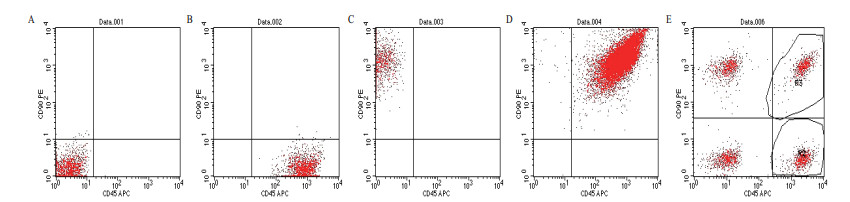
|
| A:空白细胞仅在LL区域;B:单染APC-anti-CD45(1 μg/mL)抗体在LR区域;C:单染PE-anti-CD90.2(1 μg/mL)在UL区域;D:双染APC-anti-CD45和PE-anti-CD90.2(1 μg/mL)在UR区域;E:取相同体积量的ABCD管细胞混合 图 2 流式模板初步调试过程图 |
收集突变表达21 d后的EMS细胞通过免疫磁性分离技术对突变型细胞进行富集后进行流式检测(图 3-A),通过上述流式门控策略调试进一步确定R2区域为GPI(-)区域,即CD45(+)/CD90.2(-)。

|
| A:突变体细胞富集确认突变区域R2(CD90(-)/CD45(+))、R3(CD90(+)/CD45(+));B:取空白细胞分别进行空白对照、单染APC-anti-CD45(R2区域模拟突变细胞)、单染PE-anti-CD90.2、双染APC-anti-CD45和PE-anti-CD90.2四种染色方式后定量混合均匀确定检测当日流式条件质控 图 3 流式突变区域确认与质控图 |
Pig-a基因突变频率变化见图 4,呈现先上升后下降趋势,整体在第8天左右呈现最大突变频率,最终确定细胞在受试物作用时间结束后表达8 d作为Pig-a基因突变频率检测时间终点。

|
| 图 4 Pig-a基因突突变频率表达周期 |
EMS、ENU、NaCl、Glc作用24h,B(a)P、4-NQO给药4 h表达8 d Pig-a基因突变频率检测结果(图 5-6)显示,非S9代谢活化条件下,EMS浓度大于100 μg/mL时,Pig-a基因突变率与溶媒对照组相比,存在显著性差异(*P < 0.05,**P < 0.01,***P < 0.001),且呈浓度递增趋势;ENU浓度大于125 μg/mL时,Pig-a基因突变率与溶媒对照组相比,存在显著性差异(**P < 0.01,***P < 0.001),且呈浓度递增趋势。
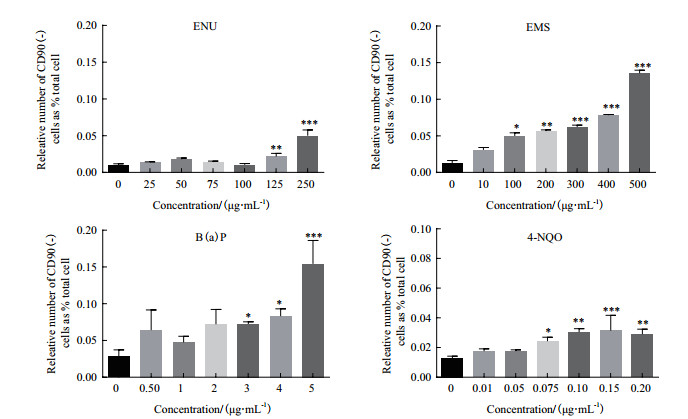
|
| 图 5 EMS、ENU给药24 h,B(a)P、4-NQO给药4 h表达8d Pig-a基因突变频率 |

|
| 图 6 NaCl、Glc给药24 h表达8 d Pig-a基因突变频率 |
S9代谢活化条件下,当B(a)P浓度大于2.0 μg/mL时,Pig-a基因突变率与溶媒对照组相比,存在显著性差异(*P < 0.05,***P < 0.001),且成浓度递增趋势。当4-NQO浓度大于0.075 μg/mL时Pig-a基因突变率与溶媒对照组相比,存在显著性差异(*P < 0.05,**P < 0.01,***P < 0.001),且成浓度递增趋势。
非S9代谢活化条件下,阴性对照Glc、NaClPig-a基因突变率与溶媒对照组相比,不存在显著性差异(P > 0.05),且无浓度梯度关系。
2.5 免疫荧光结果采用免疫荧光方法染色识别细胞核(DAPI,蓝色)、细胞表面CD45(AlexaFluor 568,橙色)和细胞表面CD90.2(AlexaFluor 488,绿色)后,可见细胞表面仅表达绿色荧光信号的突变细胞和同时表达红色及绿色荧光信号的野生型细胞(图 7)。
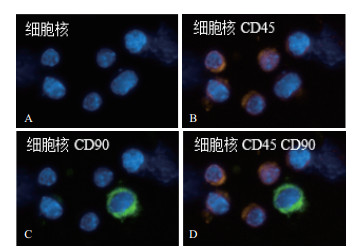
|
| A:DAPI染细胞核;B:所有细胞表面均表达CD45(Alexa Fluor® 568);C:突变型细胞表面无CD90,正常细胞表面表达CD90(Alexa Fluor® 488);D:通道叠加 图 7 免疫荧光结果图 |
PCR结果(表 2)测序显示存在4种突变类型。
已知非致突变化合物NaCl和Glc在本研究体系中均显示为阴性结果,细胞与两者共同作用24 h后表达8 d均未见CD90突变率增加。EMS属于烷化剂,在非代谢活化状态下可导致基因突变,以点突变为主。ENU则是体内Pig-a基因突变首选阳性剂,可导致DNA碱基烷化、互补碱基对横向交联等损伤,最终引发基因突变[15]。本研究结果显示,上述两种阳性剂均可在非代谢活化条件下引起L5178Y细胞Pig-a基因突变率显著性升高,大于阴性对照的10倍。阳性剂B(a)P则需经细胞微粒体中的混合功能氧化酶激活转化为环氧化物,与DNA共价结合形成加合物,从而诱发突变[16]。4-NQO在还原酶的作用下可转化为4-羟氨基喹啉-1-氧化物及4-乙酰基喹啉-1-氧化物,后者能以共价结合的方式结合核酸形成DNA加合物,引起碱基的改变(G→A)[17-18]。本研究结果显示在S9代谢活化条件下,B(a)P浓度大于2 μg/mL时与L5178Y细胞作用4 h后可引起Pig-a基因突变率显著性升高,大于阴性对照的10倍。4种阳性化合物与2种阴性化合物的试验结果与预期相符。
EMS作用于L5178Y细胞24 h后进行20 d表达培养,分别于第4、8、12、16、20天检测Pig-a基因突变频率,整体趋势呈现出先上升趋势,于8 d左右出现峰值,后随时间推移呈现下降趋势,提示最佳检测时间点为8 d。阳性剂体内Pig-a基因突变频率变化为上升而无下降趋势,体内/外Pig-a基因突变频率变化趋势存在一定差异,主要原因是受试物进入机体后产生骨髓毒性,通过不断造血将突变细胞释放至外周血,产生累积效应。但L5178Y细胞需通过稀释传代维持在合适的细胞密度,当表达峰值过后再进行稀释传代,突变细胞比例降低,故突变频率呈现下降趋势。L5178Y细胞在给药浓度设置上应明确最高剂量,细胞毒性RPD应大于或略低于50%,以排除因细胞毒性过大而造成的假阳性结果。免疫荧光结果进一步证实突变细胞表面无CD90表达。
ENU致突变细胞测序结果显示存在G→C,A→C,C→T三种不同类型的点突变。相关研究显示采用不同阳性剂有不同的突变类型。David R等[9]研究结果显示EMS作用L5178Y细胞后主要的突变类型有G→A,C→T两种突变类型;Javier Revollo等[19]发现B(a)P诱导的Pig-a基因突变主要是G:C或富含G:C序列的碱基对的取代,小插入或缺失等,主要的突变类型有G→T,G→C,G→A,A→T,T→C。
基因突变检测是遗传毒性评价的重要手段,本研究所用L5178Y细胞建立Pig-a基因突变试验,与体内Pig-a基因突变试验相比可大幅度节省试验成本,检测耗时周期短,不涉及动物伦理,符合“3R”原则。可适用于Ames试验中不适宜检测的化合物,作为后续追踪检测。相较于传统的hprt或tk基因突变存在一定的优势,试验耗时短,耗材少,可进行高通量检测。另外,L5178Y细胞Pig-a基因自发突变率较低,试验前期无需进行杂合性突变缺失检测,但tk基因突变则在试验前期进行预清除[20]。对上述4种试验方法进行对比讨论见表 3。
综上,本研究在国内首次建立并验证报道流式细胞术检测L5178Y小鼠淋巴瘤细胞中Pig-a基因突变的方法,并使用四种诱变剂检验该方法的有效性和可靠性。后期可经不同实验室进行进一步大规模联合验证,建立和规范相关试验规程操作,作为药物遗传毒性致突变评价中介于Ames试验和体内Pig-a的桥梁,进一步完善体外遗传毒性致突变组合。
4 结论本研究通过使用流式细胞术检测EMS、ENU、B(a)P、4-NQO、NaCl、Glc致小鼠淋巴瘤细胞L5178Y表面GPI连接的CD90蛋白缺失情况,建立并验证基于L5178Y细胞的体外Pig-a基因突变试验方法。
| [1] |
Kawagoe K, Takeda J, Endo Y, et al. Molecular cloning of murine Pig-a, a gene for GPI-anchor biosynthesis, and demonstration of interspecies conservation of its structure, function, and genetic locus[J]. Genomics, 1994, 23(3): 566-574. DOI:10.1006/geno.1994.1544 |
| [2] |
Araten DJ, Nafa K, Pakdeesuwan K, et al. Clonal populations of hematopoietic cells with paroxysmal nocturnal hemoglobinuria genotype and Phenotype are present in normal individuals[J]. Proc Natl Acad Sci USA, 1999, 96(9): 5209-5214. DOI:10.1073/pnas.96.9.5209 |
| [3] |
Dobrovolsky VN, ElesPuru RK, Bigger CA, et al. Monitoring humans for somatic mutation in the endogenous Pig-a gene using red blood cells[J]. Environ Mol Mutagen, 2011, 52(9): 784-794. DOI:10.1002/em.20667 |
| [4] |
Dobrovolsky VN, Miura D, Heflich RH, et al. The in vivo Pig-a gene mutation assay, a potential tool for regulatory safety assessment[J]. Environ Mol Mutagen, 2010, 51(8-9): 825-835. DOI:10.1002/em.20627 |
| [5] |
Chen GF, Wen HR, Mao ZH, et al. Assessment of the Pig-a, micronucleus, and comet assay endpoints in rats treated by acute or repeated dosing protocols with procarbazine hydrochloride and ethyl carbamate[J]. Environmental and Molecular Mutagenesis, 2019, 60(1): 56-71. DOI:10.1002/em.22227 |
| [6] |
Pu J, Deng Y, Tan X, et al. The in vivo pig-a gene mutation assay is applied to study the genotoxicity of procarbazine hydrochloride in sprague-dawley rats[J]. Fundamental Toxicological Sciences, 2016, 3(4): 167-175. DOI:10.2131/fts.3.167 |
| [7] |
McKinzie PB, Revollo JR. Whole genome sequencing of mouse lymphoma L5178Y-3. 7. 2C(TK(+/-))reveals millions of mutations and genetic markers[J]. Mutat Res, 2017, 814: 1-6. DOI:10.1016/j.mrgentox.2016.12.001 |
| [8] |
Wang Y, Revollo J, McKinzie P, et al. Establishing a novel Pig-a gene mutation assay in L5178YTk(+/-)mouse lymphoma cells[J]. Environ Mol Mutagen, 2018, 59(1): 4-17. DOI:10.1002/em.22152 |
| [9] |
David R, Talbot E, Allen B, et al. The development of an in vitro Pig-a assay in L5178Y cells[J]. Arch Toxicol, 2018, 92(4): 1609-1623. DOI:10.1007/s00204-018-2157-4 |
| [10] |
Kruger CT, Hofmann M, Hartwig A. The in vitro PIG-A gene mutation assay:mutagenicity testing via flow cytometry based on the glycosylphosphatidylinositol(GPI)status of TK6 cells[J]. Arch Toxicol, 2015, 89(12): 2429-2443. DOI:10.1007/s00204-014-1413-5 |
| [11] |
Kruger CT, Fischer BM, Armant O, et al. The in vitro PIG-A gene mutation assay:glycosylphosphatidylinositol(GPI)-related genotype-to-phenotype relationship in TK6 cells[J]. Arch Toxicol, 2016, 90(7): 1729-1736. DOI:10.1007/s00204-016-1707-x |
| [12] |
李若婉, 周长慧, 黄鹏程, 等. 基于TK6细胞的体外PIG-A基因突变检测方法的建立[J]. 癌变·畸变·突变, 2019, 31(3): 242-248. |
| [13] |
GollaPudi BB, Lynch AM, Heflich RH, et al. The in vivo Pig-a assay:A report of the international workshop on genotoxicity testing(IWGT)workgroup[J]. Mutat Res Genet Toxicol Environ Mutagen, 2015, 783: 23-35. DOI:10.1016/j.mrgentox.2014.09.007 |
| [14] |
Johnson GED, Slob W, Doak SH, et al. New approaches to advance the use of genetic toxicology analyses for human health risk assessment[J]. Toxicology Research, 2015, 4(3): 667-676. DOI:10.1039/C4TX00118D |
| [15] |
白宝清, 赵丽娇, 钟儒刚. 亚硝基脲对DNA损伤作用的研究进展[J]. 化学通报, 2010, 73(3): 212-219. |
| [16] |
Gajecka M, Kujawski LM, Gawecki J, et al. The protective effect of vitamins C and E against B(a)P-induced genotoxicity in human lymphocytes[J]. J Environ Pathol Toxicol Oncol, 1999, 18(3): 159-167. |
| [17] |
Downes DJ, Chonofsky M, Tan K, et al. Characterization of the mutagenic spectrum of 4-nitroquinoline 1-oxide(4-NQO)in Aspergillus nidulans by whole genome sequencing[J]. G3(Bethesda), 2014, 4(12): 2483-2492. |
| [18] |
Ribeiro DA, Favero SDM. Genomie instability in non-neoPlastic oral mueosa cells can predict risk during 4-nitroquinoline 1-oxide-induced rat tongue carcinogenesis[J]. 2004, 40(9): 910-915.
|
| [19] |
Revollo J, Wang Y, Mckinzie P, et al. Spectrum of benzo[a]pyrene-induced mutations in the Pig-a gene of L5178YTk+/- cells identified with next generation sequencing[J]. Mutat Res, 2017, 824: 1-8. DOI:10.1016/j.mrgentox.2017.09.003 |
| [20] |
Wang J, Sawyer JR, Chen L, et al. The mouse lymphoma assay detects recombination, deletion, and aneuploidy[J]. Toxicol Sci, 2009, 109(1): 96-105. DOI:10.1093/toxsci/kfp037 |







