2. 西南民族大学生命科学与技术学院,成都 610041;
3. 西南民族大学青藏高原动物遗传资源保护与利用重点实验室,成都 610041;
4. 四川华汉三创生物科技有限公司,成都 610093
2. College of Life Science and Technology, Southwest Minzu University, Chengdu 610041;
3. Key Laboratory of Qinghai-Tibetan Plateau Animal Genetic Resource Reservation and Utilization, Southwest Minzu University, Chengdu 610041;
4. Sichuan Hua Hansan Bio Technology Co., Ltd., Chengdu 610093
牦牛(Bos grunniens)是青藏高原的特有物种,主要分布于我国青海、西藏、四川、甘肃西北部和新疆南部[1],是藏区牧民的主要经济来源。牦牛肉的干物质、蛋白及矿物质含量均高于黄牛,且脂肪含量低于黄牛,是一种高蛋白质、低脂肪、富含矿物质的优质肉类资源[2]。且因其生长周期长和体型较小,加之高昂的运输成本,售价远高于黄牛。高原地区牧民为提高畜牧业经济效益,常将黄牛与牦牛进行种间杂交,杂交后代称为犏牛。其中以黄牛为父本,牦牛为母本的杂交后代为真犏牛;而牦牛为父本,黄牛为母本产生的杂交后代为假犏牛。相较牦牛,犏牛个体更大发育更快,耕地效率更高,产肉产奶量均优于牦牛;而相较黄牛,犏牛具有更好的高原适应性,能够很好地适应高原恶劣的环境[3-4]。利用犏牛肉制作肉干也是高原牧区获得经济收入的主要方式,对其进行鉴定具有较大的市场需求。
许多不法商家为牟取暴利,利用水牛、黄牛、猪、羊、鸡、兔、鸭、狗等肉类制造“牦牛肉干”以假乱真。据统计,国际市场中假肉干占比15%-39%,而在中国市场假牦牛肉干的比例更是高达80%[5-7]。大量假牦牛肉干的出现,严重扰乱了市场,损害了消费者权益。但假牦牛肉干更大的危害是拉低了牦牛肉类产品的市场价格,扰乱了牦牛产业链,减少了藏区牧民收益。因此,急需要发展一种适合市场和基层使用,结果判定较为简单的方法对牦牛和犏牛肉干进行鉴定。
目前,常用的肉类鉴定方法有蛋白质鉴定和DNA鉴定两类。基于蛋白质特性的鉴定方法主要包括酶联免疫吸附分析(Enzyme-1inked immunosorbnent assay,ELISA)和近红外光谱(Near infrared reflectance,NIR)分析等[8-10]。蛋白分析操作相对简便,但分析混合样品难度较大。且加工烹调会使肉制品失去原有蛋白特性,导致这类方法将无法鉴定肉源成分。因此,基于蛋白质特性的鉴定方法存在一定的局限性。基于DNA序列特异性鉴定的方法主要有普通PCR、[11-12]限制性片段长度多态性PCR(Restriction fragment length polymorphism,PCR-RFLP)[6, 13]、高分辨率熔解(High-resolution melting,HRM)分析[14-15]、荧光定量PCR(Realtime fluorescence quantitative PCR,RTFQ PCR)[16-19]。这些方法灵敏度高,特异性强,且DNA分子结构相对稳定,加工处理的肉制品中也能提取出小片DNA,弥补了蛋白质鉴定方法的不足。但这些要实现高通量鉴定,步骤较为繁琐。多重PCR能在一个体系中同时扩增多条DNA片段,可根据扩增片段大小同时鉴定多种物种,克服了以上方法的缺点,且也被广泛运用于肉源成分鉴定[20-21]。但牦牛与黄牛亲缘关系较近,遗传密码极为相似[22],增加了区分这两个物种的难度。基于国内市场以假乱真的现状,本课题组Lee等,根据不同物种在线粒体12S rRNA上的特异性变异位点,成功设计出了9对特异性引物,利用多重PCR技术,对水牛、黄牛、猪、羊、鸡、兔、鸭、狗、牦牛9个物种进行鉴定[23]。该方法操作难度高,结果读取较复杂,不适于全面推广,且未包含犏牛鉴定方法。
PCR-膜芯片技术是使用多重PCR扩增靶序列,并在尼龙膜上与固化了生物素标记的特异性探针进行杂交,一次杂交即可同时筛查被检DNA中的多种突变。该方法已广泛运用于病原体、基因突变和基因分型的检测[24-26]。目前研究显示,PCR-膜芯片技术可实现多种动物源性成分的鉴定,且该方法快捷准确,操作要求不高[27-28]。但在目前较少应用于物种鉴定。因此,本研究拟在PCR-膜芯片技术的基础上,以期建立包括牦牛及犏牛在内的11种动物源肉制品的鉴定方法。
1 材料与方法 1.1 材料 1.1.1 样品来源鸡、鸭、兔、猪、黄牛鲜肉样品各8份购自成都市洗面桥街农贸市场,牦牛与犏牛样品各8份分别采自红原及香格里拉屠宰场,国内品牌购于淘宝网站,散装肉干样品共19个购于成都市武侯大街某特产批发市场。
1.1.2 主要试剂DNA提取试剂盒(天根)、尼龙膜、SSC、去活化液(100 mmol/L,NaOH)、去活化清洗液(2×SSPE,0.1% SDS)、杂交清洗液(2×SSPE,0.5% SDS)、孵育液(碱性磷酸酶标记的链霉亲和素按1:2 000的比例加入到999.5 μL的孵育液中)、孵育清洗液1(2×SSPE,0.5% SDS)、孵育清洗液2(0.3 mol/L NaCl、20 mmol/L NaH2PO4、20 mmol/L EDTA· Na2,其pH为7.4)、显色液(BCIP/NBT、5-溴-4-氯-3-吲哚磷酸和氯化硝基四氮唑兰)、PCR引物及探针(上海生工合成并纯化)、PCR Master Buffer(100 mmol/L tris-HCl,15 mmol/L MgCl2,其pH为8.0)、dNTP、Taq Polymerase(擎科)。
1.2 方法 1.2.1 PCR引物及探针设计根据物种特异性基因片段,猪的线粒体细胞色素氧化酶亚基基因(Cytochrome oxidase subunit I,COX)、山羊的线粒体细胞色素b基因(Cytochrome b,cyt b)[28]、其他物种的线粒体12S rRNA设计引物和探针,以actin基因作为内参基因。引物和探针的设计由Primer Premier 5.0软件完成。根据膜基因芯片杂交原理,反向引物5'端以生物素标记,探针5'端以Aminolinker(C6)修饰。引物和探针序列见表 1。
去除肉干样品中的油脂和杂质等添加剂,称取大约50 mg肉样,已知鲜肉样品按物种名编号,肉干样品以S01-S21编号。使用DNA提取试剂盒提取并纯化DNA。
1.2.3 多重PCR以待测样品DNA为模板,在20 μL体系中加入Mix酶10 μL、正向引物(20 μmol/L)0.5 μL、反向引物(20 μmol/L)0.5 μL、模板DNA100 ng、ddH2O 8 μL,按以下条件进行扩增:95℃预变性10 min;94℃变性30 s、60℃退火15 s、72℃延伸15 s、33个循环;72℃延伸3 min。得到多重PCR产物。
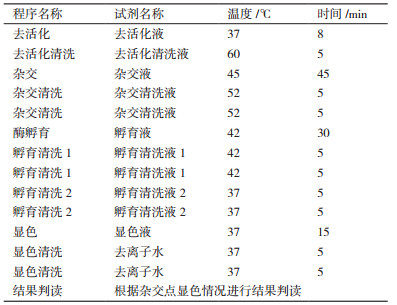
1.2.4 膜芯片制备将尼龙膜裁成1.2 cm×1.8 cm的膜条,将裁好的尼龙膜放置于双蒸水中浸泡15 min,再用15×SSC浸泡15 min,取出放在滤纸上60℃烘干,待尼龙膜降温至室温,按一定顺序点上内参探针(5 μmol/L)、检测探针(5 μmol/L)、阳性探针PC(5 μmol/L)以及阴性探针NC(5 μmol/L)(图 1)。
|
| 图 1 芯片点样模式图 |
(1)PCR产物变性:多重PCR产物95℃ 5 min使其解链,放置冰上备用。(2)膜芯片去活化:将制备好的膜芯片放入杂交盒内,芯片标签正面朝上,加入1 mL去活化液37℃孵育8 min。(3)洗膜:加入1 mL去活化清洗液60℃洗脱5 min。(4)杂交:将变性后的PCR产物与1 mL杂交液充分混匀,放入水平摇床45℃,90 r/min孵育45 min。(5)洗膜:吸除杂交体系液,加入1 mL预热好的杂交清洗液,水平摇床52℃,90 r/min,震荡清洗5 min,洗涤2次。(6)孵育:吸除杂交清洗液,加入预热好的酶孵育液,42℃水平摇床90 r/min,震荡孵育30 min。(7)洗膜:吸除酶孵育液,加入1 mL预热好的孵育清洗液1,42℃水平摇床90 r/min,震荡清洗5 min,洗涤2次;吸除孵育清洗液1,加入1 mL预热好的孵育清洗液2,37℃水平摇床90 r/min,震荡清洗5 min,洗涤2次。(8)显色:吸除孵育清洗液2,加入1 mL显色液,37℃静置15 min,吸除显色液,加入1 mL去离子水37℃洗涤2次,待膜芯片干燥后进行结果判读。具体流程如表 2所示。
将已知物种鲜肉样品通过上述步骤杂交显色,进行特异性评估;使用TE将已知物种DNA样品稀释到10 ng、1 ng、0.1 ng、0.01 ng四个浓度测试膜芯片灵敏度;肉干类样品也通过上述方法杂交显色。显色完成后,膜上内参和PC位点显色,代表此次实验结果有效,将显色位点位置与图 1相对应即可知道该样品肉源成分。
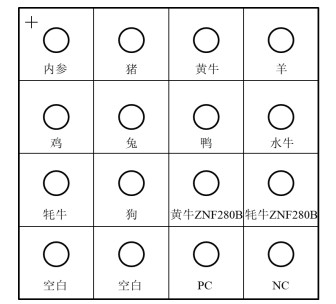
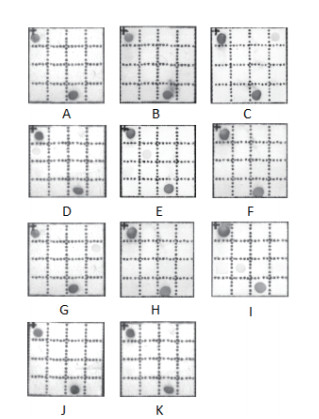
2 结果 2.1 特异性评估分别提取猪、黄牛、山羊、鸡、兔、鸭、水牛、牦牛、狗、真犏牛、假犏牛11种已知样品DNA,通过多重PCR扩增,将扩增产物与膜芯片在杂交盒中杂交显色。结果显示(图 2)11个样品都被准确识别,各阳性杂交点均清晰,无漏检,多检现象,结果说明该芯片特异性强,结果准确,可用于鉴定11种肉类成分。
|
| A:猪;B:黄牛;C:山羊;D:鸡;E:兔;F:鸭;G:水牛;H:牦牛;I:狗;J:真犏牛;K:假犏牛 图 2 特异性评估芯片扫描结果图 |
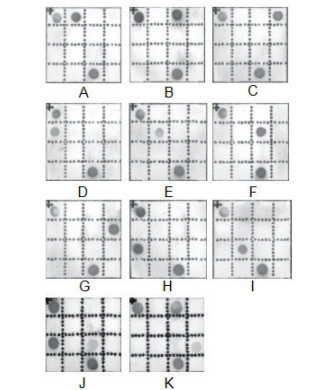
使用TE将猪、黄牛、山羊、鸡、兔、鸭、水牛、牦牛、狗、真犏牛、假犏牛11种已知样品DNA分别稀释至10 ng、1 ng、0.1 ng、0.01 ng四个浓度梯度与膜芯片进行杂交,结果显示(图 3-6),10 ng,1 ng以及0.1 ng核酸均能与探针结合,形成清晰显著的阳性斑点。将核酸稀释至0.01 ng时,只有部分物种芯片杂交结果呈弱阳性;猪,黄牛,鸭,牦牛,真犏牛,假犏牛杂交结果均呈阴性。为确定本实验的准确检出范围,我们使用0.1 ng核酸进行了60次平行实验,其检测结果均为阳性。结果说明膜芯片灵敏度较高,能准确检测0.1 ng及以上浓度的微量核酸。

|
| A:猪;B:黄牛;C:山羊;D:鸡;E:兔;F:鸭;G:水牛;H:牦牛;I:狗;J:真犏牛;K:假犏牛 图 3 灵敏度测试10 ng核酸芯片扫描结果图 |

|
| A:猪;B:黄牛;C:山羊;D:鸡;E:兔;F:鸭;G:水牛;H:牦牛;I:狗;J:真犏牛;K:假犏牛 图 4 灵敏度测试1 ng核酸芯片扫描结果 |
|
| A:猪;B:黄牛;C:山羊;D:鸡;E:兔;F:鸭;G:水牛;H:牦牛;I:狗;J:真犏牛;K:假犏牛 图 5 灵敏度测试0.1 ng核酸芯片扫描结果图 |
|
| A:猪;B:黄牛;C:山羊;D:鸡;E:兔;F:鸭;G:水牛;H:牦牛;I:狗;J:真犏牛;K:假犏牛 图 6 灵敏度测试0.01 ng核酸芯片扫描结果图 |
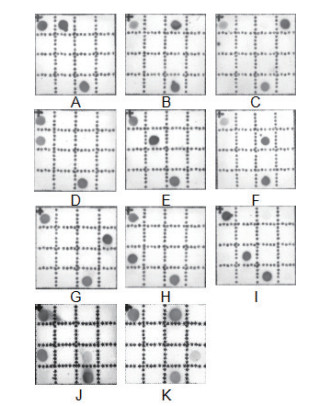
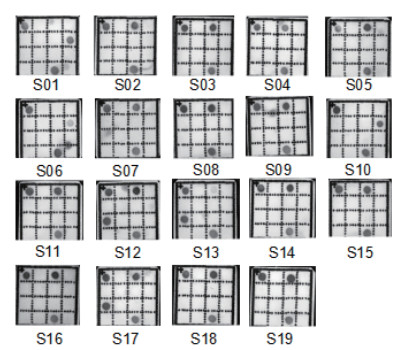
分别将S01-S19 19个待测样品DNA通过多重PCR扩增,将扩增产物与膜芯片杂交,并根据阳性斑点位置鉴定肉干肉源成分。检测结果显示(图 7),样品S01、S06、S10为水牛,样品S02、S03、S04、S05、S07、S08、S09、S11、S14、S15、S16、S18、S19为黄牛,样品S12、S13、S17为牦牛混黄牛。其中S01-S06为散卖风干牦牛肉干,S07-S19为袋装牦牛肉干。结果说明,因散卖牦牛肉干无法确定其商家及厂址,因而掺假比例高达100%,且更偏向于使用口感更差,价格较低的水牛肉造假;而袋装产品,因其注册有公司,所以假货相较散卖产品稍少,即使使用牦牛肉,也混有部分黄牛肉属于真假掺半型造假。
|
| 图 7 肉干样品检测结果图 |
青藏高原因其独特的地理环境,孕育了许多独特的物种,牦牛就是其中之一,有“高原之舟”的美誉。同时,牦牛也是藏区牧民最主要的经济来源。而牦牛肉干更是因其特有的风味口感享誉全球,但是市场大量的掺假牦牛肉干,拉低了牦牛价格,扰乱了牦牛产业链。因此,亟待开发一种专门针对牦牛肉鉴定的方法,本课题组在此前已研发的多重PCR鉴定基础上开发出了一种方便快速准确鉴定牦牛及犏牛的方法。
线粒体DNA(mt DNA)分子结构简单,基因成分相对稳定,拷贝数量大,灵敏度高,进化速度快。同时,其具有较高的种间多样性以及较低的种内变异。虽然黄牛与牦牛12S rRNA序列具有极高的相似性,但通过比对发现,牦牛mtDNA第1168位点特异性的插入有T碱基,且其他物种12S rRNA内也同样存在特异性位点。因此,根据各物种12S rRNA序列上的特异位点设计引物,用以区分各物种。由于鉴定物种种类较多,为了减小引物竞争,避免形成引物二聚体,我们将2对通用引物作为上游引物;同时为了提高PCR产物与探针的特异性杂交效果,我们根据猪的线粒体细胞色素氧化酶亚基Ⅰ基因(Cy-tochrome oxidase subunit I,COX I)及山羊线粒体细胞色素b基因(cytochrome b,cyt b)重新设计了2对引物,替换了猪和山羊的12S rRNA引物。
本课题组在多重PCR的基础上进行了改进,将多重PCR与膜芯片相结合,利用反向斑点杂交技术增加了结果的可视性,直接读取阳性杂交斑点即可识别检测样品物种,特别适合基层单位使用本技术进行鉴定。由于之前的设计使用了通用引物,不可避免的产生了一些非特异性产物,而将多重PCR产物与探针杂交,避免了非特异性引物对结果读取的影响。我们的多重PCR引物是根据各物种线粒体12S rRNA设计,能够很好地将牦牛与其他物种区分。但由于线粒体为母系遗传,故真犏牛线粒体与母本牦牛一致,假犏牛线粒体与母本黄牛一致。因此,使用此方法会将真犏牛误判为牦牛,而将假犏牛误判为黄牛从而导致误判。为规避这个问题,我们在探针上做了调整,虽然犏牛线粒体遗传于母本,但是常染色体却是来自母本和父本双方。我们通过比对牦牛和黄牛核基因ZNF280B的差异,根据其特异序列额外设计了一对特异引物,并根据扩增出的特异片段增加了牦牛ZNF280B和黄牛ZNF280B两个探针。若牦牛线粒体呈阳性,黄牛ZNF80B呈阳性,牦牛ZNF80B为阴性或阳性,则为真犏牛;若黄牛线粒体呈阳性,牦牛ZNF80B呈阳性,黄牛ZNF80B为阴性或阳性,则为假犏牛。经过实验验证,该方法成功解决了犏牛误判的问题。
4 结论通过对膜芯片特异性的评估和灵敏度的测试,充分说明PCR-膜芯片技术具有特异性强,灵敏度高的特性。利用PCR-膜芯片技术能够快速,准确的鉴定样品肉类物种。而通过对市场上售卖的牦牛肉干的鉴定,检测结果与文献报道相符,80%以上产品均为假牦牛肉干。因此,PCR-膜芯片快速鉴定牦牛及犏牛技术的推广能打击牦牛及犏牛肉干制假现象,保护消费者的权益。同时,对假牦牛肉干的打击有利于抬高牦牛肉售价,保护我国独特的肉类资源,恢复被扰乱的牦牛产业链。
| [1] |
Cai L, Wiener G. The yak[J]. Ccpa Monitor, 1995, 44(4): 57-58. |
| [2] |
张群英, 郝力壮, 刘书杰, 等. 不同地区成年牦牛肉营养成分比较[J]. 食品工业科技, 2018, 39(1): 302-307. |
| [3] |
吴周林, 左玲, 徐弘扬, 等. 犏牛杂种优势研究进展[J]. 当代畜牧, 2018(6): 23-25. |
| [4] |
付永, 魏雅萍, 孟茹. 牦牛、犏牛和黄牛生产性能、肉营养品质及风味物质的比较[J]. 动物营养学报, 2013, 25(11): 2734-2740. DOI:10.3969/j.issn.1006-267x.2013.11.029 |
| [5] |
Ballin NZ. Authentication of meat and meat products[J]. Meat Science, 2010, 86(3): 577-587. DOI:10.1016/j.meatsci.2010.06.001 |
| [6] |
Chen SY, Liu YP, Yao YG. Species authentication of commercial beef jerky based on PCR-RFLP analysis of the mitochondrial 12S rRNA gene[J]. Journal of Genetics and Genomics, 2010, 37(11): 763-769. DOI:10.1016/S1673-8527(09)60093-X |
| [7] |
Wang Y, He K, Fan L, et al. DNA barcoding reveals commercial fraud related to yak jerky sold in China[J]. Science China Life Sciences, 2016, 59(1): 106-108. DOI:10.1007/s11427-015-4979-0 |
| [8] |
水小溪, 蔡乐, 赵宝华. ELISA技术在食品安全检测中的应用[J]. 生命科学仪器, 2008, 6(10): 51-54. DOI:10.3969/j.issn.1671-7929.2008.10.014 |
| [9] |
马永征, 马冬, 白娣斯, 等. 免疫学检测肉类制品掺假研究进展[J]. 肉类研究, 2012(9): 26-29. |
| [10] |
杨志敏, 丁武. 近红外光谱技术快速鉴别原料肉掺假的可行性研究[J]. 肉类研究, 2011, 25(2): 25-28. DOI:10.3969/j.issn.1001-8123.2011.02.007 |
| [11] |
刘国强, 海小, 罗建兴, 等. PCR技术鉴定肉品中鸭源性成分的方法研究[J]. 中国家禽, 2018, 40(24): 58-60. |
| [12] |
徐慧, 马慧娟. 动物源性食品鸭血中鸭成分普通PCR检测方法探究[J]. 食品安全导刊, 2018(8): 60-61. |
| [13] |
冯海永, 刘丑生, 何建文, 等. 利用线粒体DNACyt易基因PCR-RFLP分析方法鉴别羊肉和鸭肉[J]. 食品工业科技, 2012, 33(13): 319-321. |
| [14] |
Chizuru A, Mitsuya M, Naoki A, et al. High-resolution melting(HRM)analysis of the Cu/Zn superoxide dismutase(SOD1)gene in Japanese sporadic amyotrophic Lateral Sclerosis(SALS)patients[J]. Neurol Res Int, 2011. DOI:10.1155/2011/165415 |
| [15] |
Naue J, Hansmann T, Schmidt U. High-resolution melting of 12S rRNA and cytochrome b DNA sequences for discrimination of species within distinct European animal families[J]. PLoS One, 2014, 9: e115575. DOI:10.1371/journal.pone.0115575 |
| [16] |
蒲长宇, 安得霞, 王戊腾, 等. 基于实时荧光PCR定量检测肉制品猪源性成分方法的建立[J]. 基因组学与应用生物学, 2017, 36(11): 4585-4591. |
| [17] |
周彤, 李家鹏, 李金春, 等. 一种基于多重实时荧光聚合酶链式反应熔解曲线分析的肉及肉制品掺假鉴别方法[J]. 食品科学, 2017, 38(12): 217-222. DOI:10.7506/spkx1002-6630-201712033 |
| [18] |
郭梁, 郭元晟, 钱俊平, 等. 基于TaqMan实时荧光PCR检测肉制品中羊源性成分[J]. 江苏农业学报, 2018, 34(4): 938-942. DOI:10.3969/j.issn.1000-4440.2018.04.032 |
| [19] |
徐淑菲, 孔繁德, 苗丽, 等. 鸡鸭源性成分荧光LAMP检测方法的建立及应用[J]. 中国动物检疫, 2018, 35(2): 77-81. DOI:10.3969/j.issn.1005-944X.2018.02.021 |
| [20] |
段庆梓, 尚柯, 张玉, 等. 多重PCR法用于鸡、鸭肉源性的鉴定[J]. 食品研究与开发, 2014(5): 90-93. DOI:10.3969/j.issn.1005-6521.2014.05.026 |
| [21] |
王金斌, 白蓝, 李文, 等. 同步检测动物源性成分的五重PCR的条件优化和检出限分析[J]. 核农学报, 2018, 32(3): 506-514. |
| [22] |
马云, 于波, 徐永杰, 等. 中国部分地方牛种mtDNA D-loop区全序列的遗传多样性与系统进化分析[J]. 信阳师范学院学报:自然科学版, 2012, 25(2): 202-205. |
| [23] |
Lee JN, Jiang M, Wen Y, et al. Multiplex assay for identifying animal species found in the tibetan area using the mitochondrial 12S rRNA gene[J]. Animal Biotechnology, 2017, 29(1): 1. |
| [24] |
Hidenobu S, Yoji K, Kikuya K. HLA genotyping by next-generation sequencing of complementary DNA[J]. BMC Genomics, 2017, 18(1): 914. DOI:10.1186/s12864-017-4300-7 |
| [25] |
张秀娟. PCR斑点杂交技术在肺炎支原体感染检测中的应用价值[J]. 江苏医药, 2016(24): 12-15. |
| [26] |
彭陆衡. 基于PCR-RDB技术的膜芯片开发并应用于遗传性非综合征耳聋检测[J]. 泰山医学院学报, 2017, 38(8): 935-936. DOI:10.3969/j.issn.1004-7115.2017.08.036 |
| [27] |
石丰运, 缪建锟, 等. 运用基因芯片技术检测牛、山羊、猪和鸡源性成分[J]. 生物工程学报, 2010, 26(6): 823-829. |
| [28] |
朱业培, 王玮, 等. 基于基因芯片技术检测6种动物源性成分[J]. 南京农业大学学报, 2015, 38(6): 1003-1008. |