2. 四川农业大学农学院,成都 611130;
3. 贵州省烟草科学研究院,贵阳 550081
2. College of Agronomy, Sichuan Agricultural University, Chengdu 611130;
3. Guizhou Academy of Tobacco Sciences, Guiyang 550081
转录因子是一类能够调控下游基因转录的关键DNA结合蛋白,它能够特异地结合在真核生物基因的启动子顺式作用元件区域,从而激活或者抑制基因的表达[1]。高等植物中,有众多的转录因子,其中,AP2/ERF类转录因子较为古老,为植物所特有且植物中最大的一类,。从结构上看,AP2/ERF类转录因子均含有1-2个高度保守的AP2结构域,含有1个结构域的为ERF亚家族,含有2个结构域的为AP2亚家族[2]。
AP2/ERF家族转录因子在植物的生命活动过程中发挥着十分重要的作用,参与了细胞增殖、花器管发育、植物次生代谢、植物激素应答等重要过程[3-6]。同时,越来越多的研究证明,AP2/ERF类转录因子还参与了干旱、高渗、低温、高盐等非生物胁迫和抵御微生物侵染等生物胁迫响应[3-4, 7-13]。正因为该类转录因子的多重功能,所以其在植物中的数量巨大。随着植物基因组测序工作的不断推进,以及生物信息学技术的不断进步与完善,极大地促进了AP2/ERF转录因子超级家族的研究工作。目前,模式植物拟南芥、水稻中,分别鉴定出了122、139个AP2/ERF转录因子[1];在玉米中有167个AP2/ERF转录因子[14];在辣椒中发现了123个成员[15];在葡萄、番茄、马铃薯中分别鉴定出了149、167、246个AP2/ERF类转录因子[16]。
花烟草是属于茄科烟草属的一年生草本观赏花卉,其花色种类繁多、花期较长,是优美的花坛、花镜材料,是我国许多城市的重要的绿化美化材料。研究其ERF转录因子的定位、表达模式及功能等,对于探索花烟草的生长发育的机制具有重要意义。因此,本研究利用同源克隆的方法,克隆了一个花烟草的ERF基因,采用生物信息方法对该基因进行了鉴定,并分析了其组织表达及响应非生物胁迫的表达模式,以期为花烟草ERF基因的功能研究奠定基础。
1 材料与方法 1.1 材料以花烟草(Nicotiana alata)为试验材料。
1.2 方法 1.2.1 植物材料的培养及非生物胁迫处理材料培养:挑选籽粒饱满且大小一致的种子,用次氯酸钠溶液表面消毒后,用无菌水清洗数次,随后播种于MS培养基上,并置于恒温培养室中生长(光照:16 h光/8 h暗;温度:25℃)。
非生物胁迫处理及取材:种子萌发后,在MS培养基上生长至2片真叶时,取10株幼苗,在液氮中速冻,用于目的基因的克隆。同时,挑选长势一致的烟苗,分别进行低温(4℃)、干旱(5% PEG6000)、高盐(200 mmol/L)、低钾(10 μmol/L)、ABA(1 μmol/L)及H2O2(10 mmol/L)处理。进行低温处理时,将烟苗置于4℃光照培养箱中;进行其它非生物胁迫处理时,将烟苗分别移至含有5% PEG6000、200 mmol/L Nacl、10 μmol/L钾、1 μmol/L ABA及10 mmol/L H2O2的MS培养基上,然后置于25℃光照培养箱中。每个处理设3个重复,每重复15株烟苗。在处理后0 h、3 h、6 h、12 h、24 h后,分处理、重复进行整株取样,并置于液氮中保存,用于总RNA的提取。
低钾MS培养基(钾离子终浓度为10 μmol/L)的配制,参照张雪微等方法进行[17]。
此外,采用上述种子处理方法,将种子消毒后,播种于塑料盆中进行培养。待烟苗发育到4叶1心时,分别取烟苗的根、茎、叶及开花期的烟花,保存于液氮中,用于目的基因的组织表达分析。
1.2.2 目的基因克隆总RNA的提取及反转录:采用Trizol法提取烟苗的总RNA[18],然后,参照cDNA合成试剂盒的方法,进行反转录及cDNA合成。
目的基因的克隆:参照植物的ERF基因序列,利用Primer Premier 5软件设计兼并PCR引物。在预试验的基础上,设计特异引物进行PCR扩增。引物序列如下:ERF1-F:5'-GCCCATGGAACAAGAACAAGTATATGA-3',ERF1-R:5'-GCCCTAACTAGGACAAAACATTTG-3'。以合成的cDNA为模板进行PCR扩增,反应程序为:98℃ 5 min;94℃ 30 s;53℃ 30 s;72℃ 1 min;35个循环;72℃ 10 min;12℃终止。PCR反应体系为(25 μL):cDNA模板1 μL,Taq Buffer(10x)5 μL,2.5 mol/L dNTP 2 μL,正反引物各1 μL,Taq酶1μL,ddH2O 14 μL。PCR反应结束后,将产物进行凝胶电泳,回收并纯化目的片段,连接pEASY-Blunt Simple克隆载体,转化Trans1-T1感受态细胞,在含有氨苄青霉素抗性平板上筛选,挑取阳性单克隆进行菌液PCR验证后,送至上海生工生物工程技术有限公司测序。
1.2.3 生物信息学分析利用DNAMAN 8软件对测序正确的序列进行分析,并翻译成氨基酸序列。用ProtParam(http://web.expasy.org/protparam/)对其基本理化性质进行分析,用ProtScale(http://web.expasy.org/protscale/)预测蛋白亲/疏水性。用CDD(http://smart.embl-heidelberg.de/)预测蛋白结构域。用Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)进行亚细胞定位分析,用NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)预测磷酸化位点,用SOPMA(https://npsa-prabi.ibcp.fr/)和Phyre2分别进行目的蛋白的二级结构和三级结构预测;利用MEGA7软件构建系统进化树。
1.2.4 目的基因的组织表达及响应非生物胁迫的诱导表达分析引物设计:采用Primer Premier 5软件,进行引物设计,用于目的基因的组织表达及响应非生物胁迫的诱导表达分析。正向引物:ERF1-qF:5'-GCCTCTGAACCTATTGCC-3',反向引物:ERF1-qR:5'-TGCCACGATAAAGGACTG-3';内参为18S rRNA,引物序列为18S-F:5'-CCTACGCTCTGTATACATTAGC-3';18S-R:5'-GTGTTGAGTCAAATTAAGCCGC-3'。
目的基因的组织表达及受诱导表达分析:按上述方法提取样品的总RNA,并进行反转录合成cDNA。以花烟草18S rRNA为内参,以cDNA为模板,采用qRT-PCR的方法,进行目的基因片段的PCR扩增。目的基因相对表达量的计算,采用2-△△Ct的方法进行,并利用EXCEL软件进行统计分析及做图。
2 结果 2.1 目的基因的克隆以烟草幼苗为材料提取总RNA,反转录后进行PCR扩增,用1%琼脂糖凝胶电泳,在750 bp-1 000 bp之间有一条清晰明亮的条带(图 1)。条带经回收纯化后送样测序。将测序结果用DNAMAN8进行分析并翻译成蛋白序列。同时,在NCBI网站上分析其ORF序列。结果显示,该基因ORF长度为819 bp,编码272个氨基酸。
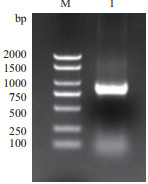
|
| 注:M:DNA Marker;1:目的基因片段 图 1 NaERF1基因克隆 |
在NCBI上,用BLAST对目的基因的CDS序列进行比对,结果表明该基因对多个物种的ERF基因同源性较高。于是,将目的基因命名为NaERF1。
2.2 NaERF1蛋白的生物信息学分析 2.2.1 理化性质分析利用Protparam(http://web.expasy.org/protparam/)对NaERF1蛋白的理化性质进行了分析,结果显示,NaERF1编码272个氨基酸,该蛋白的分子式为C1339H2064N376O431S13;分子量为30 742.16 Da;理论pI值:6.07;脂肪系数为57.72,带负电荷的残基(Asp+Glu)总数为31个,带正电荷的残基(Arg+Lys)总数为27个。NaERF1蛋白的平均亲水系数(GRAVY)为-0.659,在该蛋白氨基酸组成中丝氨酸(Ser)和苏氨酸(Thr)含量较高,分别占氨基酸总数的10.7%和9.2%;半胱甘酸(Cys)和色氨酸(Trp)含量较低,分别占氨基酸总数的1.5%和1.8%。
2.2.2 亲/疏水性分析利用ExPaSy网站的ProtScale(http://web.expasy.org/protscale/),选择Hphob./Kyte & Doolittle算法,对NaERF1蛋白的亲/疏水性进行了分析,结果如图 2所示。NaERF1蛋白序列中第13位氨基酸最高分值为1.144,第266位氨基酸最低分为-2.322。总体上看,亲水性氨基酸均匀分布在整个肽链中,且平均分值大于疏水性氨基酸,可推测NaERF1蛋白属于亲水性蛋白。

|
| 图 2 NaERF1蛋白亲/疏水性分析 |
采用ExPASy提供的TMHMM Server(http://www.cbs.dtu.dk/services/TMHMM/)软件,在线对NaERF的跨膜区进行预测,结果如图 3所示。预测结果表明,NaERF1蛋白没有跨膜区,不属于跨膜蛋白。
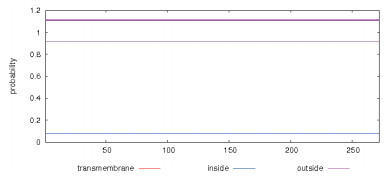
|
| 图 3 NaERF1蛋白跨膜区分析 |
利用在线软件Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/),选用Plant-mPLoc,对NaERF1蛋白进行了亚细胞定位分析。结果显示,该蛋白位于细胞质的可能性为41.5%,位于过氧化物酶体(Peroxisome)的可能性为28%,位于线粒体基质(Mitochondrial matrix space)的可能性为10%。因此,推测该蛋白可能定位于细胞质中。
2.2.5 磷酸化位点预测利用在线软件NetPhos3.1(http://www.cbs.dtu.dk/services/NetPhos/),对NaERF1蛋白进行磷酸化位点预测,结果如图 4所示。NaERF1蛋白含有21个丝氨酸(Ser)位点,9个苏氨酸(Thr)位点,4个酪氨酸(Tyr)位点,这些位点均有可能是磷酸化位点。由此说明,NaERF1蛋白能被激酶所磷酸化,从而参与花烟草的各种生理生化过程。

|
| 图 4 NaERF1蛋白磷酸化位点预测 |
通过CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)网站,对NaERF1蛋白进行了保守功能域预测。结果(图 5)显示,NaERF1蛋白的第22-83位氨基酸,存在一个典型的ERF转录因子保守结构域(AP2)。表明NaERF1蛋白属于植物的AP2/ERF家族,推测能够行使植物AP2/ERF基因的功能。

|
| 图 5 NaERF1蛋白功能域的预测 |
通过SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)软件,对NaERF1蛋白信号肽进行预测,结果(图 6)显示,该蛋白不含有信号肽。
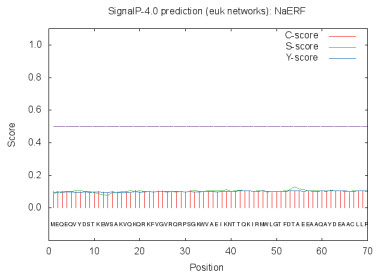
|
| 图 6 NaERF1蛋白的信号肽预测 |
利用SOPMA在线软件,对NaERF1蛋白的二级结构进行了预测,结果如图 7所示。NaERF1蛋白主要由51.10%的无规卷曲(random coil,139个氨基酸参与),38.97%的α-螺旋(alpha helix,106个氨基酸参与),7.72%的延伸链(extended strand,21个氨基酸参与),2.21% β-转角(beta turn,6个氨基酸参与)所构成。同时,利用Phyre2软件预测了NaERF1蛋白的三级结构。
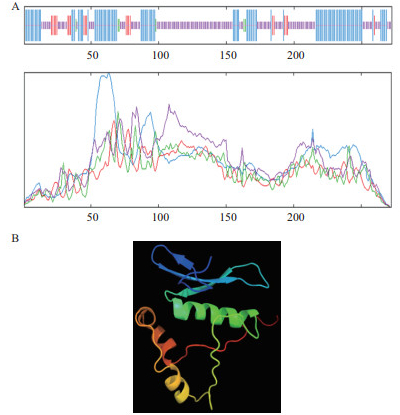
|
| A:二级结构预测;B:三级结构预测 图 7 NaERF1蛋白的二级结构与三级结构预测 |
利用NCBI Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)在线工具,在数据库中,搜索并选取与NaERF1序列相似度大于70%以上的37个同源基因,采用MEGA7软件,选用邻位相连法、Bootstrap method选择1000进行计算,构建了系统发育树(图 8)。从分析的结果可以看出,NaERF1与茄科植物的ERF基因亲缘关系较近,而与其它植物的ERF基因亲缘关系较远。其中,同源性最高的为普通烟草(Nicotiana tabacum)的ERF基因(XP 016476774.1),同源性最低的为橡胶树的ERF基因(XP 021640270.1)。
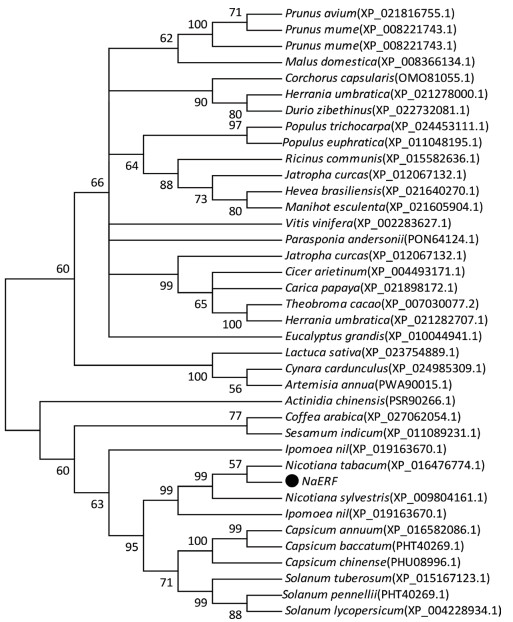
|
| 图 8 NaERF1蛋白的同源性分析 |
采用qRT-PCR的方法,对NaERF1在花烟草不同组织与器官中的表达量进行了分析。结果(图 9)显示,NaERF1基因的表达具有组织特异性,其在花中表达量最高,茎中次之,根和叶中表达量较低。该结果说明,NaERF1广泛参与了花烟草的生理生化过程,并有可能主要在花中发挥作用。

|
| 图 9 NaERF1在花烟草不同组织器官中的表达分析 |
为了探究NaERF1在花烟草适应非生物逆境中的作用,分析了NaERF1在PEG、高盐、低钾、低温、ABA和H2O2处理下,0 h、3 h、6 h、12 h及24 h的表达情况。结果(图 10)显示,NaERF1基因的表达对这6种非生物胁迫均有所响应。但其响应表达的模式,有明显不同。例如,随着胁迫时间的推移,对低温的响应呈现出先降低后升高的趋势(图 10-A);对低钾及ABA呈现先升高后降低的模式(图 10-B-C);对PEG胁迫表现为先升高再降低再升高(图 10-D);对H2O2的表现为先降低再升高又降低(图 10-E);而对高盐胁迫,NaERF1的表达则表现为持续降低(图 10-F)。本结果表明,NaERF1可能参与了花烟草对干旱、低温等非生物胁迫的响应过程。

|
| 图 10 NaERF1在非生物胁迫下的表达模式 |
植物ERF家族基因,不但数量众多,而且所行使的功能也较为复杂,例如参与植物的生长发育以及生物与非生物胁迫的应答反应等[2-4, 16, 19-20]。其功能的行使,依靠其保守的AP2/ERF结构域,该结构域由大约60个氨基酸所组成[16]。本研究所克隆的NaERF1基因所编码的蛋白,也包含了一个AP2/ERF结构域。所以,可以推测,NaERF1隶属于花烟草的ERF家族,能够行使植物ERF家族基因所具有的功能。
在拟南芥[1]、水稻[1]、玉米[14, 21]、辣椒[15]及普通烟草[20]上的研究表明,植物的ERF基因,不但成员众多,而且其亚细胞定位较为复杂,可以在细胞质、细胞核、叶绿体以及线粒体中特异表达。本研究对NaERF1进行的亚细胞定位的预测结果表明,该基因主要定位于细胞质,与前人的研究结果相一致。此外,NaERF1基因具有组织表达特异性,在花中的表达量最高,在茎、根和叶片中也均有表达。该结果与普通烟草ERF的组织表达模式较为一致[20]。这些结果暗示,NaERF1可能广泛参与了花烟草的生长发育及其生理进程。
研究表明,植物的ERF基因广泛参与了植物响应非生物胁迫的响应过程[16, 20]。例如,拟南芥的ERF基因,参与了干旱及冷害的胁迫响应[2]。过量表达TERF2能够提高转基因烟草及番茄植株对低温胁迫的耐受性[22]。对水稻而言,过量表达OsAP37[23]、OsERF48[24]及OsERF71[25-26],均能够增强转基因植物的耐旱性;而SERF1则参与了水稻耐盐性的调控,当其功能缺失时,水稻植株对盐胁迫更敏感[27]。番茄的TERF2在水稻中过表达,使得转基因水稻的耐冷性得到了提高[28]。此外,羊草LcDREB3a在拟南芥中的过量表达,可提高转基因植物的抗旱和抗盐能力[29]。本研究分析了NaERF1在PEG、高盐、低钾、低温、ABA和H2O2处理下的表达情况。结果显示,NtaERF1基因的表达,受低钾、ABA的强烈诱导;同时,也在转录水平上对PEG、高盐、低温及H2O2的胁迫有较为明显的响应。具体而言,其响应模式可以分为:(1)先降低后升高型,如在低温胁迫下的表达;(2)先升高后降低型,如在低钾及ABA胁迫下的表达;(3)先升高再降低再升高型,如响应PEG胁迫的表达;(4)先降低再升高又降低型,如响应H2O2的诱导表达;(5)持续降低型,如在高NaCl胁迫下的表达。本研究结果说明,与植物的其它AP2/ERF家族转录因子相类似,NaERF1也可能广泛参与了非生物逆境胁迫的生理响应。其中的调控机制,尚有待于进一步的探索。
4 结论花烟草NaERF1基因所编码的蛋白具有典型的AP2家族保守结构域,属于植物的ERF家族。该基因的表达具有组织特异性,并且响应非生物胁迫的诱导,能够在花烟草非生物胁迫响应中行使相应的功能。
| [1] |
Nakano T, Suzuki K, Fujimum T, et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiology, 2006, 140(2): 411-432. DOI:10.1104/pp.105.073783 |
| [2] |
Sakuma Y, Liu Q, Dubouzet JG, et al. DNA binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration and cold-inducible gene expression[J]. Biochemistry Biophysics Research Communication, 2002, 290(3): 998-1009. DOI:10.1006/bbrc.2001.6299 |
| [3] |
黄泽军, 黄荣峰, 黄大昉. ERF转录因子及其在植物防卫反应中的作用[J]. 植物病理学报, 2004, 34(3): 193-198. DOI:10.3321/j.issn:0412-0914.2004.03.001 |
| [4] |
E1iott RC, Betzner AS, Huttner E, et al. AINTEGUMENTA, an APETALA2-like gene of Arabidopsis with pleiotropic role in ovule development and flora organ growth[J]. Plant Cell, 1996, 8(2): 155-168. |
| [5] |
Jofuku KD, den Boer BG, Van Montagu M, et al. Control of Arabidopsis flower and seed development by the homeotic gene APETALA2[J]. Plant Cell, 1994, 6(9): 1211-1225. |
| [6] |
Zhang G, Chen M, Chen X, et al. Phylogeny, gene structures, and expression patterns of the ERF gene family in soybean(Glycine max L.)[J]. Journal of Experimental Botany, 2008, 59(15): 4095-4107. DOI:10.1093/jxb/ern248 |
| [7] |
Licausi F, Ohme-takage M, Perata P. APETALA2/Ethylene responsive factor(AP2/ERF)transcription factors:mediators of stress responses and developmental programs[J]. New Phytologist, 2013, 199(3): 639-649. DOI:10.1111/nph.12291 |
| [8] |
Dossa K, Wei X, Li DH, et al. Insight into the AP2/ERF transcription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J]. BMC Plant Biology, 2016, 16(1): 171. DOI:10.1186/s12870-016-0859-4 |
| [9] |
Yang Z, Tian L, Brown D, et al. Arabidopsis ERF4 is a transcriptional repressor capable of modulating ethylene and abscisic acid responses[J]. Plant Molecular Biology, 2005, 58(4): 585-596. DOI:10.1007/s11103-005-7294-5 |
| [10] |
Novillo F, Medina J, Sallnas J. Arabidopsis CBF1 and CBF3 have a different function than CBF2 in cold acclimation and define different gene classes in the CBF regulon[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(52): 21002-21007. DOI:10.1073/pnas.0705639105 |
| [11] |
Alonsoblanco C, Gomezmena C, Llorente F, et al. Genetic and molecular analyses of natural variation indicate CBF2 as a candidate gene for underlying a freezing tolerance quantitative trait locus in Arabidopsis[J]. Plant Physiology, 2005, 139(3): 1304-1312. DOI:10.1104/pp.105.068510 |
| [12] |
Yi SY, Kim JH, Joung YH, et al. The pepper transcription factor CaPF1 confers pathogen and freezing tolerance in Arabidopsis[J]. Plant Physiology, 2004, 136(1): 2862-2874. DOI:10.1104/pp.104.042903 |
| [13] |
Zhang G, Chen M, Li LC, et al. Overexpression of the soybean GmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco[J]. Journal of Experimental Botany, 2009, 60(13): 3781-3796. DOI:10.1093/jxb/erp214 |
| [14] |
Zhuang J, Deng DX, Yao QH, et al. Discovery, phylogeny and expression patterns of AP2-like genes in maize[J]. Plant Growth Regulation, 2010, 62(1): 51-58. DOI:10.1007/s10725-010-9484-7 |
| [15] |
Kim S, Park M, Yeom SI, et al. Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species[J]. Nature Genetics, 2014, 46(3): 270-278. DOI:10.1038/ng.2877 |
| [16] |
高春艳, 吴芮, 袁玉, 等. 植物AP2/ERF转录因子及其在非生物胁迫应答中的作用[J]. 江汉大学学报:自然科学版, 2017, 45(3): 236-240. |
| [17] |
张雪薇, 刘仑, 鲁黎明, 等. 烟草NtCBL1基因的克隆、表达载体构建及表达分析[J]. 植物研究, 2017, 37(3): 387-394. |
| [18] |
鲁黎明. 烟草钾转运体基因TPK1的电子克隆及生物信息学分析[J]. 中国农业科学, 2011, 44(1): 28-35. DOI:10.3864/j.issn.0578-1752.2011.01.004 |
| [19] |
高浩, 竺锡武. ERF转录因子研究进展[J]. 现代农业科技, 2017, 8: 130-132. |
| [20] |
任昂彦, 孔英珍. 普通烟草ERF转录因子亚家族成员鉴定及表达模式分析[J]. 中国烟草科学, 2017, 38(1): 18-25. |
| [21] |
史庆玲, 董永彬, 周强, 等. 玉米转录因子ZmERFl的克隆及表达分析[J]. 生物技术进展, 2017, 7(1): 38-42. |
| [22] |
Zhang Z, Huang R. Enhanced tolerance to freezing in tobacco and tomato overexpressing transcription factor TERF2/Le ERF2 is modulated by ethylene biosynthesis[J]. Plant Molecular Biology, 2010, 73(3): 241-249. DOI:10.1007/s11103-010-9609-4 |
| [23] |
Ramegowda V, Basu S, Krishnan A, et al. Rice GROWTH UNDER DROUGHT KINASE is required for drought tolerance and grain yield under normal and drought stress conditions[J]. Plant Physiology, 2014, 166(3): 1634-1645. DOI:10.1104/pp.114.248203 |
| [24] |
Jung H, Chung PJ, Park SH, et al. Overexpression of OsERF48 causes regulation of OsCML16, a calmodulin-like protein gene that enhances root growth and drought tolerance[J]. Plant Biotechnology Journal, 2017, 15(10): 1295-1308. DOI:10.1111/pbi.12716 |
| [25] |
Ahn H, Jung I, Shin SJ, et al. Transcriptional network analysis reveals drought resistance mechanisms of AP2/ERF transgenic rice[J]. Frontiers in Plant Science, 2017, 8: 1044. DOI:10.3389/fpls.2017.01044 |
| [26] |
Lee DK, Jung H, Jang G, et al. Overexpression of the OsERF71 transcription factor alters rice root structure and drought resistance[J]. Plant Physiology, 2016, 172(1): 575-588. DOI:10.1104/pp.16.00379 |
| [27] |
Schmidt R, Caldana C, Mueller- Roeber B, et al. The contribution of SERF1 to root-to-shoot signaling during salinity stress in rice[J]. Plant Signaling & Behavior, 2014, 9(1): e27540. |
| [28] |
Tian Y, Zhang H, Pan X, et al. Overexpression of ethylene response factor TERF2 confers cold tolerance in rice seedlings[J]. Transgenic Research, 2011, 20(4): 857-866. DOI:10.1007/s11248-010-9463-9 |
| [29] |
Peng X, Ma X, Fan W, et al. Improved drought and salt tolerance of Arabidopsis thaliana by transgenic expression of anovel DREB gene from Leymus chinensis[J]. Plant Cell Rep, 2011, 30(8): 1493-1502. DOI:10.1007/s00299-011-1058-2 |




