2. 北京市环境保护科学研究院,北京 100037;
3. 国家地质实验测试中心,北京100037
2. Beijing Municipal Research Institute of Environmental Protection, Beijing 100037;
3. National Research Center for Geoanalyse, Beijing 100037
养殖废水和生活污水的随意排放会导致水体的粪便污染。粪便中含有大量对人体有害的病原微生物,能够引起腹泻和急性肠胃炎等疾病[1]。许多粪源病原体可以在体外环境水体中大量生长繁殖,从而导致病原微生物的水源性传播[2-3]。目前,通常用粪便指示细菌(Fecal indicator bacteria,FIB),如大肠杆菌(Escherichia coli)、粪大肠菌群(Fecal coliforms)等来评估地表水体的粪便污染水平[4]。但有研究表明FIB与粪便污染的关联度并不高[5];并且,FIB的宿主特异性较差,不能提供有关污染源的信息[6]。准确的污染源定位对于污染防治工作至关重要。源于这个需求,各种污染溯源方法逐渐发展起来。微生物溯源(Microbial source tracking,MST)技术出现在20世纪后期,主要用于区分粪便污染的宿主来源[7]。因饮食结构和消化系统不同,不同动物消化道内的微生物表型或者基因型之间存在差异[8],MST技术即利用这种差异建立污染源指示物与动物宿主之间的特异性关系,进而来判断粪便污染源。近年来,MST技术以其客观性、低成本、检测速度快和结果准确等优点受到普遍关注[9-10]。美国环境保护署(U.S. Environmental prote-ction agency,EPA)已将MST技术应用于最大日负荷总量(Total maximum daily load,TMDL)方案制定过程中[11]。尽管如此,MST技术在实际应用过程中仍然存在地区差异性和标准化等问题。
目前有多种源指示微生物可用于MST,如双歧杆菌(Bifidobacterium)和拟杆菌(Bacteroides)等,但合适的源指示微生物及其相关检测方法的选择是开展地表水环境粪便污染溯源的关键。根据检测通量的不同,可将MST方法分为单源指示物法和多源指示物法两类。本文围绕两大类MST方法,重点综述了不同类型的指示微生物及其常用的检测方法,包括原理、优缺点等,并简要总结了MST技术在TMDL方案制定过程中的实际应用以及讨论了MST技术目前所面临的问题与挑战,最后对MST技术的发展趋势进行展望,以期促进MST技术在我国的应用,为我国地表水质管理工作提供支持。
1 MST基本原理由于不同动物饮食结构、消化系统及体内环境条件(如pH、营养条件)等的不同,经过长期的协同进化,特定微生物类群在相同物种宿主肠道中通常具有类似或相同的基因型或表型,而在不同物种宿主肠道中的基因型或表型存在差异。基于该原理,MST技术通过建立特定粪便污染指示物与动物宿主间的特异关联来判定粪便污染来源[12]。MST所检测的粪源指示微生物通常应符合以下几个特征:(1)宿主特异性;(2)在体外条件下难以繁殖;(3)一定的环境耐受性;(4)浓度足以检测[12]等。常用的粪便污染源指示物有双歧杆菌(Bifidobacterium)、拟杆菌(Bacteroides)、脆弱拟杆菌噬菌体(Bacteroides fragilis bacteriophage)、大肠杆菌噬菌体(F+ specific RNA coliphages)和人类肠道病毒(Human enteric viruses)等[13-14]。
目前,MST方法主要用于追踪地表水体的粪便污染来源。根据所检测指示物通量的不同,可分为单源指示物法和多源指示物法两大类。单源指示物法通常通过检测单一生物标记来判定污染来源;而多源指示物法则通过分析微生物群落或多种生物标记同时鉴别多个不同的污染源。
2 MST方法 2.1 单源指示物法 2.1.1 细菌指示物法 2.1.1.1 粪便大肠菌群(Fecal coliform,FC)/粪便链球菌(Fecal streptococcus,FS)法由于人类粪便中有较高数量的FC,动物粪便中含有较高数量的FS,所以,早在20世纪60年代末至70年代,有学者提出用FC与FS的比值来指示污染源。他们认为当“4 < FC/FS”时表示主要是人源粪便污染,当“FC/FS < 0.7”时表示主要是其他动物粪便污染[15-16]。之后该方法被细化为,当“4 < FC/FS”时表示主要是人源粪便污染;当“0.1 < FC/FS < 0.6”时表示主要是畜禽养殖排泄物造成的污染,而当“FC/FS < 0.1”时表示主要是野生动物粪便污染[17]。该方法能较快地产生结果,而且不需要很强的专业技能,但是由于FC和FS在环境水体中的生长速度和耐受性都不相同,并且两者的比值极易受环境中的各个因素如雨水或者工业污水的排入等的影响[18],加之大肠杆菌在环境中也是普遍存在的[19],所以FC/FS法难以准确区分人类以及其他动物的粪便污染源。
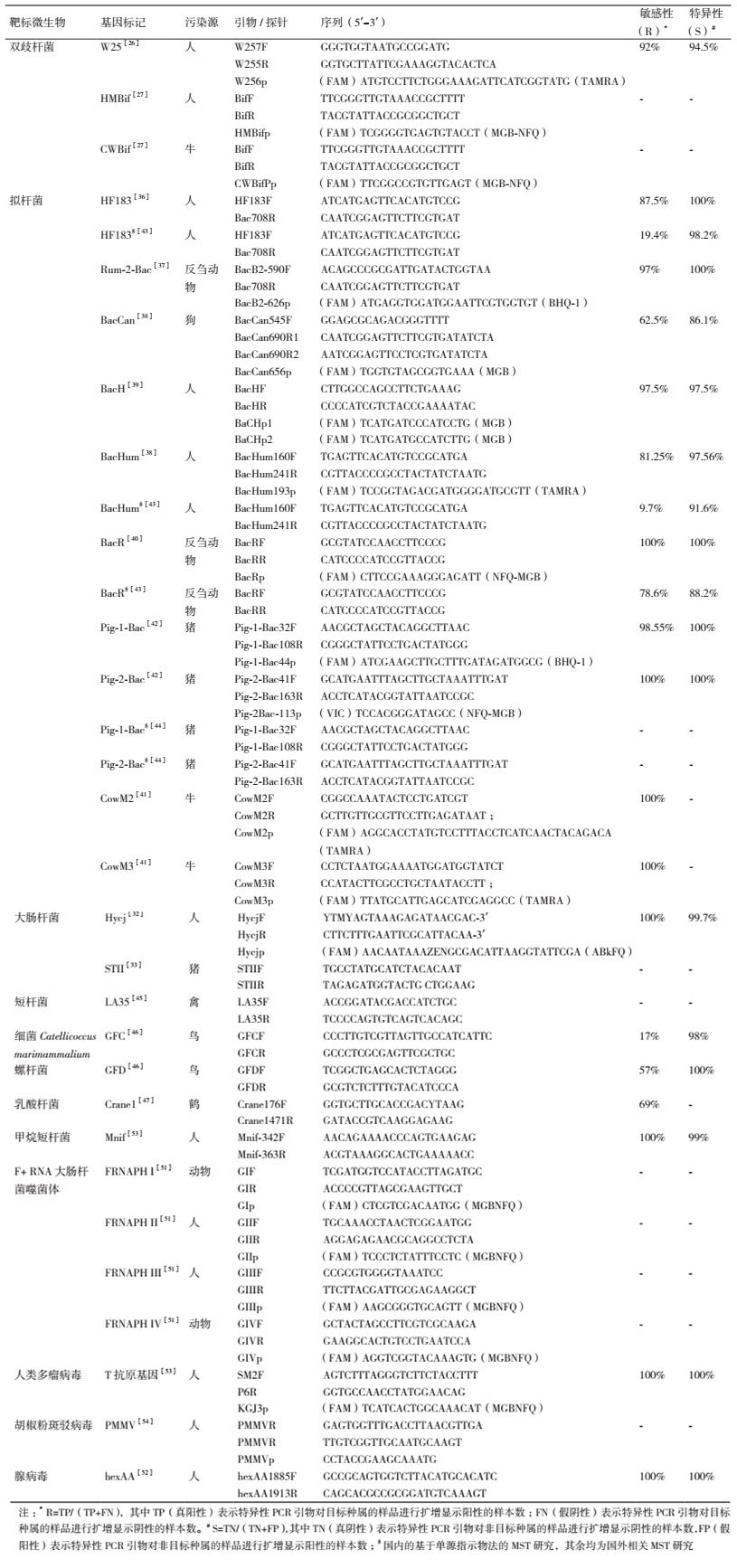
2.1.1.2 双歧杆菌(Bifidobacterium)法双歧杆菌为专性厌氧菌,在人类肠道内的浓度很高,并且在环境中难以繁殖[20],所以通常不会出现在无污染的环境水体中。有些山梨醇发酵双歧杆菌(Sorbitol-fermenting Bifidobacteria,SFB)(如B. adolescentis和B. breve)可作为人类粪便污染的指示物[21-22]。SFB可通过用含有山梨醇的琼脂培养基进行培养鉴定[23],但有研究表明基于SFB培养的MST方法的特异性并不高[24]。相比之下,利用不依赖于培养的分子检测方法,主要是基于PCR的方法,能够区分不同宿主类型中的不同双歧杆菌基因型。例如,Balleste等[25]发现在人类、禽类和牛的粪便样品中存在不同类型的双歧杆菌16S rRNA基因片段且可作为宿主特异性基因标记来指示特定来源的粪便污染。一些研究还建立了基于宿主特异性双歧杆菌16S rRNA基因标记的qPCR方法[26-27](表 1),能够进一步定量区分污染来源。
大肠杆菌为杆状的革兰氏阴性细菌,主要存在于恒温动物的肠道内,代谢类型为异养兼性厌氧型,可通过粪便或者生活污水进入环境[18]。长期以来,环境水域中的大肠杆菌被认为是粪便污染的指示微生物,并被EPA推荐作为淡水中的FIB[11]。大肠杆菌普遍存在于人和动物肠道内,无法简单地通过培养的方法区分粪便污染源,需要结合抗生素抗性分析技术、免疫学方法或DNA指纹图谱(如PFGE、rep-PCR、核糖体分型等)等技术以区分动物和人类的粪便污染[13, 21, 28]。但因携带于质粒上的抗生素抗性基因易受环境或培养条件影响而丢失、需要较大的抗血清数据库或DNA指纹图谱数据库等缺点[29-31],基于大肠杆菌培养的溯源方法已很少使用。近年来多采用分子方法检测大肠杆菌宿主特异性基因标记来鉴别粪便污染源,简化了操作步骤[32]。例如,Khatib等[33]以大肠杆菌的STII毒力基因作为猪源粪便污染的指示物,采用PCR方法可有效地检测环境水体中的猪源粪便污染。
2.1.1.4 拟杆菌(Bacteroides)法拟杆菌是肠道中的主要专性厌氧菌群,因其数量多、特异性较强且在环境中难以繁殖[34],许多研究者推荐以拟杆菌作为MST的源指示微生物。一些拟杆菌物种如脆弱拟杆菌(Bacteroides fragilis)因只存在于人类粪便中且浓度很高,故可作为人源粪便污染的指示微生物[21]。B. fragilis主要通过培养的方法进行检测以鉴别人源粪便污染,不过有研究发现从猪粪便中也能够分离培养出B. fragilis[35]。此外,拟杆菌在环境水体中通常只能存活几个小时,导致分离培养困难,因此目前基于拟杆菌分离培养的MST方法已不多见。相比之下,近年来基于拟杆菌宿主特异性基因标记的MST方法(通常为靶向拟杆菌16S rRNA基因片段的qPCR方法)因其较高的特异性和敏感性(表 1)而得到了快速发展[36-44]。
目前,利用拟杆菌特异性基因标记能够很好地区分人、猪和反刍动物的粪便污染源,但在禽类粪便污染的溯源研究中应用较少。需要注意的是,因研究区域不同,各个基因标记检测污染源的敏感性和特异性会略有差别[14]。
2.1.1.5 其他细菌指示物法除了上述常见的粪源指示细菌外,还有一些其他的粪源菌也可用于微生物溯源。例如,短杆菌(Brevibacterium)16S rRNA基因片段LA35可作为禽类粪便污染的指示物(表 1)[45];细菌Catellicoccus marimammalium 16S rRNA基因片段GFC和螺杆菌(Helicobacter)16S rRNA基因片段GFD可作为鸟类粪便污染的指示物[46];乳酸杆菌(Lactobacillales)16S rRNA基因片段Crane1可作为鹤源粪便污染指示物等[47]。
2.1.2 病毒指示物法 2.1.2.1 拟杆菌噬菌体法有些以脆弱拟杆菌(如菌株RYC 2056)为宿主的噬菌体广泛存在于受人类粪便污染的水体中,可以采用双层平板进行培养以检测人源粪便污染[48]。不过该方法的敏感性并不高,需谨慎使用[49]。
2.1.2.2 大肠杆菌噬菌体(F+RNA coliphage)法F+ RNA大肠杆菌噬菌体主要分为4种类型,即型Ⅰ、型Ⅱ、型Ⅲ和型Ⅳ。其中,型Ⅱ和型Ⅲ主要与人类粪便污染有关,型Ⅳ主要与动物粪便污染有关,而型Ⅰ与人类和动物粪便污染都有关[13, 49]。对于检测到的噬菌体可以通过免疫学方法或者遗传学方法进行鉴定,进而利用不同类型F+ RNA大肠杆菌噬菌体的宿主差异性来区分污染源[49-50]。近年来,基于分子方法(如反转录PCR等)来检测4种类型F+ RNA大肠杆菌噬菌体的MST方法不仅简化了检测过程,还提高了方法的灵敏性[51-52]。
2.1.2.3 其他病毒指示物法除了上述病毒外,还有一些其他的病毒也可作为粪源指示物(宿主多为人类)。例如,人类多瘤病毒(HPyVs)[53]、胡椒粉斑驳病毒(PMMoV)[54]等,都可以通过分子检测方法鉴别人源粪便污染。表 1列举了目前单源指示物法常用的基因标记及其对应的引物。
2.2 多源指示物法 2.2.1 DNA微阵列(Microarray)法DNA微阵列又称为DNA芯片或基因芯片,主要由硅片、玻片等材质的载体和附着其上并已知序列的基因探针分子构成。将待测样品与DNA微阵列进行杂交,通过杂交信号的有无及强弱获得待测样品中微生物群落的基因组成信息[55]。近年来DNA微阵列技术在环境监管领域日益受到关注。该方法可用于环境水体粪便污染溯源[56],能够高通量、快速地鉴别多种粪便污染源。但也存在特定污染源的基因指示物数据库缺乏、成本和复杂度较高等问题。Dubinsky等[57]通过针对16SrRNA基因的系统发育微阵列分析识别了不同动物粪便中的特征微生物类群,并且野外试验表明这种基于微阵列的MST方法能够通过一次实验追踪多种污染来源。美国国家环境保护署(U.S. Environmental protection agency,EPA)也报道了一种采用DNA微阵列技术进行溯源的方法,该微阵列由453个探针组成,能够检测粪便和宿主特异的细菌、病毒、抗生素耐药性基因以及其他与环境污染相关的基因标记,可用于娱乐水体病原菌检测和粪便污染源鉴定[58]。
2.2.2 高通量测序(High-throughput-sequencing,HTS)法高通量测序技术能一次对PCR扩增产物或来自环境样品的DNA序列信息进行大规模平行分析,从而获得海量数据。通过对微生物群落分析和开发新的MST标记,HTS-MST法提供了一个快速、有效的定性粪便污染源检测途径[59],为水体粪便污染溯源带来新的机遇。
HTS-MST方法不是依赖单个基因标记检测或者源指示微生物的培养计数,而是从微生物群落的角度,更全面地解析环境水体中的粪便污染来源[60]。近年来,主要发展起来两种HTS-MST方法:一种是经过对测序结果的聚类分析,选出某类粪便污染源(如人类和动物)特异的可操作分类单元(Operational taxonomic units,OTUs),或者是多个污染源共有的OTUs,再根据环境样品的微生物群落组成特征判断粪便污染程度和可能的污染源;另一种方法是使用基于贝叶斯算法的SourceTracker分析,通过比较环境样品与不同动物粪便中微生物群落组成特征,进而判断不同污染源对环境样品微生物群落的贡献[60],目前已被用于鉴别环境水体的粪便污染源[61]。
3 MST的实例应用——TMDL方案在环境管理和水质监测方面,MST技术已经被广泛地应用于地表水体的粪便污染鉴定及溯源,有助于管理人员针对水体污染源实施有效的治理和修复措施[62-64]。该技术可以结合TMDL方案制定流域保护措施。TMDL指水体可以接受并仍然满足水质量标准的污染物的最大日负荷量[11]。TMDL方案是EPA推行的控制水体污染源、保护地表水环境质量的计划和措施[65]。该方案已经开展了30多年,人们通过流域水质模拟、安全边际估算等积累了大量的经验,促进了水质改善和污染控制措施的实施[66]。
MST技术能够通过准确判断污染源以支持TMDL方案的制定和实施,具体体现在以下3个方面[11]:(1)源识别——在TMDL制定过程中,MST最明显的用途是鉴别水体粪便污染源,并将结果用于TMDL分析中。(2)负荷分配分析——MST有助于识别流域中最主要的污染源,其结果可用于指导开发和选择较可行的负荷分配方案。(3)实施方案制定——MST有助于将管理目标集中于其已确定的污染源上,促进制定相关的污染控制实施方案。
在TMDL方案的制定过程中采用MST技术,能够根据溯源结果更加具体地分配微生物污染负荷削减任务。例如,可以根据实际情况削减农场污染排放、减少化粪池渗漏或者是污水管道泄漏等[66]。据EPA报道,美国的爱达荷州、俄勒冈州、新墨西哥州等多个地区已将MST技术应用于当地的TMDL方案的开发和实施中,促进了当地水环境的修复[11]。
4 MST的问题在实际应用中,MST面临的一个主要问题是该技术存在地区差异性[14]。例如,研究发现在美国被推荐为人类粪便污染指示物的特异性拟杆菌基因标记HF183[67],并不适用于印度的人源粪便污染判定[64],并且在中国珠三角河网地区的MST适用性研究中发现敏感性也较低[43];而在印度能够有效鉴别人源粪便污染的拟杆菌基因标记BacHum[64],在新加坡进行MST时的敏感性并不高[14]。导致MST方法地区差异性的原因主要源自宿主本身和环境因素两个方面。
宿主方面,由于不同地区的畜禽饲养方式及人类饮食结构往往存在一定差异,微生物在适应不同地区的宿主肠道环境的过程中可能产生遗传多样性,进而使得MST指示物的表型或基因型发生变异[68],导致MST方法在不同地区的适用性问题。Shanks等[68]在美国对7个追踪牛粪便污染源的MST方法进行适用性评估时发现,由于牛粪便样品采集于不同地区并且各地喂养方法不同,使MST方法在不同地区的敏感性和特异性存在显著差异。
环境因素方面,由于指示物在不同环境条件下的衰减速率不一样,加之环境水体成分的复杂性,从而致使MST方法在不同地区的适用性存在差异。具体来说:(1)指示物排入环境水体后因受周围环境条件的影响而使其行为变的极其复杂,其衰减速率也是影响MST方法适用性的重要原因。许多学者针对指示物在不同环境条件下的衰减进行了相关研究,发现基因标记极易受环境条件的影响,如温度越低基因标记的存在时间越长[69],盐度越高基因标记的衰减速度越慢[70]等。(2)环境样品的类型及其物理和化学特征(如颗粒物、酸碱度等)直接影响MST方法的敏感性和检测限[71-72]。环境样品中通常含有腐殖酸等抑制物,难以在DNA提取过程中完全去除,从而可能导致PCR或qPCR检测的敏感性降低,检测限升高[66]。因此,在进行MST前,需要对研究区域水环境进行方法的敏感性评估,以确定方法是否受到环境样品DNA提取物中杂质成分的抑制。
5 总结及展望综上所述,环境监测和管理的需要推动了MST技术的发展。根据指示物检测通量的不同,本文把当前的MST方法分为单源指示物法和多源指示物法两大类。其中单源指示物法检测速度快、操作简单,能定量鉴别不同动物的粪便污染,但通量较低,一次只能检测一种粪便污染;而多源指示物法能同时对多个污染源进行定性解析,但尚存在成本较高、操作较复杂、难以准确定量等不足。在实际操作中可将两种方法联用,以提高溯源的效率和准确性,例如可首先通过多源指示物法定性分析环境水体中的所有可能的粪便污染来源,进而使用单源指示物法定量评估特定粪源污染程度。
MST方法目前所面临的最大挑战仍然是地区差异性问题[14],不同地区的宿主肠道菌群差异以及环境条件都可能会导致方法适用性发生改变。本课题组在评估已有的MST方法在我国华北地区的适用性时也发现,许多已报道的基于猪源特异性拟杆菌基因标记的MST方法的特异性并不高。因此在MST实际应用前,尤其是没有相关地区MST方法应用报道时,需要对MST方法的适用性进行评估,充分了解其特异性、敏感性和检测限等,以判断方法是否适用。
目前,MST技术作为一种环境监测手段在我国的应用还相对较少。未来应建立MST方法的标准化检测流程,并促进MST技术在我国水质监测和水环境保护领域的应用,以期为我国环境管理工作提供技术支持。
| [1] |
Arnold BF, Wade TJ, Benjamin-Chung J, et al. Acute gastroenteritis and recreational water:highest burden among young US children[J]. Am J Public Health, 2016, 106(9): 1690-1697. DOI:10.2105/AJPH.2016.303279 |
| [2] |
Oun A, Kumar A, Harrigan T, et al. Effects of biosolids and manure application on microbial water quality in rural areas in the US[J]. Water, 2014, 6(12): 3701-3723. DOI:10.3390/w6123701 |
| [3] |
Tremblay LA, Gadd JB, Northcott GL. Steroid estrogens and estrogenic activity are ubiquitous in dairy farm watersheds regardless of effluent management practices[J]. Agr Ecosyst Environ, 2018, 253: 48-54. DOI:10.1016/j.agee.2017.10.012 |
| [4] |
Nguyen KH, Senay C, Young S, et al. Determination of wild animal sources of fecal indicator bacteria by microbial source tracking (MST) influences regulatory decisions[J]. Water Res, 2018, 144: 424-434. DOI:10.1016/j.watres.2018.07.034 |
| [5] |
Sun H, He X, Ye L, et al. Diversity, abundance, and possible sources of fecal bacteria in the Yangtze River[J]. Appl Microbiol Biotechnol, 2017, 101(5): 2143-2152. DOI:10.1007/s00253-016-7998-2 |
| [6] |
Byappanahalli MN, Nevers MB, Korajkic A, et al. Enterococci in the environment[J]. Microbiol Molecular Biol Rev, 2012, 76(4): 685-706. DOI:10.1128/MMBR.00023-12 |
| [7] |
Parveen S, Murphree RL, Edmiston L, et al. Association of multiple-antibiotic-resistance profiles with point and nonpoint sources of Escherichia coli in Apalachicola Bay[J]. Appl Environ Microbiol, 1997, 63(7): 2607-2612. |
| [8] |
Roslev P, Bukh AS. State of the art molecular markers for fecal pollution source tracking in water[J]. Appl Microbiol Biotechnol, 2011, 89(5): 1341-1355. DOI:10.1007/s00253-010-3080-7 |
| [9] |
Converse RR, Blackwood AD, Kirs M, et al. Rapid qPCR-based assay for fecal Bacteroides spp. as a tool for assessing fecal contamination in recreational waters[J]. Water Res, 2009, 43(19): 4828-4837. DOI:10.1016/j.watres.2009.06.036 |
| [10] |
Solecki O, Jeanneau L, Jarde E, et al. Persistence of microbial and chemical pig manure markers as compared to faecal indicator bacteria survival in freshwater and seawater microcosms[J]. Water Res, 2011, 45(15): 4623-4633. DOI:10.1016/j.watres.2011.06.012 |
| [11] |
USEPA. Using microbial source tracking to support TMDL development and implementation[R]. Seattle, Washington State: USEPA, 2011. https: //www.epa.gov/sites/production/files/2015-07/documents/mst_for_tmdls_guide_04_22_11.pdf
|
| [12] |
Hagedorn C, Blanch AR, Harwood VJ. Microbial source tracking:methods, applications, and case studies[M]. New York: Springer, 2011.
|
| [13] |
Scott TM, Rose JB, Jenkins TM, et al. Microbial source tracking:current methodology and future directions[J]. Appl Environ Microbiol, 2002, 68(12): 5796-5803. DOI:10.1128/AEM.68.12.5796-5803.2002 |
| [14] |
Nshimyimana JP, Cruz MC, Thompson RJ, et al. Bacteroidales markers for microbial source tracking in Southeast Asia[J]. Water Res, 2017, 118: 239-248. DOI:10.1016/j.watres.2017.04.027 |
| [15] |
Geldreich EE. Fecal coliform and fecal streptococcus density relationships in waste discharges and receiving waters[J]. Crit Rev Env Sci Technol, 1976, 6(4): 349-369. |
| [16] |
Geldreich EE, Kenner BA. Concepts of fecal streptococci in stream pollution[J]. Journal Water Pollution Control Federation, 1969, 41(8P2): R336-R352. |
| [17] |
Edwards DR, Coyne MS, Vendrell PF, et al. Fecal coliform and streptococcus concentrations in runoff from grazed pastures in Northwest Arkansas[J]. J Am Water Resour Ass, 1997, 33(2): 413-422. DOI:10.1111/j.1752-1688.1997.tb03520.x |
| [18] |
Sinton LW, Finlay RK, Hannah DJ. Distinguishing human from animal faecal contamination in water:a review[J]. N Zealand J Marine Freshwater Res, 1998, 32(2): 323-348. DOI:10.1080/00288330.1998.9516828 |
| [19] |
Jang J, Hur HG, Sadowsky MJ, et al. Environmental Escherichia coli:ecology and public health implications-a review[J]. J Appl Microbiol, 2017, 123(3): 570-581. DOI:10.1111/jam.13468 |
| [20] |
King EL, Bachoon DS, Gates KW. Rapid detection of human fecal contamination in estuarine environments by PCR targeting of Bifidobacterium adolescentis[J]. J Microbiol Methods, 2007, 68(1): 76-81. DOI:10.1016/j.mimet.2006.06.008 |
| [21] |
Cimenti M, Hubberstey A, Bewtra JK, et al. Alternative methods in tracking sources of microbial contamination in waters[J]. Water Sa, 2007, 33(2): 183-194. |
| [22] |
Long SC, Shafer E, Arango FC, et al. Evaluation of three source tracking indicator organisms for watershed management[J]. J Water Supply:Res Technol-Aqua, 2003, 52(8): 565-575. DOI:10.2166/aqua.2003.0050 |
| [23] |
Bonjoch X, Lucena F, Blanch AR. The persistence of Bifidobacteria populations in a river measured by molecular and culture techniques[J]. J Appl Microbiol, 2009, 107(4): 1178-1185. DOI:10.1111/j.1365-2672.2009.04297.x |
| [24] |
Venegas C, Diez H, Blanch AR, et al. Microbial source markers assessment in the Bogota River basin (Colombia)[J]. J Water Health, 2015, 13(3): 801-810. DOI:10.2166/wh.2015.240 |
| [25] |
Balleste E, Blanch AR. Bifidobacterial diversity and the development of new microbial source tracking indicators[J]. Appl Environ Microbiol, 2011, 77(10): 3518-3525. DOI:10.1128/AEM.02198-10 |
| [26] |
Gourmelon M, Caprais MP, Mieszkin S, et al. Development of microbial and chemical MST tools to identify the origin of the faecal pollution in bathing and shellfish harvesting waters in France[J]. Water Res, 2010, 44(16): 4812-4824. DOI:10.1016/j.watres.2010.07.061 |
| [27] |
Gomez-Donate M, Balleste E, Muniesa M, et al. New molecular qu-antitative PCR assay for detection of host-specific Bifidobacteriaceae suitable for microbial source tracking[J]. Appl Environ Microb, 2012, 78(16): 5788-5795. DOI:10.1128/AEM.00895-12 |
| [28] |
敖静, 阮晓红, 万宇. 水环境中粪便污染的微生物溯源技术及研究进展[J]. 环境与健康杂志, 2012, 29(7): 658-662. |
| [29] |
Harwood VJ, Whitlock J, Withington V. Classification of antibiotic resistance patterns of indicator bacteria by discriminant analysis:use in predicting the source of fecal contamination in subtropical waters[J]. Appl Environ Microbiol, 2000, 66(9): 3698-3704. DOI:10.1128/AEM.66.9.3698-3704.2000 |
| [30] |
Parveen S, Hodge NC, Stall RE, et al. Phenotypic and genotypic characterization of human and nonhuman Escherichia coli[J]. Water Res, 2001, 35(2): 379-386. DOI:10.1016/S0043-1354(00)00269-4 |
| [31] |
D'Elia TV, Cooper CR, Johnston CG. Source tracking of Escherichia coli by 16S-23S intergenic spacer region denaturing gradient gel electrophoresis (DGGE) of the rrnB ribosomal operon[J]. Can J Microbiol, 2007, 53(10): 1174-1184. DOI:10.1139/W07-083 |
| [32] |
Deng D, Zhang N, Xu D, et al. Polymorphism of the glucosyltransferase gene (ycjM) in Escherichia coli and its use for tracking human fecal pollution in water[J]. Sci Total Environ, 2015, 537: 260-267. DOI:10.1016/j.scitotenv.2015.08.019 |
| [33] |
Khatib LA, Tsai YL, Olson BH. A biomarker for the identification of swine fecal pollution in water, using the STII toxin gene from enterotoxigenic Escherichia coli[J]. Appl Microbiol Biotechnol, 2003, 63(2): 231-238. DOI:10.1007/s00253-003-1373-9 |
| [34] |
郭萍, 李红娜, 李峰. MST与水环境生物源污染定量化溯源[J]. 农业环境科学学报, 2016, 35(2): 205-211. |
| [35] |
Leknoi Y, Mongkolsuk S, Sirikanchana K. Assessment of swine-specific bacteriophages of Bacteroides fragilis in swine farms with different antibiotic practices[J]. J Water Health, 2017, 15(2): 251-261. DOI:10.2166/wh.2016.069 |
| [36] |
Bernhard AE, Field KG. A PCR assay to discriminate human and ruminant feces on the basis of host differences in Bacteroides-Prevotella genes encoding 16S rRNA[J]. Appl Environ Microbiol, 2000, 66(10): 4571-4574. DOI:10.1128/AEM.66.10.4571-4574.2000 |
| [37] |
Mieszkin S, Yala JF, Joubrel R, et al. Phylogenetic analysis of Bacteroidales 16S rRNA gene sequences from human and animal effluents and assessment of ruminant faecal pollution by real-time PCR[J]. J Appl Microbiol, 2010, 108(3): 974-984. DOI:10.1111/j.1365-2672.2009.04499.x |
| [38] |
Kildare BJ, Leutenegger CM, McSwain BS, et al. 16S rRNA-based assays for quantitative detection of universal, human-, cow-, and dog-specific fecal Bacteroidales:a Bayesian approach[J]. Water Res, 2007, 41(16): 3701-3715. DOI:10.1016/j.watres.2007.06.037 |
| [39] |
Reischer GH, Kasper DC, Steinborn R, et al. A quantitative real-time PCR assay for the highly sensitive and specific detection of human faecal influence in spring water from a large alpine catchment area[J]. Lett Appl Microbiol, 2007, 44(4): 351-356. DOI:10.1111/j.1472-765X.2006.02094.x |
| [40] |
Reischer GH, Kasper DC, Steinborn R, et al. Quantitative PCR method for sensitive detection of ruminant fecal pollution in freshwater and evaluation of this method in alpine karstic regions[J]. Appl Environ Microbiol, 2006, 72(8): 5610-5614. DOI:10.1128/AEM.00364-06 |
| [41] |
Shanks OC, Atikovic E, Blackwood AD, et al. Quantitative PCR for detection and enumeration of genetic markers of bovine fecal pollution[J]. Appl Environ Microbiol, 2008, 74(3): 745-752. DOI:10.1128/AEM.01843-07 |
| [42] |
Mieszkin S, Furet J-P, Corthier G, et al. Estimation of pig fecal contamination in a river catchment by real-time PCR using two pig-specific Bacteroidales 16S rRNA genetic markers[J]. Appl Environ Microbiol, 2009, 75(10): 3045-3054. DOI:10.1128/AEM.02343-08 |
| [43] |
张杨, 吴仁人, 张一敏, 等. 珠三角河网地区粪便污染源解析[J]. 中国环境科学, 2017, 37(9): 3446-3454. DOI:10.3969/j.issn.1000-6923.2017.09.031 |
| [44] |
王显贵, 郭萍, 田云龙, 等. 利用qPCR定量检测水体中猪源拟杆菌特异性生物标记的研究[J]. 农业环境科学学报, 2013, 32(11): 2302-2308. DOI:10.11654/jaes.2013.11.028 |
| [45] |
Weidhaas JL, Macbeth TW, Olsen RL, et al. Identification of a Brevibacterium marker gene specific to poultry litter and development of a quantitative PCR assay[J]. J Appl Microbiol, 2010, 109(1): 334-347. |
| [46] |
Green HC, Dick LK, Gilpin B, et al. Genetic markers for rapid PCR-based identification of gull, Canada goose, duck, and chicken fecal contamination in water[J]. Appl Environ Microbiol, 2012, 78(2): 503-510. DOI:10.1128/AEM.05734-11 |
| [47] |
Ryu H, Lu J, Vogel J, et al. Development and evaluation of a quantitative PCR assay targeting sandhill crane (Grus canadensis) fecal pollution[J]. Appl Environ Microbiol, 2012, 78(12): 4338-4345. DOI:10.1128/AEM.07923-11 |
| [48] |
Wicki M, Auckenthaler A, Felleisen R, et al. Assessment of source tracking methods for application in spring water[J]. J Water Health, 2015, 13(2): 473-488. DOI:10.2166/wh.2014.255 |
| [49] |
Jagals P, Grabow WOK, Devilliers JC. Evaluation of indicators for assessment of human and animal fecal pollution of surface run-off[J]. Water Sci Technol, 1995, 31(5-6): 235-241. DOI:10.2166/wst.1995.0612 |
| [50] |
Lee JE, Lee H, Cho YH, et al. F+ RNA coliphage-based microbial source tracking in water resources of South Korea[J]. Sci Total Environ, 2011, 412: 127-131. |
| [51] |
Ogorzaly L, Gantzer C. Development of real-time RT-PCR methods for specific detection of F-specific RNA bacteriophage genogroups:Application to urban raw wastewater[J]. J Virol Methods, 2006, 138(1-2): 131-139. DOI:10.1016/j.jviromet.2006.08.004 |
| [52] |
Vergara GGRV, Goh SG, Rezaeinejad S, et al. Evaluation of FRNA coliphages as indicators of human enteric viruses in a tropical urban freshwater catchment[J]. Water Res, 2015, 79: 39-47. DOI:10.1016/j.watres.2015.04.022 |
| [53] |
McQuaig SM, Scott TM, Lukasik JO, et al. Quantification of human polyomaviruses JC virus and BK virus by TaqMan quantitative PCR and comparison to other water quality indicators in water and fecal samples[J]. Appl Environ Microbiol, 2009, 75(11): 3379-3388. DOI:10.1128/AEM.02302-08 |
| [54] |
Zhang T, Breitbart M, Lee WH, et al. RNA viral community in human feces:prevalence of plant pathogenic viruses[J]. PLoS Biol, 2006, 4(1): 108-118. |
| [55] |
Karakach TK, Flight RM, Douglas SE, et al. An introduction to DNA microarrays for gene expression analysis[J]. Chemometr Intell Lab, 2010, 104(1): 28-52. DOI:10.1016/j.chemolab.2010.04.003 |
| [56] |
Li X, Harwood VJ, Nayak B, et al. A novel microbial source tracking microarray for pathogen detection and fecal source identification in environmental systems[J]. Environ Sci Technol, 2015, 49(12): 7319-7329. DOI:10.1021/acs.est.5b00980 |
| [57] |
Dubinsky EA, Esmaili L, Hulls JR, et al. Application of phylogenetic microarray analysis to discriminate sources of fecal pollution[J]. Environ Sci Technol, 2012, 46(8): 4340-4347. DOI:10.1021/es2040366 |
| [58] |
Li X, Harwood V, Nayak B, et al. A customized DNA microarray for microbial source tracking in environmental systems[EB/OL]. 2016[2018-09-15]. https: //www.epa.gov/sites/production/files/2016-06/documents/customized-dna-micorarray.pdf
|
| [59] |
Tan B, Ng C, Nshimyimana JP, et al. Next-generation sequencing (NGS) for assessment of microbial water quality:current progress, challenges, and future opportunities[J]. Frontiers Microbiol, 2015, 6: 1-20. |
| [60] |
Unno T, Staley C, Brown CM, et al. Fecal pollution:new trends and challenges in microbial source tracking using next-generation sequencing[J]. Environ Microbiol, 2018, 20(9): 3132-3140. DOI:10.1111/1462-2920.14281 |
| [61] |
Brown CM, Staley C, Wang P, et al. A high-throughput DNA-sequencing approach for determining sources of fecal bacteria in a Lake Superior estuary[J]. Environ Sci Technol, 2017, 51(15): 8263-8271. DOI:10.1021/acs.est.7b01353 |
| [62] |
Devane ML, Weaver L, Singh SK, et al. Fecal source tracking methods to elucidate critical sources of pathogens and contaminant microbial transport through New Zealand agricultural watersheds-A review[J]. J Environ Manage, 2018, 222: 293-303. DOI:10.1016/j.jenvman.2018.05.033 |
| [63] |
Malla B, Shrestha RG, Tandukar S, et al. Validation of host-specific Bacteroidales quantitative PCR assays and their application to microbial source tracking of drinking water sources in the Kathmandu Valley, Nepal[J]. J Appl Microbiol, 2018, 125(2): 609-619. DOI:10.1111/jam.13884 |
| [64] |
Odagiri M, Schriewer A, Hanley K, et al. Validation of Bacteroidales quantitative PCR assays targeting human and animal fecal contamination in the public and domestic domains in India[J]. Sci Total Environ, 2015, 502: 462-470. DOI:10.1016/j.scitotenv.2014.09.040 |
| [65] |
杨龙, 王晓燕, 孟庆义. 美国TMDL计划的研究现状及其发展趋势[J]. 环境科学与技术, 2008, 31(9): 72-76. DOI:10.3969/j.issn.1003-6504.2008.09.019 |
| [66] |
魏源送, 郑嘉熹, 王光宇, 等. 地表水微生物溯源技术的开发和应用进展[J]. 水资源保护, 2016, 32(1): 1-11. |
| [67] |
Layton BA, Cao Y, Ebentier DL, et al. Performance of human fecal anaerobe-associated PCR-based assays in a multi-laboratory method evaluation study[J]. Water Res, 2013, 47(18): 6897-6908. DOI:10.1016/j.watres.2013.05.060 |
| [68] |
Shanks OC, White K, Kelty CA, et al. Performance assessment PCR-based assays targeting Bacteroidales genetic markers of bovine fecal pollution[J]. Appl Environ Microbiol, 2010, 76(5): 1359-1366. DOI:10.1128/AEM.02033-09 |
| [69] |
Dick LK, Stelzer EA, Bertke EE, et al. Relative decay of Bacteroidales microbial source tracking markers and cultivated Escherichia coli in freshwater microcosms[J]. Appl Environ Microbiol, 2010, 76(10): 3255-3262. DOI:10.1128/AEM.02636-09 |
| [70] |
Green HC, Shanks OC, Sivaganesan M, et al. Differential decay of human faecal Bacteroides in marine and freshwater[J]. Environ Microbiol, 2011, 13(12): 3235-3249. DOI:10.1111/j.1462-2920.2011.02549.x |
| [71] |
Haugland RA, Siefring SC, Wymer LJ, et al. Comparison of Enterococcus measurements in freshwater at two recreational beaches by quantitative polymerase chain reaction and membrane filter culture analysis[J]. Water Res, 2005, 39(4): 559-568. DOI:10.1016/j.watres.2004.11.011 |
| [72] |
Siefring S, Varma M, Atikovic E, et al. Improved real-time PCR assays for the detection of fecal indicator bacteria in surface waters with different instrument and reagent systems[J]. J Water Health, 2008, 6(2): 225-237. DOI:10.2166/wh.2008.022 |