作为生物生长所需的大量元素之一,氮在土壤中的含量、赋存形态直接影响着作物的产量和品质,而土壤中氮的含量、赋存形态又与土壤微生物密切相关,土壤是微生物的大本营。土壤微生物不仅种类多、数量大,且功能多样[1],通过参与土壤中的氮代谢过程共同驱动土壤氮元素生物地球化学循环[2]。
早期关于土壤中氮转化微生物的研究主要基于分离、培养等手段[3],这种研究方法不仅费时费力,且难以深入了解土壤中参与氮代谢过程不同步骤微生物的丰度及机制。现代分子生物学技术为深入了解微生物参与土壤中氮代谢过程提供了有效手段,基于分子生物学技术目前已对与氮代谢有关的土壤微生物中的固氮基因(nifH)[4-6]、氨氧化基因(amoA-AOA、amoA-AOB)[7-10]、反硝化基因(nirS、nirK和nosZ)[11-13]、厌氧氨氧化基因(HZO)[14-15]等进行了深入的研究。研究发现土壤中氮转化功能基因的多样性及丰度可以作为指示土壤中氮元素循环有效指标,揭示土壤中氮素的循环过程[16]。
土壤中参与氮转化功能基因丰度的影响因素多种多样,如偏酸性土壤中氨氧化微生物以amoA-AOA为主[17];低pH的酸性环境有利于nirS基因表达,而抑制nirK基因的表达[18];较高浓度的有效氮抑制固氮基因nifH的表达[5, 19];影响HZO基因丰度的环境因素也多种多样[20],水稻土溶氧量不足时,铁锰氧化物促进厌氧氨氧化基因表达[21];重金属促进或抑制性影响土壤中氮转化功能基因表达[22-23];剖面深度不同功能基因丰度不同等[24]。
目前,国内外关于土壤氮转化功能基因的研究多集中于农田表层土壤或沉积物中的某一类功能基因[4-15],而对极端环境下氮转化多种类型功能基因及其相互关系的研究开展较少[25-26]。土壤中的氮循环是涉及多类功能基因的一个连续过程,仅仅了解其中的某一类功能基因难以深入了解土壤中氮循环过程及其影响因素。
酸性矿业废水(Acid mine drainage,AMD)具有低pH、高盐、高重金属含量的特点[27],大量的酸性矿业废水进入农田后不仅导致土壤质量下降、粮食减产,也对土壤微生物以及土壤中的氮元素生物地球化学循环造成影响[28]。本研究以安徽省铜陵市某处受AMD污染的农田作为研究对象,基于分子生物学技术,测定受AMD污染的土壤剖面中微生物量及参与氮转化的功能基因丰度,探讨AMD污染与土壤剖面氮代谢功能基因的关系,旨为深入了解酸性矿业废水污染农田土壤中的氮循环特点提供理论支持。
1 材料与方法 1.1 材料 1.1.1 研究区概况研究区位于安徽省铜陵市某矿区附近受来自该矿酸性矿业废水污染的稻田(N 30°58'26″,E 118°4'35″),该地土壤长期受硫铁矿排放的AMD污染,对照稻田(N 30°58'38″,E 118°4'15″)则一直使用正常水源灌溉。该地区为亚热带气候,无霜期有237-258 d,年平均气温16.2℃,平均湿度在75%-81%之间,年均降雨量1 346 mm,雨季集中于7-9月[27]。
1.1.2 样品采集供试稻田包括2块,一块受硫铁矿排放的AMD污染、另一块未受AMD污染(对照)。2017年4月在每块稻田各设置5个采样点,呈梅花状分布,每个采样点利用PVC管(内径5 cm)分别采集0-10 cm、10-20 cm、20-40 cm、40-60 cm、60-80 cm和80-100 cm土壤剖面样本。野外采集的土壤样本经充分混匀后装入灭菌的密封袋,然后置于4℃冰盒保存。返回室内后,除部分新鲜样用于氨氮、硝氮含量测定和分子生物学试验外,其余样品于室内风干,研磨过筛后用于土壤pH、总氮、有机质和重金属含量分析。
1.2 方法 1.2.1 土壤理化分析方法pH值用pH计测定(土壤:去离子水=1 g:5 mL),总氮(TN)利用总有机碳分析仪(岛津TOC-L)测定;氨氮(NH4+-N)、硝态氮(NO3--N)分别采用苯酚-次氯酸钠比色法、紫外分光光度法进行测定;硫采用X-射线衍射光谱分析法测定;有机质采用烧失量法测定;土壤重金属含量采用盐酸-氢氟酸消解后,用原子吸收分光光度计(耶拿ContrAA® 700)测定。
1.2.2 总DNA提取及功能基因丰度测定微生物总DNA提取采用CTAB法提取[27],DNA样品用1%的琼脂糖凝胶电泳检测后置于零下20℃冷冻保存。参与氮转化的功能基因用上下引物在PCR仪里扩增后得到标准品,引物序列见(表 1)。经扩增后纯化并嵌入到pEASY-T3克隆载体(全式金,中国北京)中,并将连接产物转染到Trans1-T1 phage-resistant chemically competent cell(全式金,中国北京)。从阳性克隆中抽提出的质粒被用来作为标准品并进行荧光PCR(qPCR)定量,测定土壤剖面中功能基因的丰度。
qPCR定量反应在ABI step-one system扩增仪上根据KAPA SYBR FAST qPCR Kit Master Mix试剂的说明书进行。qPCR荧光定量反应体系总体积为20.0 μL:10 μL TaKaRa SYBRFAST qPCR Kit Master Mix(TaKaRa Biosystems,USA);上下游引物(10 μmol/L)各0.5 μL,样品DNA模板2 μL,7 μL ddH2O。以不同浓度梯度标准品作为模板进行荧光定量PCR扩增来绘制标准曲线,在荧光PCR定量仪器里进行采集荧光反应。产物的特异性用溶解性曲线以及凝胶电泳确定。qPCR的扩增效率是80%-90%,标准曲线的R2 > 0.99。
1.2.3 数据分析方法SPSS 20.0软件用于平均值、标准误、单因素方差分析、多因子方差分析(MANOVA)和双变量相关分析(Pearson)等的计算,显著性水平P < 0.05、P < 0.01作为显著性、极显著性判断标准;Origin 8.0软件用于趋势线的绘制。
2 结果 2.1 土壤理化性质根据相关的仪器和方法测定受AMD污染土壤的理化性质,结果如下:土壤pH为4.7-5.9,整体呈酸性,而未污染区土壤酸碱性整体呈中性;总氮含量为287.27-943.78 mg/kg,高于未污染区11.41%;氨氮含量为1.78-13.77 mg/kg,低于未污染区6.35%;硝氮1.84-5.73 mg/kg,高于未污染区55.45%;有机质含量为5.10%-5.78%,与未污染区的土壤有机质含量相似;S的含量为147.4-653.2 mg/kg,高于未污染区93.02%;Fe2O3为3.58%-5.75%,高于未污染区20.27%。土壤中的总氮、氨氮、硝氮、有机质含量有随剖面深度而降低的趋势,而Fe2O3随剖面深度的增加而富集。
2.2 土壤中细菌和古菌的数量本研究中,每克干土的细菌和古菌16S丰度分别为3.01×1013-5.80×1013copies和4.30×1011-9.06×1011copies,表明所研究土壤剖面中细菌数量高于古菌(图 1)。污染和对照的土壤剖面中,0-20 cm表层土壤细菌的数量均显著高于其他各层(P < 0.0),剖面0-80 cm范围内细菌的数量基本上呈现出随着剖面深度的增加而降低的趋势。不同于细菌,尽管污染和对照土壤剖面中古菌数量也随着剖面深度增加而降低,但整个剖面中古菌生物量的降低速率则相对较慢。与未受AMD污染的土壤剖面相比,受AMD污染的0-80 cm土壤剖面深度中细菌的生物量显著降低,而古菌仅在20-60 cm的剖面中数量显著降低(图 1)。

|
| 图 1 AMD污染和对照土壤剖面中的细菌和古菌 |
从表 2可以看出,污染和对照稻田土壤剖面中不同功能基因的丰度随剖面深度变化呈不同的趋势。在AMD污染的土壤剖面中,0-10 cm范围内的nifH、nosZ和HZO的丰度显著高于其下的其他各层(P < 0.05),而amoA-AOA、amoA-AOB、nirS、nirK等功能基因的相对丰度尽管相差一个数量级,但统计检验表明并未呈现出显著差异;对照稻田土壤剖面中,表层0-10(20)cm范围内所测定的7个功能基因的丰度均显著高于其下的其他各层(P < 0.05)。
对比AMD污染土壤和对照土壤剖面同一层次的相同功能基因的丰度可以看出,AMD污染土壤10-80 cm剖面中nifH基因丰度显著小于对照区,0-40 cm剖面中的amoA-AOB的丰度也低于对照区;而剖面中nirS(10-100 cm)和HZO(40-100 cm)则表现为相反的变化。上述结果表明,AMD对土壤剖面中功能基因nifH、nirS、amoA-AOB和HZO的影响较为显著。
AMD污染稻田土壤剖面中,参与氨氧化的功能基因(amoA-AOA、amoA-AOB)则表现为amoA-AOA的相对丰度高于amoA-AOB,而参与反硝化的功能基因(nirS、nirK)总体上表现为nirK相对丰度高于nirS。这表明amoA-AOA和nirK在AMD污染稻田土壤剖面的氨氧化和反硝化作用中扮演着更为重要的角色。
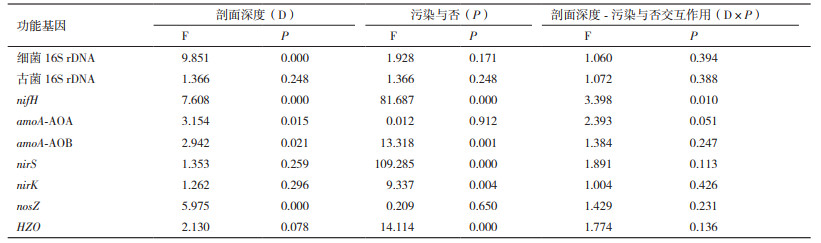
3 讨论 3.1 影响剖面中细菌和古菌数量的主要因素利用16S rDNA基因丰度作为指标表征土壤中细菌和古菌数量目前已得到广泛应用[29-30]。本研究发现无论是否受到AMD污染,土壤剖面中细菌和古菌的数量均随着剖面深度的增加而降低,且同一层次中细菌的数量高于古菌;AMD污染剖面中细菌和古菌的数量均显著低于未受污染的对照土壤,上述结果在其他研究中也得到证实[24, 27]。研究表明,土壤剖面中微生物数量随着深度增加而逐渐降低主要与养分(如有机质、总氮、氨氮和硝氮等)及溶解氧含量逐渐降低有关[24, 30],本研究中土壤总氮和硝氮含量与剖面中细菌、古菌的丰度均呈极显著的正相关(r总氮-细菌=0.615,P≤0.01;r总氮-古菌=0.525,P≤0.01;r硝氮-细菌=0.367,P≤0.01;r硝氮-古菌=0.425,P≤0.01),而土壤的细菌数量高于古菌的原因可能是由于细菌相对于古菌具有更多的代谢种类和营养类型,能够适应更多、更复杂的生态环境[31]。AMD污染的土壤剖面中细菌和古菌数量低于对照剖面则主要由于较高的重金属含量所导致[22],相关分析表明剖面中细菌和古菌丰度与土壤中铁的含量呈极显著负相关(r铁-细菌=-0.456,P≤0.01;r铁-古菌=-0.390,P≤0.01),说明随酸性矿业废水而来的铁进入土壤导致了其中细菌和古菌数量降低。尽管剖面深度和重金属含量均能导致土壤中细菌和古菌数量降低,但相对古菌而言(P=0.248)剖面深度对细菌数量的影响更为显著(P=0.000)(表 3)。
土壤中功能基因的丰度不仅受土壤微生物群落的种类组成和生物量影响[24],还受到诸多环境因子的影响。相关分析表明,剖面中细菌丰度与nifH、amoA-AOA、amoA-AOB和nosZ呈极显著正相关(相关系数分别为0.492、0.366、0.457和0.779),古菌丰度与amoA-AOA、amoA-AOB、nosZ和HZO也呈显著或极显著正相关(相关系数分别为0.569、0.270、0.673和0.476),多因素方差分析也表明(表 3)剖面深度主要影响nifH、amoA-AOA、amoA-AOB和nosZ的丰度,深度对几种功能基因丰度的影响主要与细菌和/或古菌的丰度变化有关。
本研究所分析的7个功能基因中,受AMD污染的农田土壤剖面中nifH、nirS、amoA-AOB和HZO的丰度存在显著差异;多因素方差分析也发现,AMD污染对nifH、nirS、nirK、amoA-AOB和HZO产生极显著影响(P < 0.01,表 3)。本研究中,导致农田土壤污染的酸性矿业废水来自于一处废弃的硫铁矿,低pH、高的硫酸根和铁含量是该酸性矿业废水的主要特点。双变量相关分析进一步发现,pH与nifH和amoA-AOB呈极显著的正相关,而与nirS、amoA-AOA以及HZO呈极显著负相关;总硫的含量则与nosZ、amoA-AOA以及HZO呈极显著的正相关,而铁则表现出极显著的负相关(表 4)。
amoA-AOB及amoA-AOA是驱动土壤中好氧氨氧化的功能基因。本研究中,受AMD污染稻田土壤中不仅具有较高的amoA-AOA丰度,同时氨氧化古菌的数量也显著高于氨氧化细菌(表 2),表明受AMD污染的稻田中好氧氨氧化过程主要与氨氧化古菌有关,这一结果在其他的研究中也有发现[17, 24]。研究表明,在中性和石灰性土壤中,氨氧化微生物在数量上以amoA-AOB为主导,在酸性土壤中以amoA-AOA为主导[17, 32],这也就解释了本研究中pH对amoA-AOB及amoA-AOA相对丰度有着截然不同的影响。铁和硫与amoA-AOA的相关性也是截然相反的(表 4),铁与amoA-AOA之间的负相关性可能与铁与古菌之间的显著性负相关,AMD污染土壤剖面中铁的增加导致古菌丰度降低(r铁-古菌=-0.390,P≤0.01),伴随着古菌数量的降低源于古菌的amoA-AOA丰度也相对降低;而硫与amoA-AOA极显著正相关,可能与硫酸盐还原作用-氨氧化作用之间的耦合有关[33],本研究中氨氧化功能基因amoA-AOA丰度与参与硫酸盐还原的dsrB功能基因丰度(数据在表 2中未展示)之间呈现出极显著正相关(r=0.364,P≤0.01)。由于氨氧化古菌是AMD污染稻田中好氧氨氧化过程的主要参与者,因此本研究中总氮和硝氮含量与amoA-AOA丰度之间的正相关性是容易理解的,这种关系也被其他的研究所证实[10]。
在整个反硝化过程中,nirS和nirK是将亚硝酸盐还原为NO的功能基因[32],是反硝化作用的标志性反应,也是最重要的限速步骤;而功能基因nosZ则将N2O还原为N2,是反硝化作用的最后一步。研究发现,土壤理化性质如pH、质地及含氮量等是微生物反硝化过程的重要影响因素[24, 32]。本研究中,AMD对土壤剖面中功能基因nirS的影响较为显(P < 0.05),而对nirK、nosZ等功能基因丰度的影响则相对较小或不产生影响,AMD污染土壤中nirS基因丰度显著性大于未污染区(表 2)。Samad等[18]也发现,酸性土壤环境更有利于nirS基因表达。尽管有研究发现nosZ对pH的变化十分敏感[32],但本研究中nosZ丰度与pH之间并无显著相关性。总氮和硝氮是反硝化过程的电子受体,已经得到广泛证明[32],本研究中土壤总氮和硝氮仅与nosZ具有极显著的正相关性,而与nirS和nirK并无显著相关性(表 4),这些结果表明,总氮和硝氮对反硝化过程及其基因的影响在不同的环境条件下存在有显著的差异性。
厌氧氨氧化(Anammox)过程是土壤中的微生物通过调控功能基因HZO将亚硝酸盐和NH4+氧化为N2,影响该过程的环境因素包括pH、溶解氧、NO等[20],同时剖面深度和土壤质地也对HZO功能基因丰度产生影响[34]。本研究中,pH、Fe2O3和有机质含量与HZO呈显著或极显著的负相关。Anammox过程是一个消耗H+的过程[15],AMD污染条件下较酸的土壤环境可能促进了Anammox的进程;水稻土溶解氧不足时,铁锰氧化物因充当有机质降解的氧化剂需消耗H+进行铁氨氧化[21],这可能是导致铁有机质含量与HZO呈显著或极显著的负相关主要原因。
nifH作为固氮作用的标志性功能基因主要来自固氮细菌,这些固氮细菌多偏好酸碱度为中性的土壤环境[6],土壤中较高的硝态氮含量对nifH产生不利影响,抑制了nifH功能基因的表达[5],较高的有机质含量在提供固氮细菌生长需要的同时,丰富的电子传递系统也有利于提高nifH基因的丰度与表达[24]。
土壤剖面中功能基因丰度除了受到营养状况和污染物含量影响外,还受到剖面深度的影响。表 3可以看出,剖面深度主要影响nifH、amoA-AOA、amoA-AOB和nosZ的丰度,深度对几种功能基因丰度的影响,一方面可能是随着深度增加细菌和古菌的数量逐渐降低;另一方面随着深度增加溶解氧及养分也逐渐降低,深度对功能基因丰度的影响在其他的研究中也被证实[24]。
4 结论随着剖面深度的增加,稻田土壤剖面中细菌和古菌数量以及参与N转化的部分功能基因丰度逐渐降低,AMD污染导致土壤剖面中细菌和古菌的数量显著降低,但古菌数量随剖面深度下降的速度低于细菌;AMD对土壤剖面中功能基因nifH、nirS、amoA-AOB和HZO的影响较为显著,在AMD污染土壤剖面中amoA-AOA和nirK在氨氧化和反硝化作用中扮演着更为重要的角色;酸性矿业废水污染导致的pH降低及总硫和总铁含量的增加是导致土壤剖面氨氧化和反硝化作用变化的主要原因。
| [1] |
Mendes LW, Tsai SM, Navarrete AA, et al. Soil-borne microbiome:linking diversity to function[J]. Microbial Ecology, 2015, 70(1): 255-265. DOI:10.1007/s00248-014-0559-2 |
| [2] |
Nelson MB, Martiny AC, Martiny JBH. Global biogeography of microbial nitrogen-cycling traits in soil[J]. Proceedings of the National Academy of Sciences, 2016, 113(29): 8033-8040. DOI:10.1073/pnas.1601070113 |
| [3] |
袁志辉, 王健, 杨文蛟, 等. 土壤微生物分离新技术的研究进展[J]. 土壤学报, 2014, 51(6): 1183-1191. |
| [4] |
Dahal B, Nandakafle G, Perkins L, et al. Diversity of free-Living nitrogen fixing Streptomyces in soils of the badlands of South Dakota[J]. Microbiological Research, 2017, 195: 31-39. DOI:10.1016/j.micres.2016.11.004 |
| [5] |
车荣晓, 邓永翠, 吴伊波, 等. 生物固氮与有效氮的关系:从分子到群落[J]. 生态学杂志, 2017, 36(1): 224-232. |
| [6] |
康文龙, 台喜生, 李师翁, 等. 祁连山高寒草原碱性土壤固氮微生物数量及固氮基因(nifH)群落结构研究[J]. 冰川冻土, 2013, 35(1): 208-216. |
| [7] |
Rotthauwe JH, Witzel KP, Liesack W, et al. The ammonia monooxygenase structural gene amoA as a functional marker:Molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied & Environmental Microbiology, 1998, 63(12): 4704-4712. |
| [8] |
Francis CA, Roberts KJ, Beman JM, et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688. DOI:10.1073/pnas.0506625102 |
| [9] |
侯海军, 秦红灵, 陈春兰, 等. 土壤氮循环微生物过程的分子生态学研究进展[J]. 农业现代化研究, 2014, 35(5): 588-594. |
| [10] |
Yang Y, Zhang M, Junwei HU, et al. Effects of nitrogen fertilizer application on abundance and community structure of ammonia oxidizing bacteria and archaea in a North China agricultural soil[J]. Acta Ecologica Sinica, 2017, 37(11): 3636-3646. |
| [11] |
Throbäck IN, Enwall K, Jarvis Å, et al. Reassessing PCR primers targeting nirS, nirK, and nosZ, genes for community surveys of denitrifying bacteria with DGGE[J]. FEMS Microbiology Ecology, 2004, 49(3): 401-417. DOI:10.1016/j.femsec.2004.04.011 |
| [12] |
Henry S, Baudoin E, López-Gutiérrez JC, et al. Quantification of denitrifying bacteria in soils by nirK, gene targeted real-time PCR[J]. Journal of Microbiological Methods, 2004, 59(3): 327-335. DOI:10.1016/j.mimet.2004.07.002 |
| [13] |
Henry S, Bru D, Stres B, et al. Quantitative detection of the nosZ gene, encoding nitrous oxide reductase, and comparison of the abundances of 16S rRNA, narG, nirK, and nosZ genes in soils[J]. Applied & Environmental Microbiology, 2006, 72(8): 5181-5189. |
| [14] |
Schmid MC, Hooper AB, Klotz MG, et al. Environmental detection of octahaem cytochrome c hydroxylamine/hydrazine oxidoreductase genes of aerobic and anaerobic ammonium-oxidizing bacteria[J]. Environmental Microbiology, 2008, 10(11): 3140-3149. DOI:10.1111/j.1462-2920.2008.01732.x |
| [15] |
陈宗姮, 徐杉杉, 李祥, 等. pH对厌氧氨氧化反应脱氮效能的影响[J]. 化工环保, 2015, 35(2): 121-126. DOI:10.3969/j.issn.1006-1878.2015.02.003 |
| [16] |
Wang H, Ji G, Bai X. Quantifying nitrogen transformation process rates using nitrogen functional genesin a multimedia biofilter under hydraulic loading rate constraints[J]. Ecological Engineering, 2015, 82: 323-329. DOI:10.1016/j.ecoleng.2015.05.023 |
| [17] |
Xi R, Long XE, Huang S, et al. pH rather than nitrification and urease inhibitors determines the community of ammonia oxidizers in a vegetable soil[J]. AMB Express, 2017, 7(1): 129-142. DOI:10.1186/s13568-017-0426-x |
| [18] |
Samad MS, Biswas A, Bakken LR, et al. Phylogenetic and functional potential links pH and N2O emissions in pasture soils[J]. Scientific Reports, 2016, 6(1): 35990-35998. DOI:10.1038/srep35990 |
| [19] |
Shi H, Wang L, Li X, et al. Genome-wide transcriptome profiling of nitrogen fixation in Paenibacillus sp.WLY78[J]. BMC Microbiology, 2016, 16(1): 25-25. DOI:10.1186/s12866-016-0642-6 |
| [20] |
陈重军, 冯宇, 汪瑶琪, 等. 厌氧氨氧化反应影响因素研究进展[J]. 生态环境学报, 2016, 25(2): 346-352. |
| [21] |
丁龙君.水稻土中异化铁还原与氮循环耦合的微生物机制研究[D].北京: 中国科学院大学, 2014.
|
| [22] |
Chen Y, Jiang Y, Huang H, et al. Long-term and high-concentration heavy-metal contamination strongly influences the microbiome and functional genes in Yellow River sediments[J]. Science of the Total Environment, 2018, 637-638: 1400-1412. DOI:10.1016/j.scitotenv.2018.05.109 |
| [23] |
Xue K, Nostrand JDV, Vangronsveld J, et al. Management with willow short rotation coppice increase the functional gene diversity and functional activity of a heavy metal polluted soil[J]. Chemosphere, 2015, 138: 469-477. DOI:10.1016/j.chemosphere.2015.06.062 |
| [24] |
Wang H, Li X, Li X, et al. Changes of microbial population and N-cycling function genes with depth in three Chinese paddy soils[J]. PLoS One, 2017, 12(12). DOI:10.1371/journal.pone.0189506 |
| [25] |
Cardon ZG, Stark JM, Herron PM, et al. Sagebrush carrying out hydraulic lift enhances surface soil nitrogen cycling and nitrogen uptake into inflorescences[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(47): 18988-18993. DOI:10.1073/pnas.1311314110 |
| [26] |
Stone MM, Kan J, Plante AF. Parent material and vegetation influence bacterial community structure and nitrogen functional genes along deep tropical soil profiles at the Luquillo Critical Zone Observatory[J]. Soil Biology & Biochemistry, 2015, 80(80): 273-282. |
| [27] |
杨扬.铜陵铜尾矿废弃地中参与金属硫化物氧化的微生物主要类群与分布[D].合肥: 安徽大学, 2014.
|
| [28] |
陶巍.某矿区酸性矿山废水污染土壤及湿地处理系统的调查研究[D].合肥: 合肥工业大学, 2017.
|
| [29] |
Delong EF. Archaea in coastal marine environments[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(12): 5685-5689. DOI:10.1073/pnas.89.12.5685 |
| [30] |
Li C, Yan K, Tang L, et al. Change in deep soil microbial communities due to long-term fertilization[J]. Soil Biology & Biochemistry, 2014, 75: 264-272. |
| [31] |
Ellegaard KM, Klasson L, Näslund K, et al. Comparative genomics of wolbachia and the bacterial species concept[J]. PLoS Genetics, 2013, 9(4): e1003381-e1003381. DOI:10.1371/journal.pgen.1003381 |
| [32] |
王杨.不同酸度土壤硝化和反硝化活性的差异[D].大连: 大连交通大学, 2014.
|
| [33] |
袁怡, 黄勇, 李祥, 等. 硫酸盐还原-氨氧化反应的特性研究[J]. 环境科学, 2013, 34(11): 4362-4369. |
| [34] |
Kong L, Jing H, Kataoka T, et al. Diversity and spatial distribution of hydrazine oxidoreductase (hzo) gene in the oxygen minimum zone off Costa Rica[J]. PLoS One, 2013, 8(10): e78275. DOI:10.1371/journal.pone.0078275 |