2. 中国农业科学院研究生院,北京 100081
2. Graduate School of Chinese Academy of Agricultural Sciences, Beijing 100081
硝基酚类化合物是一类重要的化工原料,是精细化工产品生产的重要中间体,被广泛地应用于染料、农药、医药、合成材料、机械和木材防腐等领域[1]。其中,单硝基酚类化合物包括邻硝基苯酚(ortho-nitrophenol,ONP)、间硝基苯酚(meta-nitrophenol,MNP)和对硝基苯酚(para-nitrophenol,PNP)3类,以PNP使用范围最广、使用量最大[2-3]。对硝基苯酚可通过不同途径进入环境,已成为环境中硝基酚类污染物典型代表之一。大多数硝基芳烃化合物有致癌、致畸及致突变作用,一些硝基化合物经过肝脏和肠道微生物代谢还可生成致癌物质和致癌物质的前体[4]。由于对硝基苯酚具有良好的水溶性,导致其很容易随水体流动进行迁移而易进入地下水系统,且在深层土壤和地下水中持留较长时间[5]。同时,由于对硝基苯酚在较低浓度(< 10 μmol/L)条件下,就可解除呼吸链氧化磷酸化过程,从而改变细胞代谢过程,因此对人体健康及环境生物具有较大威胁[6]。对硝基苯酚已被美国环保署(US EPA)列为优先控制污染物之一,在我国也被列入水中优先控制污染物黑名单[7]。
对硝基苯酚高效降解方法的探索一直是国内外研究热点之一,研究较多的包括吸附法、氧化法、生化法以及生物降解法[8-10]。对于生物降解而言,具有较好环境适应性的降解菌的分离为生物降解及环境修复等相关生物技术提供了重要的菌种资源,降解相关基因的克隆与功能验证为降解工程菌的构建及固定化酶技术等提供了重要基因资源。本研究以对硝基苯酚为底物,从长期受生活废水污染的近海滩涂分离获得了高效对硝基苯酚降解菌RL-JY1,对菌株进行了系统的鉴定与环境适应性分析,并对菌株RL-JY1降解对硝基苯酚的代谢途径及相关分子机制进行了探索,以期丰富硝基酚类降生物降解的微生物与基因资源,为硝基酚类化合物的生物降解提供参考。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒本研究所使用的降解菌RL-JY1为本实验室分离获得,目前已保存于广东省微生物菌种保藏中心(保存编号:GDMCC 60494);使用的感受态细胞Escherichia coli DH5αDTA克隆用载体(pMD19-T)均购自TaKaRa公司。
1.1.2 试剂对硝基苯酚(分析纯,95%)购于百灵威科技,溶于色谱纯甲醇(购自Fisher公司)并调整浓度至1×105 mg/L作为储备液,待使用时根据需要添加并调整至目标浓度;酵母提取物、蛋白胨、琼脂粉等配制培养基所需的主要成分均购自Oxoid公司;基因组提取试剂盒、凝胶回收试剂盒、连接转化试剂盒、质粒提取试剂盒等均购自TaKaRa公司;PCR引物合成及DNA测序均委托英潍捷基公司完成;甲醇与乙腈均为色谱纯,购自Fisher公司;其余试剂与耗材均购自国内厂家。
1.1.3 培养基无机盐离子培养基(MSM):(NH4)2SO4 2.0 g/L,MgSO4·7H2O 0.2 g/L,CaCl2 0.01 g/L,KH2PO4 1.5 g/L,K2HPO4 1.5 g/L,Na2HPO4·12H2O 1.5 g/L,NaCl 10.0 g/L,定容至指定体积后灭菌备用。
富集培养基(EM):蛋白胨5 g/L,酵母粉5 g/L,KH2PO4 1.5 g/L,K2HPO4 1.5 g/L,NaCl 10.0 g/L,定容至指定体积后灭菌备用。
LB培养基(LB):胰蛋白胨10 g/L,酵母提取物5 g/L,NaCl 10 g/L,定容至指定体积后灭菌备用。
以上培养基均使用NaOH或HCl调整pH至7.0,添加琼脂粉至1.5%(W/V)即得固体培养基,所有培养基均在121℃灭菌25 min后备用。
1.2 方法 1.2.1 对硝基苯酚降解菌的富集与分离土样和海水样品均采自广东省湛江市霞山区附近海域滩涂(北纬21° 12' 29″,东经110° 24' 55″)。取5 g土样或5 mL水样,加入100 mL新鲜的MSM培养基中,并添加对硝基苯酚至50 mg/L,培养液于30℃,180 r/min条件下培养(避光培养)。培养5 d后,取5 mL的培养液加入100 mL新鲜的MSM培养基中,添加并提高对硝基苯酚至100 mg/L,培养液于30℃,180 r/min条件下培养(避光培养)。重复此操作,每次提高对硝基苯酚浓度50 mg/L至培养液中初始对硝基苯酚浓度达到500 mg/L。
选取黄绿色消失的培养液(对硝基苯酚易溶于水,呈黄绿色),将获得的培养液划线培养至MSM固体培养基(含100 mg/L对硝基苯酚)上,获得平板于30℃条件下避光培养。每12 h对平板进行一次观测,至平板上长出较多单菌落后,挑取单菌落接种至LB液体培养基中培养24 h(30℃,180 r/min)后取1 mL菌液离心(6 000 r/min,5 min),菌体用磷酸盐缓冲液(PBS,pH 7.2)冲洗后再次离心(6 000 r/min,5 min),重复冲洗步骤3次,获得的菌体用于降解能力测定。将获得的菌体接种至10 mL新鲜的MSM培养基中,并添加对硝基苯酚至终浓度100 mg/L,作为处理组;以相同条件下的MSM并添加对硝基苯酚至相同浓度,但不添加菌液,作为对照组。将获得的培养液于30℃,180 r/min条件下培养(避光培养),培养5 d后,取培养液检测对硝基苯酚浓度。
根据测得处理组与对照组中对硝基苯酚的浓度,计算菌株对对硝基苯酚的降解率,计算公式如下:
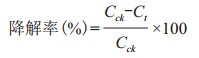
其中,Cck指t时刻对照组中对硝基苯酚的浓度,Ct指t时刻处理组中对硝基苯酚的浓度。取降解率大于90%培养液进行再次划线培养,重复上述过程至获得稳定可降解的单菌为止。
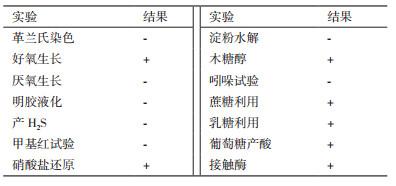
1.2.2 对硝基苯酚降解菌的鉴定基于形态学与生理生化特征分析,结合16S rRNA基因分析对获得的对硝基苯酚降解菌进行鉴定。将获得的降解菌划线接种至LB固体培养基上,于30℃恒温培养24 h后用于菌落形态观察,并参考《常见细菌系统鉴定手册》对菌株进行生理生化特征分析[11]。
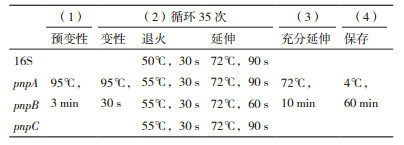
使用细菌基因组小提试剂盒(TaKaRa)提取目标菌株的基因组并用于16S rRNA基因扩增,所使用的16S rRNA基因扩增通用引物27F和1492 R引物序列如表 1所示,PCR反应条件如表 2所示。使用切胶回收试剂盒对PCR产物进行回收,将回收产物连接至pMD19-T载体后转入E. coli DH5α感受态细胞,并通过蓝白斑筛选阳性克隆;对获得的阳性克隆进行质粒提取(质粒小提试剂盒),提取后的质粒送英潍捷基进行测序;测序结果进行BLAST分析并构建系统发育树,使用MEGA 7.0进行系统发育树构建,采用NJ法进行发育树的构建(Bootstrap值为1 000)[12]。
为探索环境因素对降解率的影响,选择了典型的温度、pH及盐度作为研究对象进行探索,具体条件设置,见表 3。将获得的降解菌在LB液体培养基中培养至OD600=0.8后取1 mL菌液进行离心收集菌体(菌体离心与洗涤办法参考1.2.1进行)。所有对照组与处理均12 h取样一次进行对硝基苯酚浓度测定,且所有处理组与对照组均设3次重复,并根据测得对硝基苯酚浓度计算菌株对对硝基苯酚的降解率。
将获得的降解菌接种至含100 mg/L对硝基苯酚的MSM液体培养基(pH 7.0)中,在30℃,180 r/min条件下避光培养,每12 h取样一次进行代谢产物提取与检测。代谢产物的提取参考Samanta的方法[13],具体如下:将获得的培养液进行离心收集上清并加入等体积的乙酸乙酯充分震荡混匀后移取有机相(中性提取),再向剩余的水相中加入盐酸调整pH至2.0后再加入等体积的乙酸乙酯充分震荡混匀后移取有机相(酸性提取),最终将中性提取和酸性提取的有机相混合。通过氮吹仪将混合后的有机相吹干,将获得的沉淀用1 mL的甲醇复溶后用0.22 μm的有机相滤膜过后用于代谢产物的质谱分析。采用LC-MS进行代谢产物的质谱检测,具体条件如下:岛津LC-MS(8040),流动相为甲醇(100%),流速0.2 mL/min,采用直接进样(进样量1μL),在负离子模式下检测,毛细管电压为3.5 kV,载气为氮气(99.9%),扫描范围为100-600 m/z,使用Realtime Analysis对数据进行采集和分析。
1.2.5 对硝基苯酚降解相关基因的克隆根据已报道的对硝基苯酚降解相关基因设计引物,引物名称与序列信息见表 1,以获得的降解菌基因组为模板进PCR扩增,PCR反应条件见表 2。PCR产物经琼脂糖凝胶电泳检测后,对获得的条带进行切胶回收,连接至pMD19-T载体并转入E. coli DH5α感受态细胞,并通过蓝白斑筛选阳性克隆;对获得的阳性克隆进行质粒提取(质粒小提试剂盒,TaKaRa),提取后的质粒送英潍捷基进行测序;测序结果进行BLAST分析并与已有报道的对硝基苯酚降解目的基因进行比对与分析。
1.2.6 对硝基苯酚检测方法的建立基于高效液相色谱法(HPLC)对样品中硝基苯酚的浓度进行测定。首先,建立对硝基苯酚的HPLC的检测方法,具体如下:高效液相色谱仪为安捷伦1260(Agilent,USA),色谱柱为Eclipse Plus C18(4.6×75 mm,3.5 μm),流动相为甲醇、乙腈和水的混合物(80:10:10),流速3 mL/min,柱温30℃,进样量20 μL,检测器为可变波长检测器(VWD),检测波长为320 nm。通过配制标准浓度的对硝基苯酚溶液,并建立与320 nm处吸收值关系的标准曲线(R2均大于0.99)。对硝基苯酚的HPLC检测结果与标准曲线如图 1所示。所有样品均需经孔径为0.2 μm的滤膜过滤后,再经HPLC检测。

|
| A:5-20 mg/L间的标准曲线,B:20-100 mg/L间的标准曲线,C:HPLC对对硝基苯酚的检测结果 图 1 对硝基苯酚的HPLC检测结果与标准曲线 |
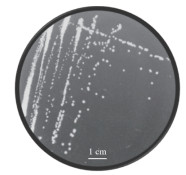
经多次富集与筛选,分离获得一株可高效降解对硝基苯酚的菌株,命名为RL-JY1,该菌株可以对硝基苯酚为唯一碳源,在72 h内对100 mg/L对硝基苯酚的降解率为100%。该菌在LB平板上呈乳白色,圆形,表面光滑湿润,菌落边缘光滑(图 2),菌株的生理生化特征如表 4所示。
|
| 图 2 菌株RL-JY1在LB平板上的菌落形态 |
经PCR扩增、连接、转化及测序后,获得了一段长度为1 495 bp的16S rRNA基因的部分序列,序列信息已递交至GenBank(登录号为:MK101056),通过BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)与已报道的序列进行比对,初步判断菌株RL-JY1为假单胞菌属(Pseudomonas sp.)细菌。根据BLAST的初步比对结果,从LPSN(List of Prokaryotic names with Standing in Nomenclature,http://www.bacterio.net/index.html)数据库下载相关菌属细菌16S rRNA基因序列,用于系统发育树的构建,16S rRNA基因序列分析结果(图 3)显示菌株RL-JY1与恶臭假单胞菌(P. putida)亲缘关系较近。根据16S rRNA基因序列分析结合生理生化特征分析,确定菌株RL-JY1为恶臭假单胞菌。
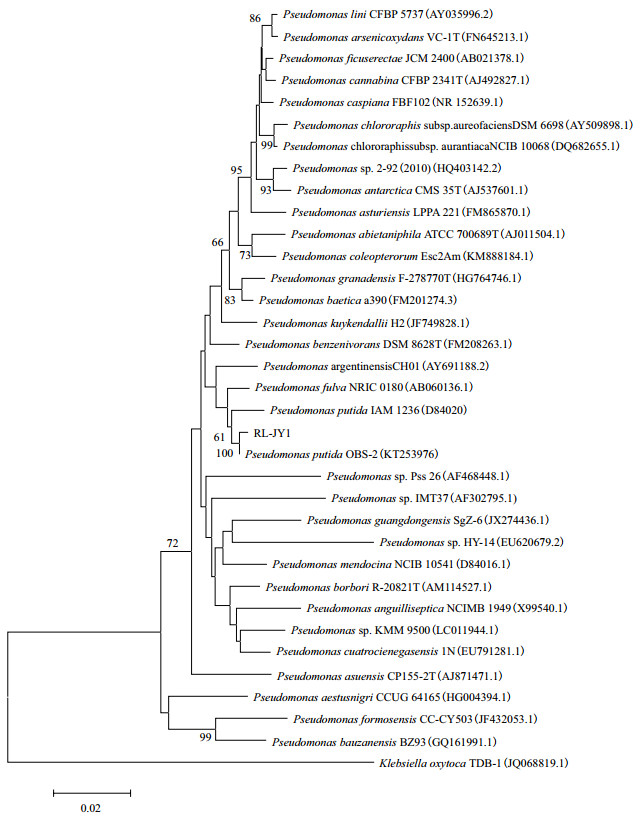
|
| 图 3 菌株RL-JY1的16S rRNA基因系统发育分析 |
温度对菌株RL-JY1降解对硝基苯酚的影响如图 4-A所示,可以看出菌株RL-JY1具有较宽的温度耐受范围,在20-40℃间,对100 mg/L对硝基苯酚的72 h降解率均大于95%(20℃时为96.3%,30℃与40℃均为100%);当温度为10℃或50℃时菌株对对硝基苯酚的降解受到显著抑制(47.3%,10℃;68.5%,50℃)。不同初始pH对菌株RL-JY1降解对硝基苯酚的影响如图 4-B所示,菌株RL-JY1在pH 5-7范围内,随着pH值的升高,菌株对对硝基苯酚的降解率(72 h,100 mg/L)逐渐升高,当pH为7.0时,72 h内对100 mg/L对硝基苯酚降解率为100%;当pH值超过7.0以后,随着pH值的上升菌株对对硝基苯酚的降解率逐渐下降,当pH值为10.0时,72 h对100 mg/L对硝基苯酚的降解率为52.7%;值得注意的是,在pH 5.0、6.0、8.0和9.0条件下,菌株72 h对对硝基苯酚(100 mg/L)的降解率均大于60%(pH为5.0时降解率为46.2%,pH为6.0时降解率为63.7%,pH为8.0时降解率为96.2%,pH为9.0时降解率为72.3%),由此可判断菌株RL-JY1对pH有较宽的耐受范围,并对酸性条件具有较好的耐受能力。盐度对菌株RL-JY1降解对硝基苯酚的影响如图 4-C所示,在NaCl浓度为0-8%范围内,对硝基苯酚的降解率无明显变化,当NaCl浓度超过8%后,随着NaCl浓度升高,降解率逐渐下降(NaCl浓度达为10%时,降解率为71.4%;NaCl浓度达为12%时,降解率为43.6%)。
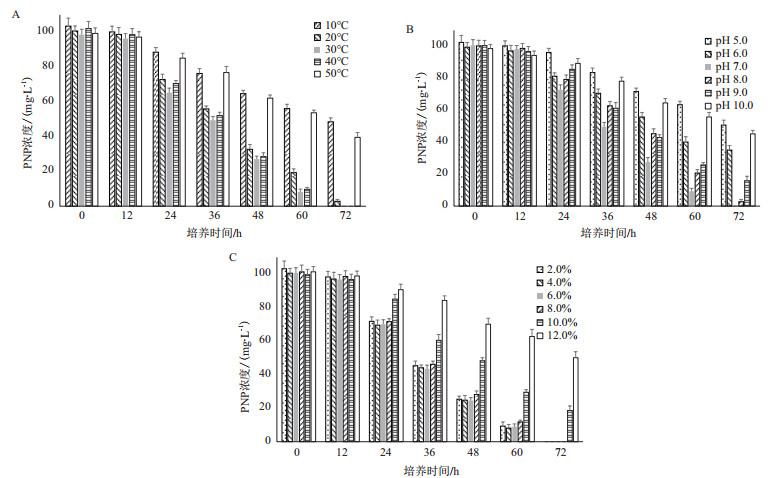
|
| A:温度对PNP降解的影响;B:初始pH对PNP降解的影响;C:盐度对PNP降解的影响 图 4 环境因素对菌株RL-JY1降解对硝基苯酚的影响 |
通过对不同培养时间点的样品进行质谱分析,获得了菌株RL-JY1降解对硝基苯酚的代谢产物信息(图 5-A);结合已有对硝基苯酚降解相关报道,推测质谱检测获得的对硝基苯酚降解产物包括4-硝基儿茶酚(m/z=153.8000)、偏苯三酚(m/z=125.1000)和马来酰乙酸(m/z=157.1000),进一步提出菌株RL-JY1降解对硝基苯酚的代谢途径如图 5-B所示,即典型的偏苯三酚途径。
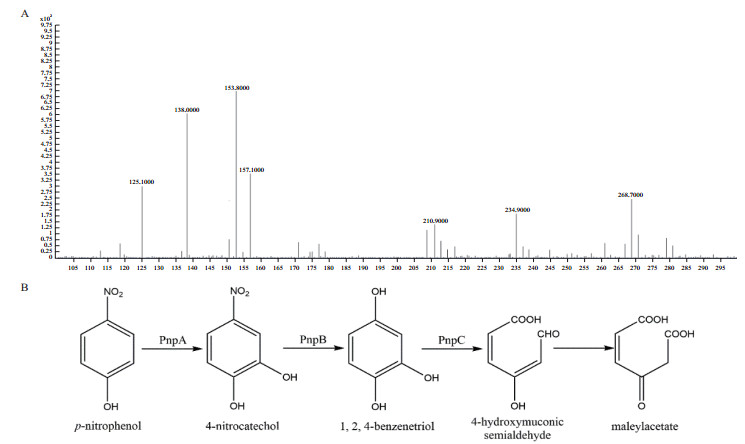
|
| A:24 h时培养液中代谢中间产物检测;B:菌株RL-JY1降解对硝基苯酚的代谢途径及相关基因 图 5 菌株RL-JY1降解对硝基苯酚代谢中间产物检测与代谢途径分析 |
以提取的菌株RL-JY1基因组为模板,通过设计的引物进行PCR扩增并对PCR产物进行电泳检测,结果如图 6所示。其中,pnpABC基因的PCR产物电泳检测结果为阳性,且条带大小与目标片段大小一致。对目标条带进行切胶回收、连接、转化并送测序。对测序片段进行BLAST比对分析,并与已有报道的pnpABC基因序列进行比对,结果显示,来自于菌株RL-JY1中的pnpA、pnpB和pnpC基因与恶臭假单胞菌DLL-E4的pnpA、pnpB和pnpC基因的序列一致性分别为99%、98%和100%,翻译后的氨基酸序列相似性均为100%。同时,基于获得的菌株RL-JY1对对硝基苯酚的代谢途径,结合已有报道,确定了获得的参与对硝基苯酚降解的相关基因的特定功能(图 5-B)。

|
| M:DL2000 DNA marker;1:pnpA,2:pnpB,3:pnpC 图 6 对硝基苯酚降解相关基因pnpABC的电泳检测 |
对硝基苯酚作为硝基酚类化合物中的典型代表,伴随其大量使用,其在环境中被广泛检出,同时,其对人体健康及环境安全的影响也得到了广泛和深入的研究。探寻高效、安全且经济的环境污染治理方法,是广大科研工作者孜孜以求的目标。针对对硝基苯酚的生物降解,目前已有较多的降解菌被分离报道,代谢途径(主要包括通过偏苯三酚和邻苯二酚介导的开环代谢途径)及相关分子机制(典型的如pnp基因簇与npd基因簇)也已得到了一定程度的研究[14-17]。但需要指出的是,具有良好环境适应性、恶劣条件耐受能力菌株的降解菌报道仍然较少。因此,分离获得具有良好环境适应性的降解菌,为环境修复及工业废水处理提供新的降解菌资源具有重要意义。同时,对于菌株降解对硝基苯酚代谢途径与相关分子机制的阐明,为菌株的改造与工程化应用提供了理论参考。
本研究从长期受城市废水污染的近海滩涂的海水与淤泥样品中,经过多次富集、驯化,分离获得一株可高效降解对硝基苯酚的细菌RL-JY1,经菌落形态、生理生化特征及16S rRNA基因分析,确定该菌为恶臭假单胞菌。目前,已报道的可降解对硝基苯酚的细菌种属包括红球菌属(Rhodococcus sp.)[18]、劳尔氏菌属(Ralstonia sp.)[19]、无色杆菌属(Achromobacter sp.)[20]、节杆菌属(Arthrobacter sp.)[21]、假单胞菌属[22]、莫拉克斯氏菌属(Moraxella sp.)[23]、芽孢杆菌属(Bacillus sp.)[24]等,其中节杆菌属和假单胞菌属分别是革兰氏阳性与革兰氏阴性细菌的典型代表。
在环境适应性方面,菌株RL-JY1对环境温度与pH具有良好的耐受能力,对温度与pH都具有较宽的耐受范围。在相对较高温度(50℃)时,菌株RL-JY1对对硝基苯酚仍具有较强降解能力(68.5%),这可能是由于菌株RL-JY1分离自常年气温较高的雷州半岛(中国大陆最南端),长期的环境驯化使得菌株对于环境温度表现出较好的耐受能力;菌株对于环境pH较宽的耐受范围对于菌株RL-JY1在环境污染修复及工业废水处理中的应用具有重要意义;同时,菌株RL-JY1表现出对盐度良好的适应性,在一定程度是由于样品采自沿海滩涂(沿海滩涂由于水分的蒸发,而盐分留在土壤中,盐度在不同时间略有变化)。目前,不论是环境污染修复还是工业废水处理,都面临着相同的问题,即环境因素复杂且处理条件恶劣,如环境温度变化较大、工业废水处理条件复杂(污染物浓度较高、高盐及极端pH)等。环境适应性分析的结果表明,菌株RL-JY1具有良好的环境适应能力与应用前景。
已有关于对硝基苯酚降解途径与相关分子机制的报道中,对节杆菌属与假单胞菌属的研究较为深入,也分别是革兰氏阳性与革兰氏阴性细菌的典型代表,且代谢途径与相关基因也可以革兰氏阳性与革兰氏阴性细菌为区分归类讨论。已报道的对硝基苯酚的降解途径主要有两类:(1)通过对苯二醌将对硝基苯酚转化为对苯二酚并进一步被开环利用,因此也被称作对苯二酚代谢途径(Hydroquinone,HQ),这种代谢途径在革兰氏阴性细菌中报道较多[25];(2)通过4-硝基儿茶酚将对硝基苯酚转化为偏苯三酚并进一步开环利用,因此也称作偏苯三酚代谢途径(1,2,4-Benzenetriol,BT),这种代谢途径在革兰氏阳性细菌与阴性细菌中均有报道[16, 26]。而对应的参与对硝基苯酚降解的相关基因也非常保守,革兰氏阳性细菌中以节杆菌为代表,由双组分的氧化还原酶将对硝基苯酚转化为偏苯三酚,偏苯三酚再由偏苯三酚双加氧酶开环生成马来酰乙酸,相关基因簇已经得到较深入的研究;同时,值得注意的是已报道的节杆菌属细菌除了可以降解对硝基苯酚,还可降解其他对位取代酚,如对氯苯酚和对氟苯酚[27-28]。而在革兰氏阴性细菌中,以假单胞菌属为代表,可以同时通过偏苯三酚和对苯二酚对对硝基苯酚进行降解,其中的氧化还原酶可将对硝基苯酚转化为偏苯三酚或对苯二酚,对苯二酚的降解主要是通过对苯二酚双加氧酶进行开环,而偏苯三酚则通过偏苯三酚双加氧酶开环,同样,相应的基因/基因簇在革兰氏阴性细菌中的也较为保守。本研究中获得的降解菌RL-JY1为具有代表性的恶臭假单胞菌,通过质谱分析与基因克隆手段明确了其遗传背景与代谢途径相,为具有良好环境适应性与高效降解潜能工程菌的构建提供了重要的理论支撑。
4 结论从长期受城市废水污染的近海滩涂分离获得了一株对硝基苯酚降解菌RL-JY1,通过生理生化特征分析与16S rRNA基因分析,确定该菌株为恶臭假单胞菌。菌株RL-JY1对于环境温度、pH及盐离子浓度都具有良好的适应性。基于代谢产物分析确定菌株RL-JY1通过偏苯三酚途径对对硝基苯酚进行降解。同时,基于对已有报道的对硝基苯酚降解相关基因设计引物,克隆获得了参与对硝基苯酚降解上游途径的相关基因。本研究从菌株特性、代谢途径及相关分子机制3个水平系统阐明了菌株RL-JY1对对硝基苯酚的降解机理,结果表明菌株对对硝基苯酚具有高效的降解能力、良好的环境适应性和较好的应用潜能。
| [1] |
佘宗莲, 饶兵, 樊玉清, 等. 废水中硝基酚类化合物生物降解的研究进展[J]. 环境工程学报, 2007, 1(7): 1-9. DOI:10.3969/j.issn.1673-9108.2007.07.001 |
| [2] |
陈晨, 张艳艳, 沈锦优. 生物电化学系统硝基酚降解与其结构关系研究[J]. 水处理技术, 2019(2): 48-50. |
| [3] |
任磊, 史延华, 贾阳, 等. 菌株Arthrobacter sp. CN2降解对硝基苯酚的特性与动力学[J]. 环境科学, 2015(5): 1757-1762. |
| [4] |
雷忻, 钟瑞, 刘利娅, 等. 对硝基苯酚对泥鳅肝组织结构及丙氨酸转氨酶活性的影响[J]. 西北农林科技大学学报:自然科学版, 2013(9): 15-20. |
| [5] |
刘星邑, 温玉娟, 刘欢, 等. 土壤中对硝基酚迁移转化和去除技术研究进展[J]. 农业环境科学学报, 2017, 11: 2161-2170. DOI:10.11654/jaes.2017-0608 |
| [6] |
万年升, 顾继东, 郝伏勤, 等. Rhodococcus sp. Ns对硝基苯酚的好氧生物降解[J]. 环境科学, 2007(2): 431-435. DOI:10.3321/j.issn:0250-3301.2007.02.039 |
| [7] |
EPA 440580063. Ambient quality for nitrophenols[S]. Washin-gton DC. 1980.
|
| [8] |
史延华, 任磊, 贾阳, 等. 施氏假单胞菌YC-YH1对甲基对硫磷的降解及其代谢产物检测[J]. 农业环境科学学报, 2015, 11: 2097-2104. DOI:10.11654/jaes.2015.11.009 |
| [9] |
洪昌红, 黄本胜, 邱静, 等. 对硝基酚在超高交联吸附树脂NDA-701上的脱附行为研究[J]. 环境科学, 2011, 5: 1388-1393. |
| [10] |
张爱丽, 邓芳芳, 周集体, 等. 粉煤灰催化H2O2氧化水中对硝基酚研究[J]. 环境科学, 2009(7): 1942-1948. DOI:10.3321/j.issn:0250-3301.2009.07.012 |
| [11] |
东秀珠. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001.
|
| [12] |
Kumar S, Stecher G, Tamura K. MEGA7:Molecular evolutionary genetics analysis version 7. 0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [13] |
Samanta SK, Bhushan B, Chauhan A, et al. Chemotaxis of a Ralstonia sp. SJ98 toward different nitroaromatic compounds and their degradation[J]. Biochemical and Biophysical Research Communications, 2000, 269(1): 117-123. DOI:10.1006/bbrc.2000.2204 |
| [14] |
Nielsen MB, Ingvorsen K. Biodegradation of para-nitrophenol by Citricoccus nitrophenolicus strain PNP1(T)at high pH[J]. Biodegradation, 2013, 24(1): 79-87. DOI:10.1007/s10532-012-9559-4 |
| [15] |
Vikram S, Pandey J, Kumar S, et al. Genes involved in degradation of para-nitrophenol are differentially arranged in form of non-contiguous gene clusters in Burkholderia sp. strain SJ98[J]. PLoS One, 2013, 8(12): e84766. DOI:10.1371/journal.pone.0084766 |
| [16] |
Zhang S, Sun W, Xu L, et al. Identification of the para-nitrophenol catabolic pathway, and characterization of three enzymes involved in the hydroquinone pathway, in Pseudomonas sp. 1-7[J]. BMC Microbiology, 2012, 12(1): 27. DOI:10.1186/1471-2180-12-27 |
| [17] |
Shen W, Liu W, Zhang J, et al. Cloning and characterization of a gene cluster involved in the catabolism of p-nitrophenol from Pseudomonas putida DLL-E4[J]. Bioresource Technology, 2010, 101(19): 7516-7522. DOI:10.1016/j.biortech.2010.04.052 |
| [18] |
Gemini VL, Gallego A, de Oliveira VM, et al. Biodegradation and detoxification of p-nitrophenol by Rhodococcus wratislaviensis[J]. International Biodeterioration & Biodegradation, 2005, 55(2): 103-108. |
| [19] |
Pandey G, Chauhan A, Samanta SK, et al. Chemotaxis of a Ralstonia sp. SJ98 toward co-metabolizable nitroaromatic compounds[J]. Biochemical and Biophysical Research Communications, 2002, 299(3): 404-409. DOI:10.1016/S0006-291X(02)02601-3 |
| [20] |
Wan N, Gu JD, Yan Y. Degradation of p-nitrophenol by Achromo-bacter xylosoxidans Ns isolated from wetland sediment[J]. International Biodeterioration & Biodegradation, 2007, 59(2): 90-96. |
| [21] |
Liu P, Zhang J, Zhou N. Characterization and mutagenesis of a two-component monooxygenase involved in para-nitrophenol degradation by an Arthrobacter strain[J]. International Biodeterioration & Biodegradation, 2010, 64(4): 293-299. |
| [22] |
Zheng YL, Liu DL, Xu H, et al. Biodegradation of p-nitrophenol by Pseudomonas aeruginosa HS-D38 and analysis of metabolites with HPLC-ESI/MS[J]. International Biodeterioration & Biodegradation, 2009, 63(8): 1125-1129. |
| [23] |
Mulchandani P, Hangarter CM, Lei Y, et al. Amperometric microbial biosensor for p-nitrophenol using Moraxella sp. -modified carbon paste electrode[J]. Biosens Bioelectron, 2005, 21(3): 523-527. DOI:10.1016/j.bios.2004.11.011 |
| [24] |
Sreenivasulu C, Megharaj M, Venkateswarlu K, et al. Degradation of p-nitrophenol by immobilized cells of Bacillus spp. isolated from soil[J]. International Biodeterioration & Biodegradation, 2012, 68: 24-27. |
| [25] |
Perry LL, Zylstra GJ. Cloning of a gene cluster involved in the catabolism of p-nitrophenol by Arthrobacter sp. strain JS443 and characterization of the p-nitrophenol monooxygenase[J]. Journal of Bacteriology, 2007, 189(21): 7563-7572. DOI:10.1128/JB.01849-06 |
| [26] |
Wang J, Ren L, Jia Y, et al. Degradation characteristics and metabolic pathway of 4-nitrophenol by a halotolerant bacterium Arthrobacter sp. CN2[J]. Toxicological and Environmental Chemistry, 2016, 98(2): 226-240. DOI:10.1080/02772248.2015.1115507 |
| [27] |
Nordin K, Unell M, Jansson JK. Novel 4-chlorophenol degradation gene cluster and degradation route via hydroxyquinol in Arthrobacter chlorophenolicus A6[J]. Appl Environ Microbiol, 2005, 71(11): 6538-6544. DOI:10.1128/AEM.71.11.6538-6544.2005 |
| [28] |
Ferreira MIM, Iida T, Hasan SA, et al. Analysis of two gene clusters involved in the degradation of 4-fluorophenol by Arthrobacter sp. strain IF1[J]. Appl Environ Microbiol, 2009, 75(24): 7767-7773. DOI:10.1128/AEM.00171-09 |