2. 南开大学环境污染过程与基准教育部重点实验室,天津 300350
2. Ministry of Education Key Laboratory of Pollution Processes and Environmental Criteria, Nankai University, Tianjin 300350
聚丙烯酰胺(Polyacrylamide,PAM)以其相对高分子量、高黏度的特性,在石油工业、水处理、造纸业和化学工业等具有广泛的应用[1-3],其中石油行业是我国PAM使用量最大的领域[4]。PAM作为三次采油的驱油剂注入地下后,产生大量含有高浓度聚合物的产出水,严重影响了后续油水分离过程[5]。虽然PAM本身无毒,但其单体丙烯酰胺能损伤人和动物神经系统[6]。
PAM在水环境中的迁移和积累对生物及人体健康具有潜在影响,因此亟待开展PAM的高效降解研究[7]。微生物降解是自然环境中有机污染物完全矿化的主要方法。微生物可通过利用或合成酶类、非蛋白质类和胞外其他物质等分解PAM[8-9]。PAM可作为氮源被微生物利用[10]。PAM也可作为碳源进行微生物生长[11-12]。本实验从受PAM污染的污泥中筛选出一株能以PAM作为唯一碳、氮源的降解菌株,研究了该菌株的降解特性,相关结果可以为受PAM污染环境的修复提供参考。
1 材料与方法 1.1 材料 1.1.1 样品及试剂菌种来源:陕蒙交界地区的大牛地油田含聚丙烯酰胺污泥。聚丙烯酰胺:分析纯,购自成都科龙化工试剂厂,分子量3×103 kD。
1.1.2 培养基营养培养基:酵母浸粉5 g/L,蛋白胨10 g/L,氯化钠5 g/L,调pH为7。基础培养基:聚丙烯酰胺,酵母浸粉0.05 g/L,氯化钠0.5 g/L,磷酸氢二钾1 g/L,七水合硫酸镁0.2 g/L,琼脂15 g/L(固体培养基),调pH为7。富集培养基:聚丙烯酰胺0.3 g/L,酵母浸粉5 g/L,胰蛋白胨10 g/L,氯化钠10 g/L,琼脂15 g/L(固体培养基),调pH为7。聚合物培养基:聚丙烯酰胺1.2 g/L,氯化钠5 g/L,磷酸氢二钾0.5 g/L,磷酸二氢钾0.5 g/L,七水合硫酸镁0.2 g/L,pH为7。牛肉膏蛋白胨培养基:牛肉膏3 g/L,蛋白胨10 g/L,氯化钠5 g/L,琼脂15 g/L(固体培养基),调pH为7。
实验所用试剂均为分析纯,购自成都科龙化工试剂厂。
1.2 方法 1.2.1 降解菌株的驯化及分离取3 g污泥样品接种于100 mL营养培养基中,在30℃、140 r/min的条件下培养48 h,富集污泥中微生物。后取5 mL菌液接种于100 mL基础培养基中,其中PAM浓度按梯度依次为0.6 g/L、0.9 g/L、1.2 g/L、1.5 g/L、1.8 g/L和2.0 g/L逐渐增大,在相同条件下培养7 d后吸取5 mL驯化液转接到新的基础培养基培养,驯化周期为7 d,连续驯化3个周期后,分别吸取1 mL菌液涂布到富集培养基平板上,30℃下倒置培养24 h-48 h,挑取平板上长出的不同菌落进行划线纯化,保存分离菌株。
在基础培养基的前提下,去掉酵母浸粉,分别加入葡萄糖、氯化铵和不加营养物质配置出限制氮源培养基、限制碳源培养基、限制营养源培养基。
1.2.2 菌株鉴定将纯化后菌液均匀涂在牛肉膏蛋白胨培养基上恒温培养24 h-48 h后,选择分布适中的单菌落进行观察:大小与形状、隆起、边缘、表面、硬度、透明度等。
使用TIANGEN细菌基因组DNA试剂盒提取菌株DNA,利用通用引物27F(5'-AGAGTTTGATCC-TGGCTCAG-3')和1492R(5'-GGTTACCTTGTTACGT- TACGACTT-3')对细菌16S rDNA基因进行PCR扩增,PCR扩增条件为94℃下预变性4 min;94℃变性1 min,53℃退火90 s,72℃延伸2 min,30个循环;72℃延伸10 min。扩增产物送至上海生工生物公司进行测序,所得结果经NBCI数据库比对后,挑选相关序列,采用MEGA 5.0软件构建系统发育树。
1.2.3 PAM浓度的影响将聚合物培养基中PAM的浓度依次调整为0.3 g/L、0.6 g/L、1.2 g/L、2.4 g/L、3.6 g/L、4.8 g/L、7.2 g/L和9.6 g/L。将挑选出的菌种接种至培养基中,放入摇床中(30℃,140 r/min)震荡培养48 h后,分别测定细菌浓度和PAM降解率。利用淀粉-碘化镉测定PAM浓度[13]。
1.2.4 培养温度的影响将菌种接种至聚合物培养基中,分别在20、25、30、35和40℃水浴恒温振荡器中培养,140 r/min,震荡培养48 h后,分别测定细菌浓度和PAM降解率。
1.2.5 培养基初始pH的影响将聚合物培养基初始pH值分别调节为3.5、4.5、5.5、6.8、7.5、8.5和9.5,并接种菌种至培养基中,放入摇床(35℃,140 r/min)震荡培养48 h后,分别测定细菌浓度和PAM降解率。
1.2.6 降解曲线的测定在上述所测量的最适PAM初始浓度和pH基础上,PCX接种到以PAM为唯一营养源的培养基中,放入摇床(35℃,140 r/min)震荡培养48 h后,分别测定细菌浓度和PAM降解率。
1.2.7 PAM降解产物分析将降解后的培养液6 000 r/min离心5 min,弃沉淀保留上清液,然后上清液和无水乙醇以1:2体积比震荡混合均匀,静置10 min后以3 000 r/min离心15 min,收集沉淀。沉淀物真空干燥后即得待测样品。凝胶渗透色谱测定降解产物的分子量,采用安捷伦PLGPC220色谱分析仪,以四氧呋喃(THF)为流动相,测定样品的分子量。利用傅里叶变换-红外光谱仪对PAM及其降解产物进行分析,分析波长介于400-4 000 cm-1之间。本实验采用费氏弧菌(Vibrio fischeri NRRL B-11177)测定聚丙烯酰胺降解产物的生物急性毒性,利用发光细菌分析仪BioFix® Lumi-10测定细菌光强度的变化,用SPSS软件的Probit工具计算PAM降解产物的半数效应浓度(EC50)。
2 结果 2.1 降解菌株的分离筛选从分离菌株中筛选出3株菌,分别命名为PCS、PCX、PW,为了研究3株菌对PAM的利用,将3种细菌的菌悬浮液分别接种到限制氮源培养基、限制碳源培养基、限制营养源(碳源、氮源)培养基。当PAM作为唯一氮源时,如图 1所示,PCS、PCX、PW对PAM的降解率分别为42%、37%和31%,普遍高于PAM为碳源和PAM为唯一营养源的降解效果;同时3株降解菌的细菌浓度分别为2.13×108 CFU/mL、2.69×108 CFU/mL、2.44×108 CFU/mL,同样高于PAM为碳源和PAM为唯一营养源的培养基。当PAM作为唯一碳源和唯一碳、氮源时仅剩菌株PCX生长情况最好,细菌浓度大于108 CFU/mL,对PAM的降解率仍大于30%,因此选择PCX作为后期实验菌种。
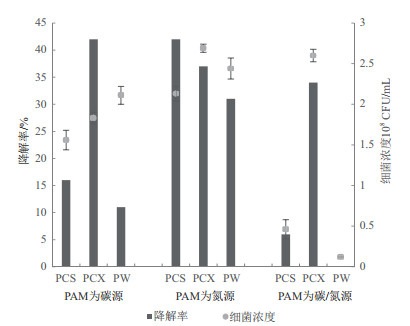
|
| 图 1 PAM为碳源、氮源、同时为碳氮源条件下3株菌的降解率 |
由菌落形态和个体形态观察发现,该细菌菌落形态特性呈现为:圆形、乳白色、有光泽、整齐、光滑及不透明;个体形态特性为:短杆状、革兰氏染色G-、无芽孢和无荚膜。进一步通过对PCX的16S rDNA检测发现:PCX的16S r DNA序列(GenBank登录号MK571603),与Klebsiella sp. NBRC 100048(登录号AB681138.1)有99.9%的相似度;根据测序结果,利用NCBI提供的Blast工具,经过16S rDNA测序及同源性对比,通过MEGA 5.5邻接法(Neighbour-joiningmethod,NJ)构建出系统发育树如图 2所示,因此鉴定菌株PCX为克雷伯氏杆菌属。
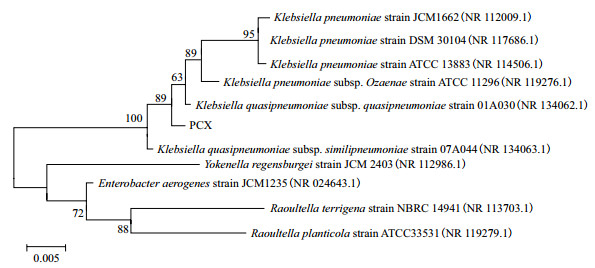
|
| 图 2 降解菌PCX基于16S rDNA序列的系统发育进化树 |
如图 3所示,当PAM初始浓度在1.2 g/L时,培养48 h后,PCX对PAM的降解率最高,达到35%。但当PAM的浓度大于3 g/L时,PAM的降解率受到抑制,基本低于20%。

|
| 图 3 PAM浓度对菌株生长和降解率的影响 |
培养温度对降解效果和菌体的生长的影响如图 4所示。在20-35℃条件下,随着温度的升高,PAM的降解率升高。在35℃时,PCX生长状况和降解效率最好,分别达到了2.44×108 CFU/mL和43%。因此选取35℃作为PCX的最适培养温度。

|
| 图 4 温度对降解的影响 |
如图 5所示,初始pH值的不同对PAM溶液中的PCX的生长产生了一定的影响,同时过高或过低的pH值都不利于PAM的降解,对于本实验的菌株PCX,适宜降解PAM的pH值范围是5.0-7.0,最适pH为7.0。
|
| 图 5 初始pH值对降解的影响 |
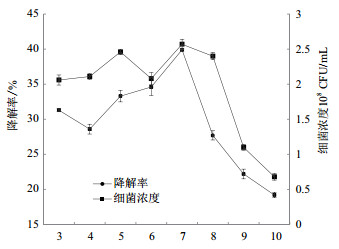
PCX接种到以PAM为唯一营养源的培养基中,随时间变化的生长曲线和降解曲线,如图 6所示。PCX在接种后2 h进入对数增长期,菌体数量不断增加,PAM降解率也随之明显升高,说明PCX能以PAM作为唯一营养源生长,同时使得PAM发生降解。培养40 h后细菌进入稳定期,细菌代谢产物不断积累,细菌繁殖速度下降,PAM降解率趋于平缓,降解率达43%左右。

|
| 图 6 降解菌PCX的降解曲线 |
聚丙烯酰胺经过降解菌PCX降解后的产物的凝胶色谱结果如表 1所示,降解产物的数均分子量(Mn)、重均分子量(Mw)和峰位分子量(Mp)分别为0.602 kD、0.647 kD和0.619 kD。聚丙烯酰胺降解前的分子量为3×103 kD,表明在微生物的作用下,PAM主碳链发生断裂,被降解成为小分子物质。
对微生物降解前后的PAM进行了红外光谱分析,如图 7所示,与生物降解前PAM样品的光谱中的峰相比,生物降解后的PAM样品的光谱中的C=O伸缩振动峰由1 665 cm-1移动到更高波数的位置1 672 cm-1处,2 925 cm-1为亚甲基反对称伸缩振动的特征吸收峰,2 854 cm-1为亚甲基对称伸缩振动的特征吸收峰。红外光谱分析表明C-N伸缩振动峰(降解前吸收峰在1 405 cm-1处)和N-H弯曲振动(降解前吸收峰在1 452 cm-1处)消失,3 300-3 500 cm-1波长范围内变成了一个宽大的吸收峰,表明PAM的酰胺基从PAM主链上剥落并被微生物利用,酰胺基通过微生物的降解完全转化为羧基。

|
| 图 7 PAM降解前后的红外光谱图 |
半数效应浓度(EC50)是导致发光细菌发光强度产生50%变化时的测试样品的浓度。用SPSS软件计算PAM降解产物的生物急性毒性,软件分析结果表明聚丙烯酰胺降解产物在30 min内对费希尔弧菌的EC50为106 mg/L,其95%置信区间为28-489。根据欧盟指令(Directive93/67/EEC)给出的化学品水生生物急性毒性分类规则,降解产物的半数致死浓度大于100 mg/g无毒性[14],可以判定克雷伯氏菌降解PAM的产物对水生生物无毒害作用,表明降解产物中可能无丙烯酰胺单体。
3 讨论从若干分离菌株中筛选出3株菌(PCS、PCX和PW),PAM作氮源时,降解菌的生长状况和对PAM的降解率普遍优于PAM做碳源和PAM为唯一营养源时;说明PAM更易于被微生物作为氮源利用。但对于PAM来说,只有当它作为碳源被利用时才更有利于聚合物的解链和彻底降解。在本研究中当菌株生长在限制碳源和限制营养源培养基中时,菌株PCX生长情况最好,细菌浓度大于108 CFU/mL,对PAM的降解率仍大于30%,说明该菌株可以将PAM既作为氮源又作为碳源而进行利用。
PCX对PAM的适宜浓度是1.2-2.4 g/L,当培养基中PAM浓度较高时,体系的黏度也会上升,影响PCX菌体的扩散,同时PAM自身的絮凝、架桥作用使得微生物更难利用[15-16]。在高PAM浓度下,PCX需要更长的时间来适应环境。相反地,当PAM的浓度低于1.2 g/L时,PCX菌体的增殖速率略缓,最终菌体数量较低。温度对PAM降解率的影响较大。随着培养温度上升使酶促反应加快,有助于菌株PCX的代谢生长,利于微生物对底物PAM的降解[17]。不同的微生物在其不同的生长阶段和生理、生化过程中,最适宜的pH值也不同[18]。pH在5.0-7.0范围内,还原酶、胞外酰胺酶活性最好,对于底物的利用率最高,从而对PAM的降解效果最好[19-20]。
通过对PAM降解产物的生物急性毒性评价发现,降解产物对水生生物无毒害作用。红外光谱分析表明聚丙烯酰胺在降解菌PCX的作用下,酰胺基已经转化为羧基被PCX作为氮源利用。PAM可同时被降解菌PCX作为碳源和氮源加以利用,将其分解代谢为小分子物质。克雷伯氏菌属在降解聚丙烯酰胺聚合物上具有一定的应用前景,为后续处理难降解聚合物提供新的思路。
4 结论本研究从聚丙烯酰胺污染的污泥中初步筛选出3株好氧的PAM降解菌种,其中PCX在限制碳源和氮源的培养基中生长情况最好,对PAM的降解率达到了43%。PCX的形态特征、生理生化鉴定和16S rDNA序列鉴定的结果表明,PCX为克雷伯氏菌属,命名为Klebsiella sp. PCX。
| [1] |
Lu S, Liu R, Sun X. A study on the synthesis and application of an inverse emulsion of amphoteric polyacrylamide as a retention aid in papermaking[J]. Journal of Applied Polymer Science, 2010, 84(2): 343-350. |
| [2] |
Montorsi L, Milani M, Venturelli M. Economic assessment of an integrated waste to energy system for an urban sewage treatment plant:a numerical approach[J]. Energy, 2018, 158: 105-110. DOI:10.1016/j.energy.2018.06.026 |
| [3] |
Biswas P, Majumdar U, Ghosh S. Analysis of reverse transcribed mRNA using PCR and polyacrylamide gel electrophoresis[J]. Methods Mol Biol, 2018, 1721: 73-87. |
| [4] |
Khadom AA, Abdul-Hadi AA. Performance of polyacrylamide as drag reduction polymer of crude petroleum flow[J]. Ain Shams Engineering Journal, 2014, 5(3): 861-865. DOI:10.1016/j.asej.2014.04.005 |
| [5] |
张建晔, 金龙渊, 张跃虎. 聚丙烯酰胺在油田三次采油的应用[J]. 化工管理, 2018, 489(18): 42. DOI:10.3969/j.issn.1008-4800.2018.18.034 |
| [6] |
苗家兵, 郭昭华, 王永旺, 等. 聚丙烯酰胺类复合材料吸附废水中Pb2+的研究进展[J]. 工业水处理, 2018, 38(6): 6-11. |
| [7] |
Gilbert WJR, Johnson SJ, Tsau JS, et al. Enzymatic degradation of polyacrylamide in aqueous solution with peroxidase and H2O2[J]. Journal of Applied Polymer Science, 2017. DOI:10.1002/app.44560 |
| [8] |
Bao M, Chen Q, Li Y, Jiang G. Biodegradation of partially hydrolyzed polyacrylamide by bacteria isolated from production water after polymer flooding in an oil field[J]. Journal of Hazardous Materials, 2010, 184(1): 105-110. |
| [9] |
Kay-Shoemake JL, Watwood ME, Sojka RE, et al. Polyacrylamide as a substrate for microbial amidase in culture and soil[J]. Soil Biology & Biochemistry, 2015, 30(13): 1647-1654. |
| [10] |
Wen Q, Chen Z, Zhao Y, et al. Biodegradation of polyacrylamide by bacteria isolated from activated sludge and oil-contaminated soil[J]. Journal of Hazardous Materials, 2010, 175(1): 955-959. |
| [11] |
Matsuoka H, Ishimura F, Takeda T, et al. Isolation of polyacryla-mide-degrading microorganisms from soil[J]. Biotechnology & Bioprocess Engineering, 2002, 7(5): 327-330. |
| [12] |
李殿杰. 聚丙烯酰胺降解菌株的分离及其油田含聚污水生物强化处理研究[J]. 环境科学与管理, 2018, 43(1): 113-117. DOI:10.3969/j.issn.1673-1212.2018.01.026 |
| [13] |
Qing Y, Caili D, Yefei W, et al. A study on mass concentration determination and property variations of produced polyacrylamide in polymer flooding[J]. Liquid Fuels Technology, 2011, 29(3): 227-235. |
| [14] |
Hernando MD, Vettori S, D e, Bueno MJ, et al. Toxicity evaluation with Vibrio fischeri test of organic chemicals used in aquaculture[J]. Chemosphere, 2007, 68(4): 724-730. DOI:10.1016/j.chemosphere.2006.12.097 |
| [15] |
Tang S, She D. Synergistic effects of rock fragment cover and polyacrylamide application on erosion of saline-sodic soils[J]. Catena, 2018, 171: 154-165. DOI:10.1016/j.catena.2018.06.033 |
| [16] |
于烽.聚丙烯酰胺降解细菌的筛选及其在脱水污泥生物干化中的应用研究[D].西安: 西北大学, 2016.
|
| [17] |
陈庆国.聚丙烯酰胺降解菌的筛选及降解含聚丙烯酰胺污水的室内研究[D].青岛: 中国海洋大学, 2009.
|
| [18] |
包木太, 彭杰, 陈庆国. 微生物对聚丙烯酰胺降解作用的研究进展[J]. 化工进展, 2011(9): 2080-2086. |
| [19] |
Yan M, Zhao L, Bao M, et al. Hydrolyzed polyacrylamide biodegradation and mechanism in sequencing batch biofilm reactor[J]. Bioresource Technology, 2016, 207: 315-321. DOI:10.1016/j.biortech.2016.01.083 |
| [20] |
Zhang C, Zhao L, Bao M, et al. Potential of hydrolyzed polyacrylamide biodegradation to final products through regulating its own nitrogen transformation in different dissolved oxygen systems[J]. Bioresource Technology, 2018, 256: 61-68. DOI:10.1016/j.biortech.2018.01.143 |





