木质素是地球上数量最多的芳香族聚合物[1],与纤维素、半纤维素共同组成木质纤维素。木质纤维素是目前生物质能源可研究和利用的主要材料。作为一种可再生能源,生物质的开发利用是解决目前人类能源危机的重要途径之一,但是天然纤维质原料的木质化很大程度上限制了其可利用性[2]。在地球碳循环中,木质素降解处于中心地位,因为大多数碳或者存在于木质素中,或者存在于受木质素保护免受酶降解的纤维素和半纤维素中[3],所以木质素能否顺利降解也极大地影响着纤维素的有效利用。此外,木质素作为一种造纸工业的副产物,由于很难被降解,排放到环境中后会导致严重的水生态污染[4]。相较于传统的物理转化和化学转化,开发低能耗、清洁度高和对环境友好的生物法降解木质素正成为研究者关注的焦点。白腐真菌是木质素降解中最主要的微生物,对其木质素降解,产酶条件的优化等方面的研究是目前生物技术应用的热点[5-6]。然而,细菌具有来源广、生长快、便于工程改造且易于工业化生产等优势,因此,细菌降解木质素的研究也具有广阔的前景。本研究利用选择培养基初步筛选具有木质素降解功能的细菌,通过16S rRNA基因序列分析鉴定菌株,进一步明确了菌株QL-Z3具有以碱木质素为唯一碳源进行生长代谢的木质素潜在降解性能。并对其相关酶学展开研究,为具有实际应用价值的木质素生物降解技术的开发提供途径。
1 材料与方法 1.1 材料 1.1.1 土壤样品供试土壤采样地位于秦岭西部的辛家山林区(海拔:1 500-2 000 m;经纬度:106°E,34°N),土样采集点的植物群落为华山松,采样深度为0-10 cm。
1.1.2 培养基(1) 菌种活化培养基:LB培养基(蛋白胨10 g/L,酵母粉5 g/L,NaCl 10 g/L,pH为7.0-7.2)。(2)液体木质素培养基:碱木质素3 g/L,K2HPO4 1 g/L,MgSO4·7H2O 0.1 g/L,CaCl2 0.08 g/L,FeSO4·7H2O 0.05 g/L,MnCl2 0.02 g/L,KH2PO4 1 g/L,蛋白胨2 g/L,固体培养基添加琼脂粉15 g/L。自然pH。(3)基础培养基(BM培养基):酵母膏10 g/L,葡萄糖20 g/L,琼脂15 g/L,pH为7.0左右。
1.2 方法 1.2.1 菌株筛选鉴别[7]称取5.0 g土壤样品于100 mL液体木质素培养液中,30℃、180 r/min振荡培养48 h。将各培养液从10-1稀释至10-6,吸取0.1 mL涂布于木质素培养基平板,30℃恒温培养。待平板上长出单菌落后,再次于木质素培养基平板划线纯化。挑取纯化后的单菌落经液体扩大培养后按1:1的体积比加入50%甘油,混匀后保存于-80℃冰箱。取-80℃保藏的菌株经过LB培养至对数期后,使用Bacterial DNA Kit(Omega)提取总DNA,以总DNA为模板,使用27F/1492R通用引物进行16S rRNA基因PCR扩增。根据电泳条带有无,挑选其中阳性克隆菌落,送西安擎科生物工程公司测序。将序列上传到NCBI数据库中比对BLAST,用MEGA6.0软件绘制系统发育树,判断菌株所属类别[8]。
1.2.2 菌株的生长测定将QL-Z3菌株接种于LB培养基中,30℃、180 r/min培养至OD600为1.2左右,5 000 r/min离心菌体5 min,用生理盐水洗涤2次后悬浮,取菌液按1%的接种量接种于液体木质素培养基中,30℃、180 r/min连续培养。通过培养液离心前后在600 nm处的吸光度差值来反应菌株的生长状况:
第n小时的OD600=OD600a-OD600b
OD600a:培养液在600 nm吸光度直接测定值;OD600b:培养液经5 000 r/min离心5 min后上清液在600 nm吸光度测定值。
1.2.3 木质素降解率的测定280 nm处的吸光度作为木质素浓度测定指标[9]:
第n小时木质素降解率(%)=(A0-An)/A0×100
A0:接菌前培养基中木质素的浓度;An:接菌培养n小时后培养基中木质素的浓度。
1.2.4 木质素降解酶的定性检测[10-11]采用苯胺兰(Azure-B)和RB亮蓝(Remazol brilliant blue)染料平板检测,在BM培养基中分别加入0.1 g/L的染料苯胺兰和RB亮蓝,铺平板后接种菌株,在30℃恒温培养箱中静置培养,每天观察,以平板培养基中菌落周围脱色圈的有无来定性检测木质素降解酶是否产生。
1.2.5 木质素酶活力的定量测定方法(1) 木质素过氧化物酶的测定[12]:反应体系为3 mL,反应混合液含有1.85 mL 0.24 mmol/L藜芦醇和1.0 mL粗酶液,预热至37℃后加入,0.1 mL 6.0 mmol/L的H2O2启动反应,并测定3 min前后在310 nm下吸光度值的增加量。一个酶活力单位(U)定义为每分钟使1 μmol藜芦醇氧化所需要的酶量。(2)漆酶的测定[13]:在25℃ 3 mL反应体系中,反应混合液含有2 mL 0.5 mmol/L的ABTS(溶于0.1 mmol/L,pH 5.0的醋酸-醋酸钠缓冲溶液中),加入1 mL粗酶液启动反应,每隔1 min测定420 nm下的吸光值,取吸光值线性变化部分,一个酶活力单位(U)定义为每分钟氧化1 μmol ABTS所需要的酶量。(3)锰过氧化物酶的测定[13]:在37℃ 3 mL反应体系,反应混合液含有2.4 mL 50 mmol/L pH 4.5的醋酸缓冲液,0.1 mL 1.6 mmol/L MnSO4溶液,0.4 mL粗酶液,37℃下加入0.1 mL 1.6 mmol/L的H2O2溶液启动反应,测定最初3 min内在240 nm下吸光度值并取线性变化部分。一个酶活力单位(U)定义为每分钟使1 μmol Mn2+转化为Mn3+所需的酶量(以上定量测定试验均做3次重复)。
1.2.6 粗酶液的制备[14]液体木质素培养基中30℃摇瓶培养的菌液8 000 r/min离心10 min所得的上清液即为粗酶液。
1.2.7 扫描电镜(SEM)观察[15]分别取30℃、180 r/min培养3 d和7 d的未接菌(空白对照)和QL-Z3接种量为1%的木质素培养液50 mL,12 000 r/min离心10 min,上清液经液氮速冻后放入真空冷冻干燥仪中干燥至恒重,干燥后的样品喷金后进行电镜扫描观察。
2 结果 2.1 菌株QL-Z3的菌种鉴定将菌株QL-Z3的16S rRNA基因序列与NCBI中现有数据库进行序列比对分析,选取结果中相似度最高的属作为对应菌株分类标准,并绘制系统进化树。由图 1可知,菌株QL-Z3的16S rRNA基因序列与登录号为AM055711的欧文氏菌属菌株Erwinia billingiae Eb661相似度最高,为99%,因此鉴定菌株QL-Z3为欧文氏菌,命名为Erwinia sp. QL-Z3。该菌株GenBank登录号MH828331;在中国微生物菌种保藏管理委员会普通微生物中心的菌株保藏编号为CGMCC 1.16623。
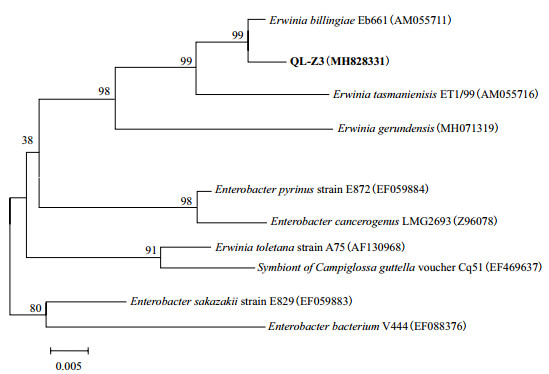
|
| 图 1 依据16S rRNA基因序列构建的菌株QL-Z3的系统进化树 |
由图 2可以看出,菌株Erwinia sp.QL-Z3能在以木质素为唯一碳源的培养基中生长,在发酵48 h后,菌株的生长量达到最大,OD600达到0.31。此外,结果显示在经过72 h培养后,培养液在280 nm处的吸光度由初始1.90降为1.63。这意味着在经过72 h的发酵,14.23%的木质素被菌株QL-Z3降解。
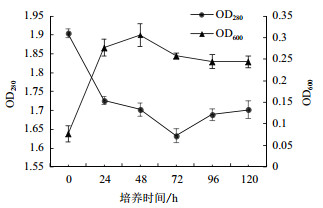
|
| 图 2 菌株生长曲线和木质素降解曲线 |
木质素的降解主要是依赖一系列酶的共同作用[16],这些酶主要包括木质素过氧化物酶(Lignin peroxidases,LiP)、锰过氧化物酶(Man-ganese peroxidases,MnP)和漆酶(Laccase,Lac)。苯胺蓝的脱色与LiP及MnP的产生有关,但不反映漆酶的产生,漆酶能使RB亮蓝脱色。结果如图 3所示。菌株Erwinia sp. QL-Z3对苯胺蓝有较强的脱色能力,对RB亮蓝有一定的脱色作用。根据平板的脱色结果,表明在无木质素诱导物存在的情况下菌株Erwinia sp. QL-Z3具有产木质素降解酶的能力,但据苯胺蓝和RB亮蓝平板脱色反应只能简单判断菌株的产酶情况,要具体确定菌株的木质素降解能力,需进行木质素降解酶活的定量测定试验。
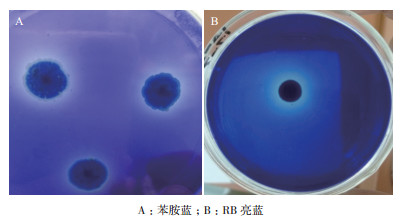
|
| 图 3 菌株Erwinia sp.QL-Z3脱色情况 |
图 4为该菌株分泌3种胞外酶的能力,从图 4中可以看出,QL-Z3具有较强的锰过氧化物酶分泌能力,在前2 d酶活增加较快,且在60 h达到最大值263.83 U/L,随后降低。然而分泌木质素过氧化物酶和漆酶的能力相对较弱,漆酶在培养48 h后达到了最高酶活,仅有77 U/L,木质素过氧化物酶也在第48小时达到最大值104.11 U/L,随后缓慢降低。这3种酶的合成均是在菌株生长对数期和稳定期内,且酶活变化趋势与菌株QL-Z3在木质素培养基中的生长规律基本一致。
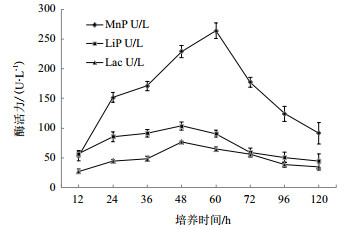
|
| 图 4 木质素降解过程中QL-Z3酶活曲线 |
利用扫描电镜观察木质素降解过程中的形貌变化,是目前证明木质素能否被微生物修饰或降解的重要技术手段之一[17],将木质素样品在Erwinia sp. QL-Z3降解前后的形态放大相同的倍数后,可以看出未经细菌处理的木质素样品(图 5-A)结构致密且呈块状,而经Erwinia sp. QL-Z3处理3 d后的样品(图 5-B)结构呈疏松多孔状,经Erwinia sp. QL-Z3处理7 d后的样品(图 5-D)结构疏松多孔更为明显。通过对比图 5-A、5-B、5-C、5-D可证明菌株Erwinia sp. QL-Z3能够对木质素样品进行解聚,破坏木质素结构,使其裂解成细小的碎片。
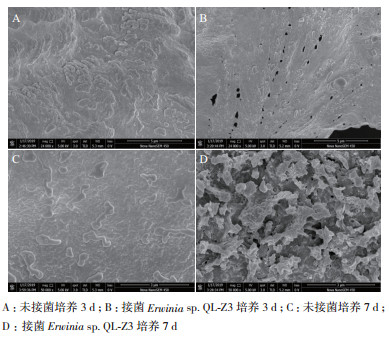
|
| 图 5 木质素样品扫描电镜图 |
由于木质素的结构复杂,作为世界上第二丰富的可再生有机资源却一直未被充分利用。研究者们对木质素综合利用方法的探索已进行了几十年,然而仍存在许多问题,如化学法易造成二次污染,物理法能耗过高,而生物法虽对环境友好,但也存在效率低的问题。目前普遍存在的燃烧或者废弃木质素的做法会极大地浪费能源并污染环境,因此筛选高效降解木质素的微生物具有现实意义,这不仅充分利用了废弃物,而且对开发以木质纤维素为原料的生物质能源也具有重要意义。
目前,国内外研究欧文氏菌主要集中在病原菌的研究,着重关注其致病性的较多。如Hirakawa等[18]研究了Erwinia carotovora在植物液泡细胞死亡过程中的作用;Antonio等[19]研究了植物对Erwinia amylovora致病性产生适应性的原因。近几十年来,木质素的微生物降解研究主要集中于白腐真菌和褐腐真菌等真菌的研究上[20],同时,也有越来越多的研究者从森林土壤或造纸废水中筛选鉴定出能降解木质素的细菌,如Streptomyces griseorubens AG-11[21]、Sphingomonas paucimobilis SYK-6[22]、Bacillus spp.[23]、Klebsiella sp. BRL6-2[24]、Rhodococcus jostii RHA1[25],这些研究表明,细菌在生物多样性和环境适应性等方面比真菌更具优势。木质素降解机制十分复杂,在降解途径上,许多研究者基于试验结果提出了相关的推测,如Shi等[26]在细菌菌株Cupriavidus basilensis B-8中推测出β-Ketoadipate代谢途径、苯酚降解途径和龙胆酸代谢途径。此外,研究者还发现苯甲酸代谢途径是木质素降解过程中的主要代谢途径,主要存在于细菌中[27]。总之,尽管对于细菌降解木质素机制已经有了大量的推测性研究[28-29],但由于细菌的种类不同,其代谢途径也不完全相同,且此类研究不够系统,尚未完整系统地描述细菌中木质素的降解途径和代谢机制。因此,建立模式细菌,利用成熟的组学技术和先进分析手段,构建细菌降解木质素的完整代谢途径是急需解决的问题。本实验的研究对象Erwinia sp. QL-Z3是一株从土壤中分离到的细菌,试验结果表明该菌株能将木质素作为唯一碳源进行生长,并且能分泌Lip、Mnp和Lac等3种重要的木质素降解酶,具备对木质素进行解聚,破坏木质素结构的能力。这表明该菌株在将来生物质能源利用及木质素污染的环境治理方面有着潜在的应用价值。国内外鲜有关于欧文氏菌降解木质素的相关文献报道,分离得到菌株Erwinia sp. QL-Z3并证明其木质素降解性能,可丰富木质素降解的细菌种类并为寻找降解利用木质素的微生物提供备选菌株,从而为开发利用以木质纤维素为主要来源的生物质能源提供更多选择。
4 结论本研究从森林土壤样品中分离得到一株具备木质素降解潜力的菌株QL-Z3,经鉴定属于欧文氏菌属(GenBank登录号MH828331;中国微生物菌种保藏管理委员会普通微生物中心的保藏编号CGMCC 1.16623)。该菌株能够利用碱木质素为唯一碳源生长,定性和定量检测均表明该菌株具备分泌木质素降解酶的能力,并且通过对木质素的解聚可使木质素形貌发生改变。在以碱木质素为唯一碳源的液体碱木质素培养基中发酵48 h,菌体生长达到稳定期,OD600达到0.31,发酵72 h,木质素降解率为14.23%。木质素降解相关酶活力在发酵48 h和60 h达到峰值:发酵60 h Mnp达到263.83 U/L,发酵48 h Lip和Lac分别达到104.11 U/L和77 U/L。本研究较为系统地揭示了Erwinia属菌株的木质素降解性能,该菌株可作为木质素降解的优良备选菌株。
| [1] |
Buswell JA, Odier E. Lignin biodegradation[J]. Critical Reviews in Biotechnology, 1987, 6(1): 1-60. |
| [2] |
Cowling EB, Kirk TK. Properties of cellulose and lignocelluosic materials as substrates for enzymatic conversion processes[J]. Biotechnology and Bioengineering Symposium, 1976(6): 95-123. |
| [3] |
Kirk TK. Lignin biodegradation:Importance and historical research perspective[J]. Recent Advances in Lignin Biodegradation Research, 1983, 17(1): 1-11. |
| [4] |
Berryman D, Houde F, DeBlois C, O'hea M. Nonylphenolic compounds in drinking and surface waters downstream of treated textile and pulp and paper effluents:a survey and preliminary assessment of their potential effects on public health and aquatic life[J]. Chemosphere, 2004, 56(3): 247-255. DOI:10.1016/j.chemosphere.2004.02.030 |
| [5] |
李慧蓉. 白腐真菌生物学和生物技术[M]. 北京: 化学工业出版社, 2005.
|
| [6] |
黄丹莲, 曾光明, 黄国和, 等. 白腐菌固态发酵条件优化及其降解植物生物质的研究[J]. 环境科学学报, 2005, 25(2): 232-237. DOI:10.3321/j.issn:0253-2468.2005.02.018 |
| [7] |
东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001.
|
| [8] |
远方, 屈淑平, 崔崇士, 等. 一株新的胡萝卜软腐欧文氏菌的分离和鉴定[J]. 微生物学报, 2004, 44(2): 136-140. DOI:10.3321/j.issn:0001-6209.2004.02.002 |
| [9] |
Lara MA, Rodríguez-Malaver AJ, Rojas OJ, et al. Black liquor lignin biodegradation by Trametes elegans[J]. International Biodeterioration & Biodegradation, 2003, 52(3): 167-173. |
| [10] |
蔡磊, 尹峻峰, 杨丽萍, 等. 几种简便的木质素降解真菌定性筛选方法[J]. 微生物学通报, 2002, 29(1): 67-69. DOI:10.3969/j.issn.0253-2654.2002.01.017 |
| [11] |
康从宝, 李清心, 刘瑞田, 等. 一株白腐菌产生的漆酶对RB亮蓝的脱色作用[J]. 应用与环境生物学报, 2002, 8(3): 298-301. DOI:10.3321/j.issn:1006-687X.2002.03.016 |
| [12] |
李越中, 高培基, 王祖农. 黄孢原毛平革菌合成木素过氧化物酶的营养调控[J]. 微生物学报, 1994, 6(1): 29-36. |
| [13] |
Chen YH, Chai LY, Zhu YH, et al. Biodegradation of kraft lignin by a bacterial strain Comamonas sp. B-9 isolated from eroded bamboo slips[J]. J Appl Microbiol, 2012, 112(5): 900-906. DOI:10.1111/j.1365-2672.2012.05275.x |
| [14] |
Sui SY, Zhen WL, Xiao P, et al. Isolation of laccase gene from Bacillus subtilis and analysis of its physicochemical properties[J]. Gene, 2011, 491(1): 49-52. |
| [15] |
张佩佩, 孙建中, 谢长校. Comamonas serinivorans C35木质素降解性能[J]. 微生物学通报, 2017, 44(5): 1131-1137. |
| [16] |
Casciello C, Tonin F, Berini F, et al. A valuable peroxidase activity from the novel species Nonomuraea gerenzanensis growing on alkali lignin[J]. Biotechnology Reports, 2017, 13: 49-57. DOI:10.1016/j.btre.2016.12.005 |
| [17] |
Zhu DC, Zhang PP, Xie CX, et al. Biodegradation of alkaline lignin by Bacillus ligniniphilus L1[J]. Biotechnology for Biofuels, 2017, 10(1): 44. |
| [18] |
Hirakawa Y, Nomura T, Hasezawa S, et al. Simplification of vacuole structure during plant cell death triggered by culture filtrates of Erwinia carotovora[J]. Journal of Integrative Plant Biology, 2015, 57(1): 127-135. |
| [19] |
Antonio C, Giampaolo B, Lorenzo R, et al. Biological relevance of volatile organic compounds emitted during the pathogenic interactions between apple plants and Erwinia amylovora[J]. Molecular Plant Pathology, 2018, 19(1): 158-168. DOI:10.1111/mpp.12509 |
| [20] |
Kudanga T, Nyanhongo GS, Guebitz GM, et al. Potential applica-tions of laccase-mediated coupling and grafting reactions:A review[J]. Enzyme Microb Technol, 2011, 48(3): 195-208. DOI:10.1016/j.enzmictec.2010.11.007 |
| [21] |
马英辉, 李利军, 卢美欢, 等. 木质素降解放线菌的鉴定及产酶特性研究[J]. 环境污染与防治, 2018, 309(8): 57-61. |
| [22] |
Masai E, Katayama Y, Fukuda M. Genetic and biochemical investigations on bacterial catabolic pathways for lignin-derived aromatic compounds[J]. Bioscience Biotechnology and Biochemistry, 2007, 71(1): 1-15. DOI:10.1271/bbb.60437 |
| [23] |
Raj A, Reddy MMK, Chandra R, et al. Biodegradation of kraft-lignin by Bacillus sp. isolated from sludge of pulp and paper mill[J]. Biodegradation, 2007, 18(6): 783-792. DOI:10.1007/s10532-007-9107-9 |
| [24] |
Woo HL, Ballor NR, Hazen TC, et al. Complete genome sequence of the lignin-degrading bacterium Klebsiella sp. strain BRL6-2[J]. Standards in Genomic Sciences, 2014, 9: 19. DOI:10.1186/1944-3277-9-19 |
| [25] |
Ahmad M, Roberts JN, Hardiman EM, et al. Identification of DypB from Rhodococcus jostii RHA1 as a lignin peroxidase[J]. Biochemistry, 2011, 50(23): 5096-5107. DOI:10.1021/bi101892z |
| [26] |
Shi Y, Chai LY, Tang CJ, et al. Characterization and genomic analysis of kraft lignin biodegradation by the beta-proteobacterium Cupriavidus basilensis B-8[J]. Biotechnology for Biofuels, 2013, 6(1): 1-14. |
| [27] |
Egland PG, Pelletier DA, Dispensa M, et al. A cluster of bacterial genes for anaerobic benzene ring biodegradation[J]. Proceedings of the National Academy of Sciences, 1997, 94(12): 6484-6489. DOI:10.1073/pnas.94.12.6484 |
| [28] |
Kamimura N, Takahashi K, Mori K., et al. Bacterial catabolism of lignin-derived aromatics:New findings in a recent decade update on bacterial lignin catabolism[J]. Environmental Microbiology Reports, 2017, 9(6): 679-705. DOI:10.1111/1758-2229.12597 |
| [29] |
de Gonzalo G, Colpa DI, Habib MH, et al. Bacterial enzymes involved in lignin degradation[J]. Journal of Biotechnology, 2016, 236: 110-119. DOI:10.1016/j.jbiotec.2016.08.011 |




