淀粉是大量存在于自然界中的植物多糖[1],是以葡萄糖为单体依次连接形成的高度聚合物。根据单糖连接方式不同,分为直链淀粉和支链淀粉[2-3]。淀粉除了可以被直接食用外,还是工业生产果葡糖浆、麦芽糖及酿酒等多种工业的主要原料[4],其在被利用过程中必须被水解为低聚糖或单糖[5]。α-淀粉酶是一类作用于淀粉内部水解其α-1,4糖苷键从而将淀粉水解为糊精、低聚糖和单糖的酶[6]。α-淀粉酶是工业上应用最早、用途最广及产量最大的酶制剂产品之一。可广泛应用于食品、动物饲料、洗涤剂、生物燃料、生物制药等多种工业生产过程中[8]。烘焙行业中,添加适量的淀粉酶可以增加面包的蓬松度,改善口感[9];在啤酒酿造工业中,添加淀粉酶可以增加麦芽的降解率,提高利用率[10];在洗涤剂行业中,淀粉酶可以有效降解淀粉类污渍[11]。α-淀粉酶可以从植物,动物和微生物中获得,同时淀粉酶的表达,纯化,发酵等技术也日益成熟。其中,微生物来源的α-淀粉酶由于其稳定性和经济可行性而被普遍应用[12]。
微生物来源的淀粉酶在生产应用中变得愈加重要,与动植物来源淀粉酶相比,真菌和细菌淀粉酶具有成本效益高、一致性好、生产所需时间和空间少、易于工艺修改和优化等优点[12]。尤其是芽孢杆菌来源的淀粉酶,已经受到全世界研究人员的广泛关注,并对其进行了深入的研究。与其他微生物相比,芽孢杆菌是表达淀粉酶的优势菌株,表达量和酶活均相对较高。同时,由于芽孢杆菌自身的分泌表达系统,使得所产生的淀粉酶直接分泌到细胞外,无需细胞破碎等过程,酶学性质检测及纯化步骤简单。目前,对芽孢杆菌来源的淀粉酶研究涉及热稳定性、水解活力及三维结构等多个方面。其中,耐热淀粉酶多来自于地衣芽孢杆菌[13]。此外,多个芽孢杆菌来源的淀粉酶晶体结构也已经被报道[14-16]。通过对结构的研究得出,大多数芽孢杆菌来源的淀粉酶属于糖苷水解酶13家族,同时具有Ca2+结合位点。同时对于这种来源淀粉酶也进行了许多突变研究,酶学性质显著提高,并可以应用到生产实践中。已有报道显示通过定向进化使淀粉酶在碱性条件下酶活提高5倍[17],通过理性设计使解淀粉芽孢杆菌淀粉酶的最适温度由83℃提高到106℃[18]。同时挖掘Ca2+非依赖型淀粉酶基因,以使在生产过程中降低成本[19]。
按照糖苷水解酶(Carbohydrate-active enzymes,CAZy))数据库(http://www.cazy.org/)的分类,α-淀粉酶主要属于糖苷水解酶第13家族(GH13)[20]。另有少部分属于GH57、GH119或者GH126家族[21-23]。目前已经有多个GH13家族的淀粉酶结构被报道[15-17]。GH13家族淀粉酶由A((β/α)8八桶状结构)、B、C(β-三明治结构)3个结构域组成。A结构域是催化结构域,天冬氨酸、谷氨酸及天冬氨酸构成了催化活性中心。B结构域的Loop区对淀粉酶的热稳定性起到重要作用[24]。C结构域呈现β片层结构,有报道指出它类似酶的底物结合域(Carbohydrate-binding module,CBM),对底物的结合起到重要作用[25]。因此对C结构域进行理性设计可能提高酶与底物的结合能力,从而提高水解活力。
具有高催化活力的淀粉酶由于其水解效率高,生产成本低,一直受到广泛的青睐。目前已经报道的淀粉酶生产菌株的产酶能力虽然能满足工业生产需求,但仍有较大的提升空间。因此,如何利用生物技术手段,改造现有的淀粉酶,提高水解效率,进一步降低生产成本依然是目前亟待解决的问题。本研究中,以目前广泛应用的来源于解淀粉芽孢杆菌的中温中性淀粉酶BAMYA为出发蛋白,通过理性设计在该淀粉酶的C端结构域设计突变位点,利用定点突变得到了比活及催化效率均大幅度提高的突变体BAMYA-L473K/K474H/N475K,并在枯草芽孢杆菌中实现高效表达。该突变体应用于工业生产可有效提高其水解效率,降低应用成本,有着非常广阔的应用前景。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒基因克隆宿主菌株大肠杆菌(Escherichia coli)Trans-T1、感受态细胞DMT购自北京全式金公司,表达宿主菌株枯草芽孢杆菌(Bacillus subtilis)SCK6及质粒pHYP16由本实验室保存。
1.1.2 培养基LB液体培养基(g/L):酵母浸提物5,胰蛋白胨10,NaCl 10。LB固体培养基(g/L):酵母浸提物5,胰蛋白胨10,NaCl 10,琼脂粉15。发酵培养基(g/L):酵母浸提物20,蛋白胨40,豆粕30,玉米粉38,NaH2PO4 2。
1.1.3 试剂及仪器直链淀粉购自Sigma公司;质粒提取试剂盒及DNA胶回收试剂盒购自OMEGA公司;FastPfu DNA聚合酶、快速突变试剂盒(Fast Mutagenesis System)、甲基化质粒模板消化酶购自北京全式金公司,BeaverBead His-tag Protein Purification磁珠(组氨酸标签蛋白纯化琼脂糖磁珠)购自苏州海狸生物科技有限公司,是专为高效、快速纯化组氨酸标签蛋白而设计的一种新型功能化材料,可通过磁性分离方式直接从生物样品中一步纯化出高纯度的目标蛋白。其他试剂均为国产分析纯。
T100 Thermal Cycler PCR与电转仪、DYY-6C凝胶成像系统(美国Bio-Rad),BioTek酶标仪(美国Thermo),HiTrap Q XL阴离子柱(美国GE)。
1.2 方法 1.2.1 穿梭质粒pHYP16-Amp-Bamy的构建本研究所用的质粒,是在芽孢杆菌表达载体pHYP16的基础之上,利用同源重组的方法,加入大肠杆菌的复制原件以及氨苄抗性基因,得到穿梭型载体pHYP16-Amp,其能同时在大肠杆菌以及枯草芽孢杆菌中操作。来源于解淀粉芽孢杆菌(Bacillus amyloliquefaciens)的淀粉酶基因Bamy是由公司合成,采用POE-PCR(Prolonged overlap extension PCR)[26]的方法,在载体上插入Bamy,得到pHYP16-Amp-Bamy。
POE-PCR程序如下:(1)扩增目的基因Bamy:反应体系50 μL:10×buffer 5 μL,dNTP 5 μL,硫酸镁2 μL,pUC57-Bamy模板1 μL,引物Bamy-F 1 μL,引物Bamy-R 1 μL,FastPfu DNA聚合酶1 μL,ddH2O 34 μL。PCR条件为:95℃反应5 min,一个循环;95℃反应30 s,60℃反应30 s,72℃反应1 min 30 s,以上3个步骤重复32个循环;72℃反应10 min,一个循环。反应产物取出后于4℃保存。(2)扩增载体pHYP16:反应体系50 μL:10×buffer 5 μL,dNTP 5 μL,硫酸镁2 μL,pHYP16模板1 μL,引物pHYP16-F 1 μL,引物pHYP16-R 1 μL,FastPfu DNA聚合酶1 μL,ddH2O 34 μL。PCR条件为:95℃反应5 min,一个循环;95℃反应30 s,60℃反应30 s,72℃反应3 min 30 s,以上3个步骤重复32个循环;72℃反应10 min,一个循环。反应产物取出后于4℃保存。
将以上两步得到的PCR产物进行胶回收,回收后进行片段和载体搭建,体系如下:10× buffer 5 μL,dNTP 5 μL,硫酸镁2 μL,pHYP16回收片段7 μL,Bamy回收片段3 μL。FastPfu DNA聚合酶1 μL,ddH2O 27 μL。PCR条件为:95℃反应5 min,一个循环;95℃反应30 s,60℃反应30 s,72℃反应5 min,以上3个步骤重复30个循环;72℃反应10 min,一个循环。反应产物取出后于4℃保存。
PCR过程中所用引物序列,见表 1。
将得到的PCR产物转化大肠杆菌Trans1-T1,涂氨苄固体LB平板,37℃恒温过夜培养,长出菌落后进行菌落PCR验证,所用引物为Bamy-F/R。根据核酸电泳检测结果,送条带正确的克隆子进行测序。得到穿梭型质粒pHYP16-Amp-Bamy。
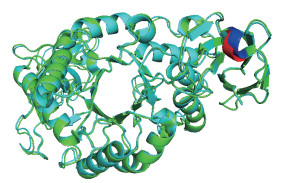
1.2.2 序列分析突变体设计来源于嗜碱芽孢杆菌KSM-1378的高比活、中温淀粉酶LAMY的比活比工业上广泛应用的、地衣芽孢杆菌来源的嗜热淀粉酶的比活高10倍[27]。将淀粉酶BAMYA与LAMY进行序列比对与结构比对(图 1和图 2),寻找有差异的位点。同时由于针对的是水解活力方面的改造,我们将突变位点主要集中在与底物结合有关的C结构域(C端),将有差异的位点进行突变,设计突变体BAMYA-L473K/K474H/N475K。

|
| 突变位点用蓝色表示 图 1 BAMYA与LAMY的序列比对结果 |
|
| 突变位点所在位置分别用红色(BAMYA)和蓝色(LAMY)标出 图 2 BAMYA(绿色)与LAMY(青色)三维结构对比图 |
利用全式金快速突变试剂盒(Fast mutagenesis system)按照操作说明进行突变。PCR体系为:2×TransStart FastPfu Fly PCR SuperMix 25 μL,突变正向引物BamyA-F 1 μL,突变反向引物BamyA-R 1 μL,模板pHYP16-Bamy质粒10 ng 1 μL,ddH2O 22 μL。PCR条件为:94℃反应2 min,一个循环;94℃反应20 s,55℃反应20 s,72℃反应4 min,以上3个步骤重复25个循环;72℃反应10 min,一个循环。反应产物取出后于4℃保存。引物序列见表 1。
PCR产物中加入1 μL DMT消化酶,在37℃水浴锅中反应1 h,取出后,10 μL产物转化大肠杆菌50 μL DMT感受态细胞中,菌液涂布于氨苄抗性平板,37℃过夜培养,待长出菌落后,进行菌落PCR验证,目的基因为1.5 kb,经核酸电泳检测后,选择条带大小正确的转化子测序。
1.2.4 重组酶BAMYA及突变体BAMYA-L473K/K474H/N475K在芽孢杆菌中的表达及纯化将序列正确的野生型及突变体质粒转化入枯草芽孢杆菌SCK6中,涂布于含有1.5%可溶性淀粉的四环素抗性平板,于37℃培养箱过夜培养。由于芽孢杆菌为分泌型表达宿主,成功表达的转化子会在平板上显示淀粉水解圈,在长出的转化子中选择菌体小、水解圈大的克隆,挑至50 mL LB培养基中,37℃,220 r/min,过夜培养,即得到种子液。将种子液以4%接种量转接至500 mL发酵培养基中,37℃,220 r/min,培养3 d。对所得到的发酵菌液离心后,收集上清,测定淀粉酶活力。
发酵上清浓缩10倍,浓缩用1 L去离子水清洗膜包,用1 L的0.5 mol/L NaOH清洗膜包中杂蛋白,最后用1 L去离子水清洗残留的NaOH,然后对发酵上清进行浓缩。在pH 7.4,20 mmol/L NaH2PO4-Na2HPO4缓冲溶液中4℃过夜透析,透析后进行磁珠亲和层析。亲和层析的纯化步骤为:配置平衡液Binding buffer为pH 7.4,20 mmol/L NaH2PO4-Na2HPO4,洗脱液为含有不同浓度咪唑(20 mmol/L、50 mmol/L、80 mmol/L、100 mmol/L、200 mmol/L、300 mmol/L、500 mmol/L)的pH 7.4,20 mmol/L NaH2PO4-Na2HPO4。吸取海狸磁珠(是专为高效、快速纯化组氨酸标签蛋白而设计的一种新型功能化材料,可通过磁性分离方式直接从生物样品中一步纯化出高纯度的目标蛋白)20 mL与磁力架上静置30 s,弃掉废液,依次用去离子水以及Binding buffer平衡磁珠,使其处于可以结合目标蛋白质的离子条件下。向平衡好的磁珠中加入50 mL上述透析好的淀粉酶液,漩涡混合2 h。取下后依次用含有20 mmol/L,50 mmol/L、80 mmol/L、100 mmol/L、200 mmol/L、300 mmol/L及500 mmol/L的洗脱液进行目的蛋白的洗脱,并收集所有洗脱液。进行后续SDS-PAGE蛋白电泳检测。对含有目的蛋白的收集液,在pH 8.0,20 mmol/L Tris-HCl缓冲液中进行过夜透析,透析后进行阴离子柱纯化,所用平衡缓冲液为pH 8.0,20 mmol/L Tris-HCl,洗脱液为在平衡液的基础上加入1 mol/L NaCl。纯化后进行峰收集,测定酶活力,并利用SDS-PAGE进行蛋白电泳检测。纯化后得到的酶液用于酶学性质分析。
1.2.5 α-淀粉酶的酶活测定α-淀粉酶的酶活测定方法采用DNS(3,5-二硝基水杨酸)定糖法[28]。将按GB/T24401-2009方法配制的2%可溶性淀粉用相应pH缓冲溶液稀释至1%终浓度淀粉溶液作为底物,测量体系包括900 μL的底物和100 μL适当稀释的酶液,在55℃水浴锅中反应20 min,加入1.5 mL的DNS试剂终止反应后,置于沸水浴中处理5 min,快速冷却至室温后取250 μL混合液用酶标仪读取在波长540 nm下的吸光值,每组反应设置1个空白对照及3个平行(突变体的酶活力测定方法相同,温度采用60℃)。
酶活单位(U)定义:在最适条件下,每分钟水解可溶性淀粉生成1 μmoL葡萄糖所需的酶量为一个酶活单位。
1.2.6 重组酶BAMYA及突变体BAMYA-L473K/K474H/N475K酶学性质的研究 1.2.6.1 最适pH和pH稳定性的测定纯化后的野生型淀粉酶BAMYA及突变体BAMYA-L473K/K474H/N475K在最适温度条件下(BAMYA为55℃,突变体为60℃),与不同pH(pH 3.0-8.0,20 mmol/L柠檬酸-磷酸盐缓冲液)的底物进行酶促反应,以测定其最适pH。将BAMYA及突变体在不同pH值(pH 3.0-8.0)的20 mmol/L柠檬酸-磷酸盐缓冲液中置于室温下处理1 h,再用pH 6.0的缓冲液作适当稀释,并在最适温度、pH 6.0的条件下测定剩余酶活,以未进行处理的样品酶活为100%对照,用于研究重组野生型BAMYA及突变体BAMYA-L473K/K474H/N475K的pH稳定性。每个反应3个平行及一个空白对照,根据测定结果绘制最适pH及pH稳定性曲线。
1.2.6.2 最适温度与温度稳定性的测定用pH 6.0的缓冲液将纯化后的BAMYA及突变体BAMYA-L473K/K474H/N475K稀释后,在不同温度下(60-80℃)进行酶促反应以确定其最适温度。将纯化后的BAMYA及突变体分别在60、70和80℃下保温,2 min、5 min、10 min、30 min及60 min取样后迅速置于冰上5 min,并在最适温度、pH 6.0的条件下测定剩余酶活性,以未热处理的酶活为100%对照,以研究野生型BAMYA及突变体的热稳定性。每个反应3个平行及一个空白对照,根据测定结果绘制最适温度及热稳定性曲线。
1.2.6.3 比活及动力学参数测定对所有需要测定酶学性质的蛋白进行定量,按照TransGene蛋白定量试剂盒的说明书进行,如表 2所示。将BSA按照表中所示浓度进行配置后,吸取20 μL BSA标准品及待定量的样品,与200 μL考马斯亮蓝显色液混合,室温放置10 min,在OD595 nm下读取吸光值,根据吸光值和蛋白浓度绘制标准曲线,拟合出蛋白含量与吸光度的线性关系。比活力单位定义为,每毫克酶蛋白所具有的酶活力单位数。
分别配制最适pH下不同浓度的底物(0.5-10.0 mg/mL),酶反应体系组成不变,但是酶反应时间10 min。在最适条件下测定酶活力,利用GraphPad Prism[29]软件分析得到BAMYA及突变体以可溶性淀粉为底物时的Vmax和Km值。
2 结果 2.1 穿梭质粒pHYP16-Amp-Bamy的构建本研究所用基因是芽孢杆菌来源的,并且芽孢杆菌对于淀粉酶的表达量较高,因此我们选择枯草芽孢杆菌作为表达宿主。此外,本研究涉及大量的定点突变工作,相对于芽孢杆菌而言,大肠杆菌感受态的转化效率高,因此,定点突变在大肠杆菌中进行,获得突变后在芽孢杆菌中进行表达。本实验所用质粒需要满足可以同时在大肠杆菌以及芽孢杆菌中操作的条件。利用同源重组的方法,在芽孢杆菌表达载体pHYP16中插入了大肠杆菌复制原件以及氨苄抗性基因,得到穿梭型载体pHYP16-Amp。构建好的质粒及过成如图 3所示。

|
| 图 3 枯草芽孢杆菌-大肠杆菌穿梭型质粒的构建(A)插入淀粉酶基因Bamy、氨苄抗性基因及其启动子前(B)插入淀粉酶基因Bamy、氨苄抗性基因及其启动子后 |
将突变位点集中在与底物结合有关的C结构域,分析并突变后,对突变体基因进行测序,得到组合突变体BAMYA-L473K/K474H/N475K。
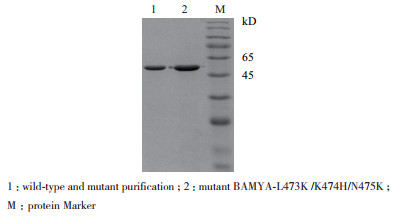
2.3 α-淀粉酶BAMYA及BAMYA-L473K/K474H/N475K在枯草芽孢杆菌中的表达及纯化对突变后的转化子进行测序后,利用Vector 10.0软件进行突变前后氨基酸及核酸序列比对,比对结果证明成功得到了突变体BAMYA-L473K/K474H/N475K,将构建的重组质粒pHYP16-BAMYA及其突变体在枯草芽孢杆菌SCK6中成功地实现异源表达,从含有可溶性淀粉的固体琼脂平板中得到水解圈大的克隆子,进行小瓶培养,而后转接发酵培养基进行摇瓶培养。离心收集发酵液上清,测定淀粉酶活力约为760 U/mL和1 260 U/mL。将得到的粗酶液用10 kD膜包过滤浓缩以初步除去酶液中的盐分及其他小分子物质,进一步用磁珠进行纯化及阴离子交换层析,进行蛋白纯化,经SDS-PAGE蛋白电泳检测,得到电泳纯酶蛋白(图 4)。分子量约为55 kD,与理论计算分子量一致。
|
| 图 4 淀粉酶野生型以及突变体纯化后SDS-PAGE分析 |
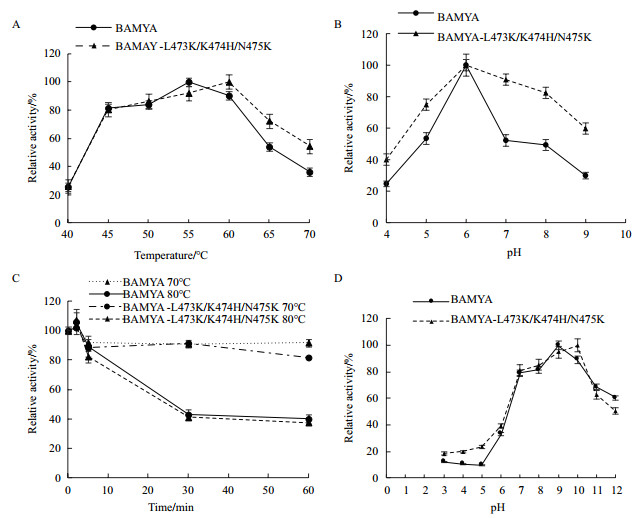
野生型淀粉酶BAMYA最适pH为6.0,最适温度为55℃。突变体最适pH 6.0,最适温度为60℃,与野生型相比最适温度提高了5℃。BAMYA和突变体的pH稳定性表现为:在pH 6.0均剩余40%左右酶活,pH 7.0-11.0稳定,pH 12.0剩余50%左右的酶活,在低于pH 5.0的条件下几乎没有酶活。BAMYA和突变体均具有很好的热稳定性,70℃处理30 min及80℃处理5 min,均剩余90%以上的酶活。
2.5 比活及动力学常数测定BAMYA的比活为6 835 U/mg,突变体BAMYA-L473K/K474H/N475K比活为10 148 U/mg,比野生型提高48%;野生型的Km值为3.58 mg/mL,突变后BAMYA-L473K/K474H/N475K的Km值为2.21 mg/mL,突变后酶对底物的亲和力提高;突变后催化效率由1 577 mL/(s·mg)提高至4 760 mL/(s·mg),较野生型相比提高了2.01倍(图 5)。
|
| 图 5 野生型BAMYA及突变体BAMYA-L473K/K474H/N475K基本性质 |
中温淀粉酶广泛应用于工业生产中,其最适温度大多在50-60℃[30],提高中温淀粉酶的水解活力及催化效率,以降低生产和使用成本,具有重要的意义。目前,关于淀粉酶水解活力的改造思路有很多,如Lu等[31]对Bacillus pseudofirmus来源的淀粉酶通过N端截短,将其比活由4.9 U/mg提高到170 U/mg,Km由11.9 mg/mL降低至3.9 mg/mL;Yang等[32]通过将Alkalimonas amylolytica来源的碱性淀粉酶末端逐一截短并与寡肽融合表达,其比活由9.4 U/mg提高到239 U/mg,Km由26.9 mg/mL降低至15.4 mg/mL;Bessler等[33]通过定向进化得到解淀粉芽孢杆菌来源淀粉酶BAA水解活力明显提高的突变体A230V/N297D/K406R/N414S,其比活由15 U/mg提高到140 U/mg。上述结果虽然酶活有一定的提高,但是仍然难以满足工业生产要求。本研究利用定点突变,将来源于解淀粉芽孢杆菌,GH13家族的淀粉酶BAMYA进行改良,所得到的淀粉酶突变体的比活达到了10 148 U/mg,并实现在枯草芽孢杆菌中的异源高效表达,突变体BAMYA-L473K/K474H/N475K比活为10 148 U/mg,酶活力达到1 260 U/mL,可以用于实际生产中,大大降低成本。
本研究的突变位点位于近C末端C结构域的473LKN475氨基酸,有文献曾报道,C端氨基酸组成会影响淀粉酶的pH稳定性[34],但相同位置的突变对比活的影响并未报道过。GH13家族的淀粉酶C结构域呈现β片层结构,属于一个未知的底物结合结构域(Carbohydrate binding modules,CBM区)对于底物的识别和吸附有重要的作用[35]。因此对于该结构域进行理性设计,可能增强其水解活力。还有报道称,在淀粉酶中还有一些具有特殊功能的结构域,大多数处于N端或者C端,在淀粉酶发挥水解活力的过程中起到重要作用。例如,来源于嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)的普鲁兰酶[36]、嗜热菌属(Thermus sp.)的麦芽淀粉酶[37]等在N端以外还含有类似的催化结构域。另外一些淀粉酶在C结构域之外还含有D、E结构域,与C结构域的功能类似,D结构域使淀粉酶有水解生淀粉的能力,如已经报道的曲霉(Aspergillus kawachii)α-淀粉酶[38]等,同时淀粉酶的C端结构域的芳香族的氨基酸(组氨酸、酪氨酸、苯丙氨酸等)对底物结合起到重要的作用[35],因此将K474H后,His的咪唑基团更有利于结合糖链。另外,参与底物结合的氨基酸的空间构象、以及氨基酸的结构大小也影响着亲和力[39],在本研究中,将BAMYA(WP_013352208.1)的第473位非极性亮氨酸(L)突变为赖氨酸(K)后,侧链为底物提供了更好的结合取向。同时有研究表明,带正电的氨基酸可降低Km,带负电的氨基酸可提高Km[39],N475K突变后可能提高了酶对底物的亲和力,从而提高了酶的比活和催化效率。本研究中,我们将BAMYA的C端进行组合突变,突变位点L473K/K474H/N475K有效提高了BAMYA的催化活性,同时该研究为其它淀粉酶突变研究提供理论指导。
根据淀粉酶应用的不同领域,对其性能的要求不尽相同。一些工业应用中,不可避免有高温过程,这就要求其酶活高的同时在高温下具有较好的稳定性。尽管有极为少数的解淀粉芽孢杆菌中温淀粉酶稳定性很好,尽管有少数的解中温淀粉酶稳定性很好,如一个Bacillus sp. I-3来源的淀粉酶,其最适温度70℃,在80℃下处理2.5 h后剩余50%酶活,但在最适条件下的表达量仅为642 U/mL[40];一个Thermobifida fusca NTU22来源的淀粉酶其最适温度为60℃,60℃处理3 h仍剩余70%酶活,但比活仅为245 U/mg[41];本研究中表达的淀粉酶BAMYA及其突变体在高温下具有很好的稳定性,同时水解活力及表达量均较高,突变体BAMYA-L473K/K474H/N475K比活为10 148 U/mg,酶活力达到1 260 U/mL,为淀粉酶生产应用提供了良好的材料。
综上,本研究通过定点突变获得一个高水解活力的α-淀粉酶基因,并在枯草芽孢杆菌中成功实现了高效异源表达。突变后得到了一个比活与催化效率明显提高,同时热稳定性没有明显下降的淀粉酶突变体BAMYA-L473K/K474H/N475K。拓宽了淀粉酶在饲料、食品及医药等领域的应用,为生产应用提供了优良的材料。通过动力学性质和氨基酸组成两个角度分析了其比活提高的分子机制,加深了中温淀粉酶在结构与功能相互关系的认识,也为淀粉酶的发展开辟了新的道路。
4 结论本研究构建了芽孢杆菌-大肠杆菌穿梭型质粒,通过理性设计对来源于解淀粉芽孢杆菌淀粉酶基因Bamy进行定点突变。在保持其热稳定性没有明显下降的前提下,使其水解活力、催化效率均大幅度提高,比活相较于野生型提高48%。通过分析序列及结构变化,对性质提高的机理进行了解释,同时该项研究为淀粉酶的生产应用提供了优良材料。
| [1] |
Reddy CK, Choi SM, Lee DJ, et al. Complex formation between starch and stearic acid:Effect of enzymatic debranching for starch[J]. Food Chem, 2018, 244(1): 136-142. |
| [2] |
Bentley IS, Williams EC. Starch conversion[C] //Godfrey T, West S(eds.)London, UK: Industrial Enzymology, The Macmillan Press Ltd, 1996: 341-357.
|
| [3] |
王丽萨.来源于Pyrococcus furiosus极端嗜热α-淀粉酶的研究[D].上海: 中国科学院研究生院, 2007.
|
| [4] |
段钢. 新型工业酶制剂的进步对生物化学品工业生产过程的影响[J]. 生物工程学报, 2009, 25(12): 1808-1818. DOI:10.3321/j.issn:1000-3061.2009.12.008 |
| [5] |
Goyal N, Gupta JK, Soni SK. A novel raw starch digesting thermostable a-amylase from Bacillus sp. I-3 and its use in the direct hydrolysis of raw potato starch[J]. Enzyme Microb Tech, 2005, 37(2): 723-734. |
| [6] |
Jens EN, Torben V. Protein engineering of bacterial amylases[J]. Biochim Biophys Acta, 2000, 1543(2): 253-274. DOI:10.1016/S0167-4838(00)00240-5 |
| [7] |
Van der Maarel MJ, Van der Veen B, Uitdehaag JCM, et al. Properties and applications of starch-converting enzymes of the α-amylase family[J]. Journal of Biotechnology, 2002, 94(2): 137-155. DOI:10.1016/S0168-1656(01)00407-2 |
| [8] |
Gupta R, Gigras P, Mohapatra H, et al. Microbial α-amylase:A biotechnological perspective[J]. Process Biochemistry, 2003, 38(11): 1599-1616. DOI:10.1016/S0032-9592(03)00053-0 |
| [9] |
Kilara A, Desai. M. Enzymes. In: Food Additives[C], Ⅲ(Eds.), New York, USA: Marcel Dekker Inc., 2002: 661-706.
|
| [10] |
Lee BH. Other microorganism based products. In: fundamentals of food biotechnology[C]. New York, USA: Wiley-VCH Inc, 1996: 291-352.
|
| [11] |
van der Maarel MJ, van der Veen B, Uitdehaag JCM, et al. Properties and applications of starch-converting enzymes of the α-amylase family[J]. Journal of Biotechnology, 2002, 94(2): 137-155. DOI:10.1016/S0168-1656(01)00407-2 |
| [12] |
Swetha S, Dhanya G, Kesavan MN, et al. α-amylase from microbial sources-an overview on recent developments[J]. Food Technol Biotechnol, 2006, 44(2): 173-184. |
| [13] |
Weemaes C, De Cordt S, Goossens K, et al. High pressure, thermal, and combined pressure-temperature stabilities of α-amylases from Bacillus species[J]. Biotechnol Bioeng, 1996, 50(1): 49-56. DOI:10.1002/(SICI)1097-0290(19960405)50:1<49::AID-BIT6>3.0.CO;2-1 |
| [14] |
Alikhajeh J, Khajeh K, Ranjbar B, et al. Structure of Bacillus amyloliquefaciens alpha-amylase at high resolution:implications for thermal stability[J]. Acta Cryst, 2010, 66(2): 121-129. |
| [15] |
Hwang KY, Song HK, Chang C, et al. Crystal structure of thermostable alpha-amylase from Bacillus licheniformis refined at 1. 7 A resolution[J]. Mol Cells, 1997, 7(2): 251-258. |
| [16] |
Shirai T, Igarashi K, Ozawa T, et al. Ancestral sequence evolutionary trace and crystal structure analyses of alkaline alpha-amylase from Bacillus sp. KSM-1378 to clarify the alkaline adaptation process of proteins[J]. Proteins, 2007, 66(3): 600-610. |
| [17] |
Cornelius B, Jutta S, Karl HM, et al. Directed evolution of a bacterial-amylase:toward enhanced pH-performance and higher specific activity[J]. Protein Science, 2003, 12(10): 2141-2149. |
| [18] |
Nathalie D, Mischa M, Philippe J, et al. Engineering the thermostability of Bacillus licheniformis α-amylase[J]. Biologia Bratislava, 2002, 57(11): 203-211. |
| [19] |
Archana S, Satyanarayana T. Cloning and expression of acidstable, high maltose-forming, Ca2+-independent α-amylase from an acidophile Bacillus acidicola and its applicability in starch hydrolysis[J]. Extremophiles, 2012, 16(3): 515-522. DOI:10.1007/s00792-012-0451-2 |
| [20] |
Azadeh E, Khosro K, Hossein NM. Thermostabilization of Bacillus amyloliquefaciens amylase by chemical cross-linking[J]. J Biotechnol, 2006, 123(4): 434-442. DOI:10.1016/j.jbiotec.2005.12.017 |
| [21] |
Janecek S, Svensson B, MacGregor EA. Alpha-amylase:an enzyme specificity found in various families of glycoside hydrolases[J]. Cellular and Molecular Life Sciences, 2014, 71(4): 1149-1170. |
| [22] |
Sumitani J, Tottori T, Kawaguchi T, et al. New type of starch-binding domain:the direct repeat motif in the C-terminal region of Bacillus sp. no. 195 alpha-amylase contributes to starch binding and raw starch degrading[J]. Biochemical Journal, 2000, 350(2): 477-484. |
| [23] |
Li Z, Duan X, Chen S, et al. Improving the reversibility of thermal denaturation and catalytic efficiency of Bacillus licheniformis α-amylase through stabilizing a long loop in domain B[J]. PLoS One, 2017, 12(3): 1-11. |
| [24] |
Nielsen JE, Borchert TV. Protein engineering of bacterial alpha-amylases[J]. Biochimica Et Biophysica Acta-Protein Structure and Molecular Enzymology, 2000, 1543(2): 253-274. DOI:10.1016/S0167-4838(00)00240-5 |
| [25] |
Robert X, Haser R, Gottschalk TE, et al. The structure of barley alpha-amylase isozyme reveals a novel role of domain C in substrate recognition and binding:A pair of sugar tongs[J]. Structure, 2003, 11(8): 973-984. DOI:10.1016/S0969-2126(03)00151-5 |
| [26] |
Wang H, Yang L, Ping YH, et al. Engineering of a Bacillus amyloliquefaciens strain with high neutral protease producing capacity and optimization of its fermentation conditions[J]. PLoS One, 2016, 11(1): 1-8. |
| [27] |
Igarashi K, Hatada Y, Hagihara H. Enzymatic properties of a novel liquefying α-amylase from an alkaliphilic Bacillus isolate and entire nucleotide and amino acid sequences[J]. Appl Environ Microbiol, 1998, 64(9): 3282-3289. |
| [28] |
Prajapati VS, Trivedi UB, Patel KC. A statistical approach for the production of thermostable and alklophilic alpha-amylase from Bacillus amyloliquefaciens KCP2 under solid-state fermentation[J]. 3 Biotech, 2015, 5(2): 211-220. DOI:10.1007/s13205-014-0213-1 |
| [29] |
Dowd JE, Riggs DS. A comparison of estimates of michaelis-menten kinetic constants from various linear transformations[J]. Journal of Biological Chemistry, 1965, 240(2): 863-869. |
| [30] |
Syu LJ, Chen YH. A study on the α-amylase fermentation performed by Bacillus amyloliquefaciens[J]. Chem Eng, 1997, 65(1): 237-247. |
| [31] |
Lu Z, Wang Q, Jiang S, et al. Truncation of the unique N-terminal domain improved the thermos-stability and specific activity of alkaline α-amylase Amy703[J]. Sci Rep, 2016, 6: 1-10. DOI:10.1038/s41598-016-0001-8 |
| [32] |
Yang H, Liu L, Shin HD, et al. Integrating terminal truncation and oligopeptide fusion for a novel protein engineering strategy to improve specific activity and catalytic efficiency:alkaline-amylase as a case study[J]. Appl Environ Microbiol, 2013, 79(20): 6429-6438. DOI:10.1128/AEM.02087-13 |
| [33] |
Bessler C, Schmitt J, Maurer KH, et al. Directed evolution of a bacterial -amylase:toward enhanced pH-performance and higher specific activity[J]. Protein Sci, 2003, 12(10): 2141-2149. |
| [34] |
Shirai T, Igarashi K, Ozawa T, et al. Ancestral sequence evolutionary trace and crystal structure analyses of alkaline alpha-amylase from Bacillus sp. KSM-1378 to clarify the alkaline adaptation process of proteins[J]. Proteins, 2007, 66(3): 600-610. |
| [35] |
Robert X, Haser R, Gottschalk TE, et al. The structure of barley alpha-amylase isozyme reveals a novel role of domain C in substrate recognition and binding:A pair of sugar tongs[J]. Structure, 2003, 11(8): 973-984. DOI:10.1016/S0969-2126(03)00151-5 |
| [36] |
Hondoh H, Kuriki T, Matsuura Y. Three-dimensional structure and substrate binding of Bacillus stearothermophilus neopullulanase[J]. J Mol Biol, 2003, 326(1): 177-188. DOI:10.1016/S0022-2836(02)01402-X |
| [37] |
Kim MS, Park JT, Kim YW, et al. Properties of a novel thermostable glucoamylase from the hyperthermophilic archaeon Sulfolobus sol-fataricus in relation to starch processing[J]. Applied Environmental Microbiology, 2004, 70(7): 3933-3940. DOI:10.1128/AEM.70.7.3933-3940.2004 |
| [38] |
Kobayashi T, Kanai H, Hayashi T, et al. Haloalkaliphilic maltot-riose-forming amylase from the archae bacterium Natronococcus sp. strain Ah-36[J]. Journal of Bacteriology, 1992, 174(11): 3439-3444. DOI:10.1128/jb.174.11.3439-3444.1992 |
| [39] |
Wang Y, Feng SY, Zhan T, et al. Improving catalytic efficiency of endo-β-1, 4-xylanase from Geobacillus stearothermophilus by directed evolution and H179 saturation mutagenesis[J]. Journal of Biotechnology, 2013, 168(4): 341-347. DOI:10.1016/j.jbiotec.2013.09.014 |
| [40] |
Goyal N, Gupta JK, Soni SK. A novel raw starch digesting thermostable a-amylase from Bacillus sp. I-3 and its use in the direct hydrolysis of raw potato starch[J]. Enzyme Microb Technol, 2005, 37(7): 723-734. DOI:10.1016/j.enzmictec.2005.04.017 |
| [41] |
Yang CH, Liu WH. Purification and properties of a maltotriose-producing a-amylase from Thermobifida fusca[J]. Enzyme Microb Technol, 2004, 35(2): 254-260. |






