2. 上海保兴生物设备工程有限公司,上海 201404
2. Shanghai Baoxing Bio-engineering Equipment Co. Ltd., Shanghai 201404
化石燃料使用带来的环境污染和能源危机等问题亟待解决,开发可再生的清洁能源正逐渐引起广泛关注。在众多的可再生能源中,生物乙醇已经成功实现了商业化。目前生物乙醇大多由玉米、甘蔗等淀粉类或糖类作物为底物[1],被称为“第一代生物乙醇”。其在美国、巴西等农业发达国家有着较好的应用,但在其他国家的发展具有很大的局限性,尤其是“与人争粮、与粮争地”的问题难以得到有效的解决。因此,以农林废弃物为代表的木质纤维素为底物的“第二代生物乙醇”应运而生[2-3]。木质纤维素由纤维素、半纤维素和木质素构成,其结构复杂,因此必须经过预处理和酶解才能释放可用于微生物发酵的糖[4]。在预处理过程中还会产生大量副产物抑制微生物的生长和乙醇的生成[5]。在稀酸预处理中,木糖会转化为糠醛,糠醛不仅消耗胞内大量还原力,还会诱发活性氧自由基(Reactive oxygen species,ROS)的产生,破坏细胞中的DNA、蛋白质、脂质成分,损伤细胞骨架,进而引发细胞程序性死亡[6]。
工业乙醇发酵主要使用酿酒酵母(Saccharomyces cerevisiae),但在过去的几十年中,运动发酵单胞菌(Zymomonas mobilis)逐渐成为研究热点。由于其特殊的ED(Entner-Doudoroff)途径[7],和酿酒酵母相比,其具有较低的产能效率,在发酵过程中会产生更少的生物质并且实现更高的葡萄糖-乙醇转化效率。但是运动发酵单胞菌对糠醛毒性十分敏感[8],因此提高运动发酵单胞菌对糠醛的耐受性将有利于该菌的工业化应用。
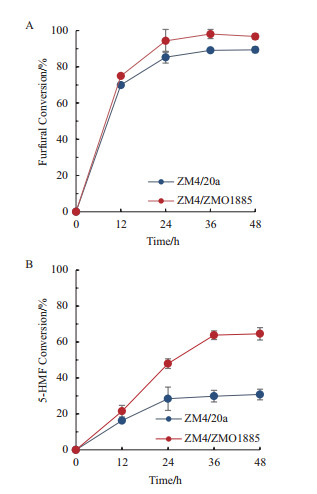
为了发掘细胞抵抗抑制物毒性的基因位点,通用的研究策略是:首先通过突变、实验室进化等方法获得具有高耐受性表型的菌株,再采用全基因组测序、RT-qPCR等方法发掘突变或者转录水平发生变化的基因,最后验证目标基因功能[9-10]。除此之外,在高浓度醛类环境下细胞中特定氧化还原酶的表达量上调,其中一些依赖于NADH、NADPH两种辅因子的酶类[10],能将醛类转化为低毒性的醇类物质,或者消除醛类引发的ROS。NADH氧化酶与细菌的抗氧化能力密切相关[11-13],如大部分乳酸菌Lactococcus lactis有氧生长过程中都会表现出该酶的活性,该酶能够催化氧气变成水分子,减轻氧气诱发的ROS[13]。在光滑球拟酵母Torulopsis glabrata中异源表达L. lactis的NADH氧化酶,可有效降低NaCl诱导的ROS,提高其高渗透压耐受性[14]。在酿酒酵母中异源表达L. lactis的NADH氧化酶也降低了其有氧发酵条件下的ROS水平,提高菌株发酵性能[15]。NADP+还原酶催化NADPH的再生,而NADPH作为一种重要的辅因子,为抗氧化系统中还原型谷胱甘肽和硫氧还蛋白的再生提供还原力[16]。谷胱甘肽还原酶是抗氧化系统中的重要成员,依赖NADPH催化氧化型谷胱甘肽向还原型谷胱甘肽转化[17-18],本研究着重研究在运动发酵单胞菌中,过表达其内源NADH氧化酶基因ZMO1885、NADP+还原酶基因ZMO1753及谷胱甘肽还原酶基因ZMO12111,探究这些具有抗氧化功能的重组菌株在糠醛胁迫条件下的生长发酵性能。
1 材料与方法 1.1 材料靶基因的选择通过在KEGG数据库(https://www.kegg.jp/)中筛选,找到运动发酵单胞菌的NADH氧化酶基因ZMO1885、NADP+还原酶基因ZMO1753和谷胱甘肽还原酶基因ZMO1211,并且对其进行过表达操作。
菌株、质粒及引物本实验所使用的菌株、质粒和引物见表 1和表 2。出发菌株为野生型运动发酵单胞菌Z. mobilis ZM4。用以表达载体构建的菌株为大肠杆菌DH5α,用以载体去甲基化的菌株为大肠杆菌JM110。
运动发酵单胞菌使用RM培养基,大肠杆菌使用LB培养基。LB培养基:10 g/L胰蛋白胨、5 g/L酵母提取物、10 g/L氯化钠,固体培养基中加入琼脂粉20 g/L。RM培养基:20 g/L葡萄糖、2 g/L磷酸二氢钾、10 g/L酵母提取物,固体培养基加入琼脂粉20 g/L。模拟木质纤维素水解液:60 g/L葡萄糖、20 g/L木糖,2 g/L磷酸二氢钾,10 g/L酵母提取物,发酵前加入0.53 g/L糠醛、0.36 g/L 5-HMF、0.34 g/L甲酸、4.33 g/L乙酸[19]。
培养含有目标表达载体的菌体,在培养基中添加盐酸四环素(Tc)20 mg/L。E.coli DH5α和JM110于1.5 mL离心管、37℃、200 r/min振荡培养,培养体积为1 mL。Z. mobilis ZM4使用250 mL摇瓶于30℃培养,培养体积为150 mL,在种子培养时静置,发酵时150 r/min振荡培养。
1.2.2 菌株构建与活化以ZM4基因组为模板获得Pgap、ZMO1885、ZMO1753、ZMO1211片段。用限制性内切酶EcoR Ⅰ和Pst Ⅰ切割pHW20a质粒,后经凝胶电泳切胶纯化后,利用同源重组的方法将目的基因和启动子Pgap连接在线性化载体pHW20a上,热激法转化大肠杆菌DH5α。为克服运动发酵单胞菌限制修饰系统对转化效率的影响,将大肠杆菌DH5α中提取的表达载体转入JM110菌株中去甲基化。每80 μL感受态细胞加入1 μg去甲基化的质粒,静置10 min后,转入预冷的1 mm电转杯中,电击转化。转化子通过PCR以及电泳验证无误后保存于-80℃备用。
重组运动发酵单胞菌活化:取-80℃保存的重组运动发酵单胞菌在含有20 mg/L Tc的RM固体培养基上三区法划线活化,置于30℃培养箱中静置培养2-3 d,待菌落长出后,挑菌至含有20 mg/L Tc的RM液体培养基中,30℃培养箱中静置培养20 h,再转接经二次活化,用于后续发酵实验。
1.2.3 重组菌株生长及耐受性能的测定于-80℃保存菌株中随机挑取3个转化子用于生长性能评定。重组运动发酵单胞菌经活化后,将OD600调为1.5以1%转接入150 mL含有20 mg/L Tc的RM液体培养基中,于30℃培养箱中静置培养约15 h,将其OD600调为1.5后10%接种于发酵培养基中,每4 h取样测量OD600,绘制生长曲线。对于耐受性能的评估,在发酵前加入糠醛2 g/L。
1.2.4 RT-qPCR验证发酵6 h后取9 mL发酵液,10 000 r/min离心2 min收集细胞,用ddH2O洗涤2次,液氮速冻用于RNA提取。使用总RNA抽提纯化试剂盒(Total RNA Extraction and Purification Kit,Sangon Biotech)提取总RNA,并利用反转录试剂盒(PrimeScript RT reagent Kit,TaKaRa)将总RNA反转成cDNA作为模板,再利用定量试剂盒(iQTM SYBR® Green,Bio-Rad)进行表达量检测,以2-ΔΔCt方法比较重组菌株和对照菌株目标基因的转录量,以ZMOr009为内参基因。本实验中使用的实时定量引物见表 3。
使用NADH氧化酶测试盒(苏州科铭生物技术有限公司)测定细胞内NADH氧化酶酶活。发酵6 h后取9 mL发酵液,离心收集细胞,用磷酸盐缓冲液(PBS)洗涤2次,根据试剂盒方法进行酶活的测定。
1.2.6 胞内活性氧检测使用探针2',7' -二氯二氢荧光素二乙酸酯(H2DCFDA)测定2 g/L糠醛条件下重组菌株和对照菌株细胞内ROS水平。发酵6 h后取9 mL发酵液,将OD600调为0.25,取4.5 mL发酵液,离心收集菌体,用PBS洗2遍菌体。后用500 μL的PBS重悬菌体,加入40 μL H2DCFDA,在黑暗条件下于30℃,200 r/min反应1 h。离心收集细胞,再用PBS洗去多余的荧光染料,后用500 μL的PBS重悬细胞,吸取200 μL于96孔板(黑色背景)中,使用全波长扫描仪(ENSPIRE 2300,PE),在485 nm下的激发光和535 nm下的发射光下测定其荧光强度,ROS的量以荧光强度表示。
1.2.7 NAD+及NADH测定使用NAD+/NADH(NAD+/NADH Assay Kit,碧云天公司)测定细胞内NADH及NAD+水平。发酵6 h后取9 mL发酵液,离心收集细胞,用PBS洗涤2次。测量NAD+的样品重悬于1 mL 0.2 mol/L HCl中,测量NADH的样品重悬于0.2 mol/L NaOH中。超声破碎细胞后将悬浮液煮沸5 min,在冰浴中快速淬火,然后在离心管中先后加入500 μL的样品和500 μL 0.2 mol/L NaOH(测量NAD+)或0.2 mol/L HCl(测量NADH)。将混合物10 000 r/min离心10 min后,收集上清液,根据NADH检测试剂盒的方案,用上清液测定NAD+和NADH的含量。
1.2.8 模拟木质纤维素水解液发酵性能检测将种子培养至OD600约2.5,20%接种于模拟木质纤维素水解液中。每12 h取一次样测量OD600,绘制生长曲线。发酵液中葡萄糖、乙醇、糠醛、5-HMF含量分析采用Waters e2695高效液相色谱(Waters,MA,USA)系统,装配Aminex HPX-87H有机酸分析柱(300 mm×7.8 mm,Hercules,Bio-Rad)和示差检测器(Waters 2414)。进样量20 μL,流动相为0.004 mol/L的硫酸溶液,流速0.6 mL/min,示差检测器温度50℃,柱温65℃。
2 结果 2.1 重组菌株生长性能评价利用质粒pHW20a分别过表达基因ZMO1885、ZMO1753、ZMO1211,探究过表达基因对于运动发酵单胞菌生长性能的影响。如图 1-A所示,过表达ZMO1885和ZMO1211对菌株的生长没有显著影响,但是过表达ZMO1753却对菌株的生长产生了明显的负面影响。菌株到达稳定期的时间延长,最大OD600降低,仅能达到对照菌株的90.03%。在耗糖能力和乙醇生产能力上,和对照菌株相比,ZMO1885(图 1-B)和ZMO1211(图 1-D)过表达菌株没有明显差异。但是,过表达ZMO1753基因的工程菌株受生长减慢的拖累,耗糖速度和乙醇的产率都明显降低(图 1-C)。运动发酵单胞菌的单基因操作可对其生长性能产生负面影响,降低的生物量进而影响到糖耗速率和乙醇生产力。因此,在获得重组菌株后有必要对其生长性能、糖耗、乙醇生产力进行评估。

|
| A:生长曲线,B、C、D分别为菌株ZM4/ZMO1885、ZM4/ZMO1211、ZM4/ZMO1753的乙醇生产及糖耗曲线 图 1 重组菌株生长性能评价 |
为考察糠醛抑制对照菌株ZM4/20a生长的效果,将ZM4/20a置于含有0.0、0.5、1.0、1.5、2.0 g/L的糠醛培养基中进行培养,测得细胞的生长曲线以及达到稳定期的OD600。
如图 2-A所示,高浓度的糠醛抑制了细胞的生长,不仅降低了细胞在对数生长期的生长速度,还延迟了细胞到达稳定期的时间。由于菌株在24 h可以达到最大OD600。对菌株最大OD600和与之对应的糠醛浓度进行拟合(图 2-B),计算得糠醛的IC50为0.865 g/L,IC75约为2 g/L。为了筛选出耐受性较强的菌株,选择IC75(~2 g/L)的糠醛浓度检测3个重组菌株的生长。选择24 h进行生物量的比较,初步评估其耐受性能(表 4)。

|
| A:不同浓度糠醛下菌株ZM4/20a生长曲线,B:不同浓度糠醛下ZM4/20a达到的最大OD600 图 2 糠醛耐受曲线 |
如表 4,在无糠醛的条件下ZM4/ZMO1885与ZM4/20a的OD600没有显著差异,但是在2 g/L糠醛的抑制下,ZM4/ZMO1885凸显出生长优势,其OD600是对照组的138.48%,表明过表达ZMO1885能够提高菌株对于糠醛的耐受性。
过表达ZMO1753基因会使得菌株在正常培养条件下生长差于对照菌株,仅能达到对照最大OD600的90.03%,但是在含有2 g/L的糠醛条件下生长时,其最大OD600略高于对照菌株ZM4/20a,能达到其最大OD600的109.07%。尽管ZMO1753基因过表达干扰了菌株的正常生长,但在糠醛抑制条件下ZMO1753编码的酶提高了糠醛耐受性,从而抵消了其对于菌株生长的负面影响。
过表达ZMO1211没有影响菌株在无糠醛培养基中的生长,但是在2 g/L糠醛存在的条件下,却降低了菌株对于糠醛的耐受性,最大OD600仅能达到对照菌株的74.1%。
对于代谢网络简单、产能效率低下的运动发酵单胞菌而言,需要考虑改造基因是否会对菌株生长造成负面影响,同时在判断基因对于胁迫耐受作用时,也必须扣除掉该基因操作对生长性能的影响。
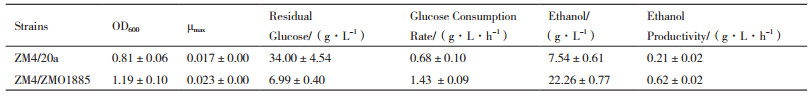
2.3 胞内活性氧自由基检测在运动发酵单胞菌中过表达ZMO1885编码的NADH氧化酶可以提高菌株对糠醛的耐受性,过表达ZMO1753也对糠醛耐受性提高有一定的效果。进一步检测ZM4/20a、ZM4/ZMO1885、ZM4/ZMO1753菌株胞内的ROS水平,探索耐受性提高与基因抗氧化功能之间的相关性。
如图 3所示,ZMO1885编码的NADH氧化酶过表达能显著降低胞内的ROS水平。和对照菌株ZM4/20a相比,菌株ZM4/ZMO1885的ROS水平降低了52.10%,减轻糠醛所引发的ROS对细胞的伤害,改善了菌株在糠醛条件下的生长状态。ZMO1753过表达菌株中ROS水平也显著下降,说明该基因也能降低糠醛引发的ROS。但该基因过表达给菌体生长带来的负面作用,抵消了部分耐受糠醛的优势,生长性能上仅略优于对照菌株。
|
| 图 3 重组菌株及对照菌株ROS水平测定 |
通过生物量和ROS实验,证明了所挑选3个基因中仅ZMO1885过表达能够显著去除胞内ROS,并显著提高菌株在糠醛抑制下的生物量。为考察该基因过表达对细胞的其他影响,表 5展示重组菌株与对照菌株的ZMO1885基因表达量、NADH氧化酶酶活及NADH/NAD+比值。尽管构建菌株中使用了强启动子Pgap,重组菌中的ZMO1885表达量仅仅比对照菌株上调了2.26倍,该基因的表达量可能受到细胞严格控制。NADH氧化酶酶活实验与转录数据相符合,实验测得的所有以NADH为辅因子的全细胞NADH氧化酶酶活仅微弱上升8.28%。一方面重组菌中的酶量和酶活改变小,另一方面,细胞内除ZMO1885编码的NADH氧化酶外,还存在其他能利用NADH作为辅因子的氧化还原酶,因此导致胞内的NADH/NAD+比例并没有显著的差异。综上所述,过表达ZMO1885并未引起运动发酵单胞菌氧化还原水平失衡的现象,不会影响其他利用NADH作为辅因子的氧化还原酶的功能。因此在无抑制物添加情况下并没有造成对菌体生长的影响。而当有糠醛加入后,又能发挥自身的功能去除ROS,避免细胞死亡。
在木质纤维素预处理水解液中,除了糠醛之外,还存在多种其他种类的重要抑制物:甲酸、乙酸、5-HMF等。因此,基于之前实验结果,挑取表型最优的菌株ZM4/ZMO1885采用模拟木质纤维素水解液进一步验证其发酵性能及耐受性能。
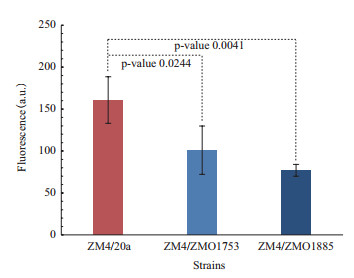
表 6为对照菌株和重组菌株在对数生长中期发酵性能上的差异。在模拟木质纤维素水解液中,ZM4/ZMO1885的生长显著优于对照菌株ZM4/20a,相应的发酵性能也有所提高。在发酵时长达到对数中期36 h,ZM4/ZMO1885的残糖量仅为ZM4/20a的20.56%,而乙醇浓度为其2.95倍。在前36 h内,菌株ZM4/ZMO1885的生物量提高了46.91%,糖耗速率提高了110.29%,乙醇生产力提高了195.24%。不仅如此,重组菌株针对水解液中的糠醛及5-HMF的去除率更高,分别为98.10%和64.55%,而对应的对照菌株仅为89.39%和30.79%(图 4)。
|
| A:糠醛转化率,B:5-HM转化率F *Furfural(5-HMF)Conversion(%)= 1-Residual Furfural(5-HMF)/ Initial Furfural(5-HMF) 图 4 模拟木质纤维素水解液中重组菌株ZM4/ZMO1885及对照菌株ZM4/20a糠醛和5-HMF转化率 |
本实验所研究的基因ZMO1885、ZMO1753、ZMO1211均为运动发酵单胞菌内源基因。其编码的酶均具有抗氧化能力,减轻由糠醛诱发的ROS对细胞的损害(图 5)。
|
| 图 5 ZMO1885、ZMO1753、ZMO1211糠醛耐受性机理图 |
对运动发酵单胞菌进行基因工程操作时,要严格考虑选取的基因并对重组菌株进行生长性能检测。ZMO1885和ZMO1211的过表达未对菌株的生长、糖耗、乙醇生产产生明显的影响。过表达ZMO1753会引起其生长的滞后以及最大OD600的降低,造成糖耗速度和乙醇生产力变低。由基因操作引起的生长性能改变归因于两方面:(1)运动发酵单胞菌代谢网络简单和生产能量效率低。运动发酵单胞菌没有完整三羧酸循环,每消耗1 mol葡萄糖,只能经由ED途径产生1 mol ATP[21]。因此,即使过表达单个基因,也可能对菌株的代谢网络造成严重的扰动。(2)运动发酵单胞菌中特定基因的具体功能。ZMO1885编码的NADH氧化酶及ZMO1211编码的谷胱甘肽还原酶的过表达均未给细胞生长造成严重的干扰,由于NADH氧化酶为非核心的细胞氧化酶,有许多同工酶发挥作用,而谷胱甘肽还原酶在没有抑制的条件下,对细胞生长并无直接关联; ZMO1753编码的NADP+还原酶的过表达显著抑制菌株生长,这可能由于其影响胞内铁氧还蛋白等重要物质的含量[22-24]。在2 g/L糠醛的条件下,若扣除基因改造对菌株生长的负面影响,ZMO1753过表达也略微提高了细胞对糠醛的耐受性。在酿酒酵母中,其磷酸戊糖途径(Pentose phosphate pathway,PPP)和抗氧化性的关联性也早已被证实,敲除PPP途径中的关键基因,会导致其抗氧化性下降[25]。过表达PPP中的限速酶,提高NADPH的含量,而NADPH作为多种过氧化酶的重要辅因子协助其清除胞内ROS,从而提高菌株的耐受性[4, 26]。
NADH氧化酶对于微生物有氧生长过程中的抗氧化性已被证实,[11-12]如乳酸菌的有氧生长中该酶发挥着重要的抗氧化作用[13]。在光滑球拟酵母Torulopsis glabrata异源表达来源于乳酸菌Lactococcus lactis NADH氧化酶,可以有效降低NaCl诱导的ROS,减少ROS给细胞带来的伤害,提高其高渗透压耐受性[14]; 而在酿酒酵母中异源表达L. lactis的NADH氧化酶也有类似的效果,降低了其有氧发酵条件下的ROS水平[15]。糠醛会诱发细胞产生大量ROS,对细胞各种微结构造成损伤[6, 27]。在2 g/L糠醛的条件下,ZMO1885过表达菌株糠醛耐受性显著提升,归因于NADH氧化酶去除ROS的能力。在含有多种抑制物的模拟木质纤维素水解液中,ZMO1885过表达菌株展示高耐受性能,与糠醛的单抑制物条件下的结论相一致。
NADH氧化酶催化NADH的氧化[15],而NADH作为一种重要的还原性辅因子参与细胞中多种氧化还原反应[28-29]。NADH/NAD+的变化会给细胞生长带来负面影响,或影响目标产物的生成[30-32]。在运动发酵单胞菌中,催化糠醛转化为糠醇的脱氢酶或醛酮还原酶需消耗NADH或NADPH[33]。因此NADH氧化酶过表达引发胞内NADH水平大幅降低可能会干扰其他氧化还原酶,进而影响细胞生长和耐受性。在本实验中,ZMO1885基因的过表达仅在转录水平发生有限倍数的上调,对胞内NADH/NAD+水平影响有限,该基因未造成胞内氧化还原失衡,并未影响细胞的生长状态,但是其催化功能降低了ROS的含量,因此提高了菌株对于糠醛的胁迫耐受性。
4 结论在运动发酵单胞菌中过表达NADH氧化酶基因ZMO1885,能够在不干扰胞内NADH/NAD+水平的前提下,通过降低胞内ROS水平、强化糠醛的去除增强了菌株的糠醛耐受性。在模拟木质纤维素水解液中,重组菌株ZM4/ZMO1885的生长发酵性能显著优于对照菌株ZM4/20a。
| [1] |
Azapagic A, Perdan S, Clift R, et al. Sustainable development in practice:case studies for engineers and scientists[M]. New York: John Wiley & Sons, 2004.
|
| [2] |
Morales M, Quintero J, Conejeros R, et al. Life cycle assessment of lignocellulosic bioethanol:environmental impacts and energy balance[J]. Renewable and Sustainable Energy Reviews, 2015, 42: 1349-1361. DOI:10.1016/j.rser.2014.10.097 |
| [3] |
曹莲莹, 李凯, 李凡, 等. 木质纤维素乙醇关键技术研究进展[J]. 生物产业技术, 2018(4): 25-32. DOI:10.3969/j.issn.1674-0319.2018.04.004 |
| [4] |
Matsufuji Y, Fujimura S, Ito T, et al. Acetaldehyde tolerance in Saccharomyces cerevisiae involves the pentose phosphate pathway and oleic acid biosynthesis[J]. Yeast, 2010, 25(11): 825-833. |
| [5] |
Wang S, Sun X, Yuan Q. Strategies for enhancing microbial tolerance to inhibitors for biofuel production:A review[J]. Bioresource Technology, 2018, 258: 302-309. DOI:10.1016/j.biortech.2018.03.064 |
| [6] |
Allen SA, Clark W, Mccaffery JM, et al. Furfural induces reactive oxygen species accumulation and cellular damage in Saccharomyces cerevisiae[J]. Biotechnology for Biofuels, 2010, 3(1): 2-11. |
| [7] |
Yang S, Fei Q, Zhang Y, et al. Zymomonas mobilis as a model system for production of biofuels and biochemical[J]. Microbial Biotechnology, 2016, 9(6): 699-717. DOI:10.1111/1751-7915.12408 |
| [8] |
Franden MA, Pilath HM, Mohagheghi A, et al. Inhibition of growth of Zymomonas mobilis model compounds found in lignocellulosic hydrolysates[J]. Biotechnology for Biofuels, 2013, 6(1): 99-113. |
| [9] |
Huang S, Xue T, Wang Z, et al. Furfural-tolerant Zymomonas mobilis derived from error-prone PCR-based whole genome shuffling and their tolerant mechanism[J]. Applied Microbiology and Biotechnology, 2018, 102(7): 3337-3347. DOI:10.1007/s00253-018-8817-8 |
| [10] |
Yi X, Gu H, Gao Q, et al. Transcriptome analysis of Zymomonas mobilis ZM4 reveals mechanisms of tolerance and detoxification of phenolic aldehyde inhibitors from lignocellulose pretreatment[J]. Biotechnology for Biofuels, 2015, 8(1): 153-167. |
| [11] |
Wang L, Chong H, Jiang R. Comparison of alkyl hydroperoxide reductase and two water-forming NADH oxidases from Bacillus cereus ATCC 14579[J]. Applied Microbiology and Biotechnology, 2012, 96(5): 1265-1273. DOI:10.1007/s00253-012-3919-1 |
| [12] |
André Jänsch, Freiding S, Jürgen Behr, et al. Contribution of the NADH-oxidase(Nox)to the aerobic life of Lactobacillus sanfranciscensis DSM20451T[J]. Food Microbiology, 2011, 28(1): 29-37. |
| [13] |
Higuchi M, Yamamoto Y, Kamio Y. Molecular biology of oxygen tolerance in lactic acid bacteria:Functions of NADH oxidases and Dpr in oxidative stress[J]. Journal of Bioscience and Bioengineering, 2000, 90(5): 484-493. DOI:10.1016/S1389-1723(01)80028-1 |
| [14] |
Xu S, Zhou J, Qin Y, et al. Water-forming NADH oxidase protects Torulopsis glabrata against hyperosmotic stress[J]. Yeast, 2010, 27(4): 207-216. |
| [15] |
Shi X, Zou Y, Chen Y, et al. Overexpression of a water-forming NADH oxidase improves the metabolism and stress tolerance of Saccharomyces cerevisiae in aerobic fermentation[J]. Frontiers in Microbiology, 2016, 7: 1427-1439. |
| [16] |
Minard KI, McAlister-Henn L. Antioxidant function of cytosolic sources of NADPH in yeast[J]. Free Radical Biology and Medicine, 2001, 31(6): 832-843. DOI:10.1016/S0891-5849(01)00666-9 |
| [17] |
Couto N, Wood J, Barber J. The role of glutathione reductase and related enzymes on cellular redox homoeostasis network[J]. Free Radical Biology and Medicine, 2016, 95: 27-42. DOI:10.1016/j.freeradbiomed.2016.02.028 |
| [18] |
Achary VMM, Reddy CS, Pandey P, et al. Glutathione reductase a unique enzyme:molecular cloning, expression and biochemical characterization from the stress adapted C 4 plant, Pennisetum glaucum(L.)R. Br[J]. Molecular Biology Reports, 2015, 42(5): 947-962. DOI:10.1007/s11033-014-3832-z |
| [19] |
Zhang MM, Zhao XQ, Cheng C, et al. Improved growth and ethanol fermentation of Saccharomyces cerevisiae in the presence of acetic acid by overexpression of SET5 and PPR1[J]. Biotechnology Journal, 2015, 10(12): 1903-1911. DOI:10.1002/biot.201500508 |
| [20] |
Dong H, Fan L, Luo Z, et al. Improvement of ethanol productivity and energy efficiency by degradation of inhibitors using recombinant Zymomonas mobilis(pHW20a-fdh)[J]. Biotechnol Bioeng, 2013, 110: 2395-2404. DOI:10.1002/bit.24897 |
| [21] |
Rutkis R, Strazdina I, Balodite E, et al. The low energy-coupling respiration in Zymomonas mobilis accelerates flux in the Entner-Doudoroff pathway[J]. PLoS One, 2016, 11(4): e0153866. DOI:10.1371/journal.pone.0153866 |
| [22] |
Schorsch M, Kramer M, Goss T, et al. A unique ferredoxin acts as a player in the low-iron response of photosynthetic organisms[J]. Proceedings of the National Academy of Sciences, 2018, 115(51): E12111-E12120. DOI:10.1073/pnas.1810379115 |
| [23] |
Schuller JM, Birrell JA, Tanaka H, et al. Structural adaptations of photosynthetic complex Ⅰ enable ferredoxin-dependent electron transfer[J]. Science, 2019, 363(6424): 257-260. DOI:10.1126/science.aau3613 |
| [24] |
Arcinas AJ, Maiocco SJ, Elliott SJ, et al. Ferredoxins as interchangeable redox components in support of MiaB, a radical S-Adenosylmethionine Methylthiotransferase[J]. Protein Science, 2019, 28(1): 267-282. DOI:10.1002/pro.3548 |
| [25] |
Juhnke H, Krems B, Kötter P, et al. Mutants that show increased sensitivity to hydrogen peroxide reveal an important role for the pentose phosphate pathway in protection of yeast against oxidative stress[J]. Molecular and General Genetics, 1996, 252(4): 456-464. DOI:10.1007/BF02173011 |
| [26] |
Gorsich SW, Dien BS, Nichols NN, et al. Tolerance to furfural-induced stress is associated with pentose phosphate pathway genes ZWF1, GND1, RPE1, and TKL1 in Saccharomyces cerevisiae[J]. Applied Microbiology and Biotechnology, 2006, 71(3): 339-349. DOI:10.1007/s00253-005-0142-3 |
| [27] |
Kim D, Hahn JS. Roles of the Yap1 transcription factor and antioxidants in Saccharomyces cerevisiae's tolerance to furfural and 5-hydroxymethylfurfural, which function as thiol-reactive electrophiles generating oxidative stress[J]. Applied and Environmental Microbiology, 2013, 79(16): 5069-5077. DOI:10.1128/AEM.00643-13 |
| [28] |
Liu CG, Xue C, Lin YH, et al. Redox potential control and applications in microaerobic and anaerobic fermentations[J]. Biotechnology Advances, 2013, 31(2): 257-265. DOI:10.1016/j.biotechadv.2012.11.005 |
| [29] |
郝学密, 杜斌, 刘黎阳, 等. ORP对酿酒酵母在木质纤维素水解液抑制物中发酵的影响[J]. 化工学报, 2015, 66(3): 1066-1071. |
| [30] |
Shi XC, Zou YN, Chen Y, et al. A water-forming NADH oxidase regulates metabolism in anaerobic fermentation[J]. Biotechnology for Biofuels, 2016, 9(1): 103-114. |
| [31] |
Heux S, Cachon R, Dequin S. Cofactor engineering in Saccharomyces cerevisiae:expression of a H2O-forming NADH oxidase and impact on redox metabolism[J]. Metabolic Engineering, 2006, 8(4): 303-314. |
| [32] |
Ji XJ, Xia ZF, Fu NH, et al. Cofactor engineering through heterologous expression of an NADH oxidase and its impact on metabolic flux redistribution in Klebsiella pneumoniae[J]. Biotechnology for Biofuels, 2013, 6(1): 7-15. |
| [33] |
Wang X, Gao Q, Bao J. Enhancement of furan aldehydes conversion in Zymomonas mobilis by elevating dehydrogenase activity and cofactor regeneration[J]. Biotechnology for Biofuels, 2017, 10(1): 24-32. |