线粒体是细胞内代谢活动的主要中枢,参与多种重要的细胞过程,包括铁硫簇的生物合成,钙稳态和细胞凋亡等。氧化磷酸化产生ATP是线粒体最重要的作用,该过程需要五个多亚基蛋白质复合物; 其中四个构成线粒体呼吸链并参与电子传递过程,电子转移在线粒体内膜上产生质子梯度,最后被复合物Ⅴ(ATP合成酶)利用以合成ATP[1-3]。由于线粒体结构和功能固有的复杂性,线粒体功能障碍与诸多的人类疾病有关,例如糖尿病、肥胖症、心血管疾病、癌症,以及线粒体功能衰老[4-7]。其中,复合体Ⅴ中ATP合酶的缺乏会导致心肌病、脑病和白内障等疾病[8-9]。
酿酒酵母的ATP合成酶由至少13种不同种类的亚基组成,5个亚基(α3β3γδ和ε)组成酵母F1,F0包含8个不同的亚基(4、OSCP、d、h和f)[10]。通过pombase数据库分析,粟酒裂殖酵母中Atp4被预测为线粒体ATP合酶F0的组成蛋白,参与线粒体的能量代谢过程并与线粒体ATP的产生密切相关。它在人和芽殖酵母中的同源蛋白,分别ATP5PB和ATP4。芽殖酵母中ATP4是核基因编码的大小为25 kD的ATP合酶亚基4。人们发现ATP4是F0的一个定子,参与F0F1-ATP合酶的组装,功能上与ATP合酶的二聚体的形成,维持内膜嵴的形状和ATP合酶的磷酸化有关[11-12]。在人中,核基因编码蛋白ATP5PB成为线粒体ATP合酶F1-F0的亚基b,与酵母线粒体ATP4的同源性为31%[13-14]。通过同源比对发现裂殖酵母Atp4与人中ATP5PB的同源性达到64%,预测Atp4参与线粒体功能发挥,而且到目前为止,在粟酒裂殖酵母中还没有Atp4功能的研究。因此本文旨在探究粟酒裂殖酵母中Atp4参与线粒体功能的机制。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒粟酒裂殖酵母单倍体菌株yHL6381(h+,his3-D1,leu1-32,ura4-D18,ade6-M210)、大肠杆菌Escherichia coli Top10、pFA6a-KanMX6、pYJ19为本实验室保存。
1.1.2 培养基YES培养基:酵母粉5 g/L,葡萄糖20 g/L,adenine、histidine、uracil、leucine各200 mg/L,2%琼脂粉(固体); YES+6%、3%甘油:每100 mL YES培养基加入12 mL、6 mL 50%甘油; LB培养基:蛋白胨10 g/L,酵母粉5 g/L,氯化钠10 g/L,1.5%琼脂粉(固体); LBAMP+培养基:每100 mL LB培养基加入氨苄青霉素钠(终浓度0.1 mg/mL); EMmLeu-(Edinburghminimal medium)培养基:邻苯二甲酸氢钾3 g/L,十二水合磷酸氢二钠5.55 g/L,氯化铵5 g/L,10 mL 100×salt stock,1 mL 1 000×vitamin,0.1 mL 10 000×mineral stock,adenine、uracil、histidine各225 mg/L,葡萄糖20 g/L,2%琼脂粉(固体)。葡萄糖单独灭菌。
1.1.3 仪器和试剂限制性内切酶、PrimeSTAR DNA Polymerase、PCR反应试剂和连接酶均购自Takara; PCR引物由南京捷瑞合成; DNA Marker购自南京百思凯; DNA纯化和割胶回收试剂盒购自南京博巧; SDS、HEPES、Sorbitol、Tris、30%丙烯酰胺等试剂购自上海生工,尼龙膜购买于GE。主要仪器有PCR仪,台式高速离心机,Odyssey双色红外激光成像系统,电泳槽等。
1.2 方法 1.2.1 Δatp4菌种构建以pFa6a-kanMX6为出发质粒,以yHL6381酵母基因组为模板扩增得到up-同源臂以及down-同源臂。将up-同源臂构建到质粒上游酶切位点Sal Ⅰ和Bgl Ⅱ间,down-同源臂构建到质粒下游EcoR Ⅰ和EcoR Ⅴ间。设计引物将up-同源臂、抗性标签、down-同源臂扩增得到单一目的条带后,醋酸锂转化到yHL6381内通过同源重组敲除基因atp4。
1.2.2 点板实验实验前3 d将菌种yHL6381和Δatp4于固体平板YES上划线活化。将活化菌种接种至培养基内,过夜培养。第二天将种子液重新转接到10 mLYES液体培养基内,并将OD都调至0.2左右,于摇床,30℃,200 r/min,培养12 h。将yHL6381和Δatp4菌液初始OD均调至3左右,按10倍梯度依次稀释为1、10-1、10-2、10-3、10-4。取2.5 μL点圈于甘油平板上。30℃,倒置平板培养3-5 d。拍照。每块平板重复3份,整个实验重复3次。
1.2.3 Atp4定位预测分析粟酒裂殖酵母数据库pombase显示Atp4定位于线粒体,通过网站Mitoprot II(https://ihg.gsf.de/ihg/mitoprot.htmL)分析Atp4的氨基酸序列是否具有线粒体定位序列,预测Atp4 N-端前29个氨基酸为线粒体定位序列:METSSKLSPVQRTTAAWQRLLPSTRFSL,序列可信度为56.13%。因此我们推测Atp4定位于线粒体内。
1.2.4 荧光显微镜观察Atp4定位取质粒pYJ19-atp4-GFP用Nru Ⅰ进行单酶切,酶切完后热失活处理,取1 μg通过LiAc导入到yHL6381内,涂布于EMM(Ade,His,Ura)筛选平板上。3-4 d后挑选大小合适的转化子在EMM筛选平板上划线。2 d后将菌株接种于相应的筛选培养基内,过夜后转接至OD600=0.2,培养至OD600=0.6左右,收菌,PBS缓冲洗一次后,加入Mitotracker染料常温染色2-3 min后,荧光显微镜观察。
1.2.5 线粒体定位实验实验以Tom20(线粒体外膜蛋白)和Hsp60(线粒体基质蛋白)为对照。分别用TritonX-100和蛋白酶K处理线粒体,TritonX-100将线粒体外膜消解而使得蛋白暴露于蛋白酶K环境下被其水解。碱性碳酸钠使线粒体膜打开释放内容物,位于基质的蛋白分布在上清中,而线粒体膜蛋白则分布在沉淀内。用不同处理并与对照蛋白相对应,从而确定目的蛋白的定位。
1.2.6 Western blotting检测线粒体蛋白表达量分别提取纯化Δatp4和yHL6381线粒体,并用BCA法测取浓度。取等量的线粒体分别进行以下处理:100℃煮10 min用以检测Cox2、Cox4、Atp6和Hsp60;45℃水浴3 min用以检测Cox1、Cob1和Cox3。将处理后的线粒体蛋白上样于12% SDS-PAGE,80 V电泳30 min,120 V电泳90 min。转膜300 mA,100 min。5%脱脂奶粉封闭1 h。一抗常温孵育2 h。二抗常温避光孵育1 h。Odyssey Infrared Imaging检测实验结果。
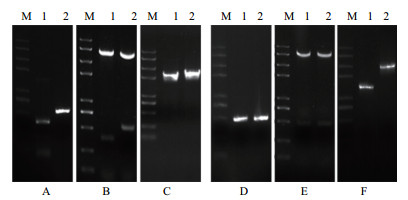
2 结果 2.1 重组质粒的构建及酶切验证Δatp4的构建和验证:选择atp4编码区前384 bp作为上游同源臂和编码区后面550 bp作为下游同源臂,进行PCR扩增和酶切验证(图 1-A-B); 将上游同源臂+KanMX+下游同源臂的融合片段导入yHL6381内,挑选转化子进行PCR验证,野生型为2 305 bp,转化子为2 504 bp(图 1-C); atp4-flag的构建和验证:选择atp4编码区前面和后面约500 bp作为上、下游同源臂(图 1-D); 上、下游构建酶切验证(图 1-E); 将上游同源臂+Flag+下游同源臂的融合片段导入yHL6381内,挑选转化子进行PCR验证,野生型为1 449 bp,转化子为3 510 bp(图 1-F)。
|
| M:DNA分子量标准(100 bp-8 kb);A、B、C是Δatp4的构建和验证,D、E、F是atp4-flag的构建和验证;A和D:(1:up-同源臂的PCR扩增片段,2:down-同源臂的PCR扩增片段);B和E:(1:up-同源臂酶切验证,2:down-同源臂酶切验证);C和F:(1:野生型的PCR验证产物,2:敲除菌的PCR验证产物) 图 1 重组质粒的构建与验证 |
为研究敲除菌的表型,构建了Δatp4菌株并在以葡萄糖或甘油为唯一碳源的YES固体培养基上点圈,以yHL6381为阴性对照。结果(图 2)显示,在YES发酵平板上,敲除菌株和野生型菌株生长状况一致。在含3%和6%甘油非发酵平板上,Δatp4菌株的生长明显减缓。说明atp4基因的敲除使得菌株呼吸缺陷,从而使线粒体蛋白功能发挥异常。

|
| 图 2 Δatp4在甘油平板上的生长情况 |
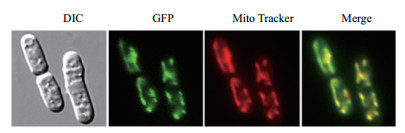
利用GFP追踪Atp4,Mitotracker追踪线粒体以确定蛋白定位。Atp4-GFP在显微镜下发出绿色荧光(图 3),Mitotracker染色显示线粒体部分为红色。将两者进行磨合后,重叠部分显示为黄色。结果表明,Atp4定位于线粒体。
|
| MitoTracker:红色荧光;GFP:绿色荧光蛋白;Merge:Red和GFP共定位 图 3 Atp4-GFP的荧光显微镜观察 |
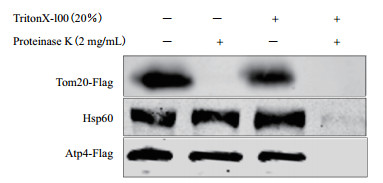
为了进一步研究Atp4定位在外膜还是内部,选择Tom20(线粒体外膜蛋白)和Hsp60(线粒体基质蛋白)作为对照。结果(图 4)显示,Atp4不定位于线粒体外膜,可能定位于线粒体基质或者内膜。
|
| 图 4 TritonX-100和蛋白酶K处理线粒体后蛋白情况 |
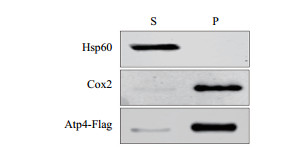
Atp4定位于线粒体基质或内膜,接下来利用碱处理线粒体确定定位。以Cox2(内膜蛋白)和Hsp60(线粒体基质蛋白)作为对照。结果(图 5)显示,Atp4主要分布在沉淀中,上清中只有少量,与内膜蛋白Cox2相同。表明Atp4定位于线粒体内膜。
|
| 图 5 碱性碳酸钠处理线粒体后蛋白情况 |
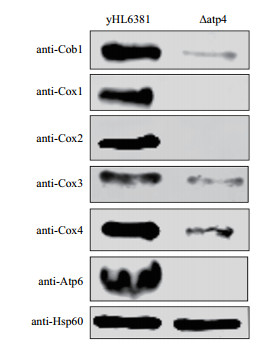
粟酒裂殖酵母的线粒体中仅合成了8种主要蛋白质,包括呼吸复合体Ⅲ(Cob1),Ⅳ(Cox1,Cox2,Cox3)和Ⅴ(Atp6,Atp8和Atp9)的亚基和核糖体蛋白小亚基(Var1)[15-16]。上述实验已经证明atp4定位于线粒体,因此我们检测了由线粒体编码的蛋白(Cob1、Cox1、Cox2、Cox3、Atp6)以及由核编码的定位于线粒体的蛋白Cox4,以内参Hsp60作为上样控制。
Western blotting结果(图 6)显示,相较于yHL6381菌株,Δatp4菌株内线粒体蛋白Cob1、Cox1、Cox2、Atp6表达量明显下降,Cox3和Cox4蛋白表达量也相应有略微下降。这表明atp4缺失影响了线粒体编码的电子传递链蛋白的表达,从而影响了线粒体呼吸链复合体的组装。结合点圈结果显示的Δatp4菌株在非发酵型培养基上出现了生长抑制的现象,判断该现象是由于该基因的缺失导致线粒体蛋白表达量降低而不能正常氧化磷酸化造成的。
|
| 图 6 Western blotting检测Δatp4菌株中线粒体相关蛋白质表达量 |
芽殖酵母缺失ATP4突变菌株,在非发酵培养基上出现了生长缺陷,这是由于影响了ATP合酶的正常组装导致的[17-18]。ATP4包括TM1(25-46 aa)和TM2(56-76 aa)以及中间环(47-55 aa)。其中,缺失中间环,引起了线粒体形态变化(嵴呈现为细长的片状)[19-20]; 破坏C-端亲水结构域,其突变菌株的氧化磷酸化功能丧失,ATP合酶与膜的结合也变得松散[21]。本文研究发现粟酒裂殖酵母Δatp4菌株在非发酵培养基上表现出呼吸缺陷; 通过荧光观察和生化处理线粒体实验得出Atp4是在线粒体内膜上发挥功能,因此推测Atp4可能参与线粒体呼吸链蛋白亚基的组装和ATP的产生。通过Western blotting实验结果表明缺失Atp4引起了Cob1、Cox1、Cox2、Atp6表达量剧烈下降,说明Atp4的缺失不仅仅影响了复合体Ⅴ的组装也使得上游呼吸链复合体Ⅲ和Ⅳ发生解离。
芽殖酵母的ATP4与裂殖酵母的Atp4为同源蛋白,通过Blast氨基酸序列比对分析得出,序列同源性大约是82%,但是N端前40个氨基酸并不保守。ATP4在芽殖酵母中是F0组成蛋白,对于ATP合酶的正常组装和氧化磷酸化的进行十分重要。在粟酒裂殖酵母内Δatp4菌株出现呼吸缺陷,线粒体蛋白表达量剧烈下降。Atp4对于粟酒裂殖酵母正常氧化磷酸化是必不可少的。但是Atp4缺失是影响蛋白的正常合成还是稳定仍需要进一步研究。
4 结论表型研究发现粟酒裂殖酵母atp4缺失后菌株出现生长缺陷。荧光显微镜观察和生化试剂TritonX-100和蛋白酶K处理以及碱性碳酸钠处理线粒体实验证明Atp4定位于线粒体内膜。Western blotting检测结果表明Atp4缺失菌株内线粒体基因组编码蛋白Cob1、Cox1、Cox2和Atp6表达量降低。
| [1] |
Castora FJ. Mitochondrial function and abnormalities implicated in the pathogenesis of ASD[J]. Progress in Neuro-Psychopharmaco-logy & Biological Psychiatry, 2018, 92: 83-108. |
| [2] |
Craven L, Alston CL, et al. Recent advances in mitochondrial dise-ase[J]. Annu Rev Genom Hum G, 2017, 18: 257-275. DOI:10.1146/annurev-genom-091416-035426 |
| [3] |
Devenish RJ, Prescott M, Roucou X, et al. Insights into ATP synthase assembly and function through the molecular genetic manipulation of subunits of the yeast mitochondrial enzyme complex[J]. Biochim Biophys Acta, 2000, 1458(2-3): 428-442. DOI:10.1016/S0005-2728(00)00092-X |
| [4] |
Malina C, Larsson C, Nielsen J. Yeast mitochondria: An overview of mitochondrial biology and the potential of mitochondrial systems biology[J]. FEMS Yeast Research, 2018, 18(5): 1-17. |
| [5] |
Herrnstadt C, Howell N. An evolutionary perspective on pathogenic mtdna mutations: Haplogroup associations of clinical disorders[J]. Mitochondrion, 2004, 4(5-6): 791-798. DOI:10.1016/j.mito.2004.07.041 |
| [6] |
Baker BM, Haynes CM. Mitochondrial protein quality control during biogenesis and aging[J]. Trends Biochem Sci, 2011, 36(5): 254-261. DOI:10.1016/j.tibs.2011.01.004 |
| [7] |
Lasserre JP, Dautant A, Aiyar RS, et al. Yeast as a system for modeling mitochondrial disease mechanisms and discovering therapies[J]. Dis Model Mech, 2015, 8(6): 509-526. DOI:10.1242/dmm.020438 |
| [8] |
Alston CL, Rocha MC, Lax NZ, et al. The genetics and pathology of mitochondrial disease[J]. J Pathol, 2017, 241(2): 236-250. DOI:10.1002/path.4809 |
| [9] |
Van Der Bliek AM, Sedensky MM, Morgan PG. Cell biology of the mitochondrion[J]. Genetics, 2017, 207(3): 843-871. DOI:10.1534/genetics.117.300262 |
| [10] |
Soubannier V, Rusconi F, Vaillier J, et al. The second stalk of the yeast atp synthase complex: Identification of subunits showing cross-links with known positions of subunit 4(subunit b)[J]. Biochemistry, 1999, 38(45): 15017-15024. DOI:10.1021/bi9916067 |
| [11] |
Mayr JA, Haack TB, Freisinger P, et al. Spectrum of combined respiratory chain defects[J]. Journal of Inherited Metabolic Disease, 2015, 38(4): 629-640. DOI:10.1007/s10545-015-9831-y |
| [12] |
Davies KM, Anselmi C, et al. Structure of the yeast f1fo-atp synthase dimer and its role in shaping the mitochondrial cristae[J]. Proc Natl Acad Sci USA, 2012, 109(34): 13602-13607. DOI:10.1073/pnas.1204593109 |
| [13] |
Vo TD, Palsson BO. Building the power house: Recent advances in mitochondrial studies through proteomics and systems biology[J]. Am J Physiol-Cell Ph, 2007, 292(1): C164-C177. DOI:10.1152/ajpcell.00193.2006 |
| [14] |
Higuti T, Tsurumi C, Osaka F, et al. Molecular cloning of cDNA for the import precursor of human subunit b of H+ synthase in mitochondria[J]. Biochemical and Biophysical Research Communications, 1991, 178(3): 1014-1020. DOI:10.1016/0006-291X(91)90993-H |
| [15] |
Foury F, Roganti T, Lecrenier N, et al. The complete sequence of the mitochondrial genome of saccharomyces cerevisiae[J]. FEBS Letters, 1998, 440(3): 325-331. DOI:10.1016/S0014-5793(98)01467-7 |
| [16] |
Andersson SG, Zomorodipour A, Andersson JO, et al. The genome sequence of rickettsia prowazekii and the origin of mitochondria[J]. Nature, 1998, 396(6707): 133-140. DOI:10.1038/24094 |
| [17] |
Kwon YY, Choi KM, Cho C, et al. Mitochondrial efficiency-dependent viability of saccharomyces cerevisiae mutants carrying individual electron transport chain component deletions[J]. Molecules and Cells, 2015, 38(12): 1054-1063. DOI:10.14348/molcells.2015.0153 |
| [18] |
Welch AK, Bostwick CJ, Cain BD. Manipulations in the peripheral stalk of the saccharomyces cerevisiae f1f0-atp synthase[J]. The Journal of Biological Chemistry, 2011, 286(12): 10155-10162. DOI:10.1074/jbc.M110.213447 |
| [19] |
Weimann T, et al. The intermembrane space loop of subunit b(4)is a major determinant of the stability of yeast oligomeric atp synthases[J]. Biochem, 2008, 47(11): 3556-3563. DOI:10.1021/bi702000g |
| [20] |
Soubannier V, Vaillier J, Paumard P, et al. In the absence of the first membrane-spanning segment of subunit 4(b), the yeast atp synthase is functional but does not dimerize or oligomerize[J]. Journal of Biological Chemistry, 2002, 277(12): 10739-10745. DOI:10.1074/jbc.M111882200 |
| [21] |
Velours J, Arselin G, et al. The yeast atp synthase subunit 4: Structure and function[J]. Biochimie, 1989, 71(8): 903-915. DOI:10.1016/0300-9084(89)90073-4 |




